Оптимизация наследованием крови — Blood inheritance optimization (BIO)
Содержание
Введение
Однажды, когда я наблюдал за тем, как медсестра берет кровь у пациентов в лаборатории, меня посетила неожиданная мысль. Группы крови — эта древняя система наследования, передающаяся из поколения в поколение по строгим генетическим законам, — вдруг предстала передо мной в совершенно новом свете. Что если эти природные качества наследования можно использовать в области оптимизационных алгоритмов?
Каждый из нас носит в своих венах уникальную комбинацию, доставшуюся от родителей. Подобно тому, как группы крови определяют совместимость при переливании, они могли бы определять способы передачи и мутации параметров в процессе оптимизации. Эта идея мне понравилась, и я решил к этому вернуться, когда будет время на исследования. Как раз представилась возможность, и после проведенных экспериментов родился алгоритм Blood Inheritance Optimization (BIO) — метод, который использует природные законы наследования групп крови, как метафору для управления эволюцией решений. В нем четыре группы крови превратились в четыре различные стратегии мутации параметров, а законы наследования определяют, как потомки получают и модифицируют характеристики своих родителей.
Как и в природе, группа крови ребенка не является простым усреднением групп крови родителей, а подчиняется генетическим законам, в BIO параметры новых решений формируются через систему наследования и мутаций. Каждая группа крови привносит свой уникальный подход к исследованию пространства решений: от консервативного сохранения лучших найденных значений, до радикальных мутаций, открывающих новые перспективные области и направления для дальнейших исследований пространства решений.
В этой статье я хочу поделиться принципами работы алгоритма BIO, который объединяет биологическое вдохновение с алгоритмической строгостью, а также предоставить результаты тестирования на уже знакомых нам функциях. Итак, приступим.
Реализация алгоритма
Для начала познакомимся с таблицей наследования группы крови потомка от родителей. Как можно заметить, наследование группы крови не является однородным. В мире существует интересная статистика распределения групп крови среди населения. Чаще всего встречается первая группа (O) — около 40% населения планеты являются её носителями. За ней следует вторая группа (A), которой обладает примерно 30% людей. Третья группа (B) встречается у 20% населения, а четвертая (AB) — самая редкая, её носителями являются лишь около 10% людей.
Изучая механизмы наследования, я узнал, что первая группа крови является рецессивной по отношению ко всем остальным. Это значит, что люди с первой группой могут передать своим детям только первую группу. В то же время, вторая и третья группы демонстрируют кодоминантность по отношению друг к другу, что приводит к появлению четвертой группы при их сочетании. Кстати, с эволюционной точки зрения четвертая группа крови — самая молодая.
Меня особенно заинтересовали некоторые уникальные свойства разных групп крови. Например, первая группа считается "универсальным донором", поскольку её можно переливать людям с любой группой крови. А четвертая группа, напротив, делает своих носителей "универсальными реципиентами" — они могут принимать кровь любой группы.
Все эти особенности системы групп крови инспирировали меня на создание соответствующих механизмов в моём алгоритме. Согласно тому, что первая группа крови является базовой и наиболее распространенной, в алгоритме она соответствует стратегии сохранения лучшего найденного решения. Таблица наследования групп крови, показывающая все возможные комбинации групп крови родителей и потенциальные группы крови их детей, легла в основу механизма определения "группы крови" нового решения на основе "групп крови" родительских решений, что напрямую влияет на способ мутации параметров в алгоритме BIO.
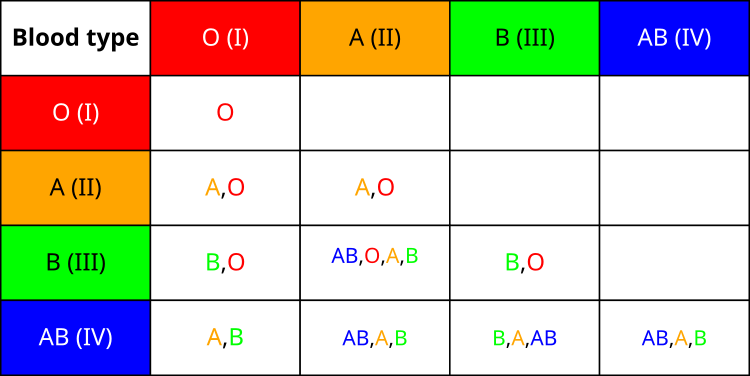
Рисунок 1. Таблица наследования группы крови
Основой алгоритма BIO является довольно простая идея: каждому решению в популяции (родительские особи) соответствует своя "группа крови" (от 1 до 4), которая определяется его порядковым номером в популяции. Когда мы создаем новое поколение решений, мы выбираем двух "родителей" из текущей популяции. При этом вероятность выбора не линейная, а квадратичная — это значит, что лучшие решения имеют заметно больше шансов стать родителями.
Дальше начинается самое интересное. На основе групп крови родителей, используя специальную матрицу наследования (она прописана в коде в методе Init), мы определяем возможные группы крови для "ребенка" — нового решения. Затем, для каждого параметра этого нового решения, если выпала первая группа крови — берем значение из лучшего найденного решения. Я сделал это по аналогии с первой группой крови как универсальным донором. Если выпала вторая группа — берем значение от одного из родителей и применяем к нему степенное распределение. Это создает тенденцию к исследованию краев диапазона параметров. При третьей группе — также берем значение от одного из родителей, но двигаем его в сторону лучшего решения на случайную величину. А при четвертой группе — берем родительское значение и отражаем его относительно границ диапазона, своего рода инверсия, что позволяет исследовать новые области поиска.
После создания нового поколения мы проверяем, не появились ли решения лучше текущего глобального решения, и сохраняем лучшие особи для следующей итерации. Вот так, используя аналогию с наследованием групп крови, мой алгоритм исследует пространство решений, комбинируя различные стратегии мутации параметров. Ниже представляю псевдокод алгоритма.
Инициализация:
- Создать популяцию агентов размером popSize (по умолчанию 50)
- Создать матрицу наследования групп крови, которая определяет возможные группы крови у детей на основе групп крови родителей (1,2,3,4)
- Инициализация диапазонов для параметров (мин., макс., значения шага)
Основной цикл:
- Если это первая итерация (ревизия = ложь):
- Случайным образом инициализировать позиции всех агентов в пределах диапазонов параметров
- Установить флаг ревизии на значение true
- Для каждого агента в популяции:
- Выбрать родительских агентов (отца и мать) с использованием квадратичного распределения вероятностей
- Определите группы крови обоих родителей, используя функцию: bloodType = 1 + (положение_в_популяции % 4)
- Для каждого параметра дочернего решения:
- Получить возможную группу крови ребенка из матрицы наследования на основе групп крови родителей
- Если у ребенка группа крови 1:
- Использовать лучшее известное решение для этого параметра.
- В противном случае:
- Случайным образом выбрать значение параметра либо отца, либо матери
- Применить мутацию на основе группы крови ребенка:
- Тип 2: Применить степенное распределение с показателем 20
- Тип 3: Переместить значение параметра в сторону наилучшего решения со случайным фактором
- Тип 4: Зеркальное значение параметра по всему диапазону параметров
- Убедиться, что параметр остается в пределах допустимого диапазона и шага
Фаза пересмотра:
- Обновить глобальное лучшее решение, если какой-либо агент имеет лучшую пригодность
- Копировать текущую популяцию во вторую половину расширенного массива популяции
- Сортировать расширенную популяцию по приспособленности
- Сохранить лучших агентов для следующего поколения
Приступим к написанию кода алгоритма. Класс "C_AO_BIO", производный от "C_AO", реализует алгоритм BIO и предполагает использование структуры данных для представления особей (агентов) в популяции, а также их контроля.
SetParams () — метод позволяет устанавливать параметры класса, в данном случае устанавливая размер популяции "popSize" из массива параметров.
Init () — метод инициализирует алгоритм, принимая минимальные и максимальные значения параметров, шаг изменения и количество эпох.
Moving () и Revision () — методы отвечают за движение (эволюцию) агентов в популяции и их ревизию (оценка производительности и выбор лучших).
S_Papa и S_Mama:
- S_Papa — представляет собой структуру, содержащую массив типов крови (bTypes).
- S_Mama — содержит массив из четырех объектов S_Papa, что предполагает наличие "родителей" для дальнейшего генетического смешивания.
Такой способ представления в виде структур позволит напрямую получить вероятную группу крови дочерней особи от родителей, указав группу крови родителей, так "ma [1].pa [2].bTypes", где "1" и "2" - группы крови матери и отца соответственно.
Метод GetBloodType () возвращает тип крови для определённого агента, а GetBloodMutation () реализует механизм мутации гена в зависимости от типа крови.
//—————————————————————————————————————————————————————————————————————————————— class C_AO_BIO : public C_AO { public: //-------------------------------------------------------------------- C_AO_BIO () { ao_name = "BIO"; ao_desc = "Blood Inheritance Optimization"; ao_link = "https://www.mql5.com/ru/articles/17246"; popSize = 50; // размер популяции ArrayResize (params, 1); params [0].name = "popSize"; params [0].val = popSize; } void SetParams () { popSize = (int)params [0].val; } bool Init (const double &rangeMinP [], // минимальные значения const double &rangeMaxP [], // максимальные значения const double &rangeStepP [], // шаг изменения const int epochsP = 0); // количество эпох void Moving (); void Revision (); private: //------------------------------------------------------------------- struct S_Papa { int bTypes []; }; struct S_Mama { S_Papa pa [4]; }; S_Mama ma [4]; S_AO_Agent p []; int GetBloodType (int ind); void GetBloodMutation (double &gene, int indGene, int bloodType); }; //——————————————————————————————————————————————————————————————————————————————
Метод "Init" инициализирует экземпляр класса "C_AO_BIO" и подготавливает его для работы, настраивая популяцию агентов и их характеристики, разберём реализацию данного метода.
Вызов метода "StandardInit" — первая строчка проверяет результат вызова метода, который проверяет/инициализирует базовые параметры, необходимые для работы алгоритма.
Инициализация массива агентов:
- Изменяет размер массива агентов "p" в два раза больше заданного размера популяции "popSize".
- В цикле "for" для каждого агента вызывается метод "Init", инициализирующий агента с помощью параметров координат.
- Далее метод размером задаёт массивы типов крови (bTypes) для структур "S_Mama" и "S_Papa".
- Для различных комбинаций (например, ma [0].pa [0], ma [1].pa [2] и т.д.) устанавливаются разные типы крови в соответствии со специальной матрицей наследования, а размер массивов указывается через "ArrayResize".
Итак, метод "Init" в классе "C_AO_BIO" выполняет важную задачу подготовки объекта к выполнению алгоритма оптимизации: создает популяцию агентов, настраивает их начальные параметры и определяет правила связывания для типов крови (наследования). Это позволяет моментально получить возможную группу крови потомка, а также использовать параметры их "крови" для дальнейшей эволюции в рамках алгоритма.
//—————————————————————————————————————————————————————————————————————————————— bool C_AO_BIO::Init (const double &rangeMinP [], const double &rangeMaxP [], const double &rangeStepP [], const int epochsP = 0) { if (!StandardInit (rangeMinP, rangeMaxP, rangeStepP)) return false; //---------------------------------------------------------------------------- ArrayResize (p, popSize * 2); for (int i = 0; i < popSize * 2; i++) p [i].Init (coords); //1-1 ArrayResize (ma [0].pa [0].bTypes, 1); ma [0].pa [0].bTypes [0] = 1; //2-2 ArrayResize (ma [1].pa [1].bTypes, 2); ma [1].pa [1].bTypes [0] = 1; ma [1].pa [1].bTypes [1] = 2; //3-3 ArrayResize (ma [2].pa [2].bTypes, 2); ma [2].pa [2].bTypes [0] = 1; ma [2].pa [2].bTypes [1] = 3; //1-2; 2-1 ArrayResize (ma [0].pa [1].bTypes, 2); ArrayResize (ma [1].pa [0].bTypes, 2); ma [0].pa [1].bTypes [0] = 1; ma [0].pa [1].bTypes [1] = 2; ma [1].pa [0].bTypes [0] = 1; ma [1].pa [0].bTypes [1] = 2; //1-3; 3-1 ArrayResize (ma [0].pa [2].bTypes, 2); ArrayResize (ma [2].pa [0].bTypes, 2); ma [0].pa [2].bTypes [0] = 1; ma [0].pa [2].bTypes [1] = 3; ma [2].pa [0].bTypes [0] = 1; ma [2].pa [0].bTypes [1] = 3; //1-4; 4-1 ArrayResize (ma [0].pa [3].bTypes, 2); ArrayResize (ma [3].pa [0].bTypes, 2); ma [0].pa [3].bTypes [0] = 2; ma [0].pa [3].bTypes [1] = 3; ma [3].pa [0].bTypes [0] = 2; ma [3].pa [0].bTypes [1] = 3; //2-3; 3-2 ArrayResize (ma [1].pa [2].bTypes, 4); ArrayResize (ma [2].pa [1].bTypes, 4); ma [1].pa [2].bTypes [0] = 1; ma [1].pa [2].bTypes [1] = 2; ma [1].pa [2].bTypes [2] = 3; ma [1].pa [2].bTypes [3] = 4; ma [2].pa [1].bTypes [0] = 1; ma [2].pa [1].bTypes [1] = 2; ma [2].pa [1].bTypes [2] = 3; ma [2].pa [1].bTypes [3] = 4; //2-4; 4-2; 3-4; 4-3; 4-4 ArrayResize (ma [1].pa [3].bTypes, 3); ArrayResize (ma [3].pa [1].bTypes, 3); ArrayResize (ma [2].pa [3].bTypes, 3); ArrayResize (ma [3].pa [2].bTypes, 3); ArrayResize (ma [3].pa [3].bTypes, 3); ma [1].pa [3].bTypes [0] = 2; ma [1].pa [3].bTypes [1] = 3; ma [1].pa [3].bTypes [2] = 4; ma [3].pa [1].bTypes [0] = 2; ma [3].pa [1].bTypes [1] = 3; ma [3].pa [1].bTypes [2] = 4; ma [2].pa [3].bTypes [0] = 2; ma [2].pa [3].bTypes [1] = 3; ma [2].pa [3].bTypes [2] = 4; ma [3].pa [2].bTypes [0] = 2; ma [3].pa [2].bTypes [1] = 3; ma [3].pa [2].bTypes [2] = 4; ma [3].pa [3].bTypes [0] = 2; ma [3].pa [3].bTypes [1] = 3; ma [3].pa [3].bTypes [2] = 4; return true; } //——————————————————————————————————————————————————————————————————————————————
Метод " Moving" выполняет эволюционные шаги в процессе оптимизации, применяя концепции наследования и мутации к популяции агентов, разберем его подробнее:
Проверка необходимости ревизии (revision) — первая часть метода проверяет, нужно ли обновлять агенты или "двигать" их и если "revision" равно "false", происходит начальная инициализация (или обновление) координат агентов (a [i] .c [j]):
- Каждый агент получает случайные значения, сгенерированные в диапазоне [rangeMin [j], rangeMax [j] с помощью метода "u.RNDfromCI".
- Затем значение приводится в необходимый диапазон с использованием "u.SeInDiSp", который применяет шаг, заданный в "rangeStep".
Переключение на состояние ревизии — после первой итерации параметр "revision" устанавливается в "true", чтобы переключиться на следующий этап, а метод завершает выполнение (return).
Инициализация переменных — вначале метода инициализируются переменные, отвечающие за случайные значения и типы крови родителей (papIND, mamIND, pBloodType, mBloodType, cBloodType и bloodIND).
Основной цикл по популяции (popSize) — метод проходит в цикле по каждому агенту в популяции:
- Генерируются два случайных индекса для родителей (papIND и mamIND) с помощью метода "u.RNDprobab ()", который генерирует случайные вероятности.
- С помощью функции "GetBloodType" извлекаются типы крови для обоих родителей.
Цикл по координатам (coords) — внутри основного цикла для каждого координата агента:
- Выбирается случайный индекс типа крови из массива "bTypes" выбранных родителей (по типу крови матери и отца).
- Если выбранный тип крови равен "1", агент получает значение от "cB [c]". В противном случае происходит смешивание:
- Значение координаты агентов выбирается случайным образом либо от отца, либо от матери.
- Применяется функция "GetBloodMutation", которая производит мутацию выбранного значения на основе типа крови.
- Значение корректируется с использованием метода "u.SeInDiSp" для обеспечения того, что оно остается в допустимых пределах.
Метод "Moving" представляет собой ключевую часть алгоритма, которая эмулирует процесс эволюции популяции агентов и включает в себя как случайную инициализацию, так и механизмы мутации и комбинирования параметров агентов на основе принципов наследования группы крови. Метод сочетает в себе аспекты случайности и наследственности, чтобы создавать новое потомство с различными значениями. Это настраивает агентов для дальнейшей оптимизации и поиска в пространстве решений.
//—————————————————————————————————————————————————————————————————————————————— void C_AO_BIO::Moving () { //---------------------------------------------------------------------------- if (!revision) { for (int i = 0; i < popSize; i++) { for (int j = 0; j < coords; j++) { a [i].c [j] = u.RNDfromCI (rangeMin [j], rangeMax [j]); a [i].c [j] = u.SeInDiSp (a [i].c [j], rangeMin [j], rangeMax [j], rangeStep [j]); } } revision = true; return; } //---------------------------------------------------------------------------- double rnd = 0.0; int papIND = 0; int mamIND = 0; int pBloodType = 0; int mBloodType = 0; int cBloodType = 0; int bloodIND = 0; for (int i = 0; i < popSize; i++) { rnd = u.RNDprobab (); rnd *= rnd; papIND = (int)u.Scale (rnd, 0.0, 1.0, 0, popSize - 1); rnd = u.RNDprobab (); rnd *= rnd; mamIND = (int)u.Scale (rnd, 0.0, 1.0, 0, popSize - 1); pBloodType = GetBloodType (papIND); mBloodType = GetBloodType (mamIND); for (int c = 0; c < coords; c++) { bloodIND = MathRand () % ArraySize (ma [mBloodType - 1].pa [pBloodType - 1].bTypes); cBloodType = ma [mBloodType - 1].pa [pBloodType - 1].bTypes [bloodIND]; if (cBloodType == 1) a [i].c [c] = cB [c]; else { if (u.RNDbool () < 0.5) a [i].c [c] = p [papIND].c [c]; else a [i].c [c] = p [mamIND].c [c]; GetBloodMutation (a [i].c [c], c, cBloodType); a [i].c [c] = u.SeInDiSp (a [i].c [c], rangeMin [c], rangeMax [c], rangeStep [c]); } } } } //——————————————————————————————————————————————————————————————————————————————
Метод "GetBloodType" определяет тип крови на основе переданного индекса "ind" - текущего положения в популяции. Таким образом, метод служит для сопоставления индексов с типами крови, используя простую арифметическую операцию с остатком. Это позволяет циклически распределить типы крови среди имеющихся индексов (0-3).
//—————————————————————————————————————————————————————————————————————————————— int C_AO_BIO::GetBloodType (int ind) { if (ind % 4 == 0) return 1; if (ind % 4 == 1) return 2; if (ind % 4 == 2) return 3; if (ind % 4 == 3) return 4; return 1; } //——————————————————————————————————————————————————————————————————————————————
Метод "GetBloodMutation" предназначен для модификации (мутации) значения генетического параметра (гена) в зависимости от его типа крови и индекса.
Параметры:
- gene — ссылка на значение гена, которое будет изменено
- indGene — индекс гена, который используется для получения диапазонов мутации
- bloodType — тип крови, что определяет логику мутации
Тип крови 2 — применяется степенное распределение "PowerDistribution" к значению гена, что меняет ген на основе заданного диапазона, вероятностно распределяя значения вокруг него.
Тип крови 3 — ген увеличивается на долю разности между лучшим текущим значением гена по популяции "cB [indGene]" и текущим значением гена. Доля смещения определяется случайным числом [0.0; 1.0].
Другие типы крови (по умолчанию) — ген изменяется таким образом, что его новое значение становится симметричным заданному диапазону (инверсионным), находясь между "rangeMin [indGene]" и "rangeMax [indGene]".
//—————————————————————————————————————————————————————————————————————————————— void C_AO_BIO::GetBloodMutation (double &gene, int indGene, int bloodType) { switch (bloodType) { case 2: gene = u.PowerDistribution (gene, rangeMin [indGene], rangeMax [indGene], 20); return; case 3: gene += (cB [indGene] - gene) * u.RNDprobab (); return; default: { gene = rangeMax [indGene] - (gene - rangeMin [indGene]); } } } //——————————————————————————————————————————————————————————————————————————————
Метод "Revision" отвечает за обновление и сортировку популяции в алгоритме BIO. В первом цикле "for" (от 0 до popSize) метод проходит по всем членам популяции "a [i]". Если значение функции приспособленности "f" текущего члена популяции "a[i].f" превышает текущее лучшее значение "fB", то обновляется "fB" новым значением, и копируются координаты "c" текущего члена популяции в массив "cB". Во втором цикле "for" текущие члены популяции "a [i]" копируются в конец массива "p", начиная с индекса "popSize". Затем создается временный массив "pT", который вдвое больше текущей популяции "popSize * 2". Вызывается метод сортировки "u.Sorting" для того, чтобы отсортировать объединенный массив "p", сохранив результаты в "pT".
//—————————————————————————————————————————————————————————————————————————————— void C_AO_BIO::Revision () { //---------------------------------------------------------------------------- for (int i = 0; i < popSize; i++) { // Обновляем лучшее глобальное решение if (a [i].f > fB) { fB = a [i].f; ArrayCopy (cB, a [i].c, 0, 0, WHOLE_ARRAY); } } //---------------------------------------------------------------------------- for (int i = 0; i < popSize; i++) { p [popSize + i] = a [i]; } S_AO_Agent pT []; ArrayResize (pT, popSize * 2); u.Sorting (p, pT, popSize * 2); } //——————————————————————————————————————————————————————————————————————————————
Результаты тестов
Алгоритм тестировался на трех различных тестовых функциях (Hilly, Forest и Megacity) с разными размерностями пространства поиска (5*2, 25*2 и 500*2 измерений) при 10 000 вычислений целевой функции. Общий результат в 53.80% говорит о том, что BIO занимает среднюю позицию среди популяционных алгоритмов оптимизации, что весьма неплохо для нового метода.
=============================
5 Hilly's; Func runs: 10000; result: 0.8156790458423091
25 Hilly's; Func runs: 10000; result: 0.6533623929914842
500 Hilly's; Func runs: 10000; result: 0.3087659267627686
=============================
5 Forest's; Func runs: 10000; result: 0.8993708810337727
25 Forest's; Func runs: 10000; result: 0.6531872390668734
500 Forest's; Func runs: 10000; result: 0.21759965952460583
=============================
5 Megacity's; Func runs: 10000; result: 0.6784615384615384
25 Megacity's; Func runs: 10000; result: 0.4763076923076923
500 Megacity's; Func runs: 10000; result: 0.13901538461538585
=============================
All score: 4.84175 (53.80%)
Единственная проблема, которую можно заметить на визуализации работы алгоритма, это тенденция к застреванию в локальных оптимумах на задачах малых размерностей, что встречается достаточно часто у популяционных алгоритмов.
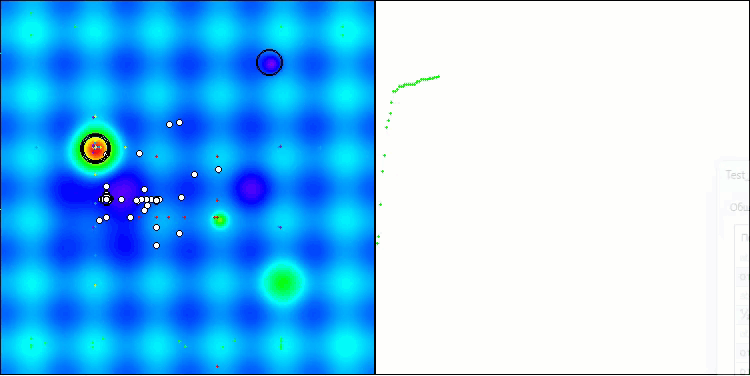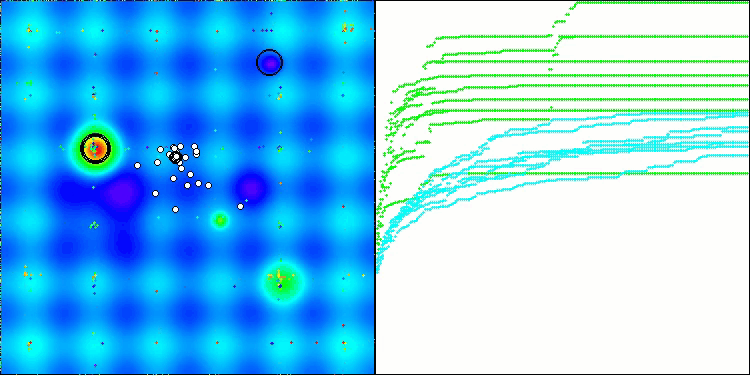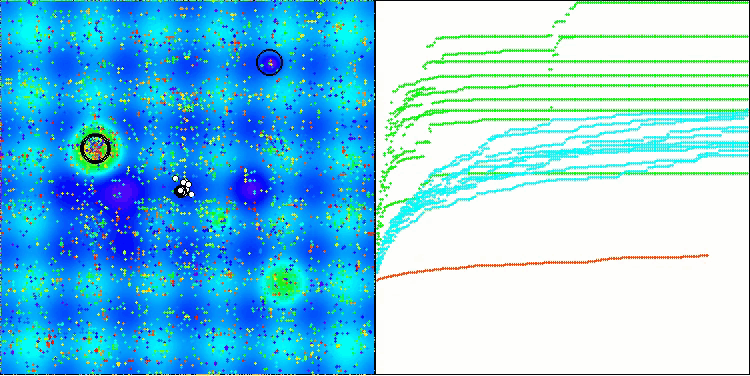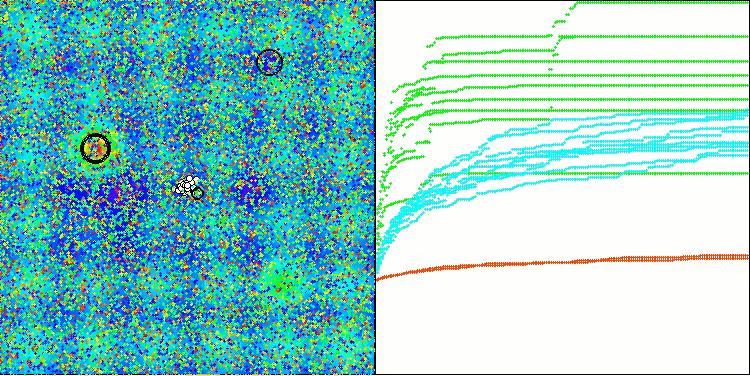
BIO на тестовой функции Hilly
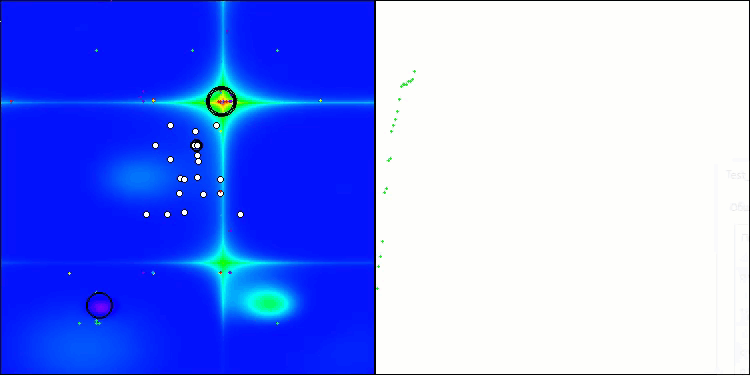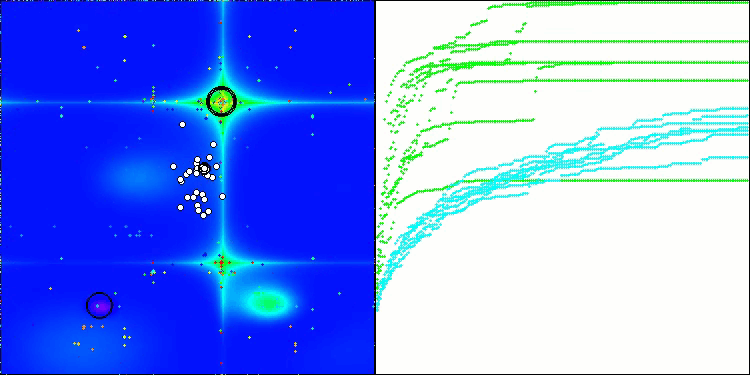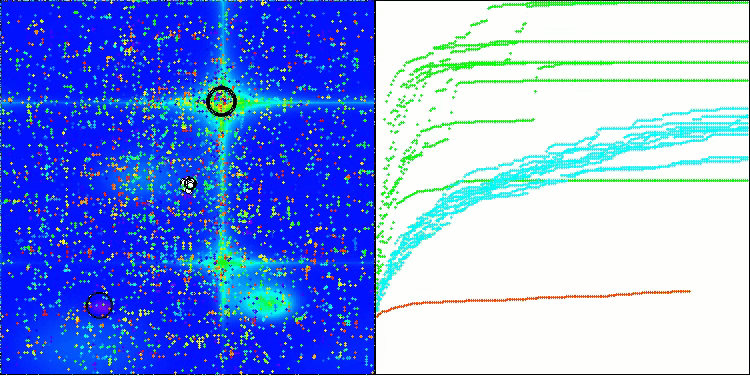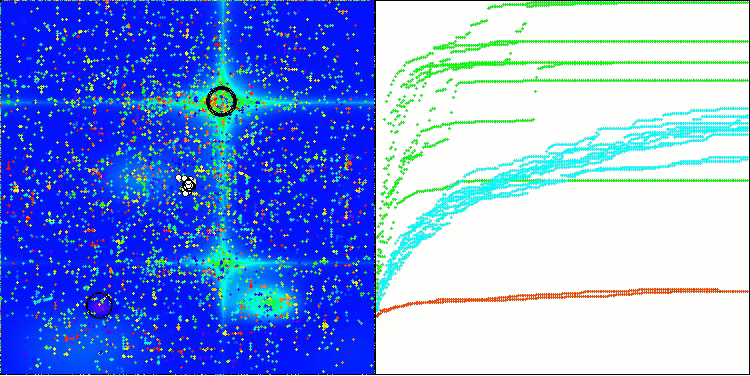
BIO на тестовой функции Forest
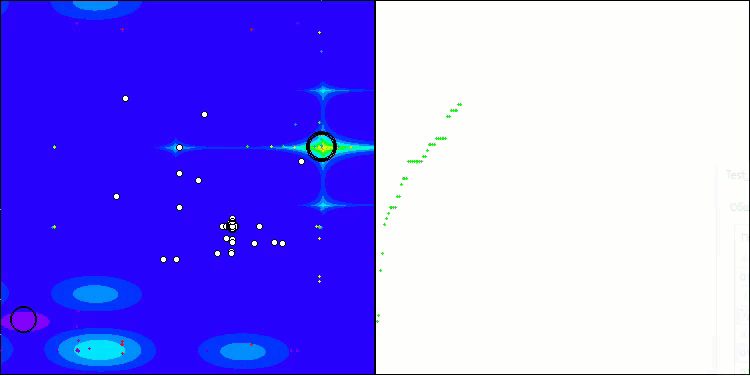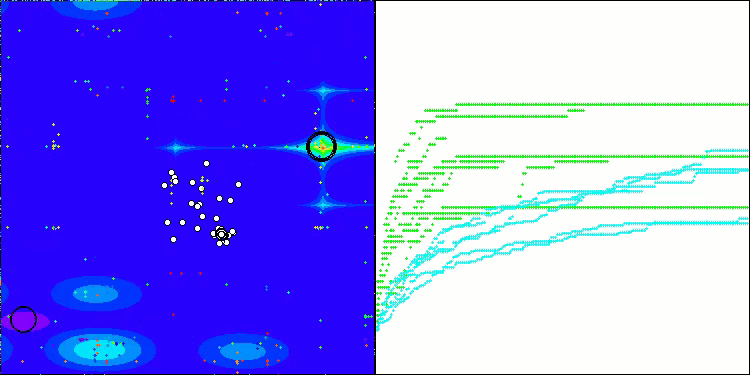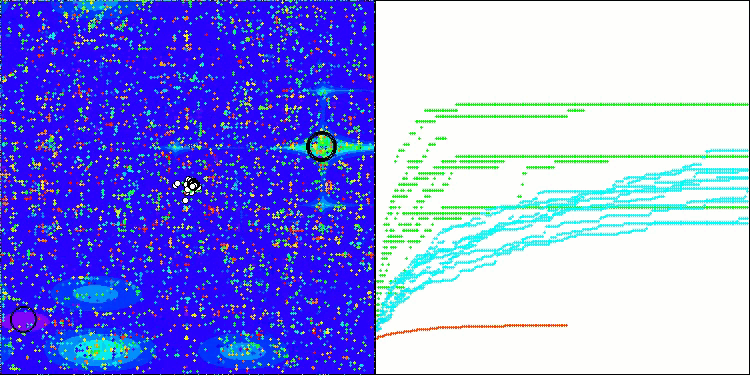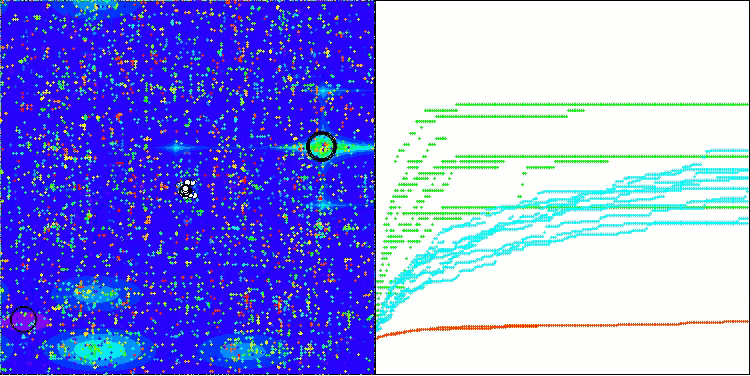
BIO на тестовой функции Megacity
По итогам тестирования алгоритм BIO занимает 20-ю позицию в рейтинговой таблице популяционных алгоритмов оптимизации.
| № | AO | Description | Hilly | Hilly final | Forest | Forest final | Megacity (discrete) | Megacity final | Final result | % of MAX | ||||||
| 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | ||||||||
| 1 | ANS | across neighbourhood search | 0,94948 | 0,84776 | 0,43857 | 2,23581 | 1,00000 | 0,92334 | 0,39988 | 2,32323 | 0,70923 | 0,63477 | 0,23091 | 1,57491 | 6,134 | 68,15 |
| 2 | CLA | code lock algorithm (joo) | 0,95345 | 0,87107 | 0,37590 | 2,20042 | 0,98942 | 0,91709 | 0,31642 | 2,22294 | 0,79692 | 0,69385 | 0,19303 | 1,68380 | 6,107 | 67,86 |
| 3 | AMOm | animal migration ptimization M | 0,90358 | 0,84317 | 0,46284 | 2,20959 | 0,99001 | 0,92436 | 0,46598 | 2,38034 | 0,56769 | 0,59132 | 0,23773 | 1,39675 | 5,987 | 66,52 |
| 4 | (P+O)ES | (P+O) evolution strategies | 0,92256 | 0,88101 | 0,40021 | 2,20379 | 0,97750 | 0,87490 | 0,31945 | 2,17185 | 0,67385 | 0,62985 | 0,18634 | 1,49003 | 5,866 | 65,17 |
| 5 | CTA | comet tail algorithm (joo) | 0,95346 | 0,86319 | 0,27770 | 2,09435 | 0,99794 | 0,85740 | 0,33949 | 2,19484 | 0,88769 | 0,56431 | 0,10512 | 1,55712 | 5,846 | 64,96 |
| 6 | TETA | time evolution travel algorithm (joo) | 0,91362 | 0,82349 | 0,31990 | 2,05701 | 0,97096 | 0,89532 | 0,29324 | 2,15952 | 0,73462 | 0,68569 | 0,16021 | 1,58052 | 5,797 | 64,41 |
| 7 | SDSm | stochastic diffusion search M | 0,93066 | 0,85445 | 0,39476 | 2,17988 | 0,99983 | 0,89244 | 0,19619 | 2,08846 | 0,72333 | 0,61100 | 0,10670 | 1,44103 | 5,709 | 63,44 |
| 8 | AAm | archery algorithm M | 0,91744 | 0,70876 | 0,42160 | 2,04780 | 0,92527 | 0,75802 | 0,35328 | 2,03657 | 0,67385 | 0,55200 | 0,23738 | 1,46323 | 5,548 | 61,64 |
| 9 | ESG | evolution of social groups (joo) | 0,99906 | 0,79654 | 0,35056 | 2,14616 | 1,00000 | 0,82863 | 0,13102 | 1,95965 | 0,82333 | 0,55300 | 0,04725 | 1,42358 | 5,529 | 61,44 |
| 10 | SIA | simulated isotropic annealing (joo) | 0,95784 | 0,84264 | 0,41465 | 2,21513 | 0,98239 | 0,79586 | 0,20507 | 1,98332 | 0,68667 | 0,49300 | 0,09053 | 1,27020 | 5,469 | 60,76 |
| 11 | ACS | artificial cooperative search | 0,75547 | 0,74744 | 0,30407 | 1,80698 | 1,00000 | 0,88861 | 0,22413 | 2,11274 | 0,69077 | 0,48185 | 0,13322 | 1,30583 | 5,226 | 58,06 |
| 12 | DA | dialectical algorithm | 0,86183 | 0,70033 | 0,33724 | 1,89940 | 0,98163 | 0,72772 | 0,28718 | 1,99653 | 0,70308 | 0,45292 | 0,16367 | 1,31967 | 5,216 | 57,95 |
| 13 | BHAm | black hole algorithm M | 0,75236 | 0,76675 | 0,34583 | 1,86493 | 0,93593 | 0,80152 | 0,27177 | 2,00923 | 0,65077 | 0,51646 | 0,15472 | 1,32195 | 5,196 | 57,73 |
| 14 | ASO | anarchy society optimization | 0,84872 | 0,74646 | 0,31465 | 1,90983 | 0,96148 | 0,79150 | 0,23803 | 1,99101 | 0,57077 | 0,54062 | 0,16614 | 1,27752 | 5,178 | 57,54 |
| 15 | RFO | royal flush optimization (joo) | 0,83361 | 0,73742 | 0,34629 | 1,91733 | 0,89424 | 0,73824 | 0,24098 | 1,87346 | 0,63154 | 0,50292 | 0,16421 | 1,29867 | 5,089 | 56,55 |
| 16 | AOSm | atomic orbital search M | 0,80232 | 0,70449 | 0,31021 | 1,81702 | 0,85660 | 0,69451 | 0,21996 | 1,77107 | 0,74615 | 0,52862 | 0,14358 | 1,41835 | 5,006 | 55,63 |
| 17 | TSEA | turtle shell evolution algorithm (joo) | 0,96798 | 0,64480 | 0,29672 | 1,90949 | 0,99449 | 0,61981 | 0,22708 | 1,84139 | 0,69077 | 0,42646 | 0,13598 | 1,25322 | 5,004 | 55,60 |
| 18 | DE | differential evolution | 0,95044 | 0,61674 | 0,30308 | 1,87026 | 0,95317 | 0,78896 | 0,16652 | 1,90865 | 0,78667 | 0,36033 | 0,02953 | 1,17653 | 4,955 | 55,06 |
| 19 | CRO | chemical reaction optimisation | 0,94629 | 0,66112 | 0,29853 | 1,90593 | 0,87906 | 0,58422 | 0,21146 | 1,67473 | 0,75846 | 0,42646 | 0,12686 | 1,31178 | 4,892 | 54,36 |
| 20 | BIO | blood inheritance optimization (joo) | 0,81568 | 0,65336 | 0,30877 | 1,77781 | 0,89937 | 0,65319 | 0,21760 | 1,77016 | 0,67846 | 0,47631 | 0,13902 | 1,29378 | 4,842 | 53,80 |
| 21 | BSA | bird swarm algorithm | 0,89306 | 0,64900 | 0,26250 | 1,80455 | 0,92420 | 0,71121 | 0,24939 | 1,88479 | 0,69385 | 0,32615 | 0,10012 | 1,12012 | 4,809 | 53,44 |
| 22 | HS | harmony search | 0,86509 | 0,68782 | 0,32527 | 1,87818 | 0,99999 | 0,68002 | 0,09590 | 1,77592 | 0,62000 | 0,42267 | 0,05458 | 1,09725 | 4,751 | 52,79 |
| 23 | SSG | saplings sowing and growing | 0,77839 | 0,64925 | 0,39543 | 1,82308 | 0,85973 | 0,62467 | 0,17429 | 1,65869 | 0,64667 | 0,44133 | 0,10598 | 1,19398 | 4,676 | 51,95 |
| 24 | BCOm | bacterial chemotaxis optimization M | 0,75953 | 0,62268 | 0,31483 | 1,69704 | 0,89378 | 0,61339 | 0,22542 | 1,73259 | 0,65385 | 0,42092 | 0,14435 | 1,21912 | 4,649 | 51,65 |
| 25 | ABO | african buffalo optimization | 0,83337 | 0,62247 | 0,29964 | 1,75548 | 0,92170 | 0,58618 | 0,19723 | 1,70511 | 0,61000 | 0,43154 | 0,13225 | 1,17378 | 4,634 | 51,49 |
| 26 | (PO)ES | (PO) evolution strategies | 0,79025 | 0,62647 | 0,42935 | 1,84606 | 0,87616 | 0,60943 | 0,19591 | 1,68151 | 0,59000 | 0,37933 | 0,11322 | 1,08255 | 4,610 | 51,22 |
| 27 | TSm | tabu search M | 0,87795 | 0,61431 | 0,29104 | 1,78330 | 0,92885 | 0,51844 | 0,19054 | 1,63783 | 0,61077 | 0,38215 | 0,12157 | 1,11449 | 4,536 | 50,40 |
| 28 | BSO | brain storm optimization | 0,93736 | 0,57616 | 0,29688 | 1,81041 | 0,93131 | 0,55866 | 0,23537 | 1,72534 | 0,55231 | 0,29077 | 0,11914 | 0,96222 | 4,498 | 49,98 |
| 29 | WOAm | wale optimization algorithm M | 0,84521 | 0,56298 | 0,26263 | 1,67081 | 0,93100 | 0,52278 | 0,16365 | 1,61743 | 0,66308 | 0,41138 | 0,11357 | 1,18803 | 4,476 | 49,74 |
| 30 | AEFA | artificial electric field algorithm | 0,87700 | 0,61753 | 0,25235 | 1,74688 | 0,92729 | 0,72698 | 0,18064 | 1,83490 | 0,66615 | 0,11631 | 0,09508 | 0,87754 | 4,459 | 49,55 |
| 31 | AEO | artificial ecosystem-based optimization algorithm | 0,91380 | 0,46713 | 0,26470 | 1,64563 | 0,90223 | 0,43705 | 0,21400 | 1,55327 | 0,66154 | 0,30800 | 0,28563 | 1,25517 | 4,454 | 49,49 |
| 32 | ACOm | ant colony optimization M | 0,88190 | 0,66127 | 0,30377 | 1,84693 | 0,85873 | 0,58680 | 0,15051 | 1,59604 | 0,59667 | 0,37333 | 0,02472 | 0,99472 | 4,438 | 49,31 |
| 33 | BFO-GA | bacterial foraging optimization - ga | 0,89150 | 0,55111 | 0,31529 | 1,75790 | 0,96982 | 0,39612 | 0,06305 | 1,42899 | 0,72667 | 0,27500 | 0,03525 | 1,03692 | 4,224 | 46,93 |
| 34 | SOA | simple optimization algorithm | 0,91520 | 0,46976 | 0,27089 | 1,65585 | 0,89675 | 0,37401 | 0,16984 | 1,44060 | 0,69538 | 0,28031 | 0,10852 | 1,08422 | 4,181 | 46,45 |
| 35 | ABHA | artificial bee hive algorithm | 0,84131 | 0,54227 | 0,26304 | 1,64663 | 0,87858 | 0,47779 | 0,17181 | 1,52818 | 0,50923 | 0,33877 | 0,10397 | 0,95197 | 4,127 | 45,85 |
| 36 | ACMO | atmospheric cloud model optimization | 0,90321 | 0,48546 | 0,30403 | 1,69270 | 0,80268 | 0,37857 | 0,19178 | 1,37303 | 0,62308 | 0,24400 | 0,10795 | 0,97503 | 4,041 | 44,90 |
| 37 | ADAMm | adaptive moment estimation M | 0,88635 | 0,44766 | 0,26613 | 1,60014 | 0,84497 | 0,38493 | 0,16889 | 1,39880 | 0,66154 | 0,27046 | 0,10594 | 1,03794 | 4,037 | 44,85 |
| 38 | ATAm | artificial tribe algorithm M | 0,71771 | 0,55304 | 0,25235 | 1,52310 | 0,82491 | 0,55904 | 0,20473 | 1,58867 | 0,44000 | 0,18615 | 0,09411 | 0,72026 | 3,832 | 42,58 |
| 39 | ASHA | artificial showering algorithm | 0,89686 | 0,40433 | 0,25617 | 1,55737 | 0,80360 | 0,35526 | 0,19160 | 1,35046 | 0,47692 | 0,18123 | 0,09774 | 0,75589 | 3,664 | 40,71 |
| 40 | ASBO | adaptive social behavior optimization | 0,76331 | 0,49253 | 0,32619 | 1,58202 | 0,79546 | 0,40035 | 0,26097 | 1,45677 | 0,26462 | 0,17169 | 0,18200 | 0,61831 | 3,657 | 40,63 |
| 41 | MEC | mind evolutionary computation | 0,69533 | 0,53376 | 0,32661 | 1,55569 | 0,72464 | 0,33036 | 0,07198 | 1,12698 | 0,52500 | 0,22000 | 0,04198 | 0,78698 | 3,470 | 38,55 |
| 42 | CSA | circle search algorithm | 0,66560 | 0,45317 | 0,29126 | 1,41003 | 0,68797 | 0,41397 | 0,20525 | 1,30719 | 0,37538 | 0,23631 | 0,10646 | 0,71815 | 3,435 | 38,17 |
| 43 | IWO | invasive weed optimization | 0,72679 | 0,52256 | 0,33123 | 1,58058 | 0,70756 | 0,33955 | 0,07484 | 1,12196 | 0,42333 | 0,23067 | 0,04617 | 0,70017 | 3,403 | 37,81 |
| 44 | Micro-AIS | micro artificial immune system | 0,79547 | 0,51922 | 0,30861 | 1,62330 | 0,72956 | 0,36879 | 0,09398 | 1,19233 | 0,37667 | 0,15867 | 0,02802 | 0,56335 | 3,379 | 37,54 |
| 45 | COAm | cuckoo optimization algorithm M | 0,75820 | 0,48652 | 0,31369 | 1,55841 | 0,74054 | 0,28051 | 0,05599 | 1,07704 | 0,50500 | 0,17467 | 0,03380 | 0,71347 | 3,349 | 37,21 |
| RW | random walk | 0,48754 | 0,32159 | 0,25781 | 1,06694 | 0,37554 | 0,21944 | 0,15877 | 0,75375 | 0,27969 | 0,14917 | 0,09847 | 0,52734 | 2,348 | 26,09 | |
Выводы
В процессе разработки и тестирования алгоритма Blood Inheritance Optimization (BIO), я пришел к нескольким важным выводам. Прежде всего, использование ассоциации наследования групп крови оказалось удачным подходом к организации различных стратегий мутации в популяционном алгоритме оптимизации. Тестирование на различных функциях и размерностях показало, что алгоритм достаточно универсален и способен эффективно работать как с простыми задачами малой размерности, так и с более сложными многомерными проблемами.
Особенно важно отметить, что представленная реализация BIO — это лишь базовая версия, демонстрирующая концепцию. Ключевая идея алгоритма заключается не столько в конкретных операторах мутации (которые могут быть заменены на любые другие), сколько в самой структуре наследования стратегий изменения параметров через аналогию с группами крови. Это открывает широкие возможности для модификации и расширения алгоритма. Каждая "группа крови" может быть ассоциирована с любыми другими операторами мутации, заимствованными из других алгоритмов или созданными специально под конкретную задачу. Более того, можно экспериментировать с количеством "групп крови", добавляя новые стратегии или комбинируя существующие.
Текущие результаты тестирования, показывающие достойную позицию в рейтинге популяционных алгоритмов (с результатом около 54%), говорят о работоспособности подхода даже в его базовой реализации. При этом замеченная тенденция к застреванию в локальных оптимумах может быть преодолена путем модификации операторов мутации или добавления новых стратегий исследования пространства решений.
Наиболее перспективным направлением развития алгоритма я вижу в создании адаптивных версий, где операторы мутации для каждой "группы крови" могут динамически меняться в процессе оптимизации, подстраиваясь под ландшафт целевой функции. Также интересным представляется исследование возможности применения различных схем наследования, отличных от классической системы групп крови "ABO", что может привести к созданию целого семейства алгоритмов, основанных на различных биологических системах наследования.
Таким образом, BIO представляет собой не просто еще один алгоритм оптимизации, а гибкую концептуальную основу для создания семейства алгоритмов, объединенных общей идеей наследования стратегий поиска решений через метафору групп крови и открывает широкие возможности для дальнейших исследований и модификаций, направленных на повышение эффективности алгоритма в различных областях применения.
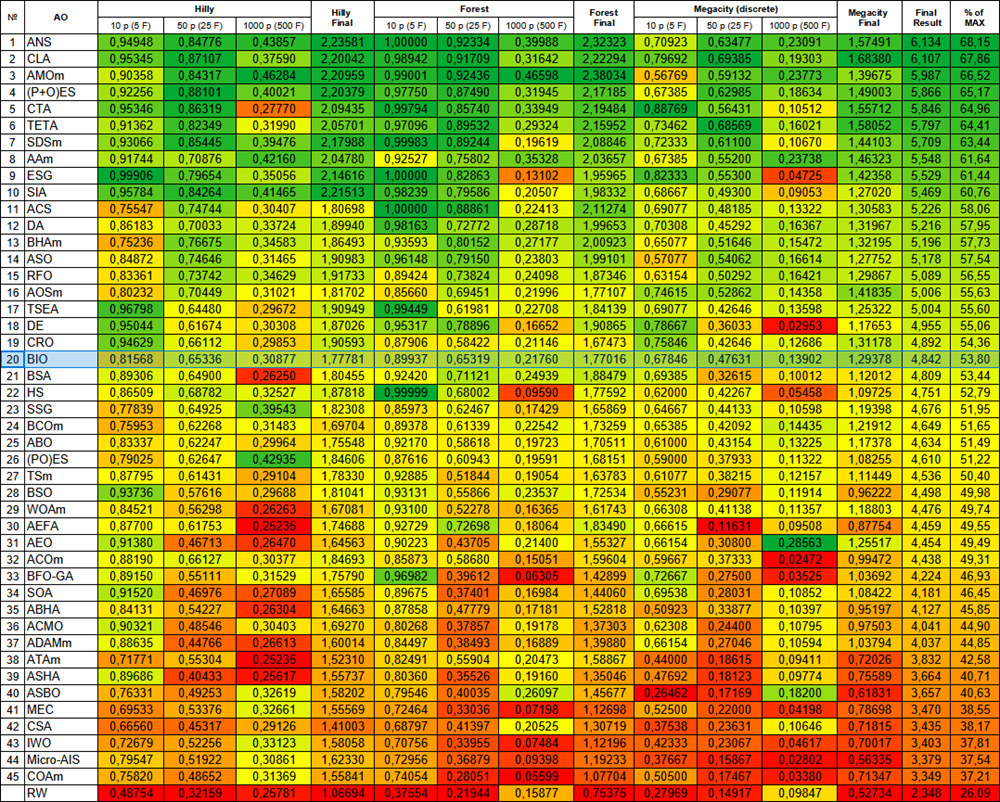
Рисунок 2. Цветовая градация алгоритмов по соответствующим тестам
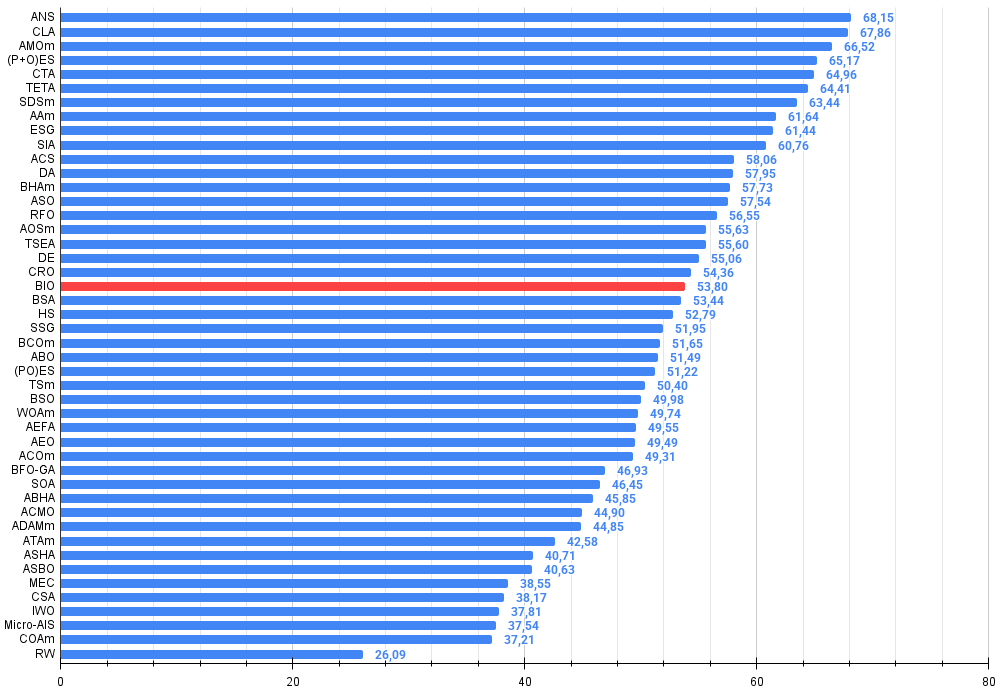
Рисунок 3. Гистограмма результатов тестирования алгоритмов (по шкале от 0 до 100, чем больше, тем лучше, где 100 — максимально возможный теоретический результат, в архиве скрипт для расчета рейтинговой таблицы)
Плюсы и минусы алгоритма BIO:
Плюсы:
- Отсутствуют внешние параметры
- Интересная идея наследования по группам крови
- Хорошая сходимость на функциях высокой и средней размерности
Минусы:
- Застревает в локальных экстремумах на задачах малой размерности.
К статье прикреплён архив с актуальными версиями кодов алгоритмов. Автор статьи не несёт ответственности за абсолютную точность в описании канонических алгоритмов, во многие из них были внесены изменения для улучшения поисковых возможностей. Выводы и суждения, представленные в статьях, основываются на результатах проведённых экспериментов.
Программы, используемые в статье
| # | Имя | Тип | Описание |
|---|---|---|---|
| 1 | #C_AO.mqh | Включаемый файл | Родительский класс популяционных алгоритмов оптимизации |
| 2 | #C_AO_enum.mqh | Включаемый файл | Перечисление популяционных алгоритмов оптимизации |
| 3 | TestFunctions.mqh | Включаемый файл | Библиотека тестовых функций |
| 4 | TestStandFunctions.mqh | Включаемый файл | Библиотека функций тестового стенда |
| 5 | Utilities.mqh | Включаемый файл | Библиотека вспомогательных функций |
| 6 | CalculationTestResults.mqh | Включаемый файл | Скрипт для расчета результатов в сравнительную таблицу |
| 7 | Testing AOs.mq5 | Скрипт | Единый испытательный стенд для всех популяционных алгоритмов оптимизации |
| 8 | Simple use of population optimization algorithms.mq5 | Скрипт | Простой пример использования популяционных алгоритмов оптимизации без визуализации |
| 9 | Test_AO_BIO.mq5 | Скрипт | Испытательный стенд для BIO |
Предупреждение: все права на данные материалы принадлежат MetaQuotes Ltd. Полная или частичная перепечатка запрещена.
Данная статья написана пользователем сайта и отражает его личную точку зрения. Компания MetaQuotes Ltd не несет ответственности за достоверность представленной информации, а также за возможные последствия использования описанных решений, стратегий или рекомендаций.
 Как функции столетней давности могут обновить ваши торговые стратегии
Как функции столетней давности могут обновить ваши торговые стратегии
 Нейросети в трейдинге: Гибридные модели последовательностей графов (GSM++)
Нейросети в трейдинге: Гибридные модели последовательностей графов (GSM++)
 Распознавание паттернов с использованием динамической трансформации временной шкалы в MQL5
Распознавание паттернов с использованием динамической трансформации временной шкалы в MQL5
 Интеграция MQL5: Python
Интеграция MQL5: Python
- Бесплатные приложения для трейдинга
- 8 000+ сигналов для копирования
- Экономические новости для анализа финансовых рынков
Вы принимаете политику сайта и условия использования