
Алгоритм миграции животных — Animal Migration Optimization (AMO)
Введение
Миграция животных: гармония и стратегия природы. Животные обычно мигрируют между местами зимовки и размножения, следуя пройденным путям, выработанным веками эволюции. Эти сезонные путешествия не являются случайными блужданиями, а представляют собой тщательно спланированные маршруты, ведущие к районам, наиболее благоприятным для их выживания и размножения. В зависимости от времени года, животные перемещаются в поисках пищи, укрытия и подходящих условий для воспроизводства, руководствуясь естественными потребностями своей стаи и инстинктами. Каждое такое путешествие — это не только борьба за существование, но и проявление гармонии с природой, где каждая особь играет уникальную роль в общей экосистеме.
Например, северные олени мигрируют на огромные расстояния в поисках лучших пастбищ, а птицы, такие как журавли и гуси, совершают долгие перелеты между зимовками и местами гнездования, используя определенные маршруты, которые передаются из поколения в поколение. Эти миграции не только обеспечивают выживание отдельных видов, но и поддерживают экосистему в целом, способствуя опылению растений и распространению семян.
Вдохновение от природы. Алгоритм AMO (Animal Migration Optimization) был предложен в 2013 году исследователем Сяньтао Ли. Основная идея этого алгоритма заключается в моделировании процесса сезонной миграции животных в поисках оптимальных условий для жизни и размножения в природе. Вдохновением для создания алгоритма послужило наблюдение за поведением мигрирующих животных, таких как птицы, рыбы и млекопитающие. Эти животные совершают сезонные перемещения между местами зимовки и размножения, следуя определенным выработанным природой правилам взаимодействия во время миграции.
Алгоритм AMO имитирует три основных составляющих при передвижении животных на длительные расстояния: исключение столкновений с соседними особями, движение в том же направлении, что и стая (группа), и поддержание достаточного расстояния между собой. Эти принципы помогают не только избежать конфликтов, но и поддерживать коллективное поведение, что критически важно для выживания в дикой природе.
Этапы оптимизации в алгоритме AMO. В алгоритм заложены два ключевых этапа оптимизации на одной итерации:
- Процесс миграции: во время этого этапа происходит обновление позиции особи с учетом ее соседей.
- Обновление популяции: на этом этапе происходит частичная замена особей на новых, с вероятностью, зависящей от положения особи в стае.
Моделирование коллективного поведения мигрирующих животных может быть эффективным подходом к решению сложных оптимизационных задач. Алгоритм пытается сбалансировать исследование пространства поиска и эксплуатацию лучших найденных решений, подражая естественным процессам, происходящим в природе.
2. Реализации алгоритма
В данной алгоритмической модели миграции животных основная концепция заключается в создании концентрических "зон" вокруг каждого животного. В зоне отталкивания фокусное животное стремится удалиться от своих соседей, чтобы избежать столкновений. Алгоритм миграции животных по авторской мысли делится на два основных процесса:
1. Процесс миграции животных. Для описания ограниченного соседства индивидов используется топологическое кольцо. Для удобства длина соседства устанавливается равной пяти для каждого измерения. Топология соседства остается стационарной и определяется на основе индексов особи в популяции. Если индекс животного равен "j", то его соседи имеют индексы [j - 2, j - 1, j, j + 1 и j + 2]. В случае, если индекс животного равен "1", его соседи будут иметь индексы [N - 1, N, 1, 2, 3] и так далее. После формирования топологии соседства случайным образом выбирается один сосед, и позиция индивида обновляется с корректировкой на положение этого соседа. Такое описание дают авторы оригинального алгоритма, в данном случае ограничение по количеству соседей можно вынести в параметры алгоритма, для того чтобы с помощью экспериментов найти наилучшее количество соседей для обеспечения максимально возможной результативности алгоритма.
2. Процесс обновления популяции. В процессе обновления популяции алгоритм моделирует, как некоторые животные покидают группу, а другие присоединяются к популяции. Индивиды могут быть заменены новыми животными с вероятностью "p", которая определяется на основе качества функции приспособленности. Мы сортируем популяцию в порядке убывания значений фитнес-функции, что означает, что вероятность изменения индивида с наилучшей приспособленностью составляет "1/N", в то время как у индивида с наихудшим значением она равна "1".
Процесс миграции животных и процесс обновления популяции, согласно версии автора, можно расписать алгоритмически, как показано ниже.
Процесс миграции животных:
1. Для каждого животного: a. Определение топологического соседства животного (5 ближайших соседей).b. Выбор случайного соседа из списка соседей.
c. Обновление позиции животного в направлении выбранного соседа по формуле:
x_j_new = x_j_old + r * (x_neighbor - x_j_old), где:
- x_j_new — новая позиция j-го животного,
- x_j_old — текущая позиция,
- x_neighbor — позиция выбранного соседа,
- r — случайное число из нормального распределения.
d. Оценка новой позиции животного с помощью целевой функции.
Процесс обновления популяции:
1. Сортировка животных по значению целевой функции в порядке убывания. 2. Для каждого животного: a. Вычисление вероятности замены животного новым случайным животным:p = 1.0 - (1.0 / (x + 1)), где x — ранг (индекс) i-го животного в отсортированном списке.
b. Если случайное число меньше p, то заменить животное (изменить координату на среднее значение координат двух случайно выбранных из популяции животных). c. Иначе, оставить животное без изменений.3. Оценка новой популяции с помощью целевой функции.
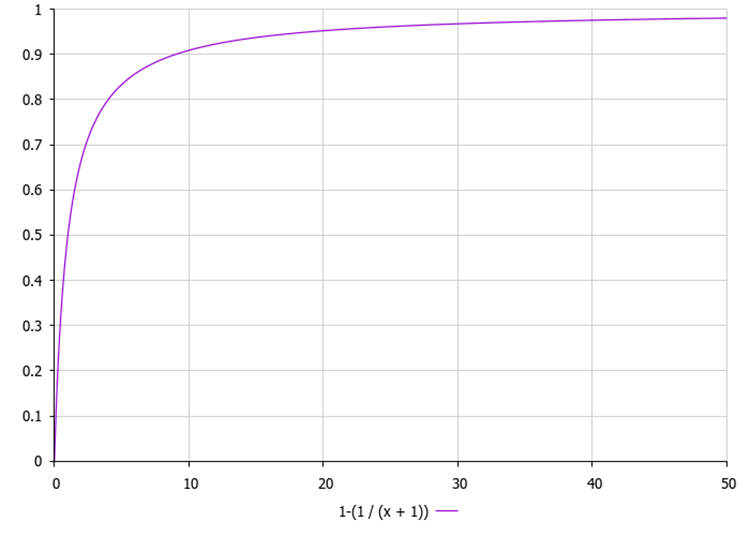
Рисунок 1. График вероятности изменения для особи, в зависимости от ее положения в популяции, где "x" - индекс особи в популяции
Перейдем к написанию псевдокода алгоритма миграции животных AMO.
1. Инициализация популяции животных со случайными позициями.
2. Основной цикл:
a. Для каждого животного:
Определение топологического соседства.Выбор случайного соседа.
Обновление позиции животного в направлении соседа.
Оценка новой позиции.
b. Сортировка популяции по значениям целевой функции.
c. Вероятностная замена худших животных новыми.
d. Оценка обновленной популяции.
e. Обновление лучшего решения.
3. Повторять с п.2 до выполнения критерия останова.
Теперь, когда мы ознакомились с алгоритмом, можем перейти к написанию кода. Давайте подробно рассмотрим код класса "C_AO_AMO":
1. Класс "C_AO_AMO" наследуется от базового класса "C_AO", что позволяет использовать его функциональность и расширять её.
2. В конструкторе задаются основные характеристики алгоритма, такие как имя, описание и ссылка на статью. Также инициализируются параметры алгоритма, включая размер популяции, отклонение и количество соседей.
3. popSize, deviation, neighborsNumberOnSide — переменные определяют размер популяции, стандартное отклонение и количество соседей с одной стороны, которые будут учитываться при миграции.
4. SetParams — метод отвечает за обновление параметров алгоритма на основе значений, хранящихся в массиве "params".
5. Init — метод инициализации, который принимает массивы для минимальных и максимальных значений диапазона, шагов и количество эпох.
6. Moving () — метод отвечает за перемещение агентов в пространстве поиска, "Revision ()" — метод проверяет и обновляет состояние агентов в популяции.
7. S_AO_Agent population [] — массив хранит текущую популяцию агентов (животных), "S_AO_Agent pTemp []" — временный массив для использования при сортировке популяции.
8. GetNeighborsIndex — приватный метод, используется для получения индексов соседей для конкретного агента.
Класс "C_AO_AMO" реализует алгоритм оптимизации на основе миграции животных, предоставляя необходимые методы и параметры для настройки и выполнения алгоритма. Он наследует функциональность от базового класса, что позволяет расширять и модифицировать его поведение в зависимости от требований задачи.
//—————————————————————————————————————————————————————————————————————————————— class C_AO_AMOm : public C_AO { public: //-------------------------------------------------------------------- ~C_AO_AMOm () { } C_AO_AMOm () { ao_name = "AMOm"; ao_desc = "Animal Migration Optimization M"; ao_link = "https://www.mql5.com/ru/articles/15543"; popSize = 50; // Размер популяции deviation = 8; neighborsNumberOnSide = 10; ArrayResize (params, 3); params [0].name = "popSize"; params [0].val = popSize; params [1].name = "deviation"; params [1].val = deviation; params [2].name = "neighborsNumberOnSide"; params [2].val = neighborsNumberOnSide; } void SetParams () { popSize = (int)params [0].val; deviation = params [1].val; neighborsNumberOnSide = (int)params [2].val; } bool Init (const double &rangeMinP [], const double &rangeMaxP [], const double &rangeStepP [], const int epochsP = 0); void Moving (); void Revision (); //---------------------------------------------------------------------------- double deviation; int neighborsNumberOnSide; S_AO_Agent population []; // Популяция животных S_AO_Agent pTemp []; // Временная популяция животных private: //------------------------------------------------------------------- int GetNeighborsIndex (int i); }; //——————————————————————————————————————————————————————————————————————————————
Далее рассмотрим код метода "Init" класса "C_AO_AMO". Описание каждой части метода:
1. rangeMinP [], rangeMaxP [], rangeStepP [] — массивы для определения минимальных, максимальных диапазонов оптимизируемых параметров и их шагов.
2. Метод "StandardInit" выполняет стандартную инициализацию на основе переданных диапазонов.
3. Изменение размера массивов "population" и "pTemp" на "popSize".
4. Инициализация агентов проходит по всем элементам массива "population" и инициализирует каждого агента с помощью метода "Init", передавая ему количество координат "coords".
5. Если все операции выполнены успешно, метод возвращает "true".
Метод "Init" класса "C_AO_AMO" отвечает за инициализацию популяции агентов с учетом заданных диапазонов и параметров.
//—————————————————————————————————————————————————————————————————————————————— bool C_AO_AMO::Init (const double &rangeMinP [], const double &rangeMaxP [], const double &rangeStepP [], const int epochsP = 0) { if (!StandardInit (rangeMinP, rangeMaxP, rangeStepP)) return false; //---------------------------------------------------------------------------- ArrayResize (population, popSize); ArrayResize (pTemp, popSize); for (int i = 0; i < popSize; i++) population [i].Init (coords); return true; } //——————————————————————————————————————————————————————————————————————————————
Следующим разберем метод "Moving" класса "C_AO_AMO", который отвечает за движение агентов в популяции. Основные части кода:
1. Проверка состояния "revision":
- Если "revision" равно "false", это означает, что метод вызывается в первый раз или после сброса. В этом случае происходит инициализация популяции.
- Для каждого индивидуума популяции (от "0" до "popSize") и для каждой координаты (от "0" до "coords") генерируются случайные значения в заданном диапазоне ("rangeMin" и "rangeMax").
- Затем эти значения обрабатываются функцией "SeInDiSp", которая корректирует их с учетом заданного шага ("rangeStep").
2. Установка флага "revision":
- После инициализации "revision" устанавливается в "true", и метод завершается.
3. Основной цикл миграции:
- Если "revision" равно "true", метод переходит к основной логике миграции.
- Для каждого индивидуума снова происходит итерация по координатам.
- GetNeighborsIndex(i) используется для получения индекса соседа, с которым будет сравниваться текущий индивидуум.
- Вычисляется расстояние "dist" между значениями координат соседа и текущего индивидуума.
- На основе этого расстояния определяются минимальные и максимальные границы ("min" и "max"), в которых находится новое значение координаты.
- Если вычисленные границы выходят за пределы допустимого диапазона, они корректируются с учетом "rangeMin" и "rangeMax".
- Затем новое значение координаты рассчитывается с использованием нормального распределения (функция "GaussDistribution"), что позволяет учитывать стандартное отклонение ("deviation").
- Как и в первом случае, новое значение также обрабатывается с помощью "SeInDiSp".
Метод "Moving" отвечает за обновление позиций агентов в зависимости от их соседей и случайных факторов.
//—————————————————————————————————————————————————————————————————————————————— void C_AO_AMO::Moving () { //---------------------------------------------------------------------------- if (!revision) { for (int i = 0; i < popSize; i++) { for (int c = 0; c < coords; c++) { a [i].c [c] = u.RNDfromCI (rangeMin [c], rangeMax [c]); a [i].c [c] = u.SeInDiSp (a [i].c [c], rangeMin [c], rangeMax [c], rangeStep [c]); } } revision = true; return; } //---------------------------------------------------------------------------- int ind1 = 0; double dist = 0.0; double x = 0.0; double min = 0.0; double max = 0.0; for (int i = 0; i < popSize; i++) { for (int c = 0; c < coords; c++) { //------------------------------------------------------------------------ ind1 = GetNeighborsIndex (i); dist = fabs (population [ind1].c [c] - a [i].c [c]); x = a [i].c [c]; min = x - dist; max = x + dist; if (min < rangeMin [c]) min = rangeMin [c]; if (max > rangeMax [c]) max = rangeMax [c]; a [i].c [c] = u.GaussDistribution (x, min, max, deviation); a [i].c [c] = u.SeInDiSp (a [i].c [c], rangeMin [c], rangeMax [c], rangeStep [c]); } } } //——————————————————————————————————————————————————————————————————————————————
Далее код представляет собой метод "Revision" класса "C_AO_AMO". Рассмотрим его по частям:
1. Поиск лучшего индивидуума:
- Переменная "ind" используется для хранения индекса индивидуума с наилучшей функцией ("f").
- Проходя по всей популяции (от "0" до "popSize"), код обновляет значение "fB", если текущий индивидуум имеет лучшее значение функции.
- Если найден лучший индивидуум, его характеристики (координаты) копируются в массив "cB".
- Для каждого индивидуума в популяции (от "0" до "popSize") вычисляется вероятность "prob", которая зависит от индекса "i".
- Для каждой координаты (от "0" до "coords") происходит случайное сравнение с вероятностью "prob".
- Если случайное число меньше "prob", выбираются два случайных индивидуума "ind1" и "ind2".
- Если оба индивидуума совпадают, "ind2" увеличивается, чтобы избежать выбора одного и того же индивидуума.
- Новое значение координаты текущего индивидуума вычисляется как среднее значение координат двух случайных индивидуумов, а затем корректируется с помощью функции "SeInDiSp", которая ограничивает значение в заданном диапазоне.
- После завершения изменений, вся популяция обновляется, копируя значения из массива "a".
- Затем вызывается функция "Sorting", которая сортирует популяцию по значению функции "f".
//—————————————————————————————————————————————————————————————————————————————— void C_AO_AMO::Revision () { //---------------------------------------------------------------------------- int ind = -1; for (int i = 0; i < popSize; i++) { if (a [i].f > fB) { fB = a [i].f; ind = i; } } if (ind != -1) ArrayCopy (cB, a [ind].c, 0, 0, WHOLE_ARRAY); //---------------------------------------------------------------------------- int ind1 = 0; int ind2 = 0; double dist = 0.0; double x = 0.0; double min = 0.0; double max = 0.0; double prob = 0.0; for (int i = 0; i < popSize; i++) { prob = 1.0 - (1.0 / (i + 1)); for (int c = 0; c < coords; c++) { if (u.RNDprobab() < prob) { ind1 = u.RNDminusOne (popSize); ind2 = u.RNDminusOne (popSize); if (ind1 == ind2) { ind2++; if (ind2 > popSize - 1) ind2 = 0; } a [i].c [c] = (population [ind1].c [c] + population [ind2].c [c]) * 0.5; a [i].c [c] = u.SeInDiSp (a [i].c [c], rangeMin [c], rangeMax [c], rangeStep [c]); } } } //---------------------------------------------------------------------------- for (int i = 0; i < popSize; i++) population [i] = a [i]; u.Sorting (population, pTemp, popSize); } //——————————————————————————————————————————————————————————————————————————————
Последним разберем код метода "GetNeighborsIndex" класса "C_AO_AMO", который отвечает за получение индекса случайного соседа для заданного индекса "i" с учетом границ массива.
1. Инициализация переменных:
- Ncount — количество соседей, определяемое переменной "neighborsNumberOnSide".
- N — общее количество соседей, включая сам элемент, определяется как "Ncount * 2 + 1".
2. Метод использует условные операторы для проверки положения индекса "i" относительно границ массива.
3. Обработка первых "Ncount" элементов (границ слева). Если индекс "i" меньше "Ncount", это означает, что он находится в начале массива. В этом случае метод генерирует случайный индекс соседа от "0" до "N-1".
4. Обработка последних "Ncount" элементов (границ справа). Если индекс "i" больше или равен "popSize - Ncount", это означает, что он находится в конце массива. Метод генерирует индекс соседа, начиная с "popSize - N", чтобы учесть границы.
5. Обработка всех остальных элементов. Если индекс "i" находится где-то в середине массива, метод генерирует индекс соседа, который смещается на "Ncount" влево и добавляет случайное значение от "0" до "N-1".
6. В конце метод возвращает сгенерированный индекс соседа.
Метод "GetNeighborsIndex" позволяет получить индекс случайного соседа для заданного индекса "i" с учетом границ массива.
//—————————————————————————————————————————————————————————————————————————————— int C_AO_AMO::GetNeighborsIndex (int i) { int Ncount = neighborsNumberOnSide; int N = Ncount * 2 + 1; int neighborIndex; // Выбор случайного соседа с учетом границ массива if (i < Ncount) { // Для первых Ncount элементов neighborIndex = MathRand () % N; } else { if (i >= popSize - Ncount) { // Для последних Ncount элементов neighborIndex = (popSize - N) + MathRand () % N; } else { // Для всех остальных элементов neighborIndex = i - Ncount + MathRand () % N; } } return neighborIndex; } //——————————————————————————————————————————————————————————————————————————————
Теперь, как только мы закончили написание алгоритма, проверим как он работает. Результаты оригинальной версии алгоритма:
AMO|Animal Migration Optimization|50.0|1.0|2.0|
=============================
5 Hilly's; Func runs: 10000; result: 0.43676335174918435
25 Hilly's; Func runs: 10000; result: 0.28370099295372453
500 Hilly's; Func runs: 10000; result: 0.25169663266864406
=============================
5 Forest's; Func runs: 10000; result: 0.312993148861033
25 Forest's; Func runs: 10000; result: 0.23960515885149344
500 Forest's; Func runs: 10000; result: 0.18938496103401775
=============================
5 Megacity's; Func runs: 10000; result: 0.18461538461538463
25 Megacity's; Func runs: 10000; result: 0.12246153846153851
500 Megacity's; Func runs: 10000; result: 0.10223076923076994
=============================
All score: 2.12345 (23.59%)
К сожалению, оригинальная версия показывает слабые поисковые качества, такие показатели не будем заносить в рейтинговую таблицу. Анализ результатов выявил существенное отставание от других участников, что побудило меня к глубокому переосмыслению алгоритма.
При детальном рассмотрении стратегии, обнаружился ключевой недостаток: сортировка популяции не способствовала накоплению генетического материала лучших особей. Она лишь изменяла их топологическое расположение, не затрагивая сущность. Влияние сортировки ограничивалось лишь корректировкой вероятности изменения координат особей в пространстве поиска, причем эта вероятность обратно пропорциональна их приспособленности.
Примечательно, что новые координаты формировались исключительно на основе уже существующих в популяции, путем усреднения значений двух случайно выбранных особей. Осознание этих нюансов привело к идее расширения популяции с целью интеграции дочерних особей в родительскую группу перед проведением сортировки. Такой подход позволит не только сохранять лучшие генетические комбинации, но и естественным образом вытеснять менее приспособленные особи. Несомненно, проблема обновления генофонда популяции остается актуальной, однако предложенная модификация должна значительно повысить динамику эволюционного процесса. Для реализации этой идеи начнем с изменения метода инициализации, увеличив размер родительской популяции вдвое.
Приведем полностью код инициализации, где видно увеличение родительской популяции вдвое.
//—————————————————————————————————————————————————————————————————————————————— bool C_AO_AMOm::Init (const double &rangeMinP [], const double &rangeMaxP [], const double &rangeStepP [], const int epochsP = 0) { if (!StandardInit (rangeMinP, rangeMaxP, rangeStepP)) return false; //---------------------------------------------------------------------------- ArrayResize (population, popSize * 2); ArrayResize (pTemp, popSize * 2); for (int i = 0; i < popSize * 2; i++) population [i].Init (coords); return true; } //——————————————————————————————————————————————————————————————————————————————
Соответственно необходимо поправить метод "Revision":
//---------------------------------------------------------------------------- for (int i = 0; i < popSize; i++) population [i + popSize] = a [i]; u.Sorting (population, pTemp, popSize * 2);
После соответствующих поправок еще раз протестируем алгоритм и сравним результаты:
AMOm|Animal Migration Optimization M|50.0|1.0|2.0|
=============================
5 Hilly's; Func runs: 10000; result: 0.4759595972704031
25 Hilly's; Func runs: 10000; result: 0.31711543296080447
500 Hilly's; Func runs: 10000; result: 0.2540492181444619
=============================
5 Forest's; Func runs: 10000; result: 0.40387880560608347
25 Forest's; Func runs: 10000; result: 0.27049305409901064
500 Forest's; Func runs: 10000; result: 0.19135802944407254
=============================
5 Megacity's; Func runs: 10000; result: 0.23692307692307696
25 Megacity's; Func runs: 10000; result: 0.14461538461538465
500 Megacity's; Func runs: 10000; result: 0.10109230769230851
=============================
All score: 2.39548 (26.62%)
В данном случае видим улучшение общего результата на 3%, что говорит о шансах к успеху на выбранном пути.
Далее, попробуем перенести вероятностное изменение особей в зависимости от ранга в метод "Moving", это позволит производить изменения координат особей сразу же после получения новых координат от ближайших соседей.
//---------------------------------------------------------------------------- int ind1 = 0; int ind2 = 0; double dist = 0.0; double x = 0.0; double min = 0.0; double max = 0.0; double prob = 0.0; for (int i = 0; i < popSize; i++) { prob = 1.0 - (1.0 / (i + 1)); for (int c = 0; c < coords; c++) { //------------------------------------------------------------------------ ind1 = GetNeighborsIndex (i); dist = fabs (population [ind1].c [c] - a [i].c [c]); x = a [i].c [c]; min = x - dist; max = x + dist; if (min < rangeMin [c]) min = rangeMin [c]; if (max > rangeMax [c]) max = rangeMax [c]; a [i].c [c] = u.GaussDistribution (x, min, max, deviation); //---------------------------------------------------------------------------- if (u.RNDprobab() < prob) { ind1 = u.RNDminusOne (popSize); ind2 = u.RNDminusOne (popSize); if (ind1 == ind2) { ind2++; if (ind2 > popSize - 1) ind2 = 0; } a [i].c [c] = (population [ind1].c [c] + population [ind2].c [c]) * 0.5; } a [i].c [c] = u.SeInDiSp (a [i].c [c], rangeMin [c], rangeMax [c], rangeStep [c]); } }
Снова проверим результаты:
AMO|Animal Migration Optimization|50.0|1.0|2.0|
=============================
5 Hilly's; Func runs: 10000; result: 0.7204154413083147
25 Hilly's; Func runs: 10000; result: 0.4480389094268583
500 Hilly's; Func runs: 10000; result: 0.25286213277651365
=============================
5 Forest's; Func runs: 10000; result: 0.7097109421461968
25 Forest's; Func runs: 10000; result: 0.3299544372347476
500 Forest's; Func runs: 10000; result: 0.18667655927410348
=============================
5 Megacity's; Func runs: 10000; result: 0.41076923076923083
25 Megacity's; Func runs: 10000; result: 0.20400000000000001
500 Megacity's; Func runs: 10000; result: 0.09586153846153929
=============================
All score: 3.35829 (37.31%)
Значительно лучше, стоит продолжить. И после некоторых экспериментов с кодом, у нас получился окончательный вариант метода "Moving":
//---------------------------------------------------------------------------- int ind1 = 0; int ind2 = 0; double dist = 0.0; double x = 0.0; double min = 0.0; double max = 0.0; double prob = 0.0; for (int i = 0; i < popSize; i++) { prob = 1.0 - (1.0 / (i + 1)); for (int c = 0; c < coords; c++) { //------------------------------------------------------------------------ ind1 = GetNeighborsIndex (i); dist = fabs (population [ind1].c [c] - a [i].c [c]); x = population [ind1].c [c]; min = x - dist; max = x + dist; if (min < rangeMin [c]) min = rangeMin [c]; if (max > rangeMax [c]) max = rangeMax [c]; a [i].c [c] = u.GaussDistribution (x, min, max, deviation); a [i].c [c] = u.SeInDiSp (a [i].c [c], rangeMin [c], rangeMax [c], rangeStep [c]); //------------------------------------------------------------------------ if (u.RNDprobab() < prob) { if (u.RNDprobab() <= 0.01) { ind1 = u.RNDminusOne (popSize); ind2 = u.RNDminusOne (popSize); //if (ind1 == ind2) { //ind2++; //if (ind2 > popSize - 1) ind2 = 0; a [i].c [c] = (population [ind1].c [c] + population [ind2].c [c]) * 0.5; a [i].c [c] = u.SeInDiSp (a [i].c [c], rangeMin [c], rangeMax [c], rangeStep [c]); } } //ind1 = u.RNDminusOne (popSize); //a [i].c [c] = population [ind1].c [c]; } } } } //——————————————————————————————————————————————————————————————————————————————
Переходим от метода "Moving" к окончательному варианту метода "Revision" класса "C_AO_AMO", который отвечает за обновление и сортировку популяции агентов.
//—————————————————————————————————————————————————————————————————————————————— void C_AO_AMO::Revision () { //---------------------------------------------------------------------------- int ind = -1; for (int i = 0; i < popSize; i++) { if (a [i].f > fB) { fB = a [i].f; ind = i; } } if (ind != -1) ArrayCopy (cB, a [ind].c, 0, 0, WHOLE_ARRAY); //---------------------------------------------------------------------------- for (int i = 0; i < popSize; i++) population [popSize + i] = a [i]; u.Sorting (population, pTemp, popSize * 2); } //——————————————————————————————————————————————————————————————————————————————По окончательному формированию кода, снова переходим к испытаниям.
3. Результаты тестов
Распечатка работы алгоритма AMO на стенде с тестовыми функциями:
AMOm|Animal Migration Optimization|50.0|8.0|10.0|
=============================
5 Hilly's; Func runs: 10000; result: 0.9627642143272663
25 Hilly's; Func runs: 10000; result: 0.8703754433240446
500 Hilly's; Func runs: 10000; result: 0.467183248460726
=============================
5 Forest's; Func runs: 10000; result: 0.9681183408862706
25 Forest's; Func runs: 10000; result: 0.9109372988714968
500 Forest's; Func runs: 10000; result: 0.4719026790932256
=============================
5 Megacity's; Func runs: 10000; result: 0.6676923076923076
25 Megacity's; Func runs: 10000; result: 0.5886153846153845
500 Megacity's; Func runs: 10000; result: 0.23546153846153978
=============================
All score: 6.14305 (68.26%)
Высокие результаты на лидерство в рейтинговой таблице, однако ценой их является высокий разброс итоговых значений на функциях малой размерности. Сделаем 50 испытаний, вместо обычных 10.
AMOm|Animal Migration Optimization|50.0|8.0|10.0|
=============================
5 Hilly's; Func runs: 10000; result: 0.903577388020872
25 Hilly's; Func runs: 10000; result: 0.8431723262743616
500 Hilly's; Func runs: 10000; result: 0.46284266807030283
=============================
5 Forest's; Func runs: 10000; result: 0.9900061169785055
25 Forest's; Func runs: 10000; result: 0.9243600311397848
500 Forest's; Func runs: 10000; result: 0.4659761237381695
=============================
5 Megacity's; Func runs: 10000; result: 0.5676923076923077
25 Megacity's; Func runs: 10000; result: 0.5913230769230771
500 Megacity's; Func runs: 10000; result: 0.23773230769230896
=============================
All score: 5.98668 (66.52%)
Теперь результаты более реалистичные, но слегка снизилась и результативность.
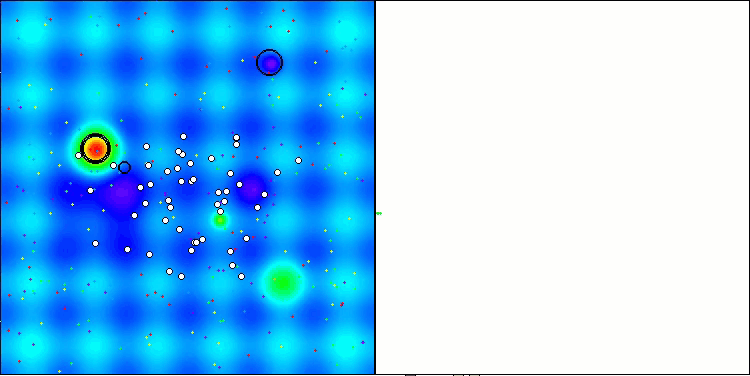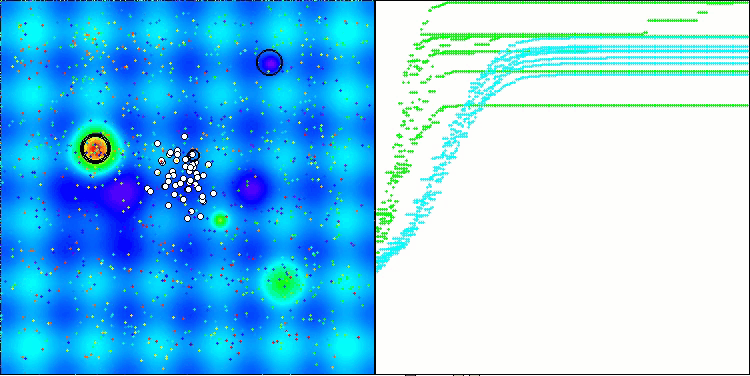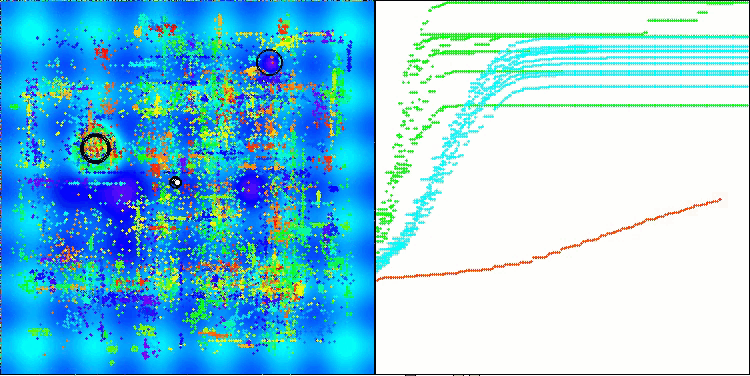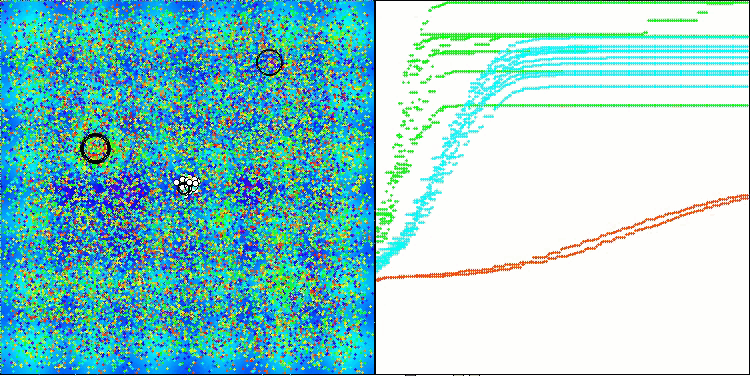
AMOm на тестовой функции Hilly
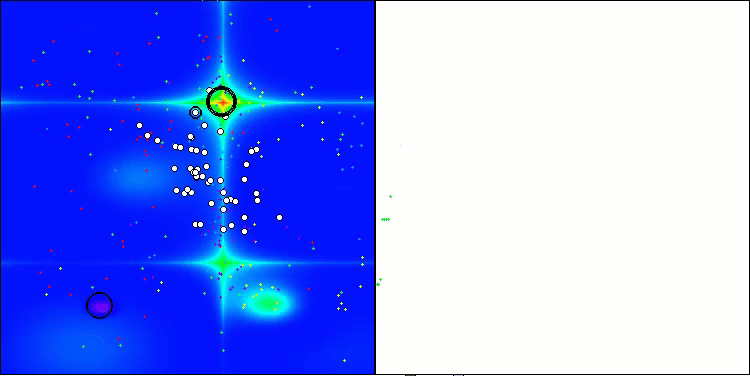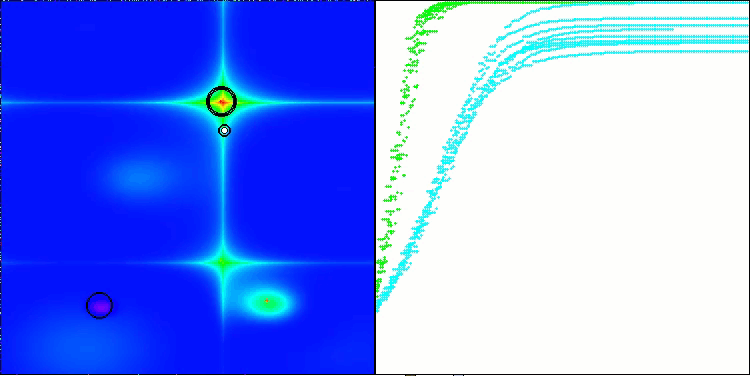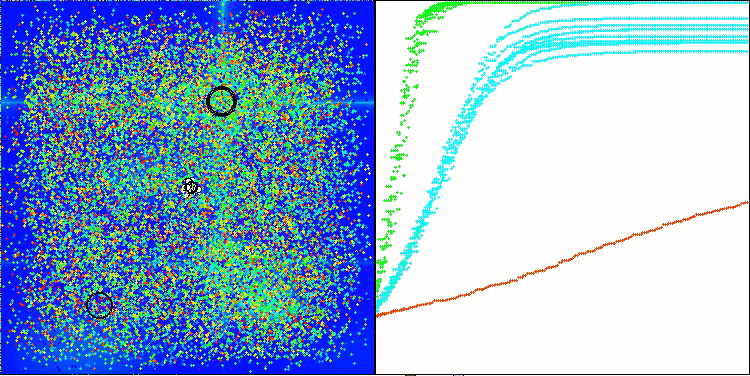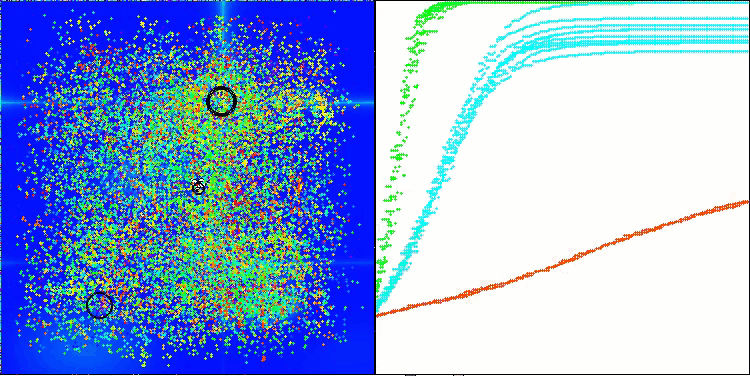
AMOm на тестовой функции Forest
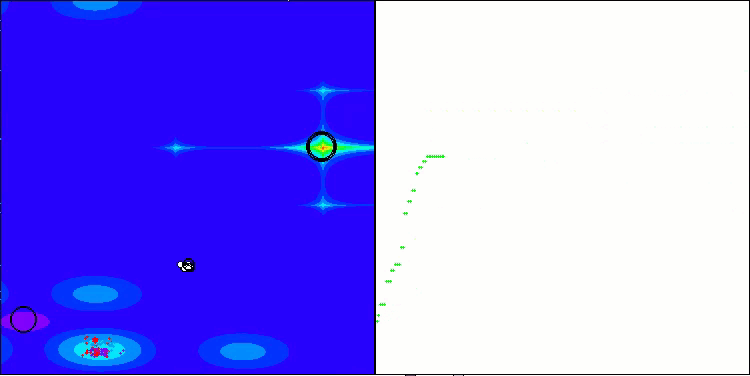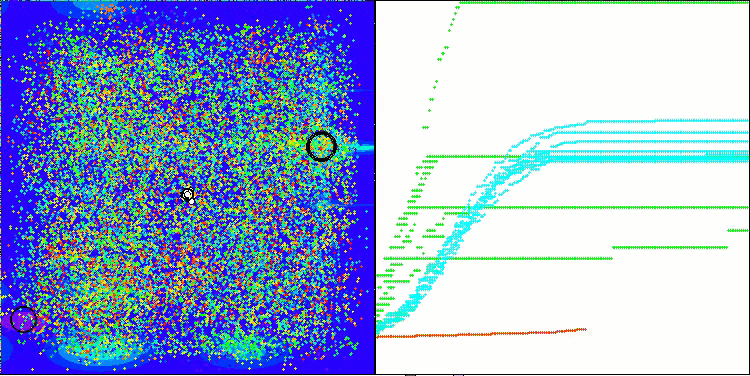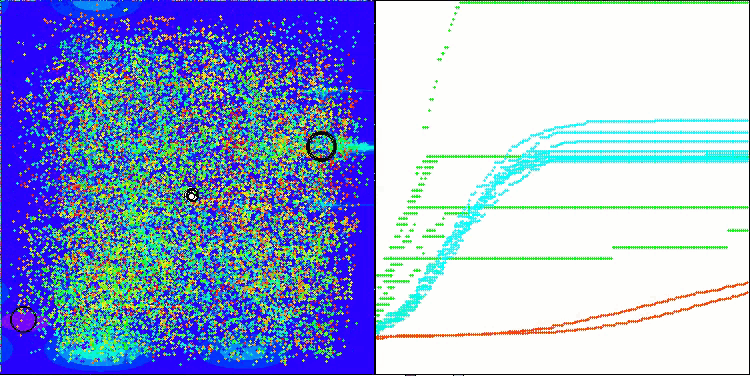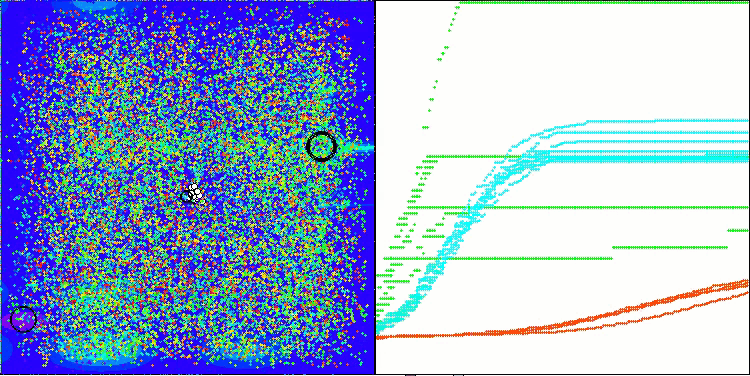
AMOm на тестовой функции Megacity
После удивительных превращений (помните, как в старом добром фильме "брюки превращаются...в элегантные шорты"?), алгоритм по результатам занимает уверенное 3 место в рейтинговой таблице.
| № | AO | Description | Hilly | Hilly final | Forest | Forest final | Megacity (discrete) | Megacity final | Final result | % of MAX | ||||||
| 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | ||||||||
| 1 | ANS | across neighbourhood search | 0,94948 | 0,84776 | 0,43857 | 2,23581 | 1,00000 | 0,92334 | 0,39988 | 2,32323 | 0,70923 | 0,63477 | 0,23091 | 1,57491 | 6,134 | 68,15 |
| 2 | CLA | code lock algorithm | 0,95345 | 0,87107 | 0,37590 | 2,20042 | 0,98942 | 0,91709 | 0,31642 | 2,22294 | 0,79692 | 0,69385 | 0,19303 | 1,68380 | 6,107 | 67,86 |
| 3 | AMOm | animal migration ptimization M | 0,90358 | 0,84317 | 0,46284 | 2,20959 | 0,99001 | 0,92436 | 0,46598 | 2,38034 | 0,56769 | 0,59132 | 0,23773 | 1,39675 | 5,987 | 66,52 |
| 4 | (P+O)ES | (P+O) evolution strategies | 0,92256 | 0,88101 | 0,40021 | 2,20379 | 0,97750 | 0,87490 | 0,31945 | 2,17185 | 0,67385 | 0,62985 | 0,18634 | 1,49003 | 5,866 | 65,17 |
| 5 | CTA | comet tail algorithm | 0,95346 | 0,86319 | 0,27770 | 2,09435 | 0,99794 | 0,85740 | 0,33949 | 2,19484 | 0,88769 | 0,56431 | 0,10512 | 1,55712 | 5,846 | 64,96 |
| 6 | SDSm | stochastic diffusion search M | 0,93066 | 0,85445 | 0,39476 | 2,17988 | 0,99983 | 0,89244 | 0,19619 | 2,08846 | 0,72333 | 0,61100 | 0,10670 | 1,44103 | 5,709 | 63,44 |
| 7 | ESG | evolution of social groups | 0,99906 | 0,79654 | 0,35056 | 2,14616 | 1,00000 | 0,82863 | 0,13102 | 1,95965 | 0,82333 | 0,55300 | 0,04725 | 1,42358 | 5,529 | 61,44 |
| 8 | SIA | simulated isotropic annealing | 0,95784 | 0,84264 | 0,41465 | 2,21513 | 0,98239 | 0,79586 | 0,20507 | 1,98332 | 0,68667 | 0,49300 | 0,09053 | 1,27020 | 5,469 | 60,76 |
| 9 | ACS | artificial cooperative search | 0,75547 | 0,74744 | 0,30407 | 1,80698 | 1,00000 | 0,88861 | 0,22413 | 2,11274 | 0,69077 | 0,48185 | 0,13322 | 1,30583 | 5,226 | 58,06 |
| 10 | ASO | anarchy society optimization | 0,84872 | 0,74646 | 0,31465 | 1,90983 | 0,96148 | 0,79150 | 0,23803 | 1,99101 | 0,57077 | 0,54062 | 0,16614 | 1,27752 | 5,178 | 57,54 |
| 11 | TSEA | turtle shell evolution algorithm | 0,96798 | 0,64480 | 0,29672 | 1,90949 | 0,99449 | 0,61981 | 0,22708 | 1,84139 | 0,69077 | 0,42646 | 0,13598 | 1,25322 | 5,004 | 55,60 |
| 12 | DE | differential evolution | 0,95044 | 0,61674 | 0,30308 | 1,87026 | 0,95317 | 0,78896 | 0,16652 | 1,90865 | 0,78667 | 0,36033 | 0,02953 | 1,17653 | 4,955 | 55,06 |
| 13 | CRO | chemical reaction optimisation | 0,94629 | 0,66112 | 0,29853 | 1,90593 | 0,87906 | 0,58422 | 0,21146 | 1,67473 | 0,75846 | 0,42646 | 0,12686 | 1,31178 | 4,892 | 54,36 |
| 14 | BSA | bird swarm algorithm | 0,89306 | 0,64900 | 0,26250 | 1,80455 | 0,92420 | 0,71121 | 0,24939 | 1,88479 | 0,69385 | 0,32615 | 0,10012 | 1,12012 | 4,809 | 53,44 |
| 15 | HS | harmony search | 0,86509 | 0,68782 | 0,32527 | 1,87818 | 0,99999 | 0,68002 | 0,09590 | 1,77592 | 0,62000 | 0,42267 | 0,05458 | 1,09725 | 4,751 | 52,79 |
| 16 | SSG | saplings sowing and growing | 0,77839 | 0,64925 | 0,39543 | 1,82308 | 0,85973 | 0,62467 | 0,17429 | 1,65869 | 0,64667 | 0,44133 | 0,10598 | 1,19398 | 4,676 | 51,95 |
| 17 | (PO)ES | (PO) evolution strategies | 0,79025 | 0,62647 | 0,42935 | 1,84606 | 0,87616 | 0,60943 | 0,19591 | 1,68151 | 0,59000 | 0,37933 | 0,11322 | 1,08255 | 4,610 | 51,22 |
| 18 | BSO | brain storm optimization | 0,93736 | 0,57616 | 0,29688 | 1,81041 | 0,93131 | 0,55866 | 0,23537 | 1,72534 | 0,55231 | 0,29077 | 0,11914 | 0,96222 | 4,498 | 49,98 |
| 19 | WOAm | wale optimization algorithm M | 0,84521 | 0,56298 | 0,26263 | 1,67081 | 0,93100 | 0,52278 | 0,16365 | 1,61743 | 0,66308 | 0,41138 | 0,11357 | 1,18803 | 4,476 | 49,74 |
| 20 | AEFA | artificial electric field algorithm | 0,87700 | 0,61753 | 0,25235 | 1,74688 | 0,92729 | 0,72698 | 0,18064 | 1,83490 | 0,66615 | 0,11631 | 0,09508 | 0,87754 | 4,459 | 49,55 |
| 21 | ACOm | ant colony optimization M | 0,88190 | 0,66127 | 0,30377 | 1,84693 | 0,85873 | 0,58680 | 0,15051 | 1,59604 | 0,59667 | 0,37333 | 0,02472 | 0,99472 | 4,438 | 49,31 |
| 22 | BFO-GA | bacterial foraging optimization - ga | 0,89150 | 0,55111 | 0,31529 | 1,75790 | 0,96982 | 0,39612 | 0,06305 | 1,42899 | 0,72667 | 0,27500 | 0,03525 | 1,03692 | 4,224 | 46,93 |
| 23 | ABHA | artificial bee hive algorithm | 0,84131 | 0,54227 | 0,26304 | 1,64663 | 0,87858 | 0,47779 | 0,17181 | 1,52818 | 0,50923 | 0,33877 | 0,10397 | 0,95197 | 4,127 | 45,85 |
| 24 | ASBO | adaptive social behavior optimization | 0,76331 | 0,49253 | 0,32619 | 1,58202 | 0,79546 | 0,40035 | 0,26097 | 1,45677 | 0,26462 | 0,17169 | 0,18200 | 0,61831 | 3,657 | 40,63 |
| 25 | MEC | mind evolutionary computation | 0,69533 | 0,53376 | 0,32661 | 1,55569 | 0,72464 | 0,33036 | 0,07198 | 1,12698 | 0,52500 | 0,22000 | 0,04198 | 0,78698 | 3,470 | 38,55 |
| 26 | IWO | invasive weed optimization | 0,72679 | 0,52256 | 0,33123 | 1,58058 | 0,70756 | 0,33955 | 0,07484 | 1,12196 | 0,42333 | 0,23067 | 0,04617 | 0,70017 | 3,403 | 37,81 |
| 27 | Micro-AIS | micro artificial immune system | 0,79547 | 0,51922 | 0,30861 | 1,62330 | 0,72956 | 0,36879 | 0,09398 | 1,19233 | 0,37667 | 0,15867 | 0,02802 | 0,56335 | 3,379 | 37,54 |
| 28 | COAm | cuckoo optimization algorithm M | 0,75820 | 0,48652 | 0,31369 | 1,55841 | 0,74054 | 0,28051 | 0,05599 | 1,07704 | 0,50500 | 0,17467 | 0,03380 | 0,71347 | 3,349 | 37,21 |
| 29 | SDOm | spiral dynamics optimization M | 0,74601 | 0,44623 | 0,29687 | 1,48912 | 0,70204 | 0,34678 | 0,10944 | 1,15826 | 0,42833 | 0,16767 | 0,03663 | 0,63263 | 3,280 | 36,44 |
| 30 | NMm | Nelder-Mead method M | 0,73807 | 0,50598 | 0,31342 | 1,55747 | 0,63674 | 0,28302 | 0,08221 | 1,00197 | 0,44667 | 0,18667 | 0,04028 | 0,67362 | 3,233 | 35,92 |
| 31 | FAm | firefly algorithm M | 0,58634 | 0,47228 | 0,32276 | 1,38138 | 0,68467 | 0,37439 | 0,10908 | 1,16814 | 0,28667 | 0,16467 | 0,04722 | 0,49855 | 3,048 | 33,87 |
| 32 | GSA | gravitational search algorithm | 0,64757 | 0,49197 | 0,30062 | 1,44016 | 0,53962 | 0,36353 | 0,09945 | 1,00260 | 0,32667 | 0,12200 | 0,01917 | 0,46783 | 2,911 | 32,34 |
| 33 | BFO | bacterial foraging optimization | 0,61171 | 0,43270 | 0,31318 | 1,35759 | 0,54410 | 0,21511 | 0,05676 | 0,81597 | 0,42167 | 0,13800 | 0,03195 | 0,59162 | 2,765 | 30,72 |
| 34 | ABC | artificial bee colony | 0,63377 | 0,42402 | 0,30892 | 1,36671 | 0,55103 | 0,21874 | 0,05623 | 0,82600 | 0,34000 | 0,14200 | 0,03102 | 0,51302 | 2,706 | 30,06 |
| 35 | BA | bat algorithm | 0,59761 | 0,45911 | 0,35242 | 1,40915 | 0,40321 | 0,19313 | 0,07175 | 0,66810 | 0,21000 | 0,10100 | 0,03517 | 0,34617 | 2,423 | 26,93 |
| 36 | SA | simulated annealing | 0,55787 | 0,42177 | 0,31549 | 1,29513 | 0,34998 | 0,15259 | 0,05023 | 0,55280 | 0,31167 | 0,10033 | 0,02883 | 0,44083 | 2,289 | 25,43 |
| 37 | IWDm | intelligent water drops M | 0,54501 | 0,37897 | 0,30124 | 1,22522 | 0,46104 | 0,14704 | 0,04369 | 0,65177 | 0,25833 | 0,09700 | 0,02308 | 0,37842 | 2,255 | 25,06 |
| 38 | PSO | particle swarm optimisation | 0,59726 | 0,36923 | 0,29928 | 1,26577 | 0,37237 | 0,16324 | 0,07010 | 0,60572 | 0,25667 | 0,08000 | 0,02157 | 0,35823 | 2,230 | 24,77 |
| 39 | Boids | boids algorithm | 0,43340 | 0,30581 | 0,25425 | 0,99346 | 0,35718 | 0,20160 | 0,15708 | 0,71586 | 0,27846 | 0,14277 | 0,09834 | 0,51957 | 2,229 | 24,77 |
| 40 | MA | monkey algorithm | 0,59107 | 0,42681 | 0,31816 | 1,33604 | 0,31138 | 0,14069 | 0,06612 | 0,51819 | 0,22833 | 0,08567 | 0,02790 | 0,34190 | 2,196 | 24,40 |
| 41 | SFL | shuffled frog-leaping | 0,53925 | 0,35816 | 0,29809 | 1,19551 | 0,37141 | 0,11427 | 0,04051 | 0,52618 | 0,27167 | 0,08667 | 0,02402 | 0,38235 | 2,104 | 23,38 |
| 42 | FSS | fish school search | 0,55669 | 0,39992 | 0,31172 | 1,26833 | 0,31009 | 0,11889 | 0,04569 | 0,47467 | 0,21167 | 0,07633 | 0,02488 | 0,31288 | 2,056 | 22,84 |
| 43 | RND | random | 0,52033 | 0,36068 | 0,30133 | 1,18234 | 0,31335 | 0,11787 | 0,04354 | 0,47476 | 0,25333 | 0,07933 | 0,02382 | 0,35648 | 2,014 | 22,37 |
| 44 | GWO | grey wolf optimizer | 0,59169 | 0,36561 | 0,29595 | 1,25326 | 0,24499 | 0,09047 | 0,03612 | 0,37158 | 0,27667 | 0,08567 | 0,02170 | 0,38403 | 2,009 | 22,32 |
| 45 | CSS | charged system search | 0,44252 | 0,35454 | 0,35201 | 1,14907 | 0,24140 | 0,11345 | 0,06814 | 0,42299 | 0,18333 | 0,06300 | 0,02322 | 0,26955 | 1,842 | 20,46 |
Выводы
Исходя из результатов работы алгоритма AMOm на тестовых функциях, можно сделать следующие выводы: несмотря на разброс значений на функциях малой размерности, алгоритм показывает отличную масштабируемость на функциях с большой размерностью. Основные изменения, внесенные в оригинальную версию алгоритма, значительно улучшили показатели его работы. В данном случае увеличение родительской популяции в два раза (для сортировки вместе с дочерними особями) и изменение последовательности выполнения этапов алгоритма дало возможность получения более широкого спектра разнообразных решений. Данный алгоритм стал ярким примером возможностей применения дополнительных приемов для его модификации, которые привели к значительным улучшениям, что следовало из продолжения улучшения самой логики алгоритма, что не всегда работает в отношении других алгоритмов оптимизации.
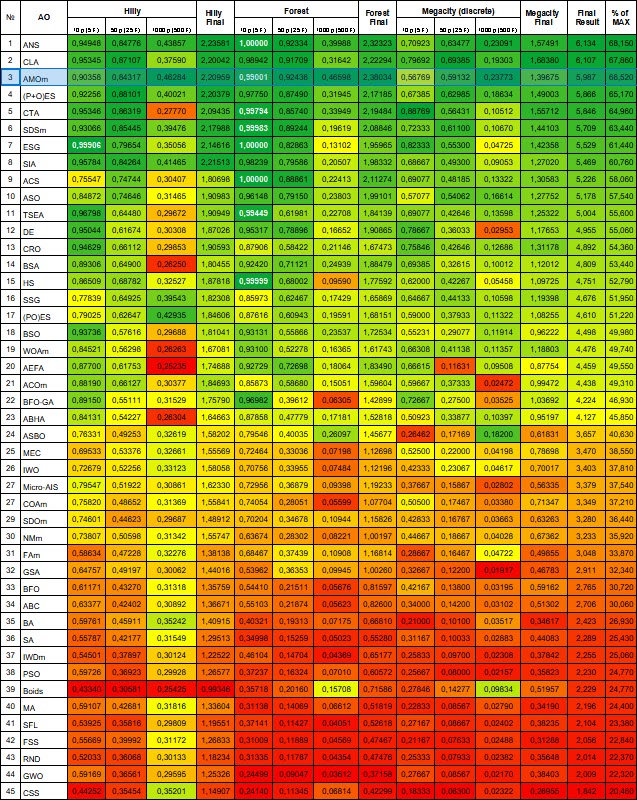
Рисунок 2. Цветовая градация алгоритмов по соответствующим тестам. Белым цветом подсвечены результаты больше или равные 0.99
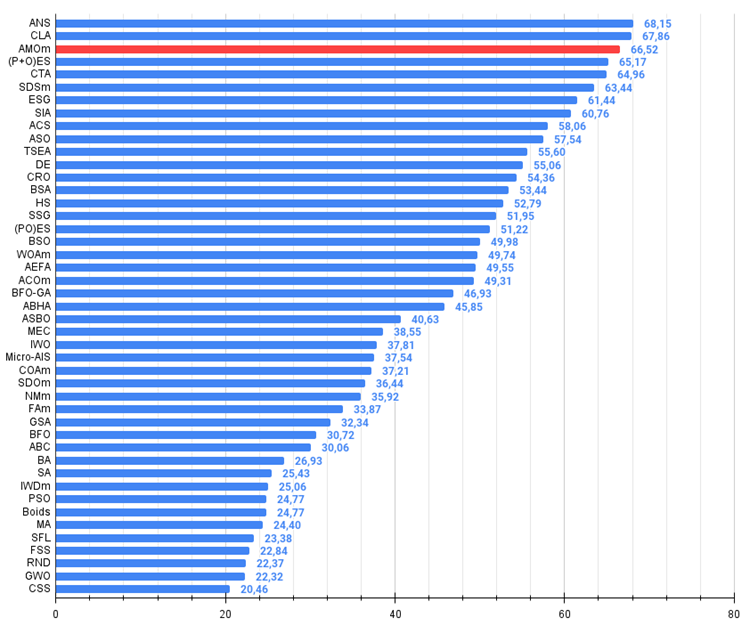
Рисунок 3. Гистограмма результатов тестирования алгоритмов (по шкале от 0 до 100, чем больше, тем лучше,
где 100 - максимально возможный теоретический результат, в архиве скрипт для расчета рейтинговой таблицы)
Плюсы и минусы алгоритма AMOm:
Плюсы:
- Отличная сходимость.
- Высокая масштабируемость.
- Небольшое количество параметров.
- Простая реализация.
Минусы:
- Разброс результатов на функциях малой размерности.
К статье прикреплён архив с актуальными версиями кодов алгоритмов. Автор статьи не несёт ответственности за абсолютную точность в описании канонических алгоритмов, во многие из них были внесены изменения для улучшения поисковых возможностей. Выводы и суждения, представленные в статьях, основываются на результатах проведённых экспериментов.
Предупреждение: все права на данные материалы принадлежат MetaQuotes Ltd. Полная или частичная перепечатка запрещена.
Данная статья написана пользователем сайта и отражает его личную точку зрения. Компания MetaQuotes Ltd не несет ответственности за достоверность представленной информации, а также за возможные последствия использования описанных решений, стратегий или рекомендаций.
 Методы Уильяма Ганна (Часть I): Создаем индикатор углов Ганна
Методы Уильяма Ганна (Часть I): Создаем индикатор углов Ганна
 Разработка системы репликации (Часть 44): Проект Chart Trade (III)
Разработка системы репликации (Часть 44): Проект Chart Trade (III)
 Упрощаем торговлю на новостях (Часть 1): Создаем базу данных
Упрощаем торговлю на новостях (Часть 1): Создаем базу данных
 Разбираем примеры торговых стратегий в клиентском терминале
Разбираем примеры торговых стратегий в клиентском терминале
- Бесплатные приложения для трейдинга
- 8 000+ сигналов для копирования
- Экономические новости для анализа финансовых рынков
Вы принимаете политику сайта и условия использования