Оптимизация бактериальным хемотаксисом — Bacterial Chemotaxis Optimization (BCO)
Содержание
Введение
В области оптимизации многие исследователи и разработчики черпают вдохновение из биологических процессов происходящих в природе, таких как эволюция, социальные взаимосвязи или поведение живых организмов в поисках пищи. Это приводит к разработке совершенно новых инновационных методов оптимизации. Исследования показали, что эти методы зачастую превосходят классические эвристические и градиентные подходы, особенно при решении мультимодальных, недифференцируемых и дискретных задач. Одним из таких методов является алгоритм хемотаксиса, впервые предложенный Бремерманом и его коллегами, мы уже рассматривали алгоритм оптимизации бактериального поиска пищи (BFO). Он моделирует реакцию бактерий на хемоаттрактанты в градиентах концентрации. Бремерман провел анализ хемотаксиса в трехмерных градиентах и применил его для обучения нейронных сетей. Хотя этот алгоритм основан на принципах бактериального хемотаксиса, новые биологические открытия позволили создать более детализированную модель этого процесса.
В данной статье мы и постараемся рассмотреть эту модель как новую стратегию оптимизации. Основные отличия новой модели от традиционного алгоритма хемотаксиса заключаются в следующем:
- Бактерии в новой модели используют информацию о значениях концентрации.
- Они не продолжают движение в одном направлении при увеличении концентраций хемоаттрактантов.
Бактерии — это одноклеточные организмы, одна из самых простых форм жизни на Земле. Несмотря на свою простоту, они способны получать информацию об окружающей среде, ориентироваться в ней и эффективно использовать эту информацию для выживания. Реакция бактерий на изменения в окружающей среде была предметом интенсивных исследований в последние десятилетия, что также привлекло внимание ученых, работающих в области оптимизации. Алгоритмы оптимизации можно рассматривать как системы, которые собирают информацию о функциональном ландшафте и используют её для достижения оптимума. Простота идеи бактериального хемотаксиса делает его привлекательной отправной точкой для построения такого типа алгоритмов.
В разных исследованиях было установлено, что бактерии обмениваются информацией друг с другом, хотя о механизмах этой коммуникации известно не так много. Обычно бактерии рассматриваются как индивидуумы, и социальное взаимодействие в моделях не учитывается. Это отличает их от моделей взаимодействия, описывающих поведение общественных насекомых (таких, как муравьи, пчелы, осы или термиты), которые действуют как системы с коллективным интеллектом, что открывает иные возможности для решения различных задач.
Адаптация — еще один важный аспект хемотаксиса. Бактерии способны изменять свою чувствительность к постоянным химическим условиям, что позволяет им эффективно реагировать на изменения в окружающей среде. Это качество делает их не только выносливыми, но и высокоэффективными в поиске ресурсов.
В данном исследовании авторы сосредоточились на микроскопических моделях, учитывающих хемотаксис отдельных бактерий, в отличие от макроскопических моделей, анализирующих движение колоний. Алгоритм был разработан С. Д. Мюллером и П. Коумацакасом, и его основные идеи были представлены и опубликованы в 2002 году.
Реализация алгоритма
Идея алгоритма бактериального хемотаксиса (BCO) заключается в использовании биологических принципов, наблюдаемых в поведении бактерий, для решения задач оптимизации. Алгоритм моделирует, как бактерии реагируют на градиенты химических веществ в окружающей среде, что позволяет им находить более благоприятные условия для жизни. Основные идеи алгоритма:
- Алгоритм описывает движение бактерий как последовательность прямолинейных траекторий, соединенных мгновенными поворотами. Каждое движение характеризуется скоростью, направлением и продолжительностью.
- Направление поворота бактерии определяется вероятностным распределением, что позволяет учитывать случайные изменения в движении.
- Алгоритм использует информацию о градиентах функции, чтобы направлять бактерию к оптимальному решению, минимизируя количество итераций, необходимых для достижения цели.
Подробный псевдокод алгоритма бактериальной хемотаксической оптимизации (BCO) описывает основные шаги алгоритма, включая инициализацию, основной цикл оптимизации с этапами движения и ревизии, а также вспомогательные функции.
Инициализация.
1. Задать параметры:
- popSize: размер популяции (количество бактерий)
- hs: количество шагов для расчета среднего изменения
- T0: начальная температура
- b: параметр
- tau_c: параметр
- v: скорость
2. Создать массив бактерий размером popSize
3. Для каждой бактерии i от 0 до popSize-1:
- Инициализировать fPrev значением -DBL_MAX
- Создать массив fHistory размером hs и заполнить его нулями
Основной цикл оптимизации. Повторять до достижения условия остановки:
Этап движения для каждой бактерии i от 0 до popSize - 1:
1. Получить текущее значение целевой функции f_tr
2. Получить предыдущее значение целевой функции f_pr из bacterium [i].fPrev
3. Если f_tr <= f_pr: T = T0
Иначе: вычислить b_corr = CalculateCorrectedB (f_tr, f_pr, i)
- T = T0 * exp (b_corr * (f_tr - f_pr))
4. Сгенерировать tau из экспоненциального распределения с параметром T.
5. Вычислить новые углы new_angles [] для coords - 1 измерений: для каждого угла j от 0 до coords - 2:
- theta = CalculateRotationAngle ()
- mu = 62 * (1 - cos (theta)) * π / 180
- sigma = 26 * (1 - cos (theta)) * π / 180
- new_angles [j] = случайное число из гауссовского распределения с параметрами (mu, mu-π, mu+π)
6. Вычислить новую позицию:
- l = v * tau
- CalculateNewPosition (l, new_angles, new_position, current_position)
7. Обновить позицию бактерии, ограничивая значения в пределах rangeMin и rangeMax
8. Обновить bacterium [i].fPrev значением f_tr
Этап ревизии.
1. Обновить глобальное лучшее решение cB со значением приспособленности fB
2. Для каждой бактерии i от 0 до popSize - 1. Обновить историю значений целевой функции fHistory:
- Сдвинуть все значения на одну позицию влево
- Добавить текущее значение целевой функции в конец истории
Вспомогательные функции:
CalculateCorrectedB (f_tr, f_pr, bacteriumIndex)
1. Вычислить delta_f_tr = f_tr - f_pr
2. Вычислить delta_f_pr = среднее изменение за последние hs шагов
3. Если |delta_f_pr| < epsilon: Вернуть b
Иначе: Вернуть b * (1 / (|delta_f_tr / delta_f_pr| + 1) + 1 / (|f_pr| + 1))
CalculateRotationAngle ()
1. Вычислить cos_theta = exp (-tau_c / T0)
2. Вернуть arccos (cos_theta)
CalculateNewPosition (l, angles, new_position, current_position)
1. Вычислить new_position [0] с учетом всех углов
2. Для каждой координаты i от 1 до coords - 1:
Вычислить new_position [i] с учетом соответствующих углов
3. Применить случайное направление (1 или -1) к каждой координате
GenerateFromExponentialDistribution (T)
Вернуть -T * ln (случайное число от 0 до 1)
Переходим к написанию кода алгоритма. Для представления бактерии как решение задачи оптимизации опишем структуру "S_BCO_Bacterium".
1. Поля структуры:
- fPrev - предыдущее значение целевой функции.
- fHistory [] - массив истории значений целевой функции.
2. Init - метод инициализации выполняет действия:
- изменяется размер массива "fHistory" до значения "historySize".
- инициализируются все элементы массива "fHistory" значением "0.0".
- fPrev - устанавливается предыдущее значение целевой функции в минимально возможное значение.
Бактерия в виде структуры, предусматривающей отслеживание изменения значений целевой функции на протяжении заданного количества итераций (отдельных перемещений).
//—————————————————————————————————————————————————————————————————————————————— struct S_BCO_Bacterium { double fPrev; // previous value of the objective function double fHistory []; // history of objective function values void Init (int coords, int historySize) { ArrayResize (fHistory, historySize); ArrayInitialize (fHistory, 0.0); fPrev = -DBL_MAX; } }; //——————————————————————————————————————————————————————————————————————————————
Опишем класс алгоритма "C_AO_BCO". Давайте разберем его по частям.
1. В конструкторе инициализируются внешние параметры алгоритма.
2. Метод "SetParams" - обновляет значения параметров из массива "params".
3. Методы "Moving" и "Revision"- отвечают за движение бактерий и пересмотр их позиций.
4. В классе определены несколько приватных методов, которые используются для различных расчетов, связанных с алгоритмом. "CalculateAverageDeltaFpr", "CalculateNewAngles", "CalculateNewPosition", "GenerateFromExponentialDistribution", "CalculateCorrectedB", "CalculateRotationAngle", "RNDdir", bacterium - массив бактерий (популяция). Параметры класса:
- hs - количество шагов для расчета среднего изменения.
- T0 - начальная температура.
- b - параметр.
- tau_c - параметр.
- v - скорость.
//—————————————————————————————————————————————————————————————————————————————— class C_AO_BCO : public C_AO { public: //-------------------------------------------------------------------- ~C_AO_BCO () { } C_AO_BCO () { ao_name = "BCO"; ao_desc = "Bacterial Chemotaxis Optimization"; ao_link = "https://www.mql5.com/ru/articles/15711"; popSize = 50; // population size (number of bacteria) hs = 10; // number of steps to calculate average change T0 = 1.0; // initial temperature b = 0.5; // parameter b tau_c = 1.0; // parameter tau_c v = 1.0; // velocity ArrayResize (params, 6); params [0].name = "popSize"; params [0].val = popSize; params [1].name = "hs"; params [1].val = hs; params [2].name = "T0"; params [2].val = T0; params [3].name = "b"; params [3].val = b; params [4].name = "tau_c"; params [4].val = tau_c; params [5].name = "v"; params [5].val = v; } void SetParams () { popSize = (int)params [0].val; hs = (int)params [1].val; T0 = params [2].val; b = params [3].val; tau_c = params [4].val; v = params [5].val; } bool Init (const double &rangeMinP [], //minimum search range const double &rangeMaxP [], //maximum search range const double &rangeStepP [], //step search const int epochsP = 0); //number of epochs void Moving (); void Revision (); //---------------------------------------------------------------------------- int hs; double T0; double b; double tau_c; double v; S_BCO_Bacterium bacterium []; private: //------------------------------------------------------------------- double CalculateAverageDeltaFpr (int bacteriumIndex); void CalculateNewAngles (double &angles []); void CalculateNewPosition (double l, const double &angles [], double &new_position [], const double ¤t_position []); double GenerateFromExponentialDistribution (double T); double CalculateCorrectedB (double f_tr, double f_pr, int bacteriumIndex); double CalculateRotationAngle (); int RNDdir (); }; //—————————————————————————————————————————————————————————————————————————————— bool C_AO_BCO::Init (const double &rangeMinP [], //minimum search range const double &rangeMaxP [], //maximum search range const double &rangeStepP [], //step search const int epochsP = 0) //number of epochs { if (!StandardInit (rangeMinP, rangeMaxP, rangeStepP)) return false; //---------------------------------------------------------------------------- ArrayResize (bacterium, popSize); for (int i = 0; i < popSize; i++) bacterium [i].Init (coords, hs); return true; } //——————————————————————————————————————————————————————————————————————————————
Метод "Moving" класса "C_AO_BCO" отвечает за перемещение бактерий в пространстве поиска. Давайте разберем как он работает:
1. Если "revision" равно "false", это означает, что бактерии имеют начальную позицию.
- Для каждой бактерии "a [i]" генерируются случайные координаты в пределах заданного диапазона.
- Функция "u.RNDfromCI" генерирует случайное значение, а "u.SeInDiSp" корректирует ее с учетом заданного шага.
2. Основной цикл перемещения, если "revision" равно "true". Метод выполняет основную логику перемещения бактерий. Определение температуры "T":
- Если текущее значение функции "f_tr" лучше или равно предыдущему "f_pr", используется начальная температура "T0".
- В противном случае температура корректируется с помощью функции "CalculateCorrectedB", которая учитывает разницу между текущим и предыдущим значением функции приспособленности.
- Генерация времени перемещения "tau": используется экспоненциальное распределение для генерации времени перемещения.
- Вычисляются новые углы перемещения и новая позиция на основе длины перемещения "l" и новых углов.
- Новая позиция корректируется с учетом заданного диапазона и шага.
- В конце цикла обновляется предыдущее значение функции приспособленности для каждой бактерии.
Метод "Moving" реализует основную логику перемещения бактерий в пространстве поиска, адаптируя их поведение в зависимости от результатов предыдущих итераций. Он использует случайные элементы и адаптивные механизмы в поисках оптимального решения.
//—————————————————————————————————————————————————————————————————————————————— void C_AO_BCO::Moving () { //---------------------------------------------------------------------------- if (!revision) { for (int i = 0; i < popSize; i++) { for (int c = 0; c < coords; c++) { a [i].c [c] = u.RNDfromCI (rangeMin [c], rangeMax [c]); a [i].c [c] = u.SeInDiSp (a [i].c [c], rangeMin [c], rangeMax [c], rangeStep [c]); } } revision = true; return; } //---------------------------------------------------------------------------- for (int i = 0; i < popSize; i++) { double f_tr = a [i].f; double f_pr = bacterium [i].fPrev; double T; if (f_tr <= f_pr) { T = T0; } else { double b_corr = CalculateCorrectedB (f_tr, f_pr, i); T = T0 * exp (b_corr * (f_tr - f_pr)); } double tau = GenerateFromExponentialDistribution (T); double new_angles []; ArrayResize (new_angles, coords - 1); CalculateNewAngles (new_angles); double l = v * tau; double new_position []; ArrayResize (new_position, coords); CalculateNewPosition (l, new_angles, new_position, a [i].c); for (int c = 0; c < coords; c++) { a [i].c [c] = u.SeInDiSp (new_position [c], rangeMin [c], rangeMax [c], rangeStep [c]); } bacterium [i].fPrev = a [i].f; } } //——————————————————————————————————————————————————————————————————————————————
Метод "Revision" класса "C_AO_BCO" отвечает за обновление информации о лучших найденных решениях и истории значений функции приспособленности для каждой бактерии.
1. Переменная "ind" инициализируется значением "-1". Она будет использоваться для хранения индекса бактерии, у которой найдено лучшее значение функции.
2. Поиск лучшего значения функции:
- Метод проходит через все бактерии в популяции "popSize" и ищет ту, у которой значение функции "f" больше текущего лучшего значения "fB".
- Если находится бактерия с более высоким значением функции, то "fB" обновляется, и индекс этой бактерии сохраняется в "ind".
3. Если была найдена бактерия с индексом "ind" (т.е. "ind" не равен "-1"), то координаты этой бактерии копируются в массив "cB", который, представляет собой координаты текущего лучшего решения.
4. Для каждой бактерии обновляется история значений функции. Метод проходит по каждому элементу "fHistory", сдвигая значения на одну позицию влево, чтобы освободить место для нового значения. В конце каждой итерации в последний элемент массива "fHistory" записывается текущее значение приспособленности "a [i].f" для каждой бактерии.
Таким образом, метод "Revision" выполняет две основные функции:
- Обновляет лучшее значение функции приспособленности и соответствующие координаты.
- Обновляет историю значений функции приспособленности для каждой бактерии, что позволяет отслеживать изменения в их состоянии на протяжении истории их перемещений.
//—————————————————————————————————————————————————————————————————————————————— void C_AO_BCO::Revision () { int ind = -1; for (int i = 0; i < popSize; i++) { if (a [i].f > fB) { fB = a [i].f; ind = i; } } if (ind != -1) { ArrayCopy (cB, a [ind].c, 0, 0, WHOLE_ARRAY); } for (int i = 0; i < popSize; i++) { for (int j = 1; j < hs; j++) { bacterium [i].fHistory [j - 1] = bacterium [i].fHistory [j]; } bacterium [i].fHistory [hs - 1] = a [i].f; } } //——————————————————————————————————————————————————————————————————————————————
Метод "CalculateAverageDeltaFpr" класса "C_AO_BCO" предназначен для вычисления средних изменений значений фитнес-функции (дельты) для конкретной бактерии между двумя её соседними положениями, основываясь на истории значений приспособленности.
//—————————————————————————————————————————————————————————————————————————————— double C_AO_BCO::CalculateAverageDeltaFpr (int bacteriumIndex) { double sum = 0; for (int i = 1; i < hs; i++) { sum += bacterium [bacteriumIndex].fHistory [i] - bacterium [bacteriumIndex].fHistory [i - 1]; } return sum / (hs - 1); } //——————————————————————————————————————————————————————————————————————————————
Метод "CalculateNewAngles" класса "C_AO_BCO" предназначен для вычисления новых углов на основе логики, связанной с вращением и распределением, выполняет следующие действия:
- Проходит по массиву для новых углов и для каждого угла вычисляет новое значение.
- Использует параметры, зависящие от косинуса угла, для генерации значений, которые используют гауссовское распределение.
//—————————————————————————————————————————————————————————————————————————————— void C_AO_BCO::CalculateNewAngles (double &angles []) { for (int i = 0; i < coords - 1; i++) { double theta = CalculateRotationAngle (); double mu = 62 * (1 - MathCos (theta)) * M_PI / 180.0; double sigma = 26 * (1 - MathCos (theta)) * M_PI / 180.0; angles [i] = u.GaussDistribution (mu, mu - M_PI, mu + M_PI, 8); } } //——————————————————————————————————————————————————————————————————————————————
Метод "CalculateNewPosition" класса "C_AO_BCO" предназначен для вычисления новых координат позиции на основе текущих координат, углов и параметра "l".
1. Входные параметры метода:
- l - коэффициент, влияющий на изменение позиции.
- angles [] - массив углов, которые используются для вычисления новой позиции.
- new_position [] - массив, в который будут записаны новые координаты.
- current_position [] - массив текущих координат.
2. Первая координата "new_position [0]" вычисляется как сумма текущей координаты "current_position [0]" и произведения "l" на разницу между "rangeMax [0]" и "rangeMin [0]".
3. Затем первая координата умножается на косинусы углов из массива "angles", начиная с первого до предпоследнего.
4. Результат умножается на значение, возвращаемое функцией "RNDdir ()", которая генерирует случайное направление "-1" или "1".
5. Для каждой следующей координаты "new_position [i]", где "i" от "1" до "coords - 2" вычисляется новая позиция на основе текущей позиции и синуса соответствующего угла.
6. Каждая новая координата также умножается на косинусы углов, начиная с текущего индекса "i" до предпоследнего.
7. Случайное направление для остальных координат, результат также умножается на значение, возвращаемое "RNDdir ()".
8. Обработка последней координаты, если количество координат больше 1, для последней координаты "new_position [coords - 1]" вычисляется новая позиция на основе текущей позиции и синуса последнего угла.
Таким образом метод "CalculateNewPosition" выполняет следующие действия:
- Вычисляет новые координаты на основе текущих координат и углов.
- Учитывает влияние случайного направления на каждую координату.
- Применяет тригонометрические функции (синус и косинус) для учета углов.
Таким образом, этот метод используется для моделирования движения бактерий в пространстве с учетом их текущего положения и заданных углов.
//—————————————————————————————————————————————————————————————————————————————— void C_AO_BCO::CalculateNewPosition (double l, const double &angles [], double &new_position [], const double ¤t_position []) { new_position [0] = current_position [0] + l * (rangeMax [0] - rangeMin [0]); for (int k = 0; k < coords - 1; k++) { new_position [0] *= MathCos (angles [k]); } new_position [0] *= RNDdir (); for (int i = 1; i < coords - 1; i++) { new_position [i] = current_position [i] + l * MathSin (angles [i - 1]) * (rangeMax [0] - rangeMin [0]); for (int k = i; k < coords - 1; k++) { new_position [i] *= MathCos (angles [k]); } new_position [i] *= RNDdir (); } if (coords > 1) { new_position [coords - 1] = current_position [coords - 1] + l * MathSin (angles [coords - 2]); } } //——————————————————————————————————————————————————————————————————————————————
Далее кратко опишем метод "RNDdir" класса "C_AO_BCO", который предназначен для генерации случайного направления, которое может принимать одно из двух значений: -1 или 1.
//—————————————————————————————————————————————————————————————————————————————— int C_AO_BCO::RNDdir () { if (u.RNDbool () < 0.5) return -1; return 1; } //——————————————————————————————————————————————————————————————————————————————
Сделаем краткий обзор методов класса "C_AO_BCO".
Метод "GenerateFromExponentialDistribution" выполняет:
- генерацию случайного числа по экспоненциальному распределению с параметром "T".
- далее использует случайное число из диапазона (0, 1), вычисляет его логарифм и умножает на "-T".
- получаем случайное число, распределенное по экспоненциальному закону.
Метод "CalculateCorrectedB" выполняет:
- расчет скорректированного значения "b" на основе разницы между "f_tr" и "f_pr" (текущей и предыдущей приспособленности).
- вычисляет разницу между "f_tr" и "f_pr", получает среднее значение для бактерии, и в зависимости от этого возвращает скорректированное значение "b".
//—————————————————————————————————————————————————————————————————————————————— double C_AO_BCO::GenerateFromExponentialDistribution (double T) { return -T * MathLog (u.RNDprobab ()); } double C_AO_BCO::CalculateCorrectedB (double f_tr, double f_pr, int bacteriumIndex) { double delta_f_tr = f_tr - f_pr; double delta_f_pr = CalculateAverageDeltaFpr (bacteriumIndex); if (MathAbs (delta_f_pr) < DBL_EPSILON) { return b; } else { return b * (1 / (MathAbs (delta_f_tr / delta_f_pr) + 1) + 1 / (MathAbs (f_pr) + 1)); } } //——————————————————————————————————————————————————————————————————————————————
Последним следует метод "CalculateRotationAngle" класса "C_AO_BCO". Метод вычисляет угол вращения на основе заданных параметров и возвращает значение в радианах.
//—————————————————————————————————————————————————————————————————————————————— double C_AO_BCO::CalculateRotationAngle () { double cos_theta = MathExp (-tau_c / T0); return MathArccos (cos_theta); } //——————————————————————————————————————————————————————————————————————————————
Протестируем оригинальную версию алгоритма и смотрим результаты:
=============================
5 Hilly's; Func runs: 10000; result: 0.42924491510564006
25 Hilly's; Func runs: 10000; result: 0.282259866768426
500 Hilly's; Func runs: 10000; result: 0.2515386629014219
=============================
5 Forest's; Func runs: 10000; result: 0.2476662231845009
25 Forest's; Func runs: 10000; result: 0.17824381036550777
500 Forest's; Func runs: 10000; result: 0.15324081202657283
=============================
5 Megacity's; Func runs: 10000; result: 0.2430769230769231
25 Megacity's; Func runs: 10000; result: 0.11415384615384619
500 Megacity's; Func runs: 10000; result: 0.09444615384615461
=============================
All score: 1.99387 (22.15%)
Полученные результаты настолько слабые, что не имеет смысла заносить их в рейтинговую таблицу. При проектировании алгоритма и написании кода по текстовому описанию автора, мне пришлось скорректировать некоторые формулы: одни из них не имели математического смысла (например, деление числа на вектор), а другие вырождались либо в крайне малые, или крайне большие значения, несопоставимые с размерностями задачи, при этом я распечатывал результаты функций по каждой из представленных формул и анализировал. Таким образом, в том виде, в котором он описан авторами, алгоритм не может функционировать в принципе.
Кроме того, операции с углами, в расчетах которых используется нормальное распределение, можно существенно упростить, так как физический смысл этих операций сводится к тому, что на каждой координате можно просто отложить новое приращение от текущего местоположения бактерии. Поэтому я решил разработать свою реализацию бактериального хемотаксиса, по возможности придерживаясь основных концепций и идей алгоритма.
Итак, представляю псевдокод моей версии алгоритма:
Инициализация:
1. Создать популяцию из popSize бактерий
2. Для каждой бактерии i:
Инициализировать нулём историю значений целевой функции f_history [i] размера hs
Установить начальное значение f_prev [i] = -DBL_MAX
Основной цикл:
Пока не достигнуто условие остановки:
1. Если это первая итерация:
Для каждой бактерии i:
Случайно инициализировать позицию x [i] в пространстве поиска
x [i].[j] ∈ [x_min [j], x_max [j]] для каждой координаты j
2. Иначе:
Для каждой бактерии i:
a. Вычислить среднее изменение целевой функции:
delta_ave [i] = (1 / (hs - 1)) * sum (f_history [i].[k] - f_history [i].[k-1] for k in 1..hs-1) + epsilon
b. Вычислить относительное изменение приспособленности:
delta [i] = 1 - |f (x [i]) - f_prev [i]| / delta_ave [i]
delta [i] = max (delta [i], 0.0001)
c. Для каждой координаты j:
С вероятностью 0.5:
dist [j] = (x_max [j] - x_min [j]) * delta [i]
x [i].[j] = N (x [i].[j], x [i].[j] - dist [j], x [i].[j] + dist [j])
Ограничить x [i].[j] в пределах [x_min [j], x_max [j]]
Иначе:
x [i].[j] = x_best [j]
d. Обновить f_prev [i] = f (x [i])
3. Оценить целевую функцию f (x [i]) для каждой бактерии
4. Обновить лучшее найденное решение:
Если существует i: f (x [i]) > f (x_best), то x_best = x [i]
5. Обновить историю значений целевой функции для каждой бактерии:
Сдвинуть значения в f_history [i]
Добавить новое значение: f_history [i].[hs - 1] = f (x [i])
Завершение:
Вернуть лучшее найденное решение x_best
Где:
- x [i] - позиция i-й бактерии
- f (x) - целевая функция
- hs - размер истории
- epsilon - малая константа для предотвращения деления на ноль
- N (μ, a, b) - усеченное нормальное распределение со средним μ и границами [a, b]
Таким образом, мой модифицированный псевдокод отражает основную структуру и логику алгоритма BCO. Заострим внимание на основных моментах:
- Алгоритм использует популяцию бактерий, каждая из которых представляет потенциальное решение.
- На каждой итерации бактерии двигаются в пространстве поиска, используя информацию о своих предыдущих результатах.
- Движение основано на относительном изменении целевой функции, что позволяет алгоритму адаптироваться к ландшафту оптимизации.
- Алгоритм сохраняет историю значений целевой функции для каждой бактерии, что используется для расчета среднего изменения.
- С некоторой вероятностью бактерия будет двигаться к лучшему известному решению вместо исследования новой области (аналог наследования признаков в генетике).
- После каждого движения производится оценка новых позиций и обновление лучшего найденного решения.
BCOm версия сочетает в себе элементы локального поиска (движение отдельных бактерий) и глобального поиска (обмен информацией через лучшее известное решение).
Давайте рассмотрим основные отличия двух версий алгоритма. Во-первых, упрощен механизм движения бактерий, я отказался от сложной системы с углами поворота и температурой. Во-вторых, новая версия меньше полагается на историю изменений для корректировки поведения бактерий, используя более прямолинейный подход к обновлению позиций. Вместо комбинации экспоненциального и нормального распределений, новый алгоритм использует нормальное распределение для обновления координат. Следствием внесенных изменений стало сокращение количества параметров до одного (не считая размер популяции), контролирующих поведение алгоритма, что изменило способ настройки и упростило управление процессом оптимизации.
В целом новый псевдокод предполагает более простые вычисления на каждом шаге оптимизации, что также должно позитивно влиять на скорость выполнения задачи алгоритмом (в оригинальной версии производились многократные вычисления синуса и косинуса углов поворота для каждой координаты каждой бактерии). Мой подход несколько иначе балансирует между исследованием нового пространства решений и эксплуатацией уже найденных хороших решений.
Эти изменения в логике привели к созданию более простого и, предположительно (до получения результатов тестов), более производительного алгоритма. Переходим к написанию кода.
Структуру "S_BCO_Bacterium", представляющую каждую бактерию, оставим без изменений. Она предназначена для хранения информации о бактерии и ее истории значений целевой функции.
В метод "Init" класса "C_AO_BCOm", отвечающий за инициализацию параметров алгоритма, добавим определение расстояние допустимого перемещения по каждой координате.
Таким образом, метод "Init" класса "C_AO_BCOm" отвечает за инициализацию параметров алгоритма оптимизации. Он проверяет стандартные условия инициализации, создает необходимые массивы и заполняет их значениями.
//—————————————————————————————————————————————————————————————————————————————— bool C_AO_BCOm::Init (const double &rangeMinP [], //minimum search range const double &rangeMaxP [], //maximum search range const double &rangeStepP [], //step search const int epochsP = 0) //number of epochs { if (!StandardInit (rangeMinP, rangeMaxP, rangeStepP)) return false; //---------------------------------------------------------------------------- ArrayResize (bacterium, popSize); for (int i = 0; i < popSize; i++) bacterium [i].Init (coords, hs); ArrayResize (allowedDispl, coords); for (int c = 0; c < coords; c++) allowedDispl [c] = rangeMax [c] - rangeMin [c]; return true; } //——————————————————————————————————————————————————————————————————————————————
Разберем метод "Moving" класса "C_AO_BCOm", который отвечает за перемещение бактерий в пространстве поиска. Давайте разберем его по частям.
1. На первой итерации действия метода не изменились: инициализация координат бактерий случайными значения в заданном диапазоне.
2. Основной алгоритм перемещения: объявляются переменные для хранения значений, таких, как "Δ", "ΔAve", и "dist" и для каждого индивидуума в популяции:
- Вычисляется среднее значение "ΔAve" с использованием функции "CalculateAverageDeltaFpr (i)".
- Вычисляется относительное изменение "Δ", которое используется для определения степени изменения положения бактерий.
- Если "Δ" слишком мал, оно устанавливается в 0.0001.
3. Изменение координат:
- Для каждой координаты проверяется случайная вероятность (50%).
- Если условие выполняется, вычисляется расстояние "dist", которое зависит от "allowedDispl [c]" и "Δ".
- Новое значение "x" вычисляется с помощью функции "GaussDistribution", с учетом границ "xMin" и "xMax".
- Если "x" выходит за пределы диапазона, оно корректируется с использованием "RNDfromCI".
- Наконец, новое значение координаты сохраняется с учетом шага "rangeStep".
4. Сохраняется предыдущее значение функции приспособленности "f" для каждого индивидуума в массиве "bacterium". Используются массивы "a" и "bacterium". Функции "RNDfromCI", "SeInDiSp", "GaussDistribution" отвечают за генерацию случайных чисел и распределений, а также за нормализацию значений координат.
Таким образом, функция "Moving ()" отвечает за инициализацию и обновление положения особей в популяции в рамках алгоритма. Она использует случайные вероятности и распределения для управления перемещением бактерий. Однако ключевым отличием от оригинальной версии является более простой и эффективный способ реализации градиента функции приспособленности. При уменьшении разницы в самочувствии бактерии на текущем шаге по сравнению с предыдущим, её движение ускоряется. Напротив, при нахождении в благоприятной внешней среде бактерия замедляется. Это противоречит естественному поведению бактерий, которые в агрессивной среде впадают в депрессию и анабиоз.
//—————————————————————————————————————————————————————————————————————————————— void C_AO_BCOm::Moving () { //---------------------------------------------------------------------------- if (!revision) { for (int i = 0; i < popSize; i++) { for (int c = 0; c < coords; c++) { a [i].c [c] = u.RNDfromCI (rangeMin [c], rangeMax [c]); a [i].c [c] = u.SeInDiSp (a [i].c [c], rangeMin [c], rangeMax [c], rangeStep [c]); } } revision = true; return; } //---------------------------------------------------------------------------- double x = 0.0; double xMin = 0.0; double xMax = 0.0; double Δ = 0.0; double ΔAve = 0.0; double dist = 0.0; for (int i = 0; i < popSize; i++) { ΔAve = CalculateAverageDeltaFpr (i) + DBL_EPSILON; Δ = fabs (a [i].f - bacterium [i].fPrev) / ΔAve; Δ = 1.0 - Δ; if (Δ < 0.0001) Δ = 0.0001; for (int c = 0; c < coords; c++) { if (u.RNDprobab () < 0.5) { dist = allowedDispl [c] * Δ; x = a [i].c [c]; xMin = x - dist; xMax = x + dist; x = u.GaussDistribution (x, xMin, xMax, 8); if (x > rangeMax [c]) x = u.RNDfromCI (xMin, rangeMax [c]); if (x < rangeMin [c]) x = u.RNDfromCI (rangeMin [c], xMax); a [i].c [c] = u.SeInDiSp (x, rangeMin [c], rangeMax [c], rangeStep [c]); } else a [i].c [c] = cB [c]; } bacterium [i].fPrev = a [i].f; } } //——————————————————————————————————————————————————————————————————————————————
Метод "Revision ()" в классе "C_AO_BCOm", который отвечает за обновление информации о популяции и истории значений целевой функции, не изменился.
Результаты тестов
Теперь посмотрим, как работает новая версия алгоритма BCOm:
BCO|Bacterial Chemotaxis Optimization|50.0|10.0|
=============================
5 Hilly's; Func runs: 10000; result: 0.759526049526603
25 Hilly's; Func runs: 10000; result: 0.6226756163411526
500 Hilly's; Func runs: 10000; result: 0.31483373090540534
=============================
5 Forest's; Func runs: 10000; result: 0.8937814268120954
25 Forest's; Func runs: 10000; result: 0.6133909133246214
500 Forest's; Func runs: 10000; result: 0.22541842289630293
=============================
5 Megacity's; Func runs: 10000; result: 0.653846153846154
25 Megacity's; Func runs: 10000; result: 0.42092307692307684
500 Megacity's; Func runs: 10000; result: 0.14435384615384755
=============================
All score: 4.64875 (51.65%)
Вероятно, накопленный опыт и критический взгляд на доступную информацию об оригинальной версии сделали свое дело, и новая версия алгоритма показала себя более мощной в практическом применении, чем оригинальная.
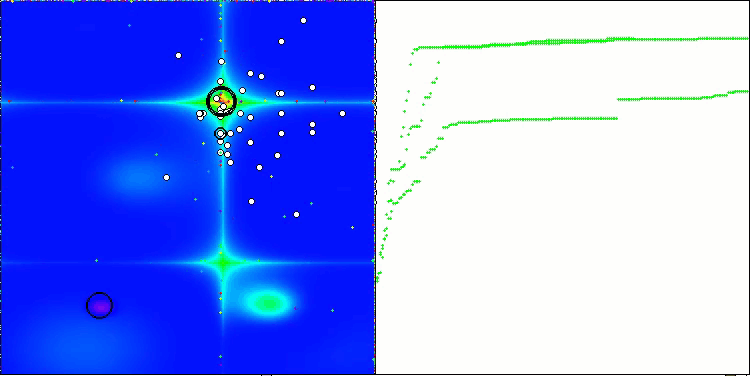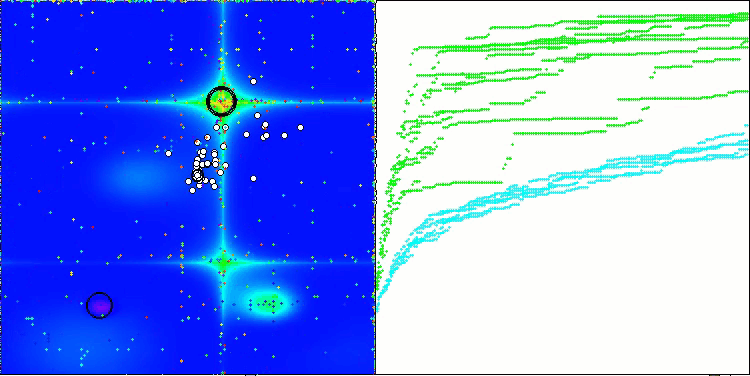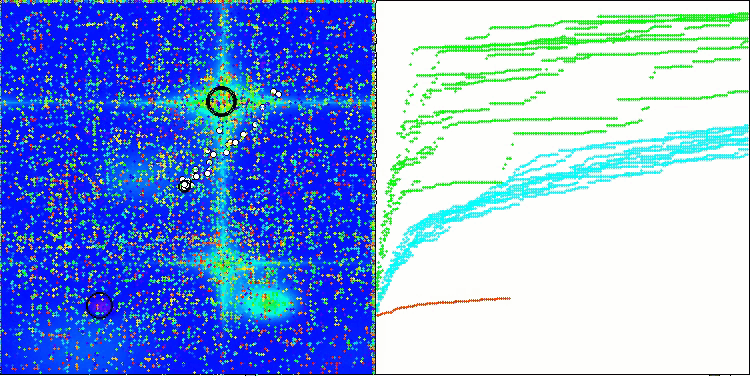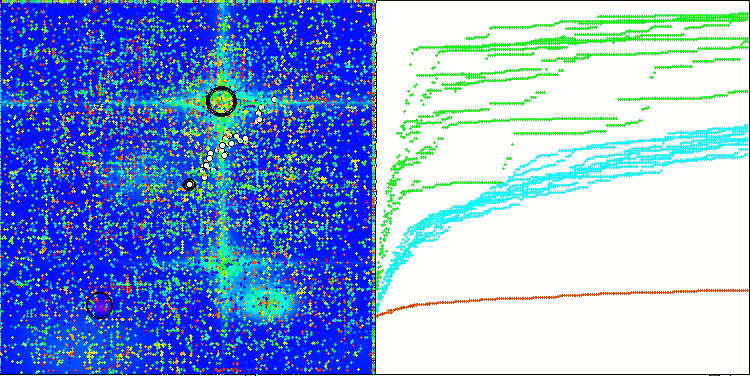
На визуализации работы алгоритма BCOm можно отметить хорошую проработку значимых участков гиперпространства, что говорит о высокой способности к исследованию поверхности оптимизируемой функции.
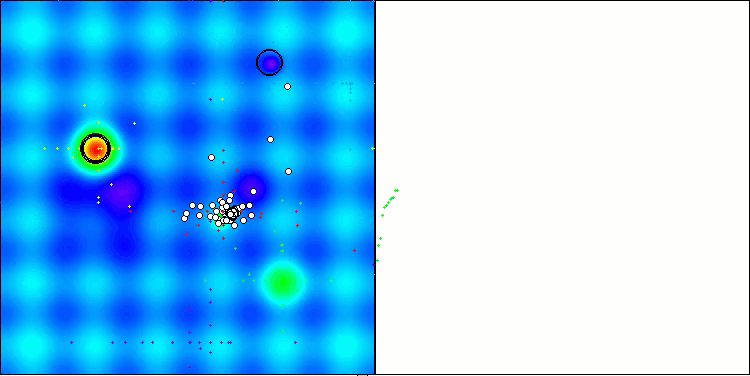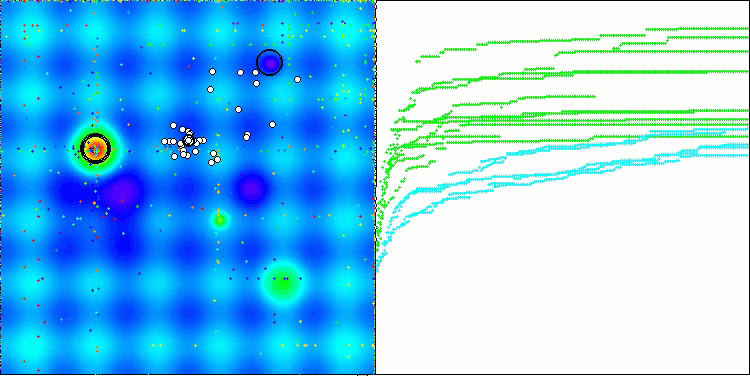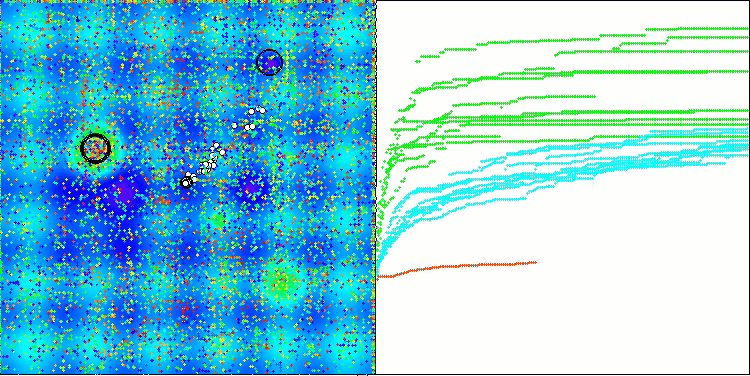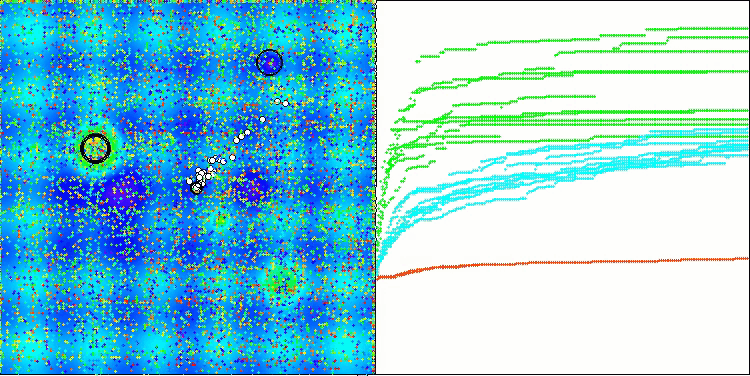
BCOm на тестовой функции Hilly
BCOm на тестовой функции Forest
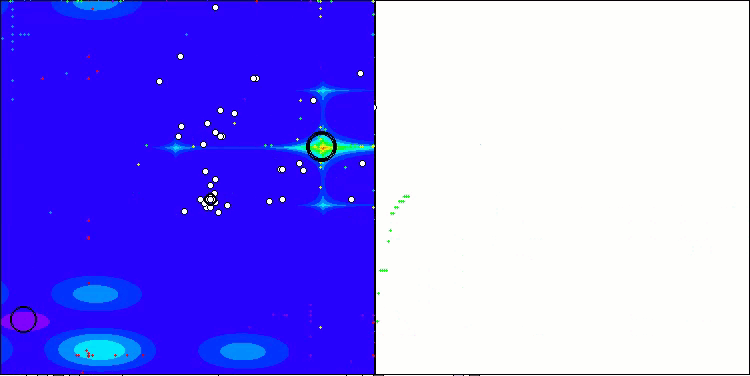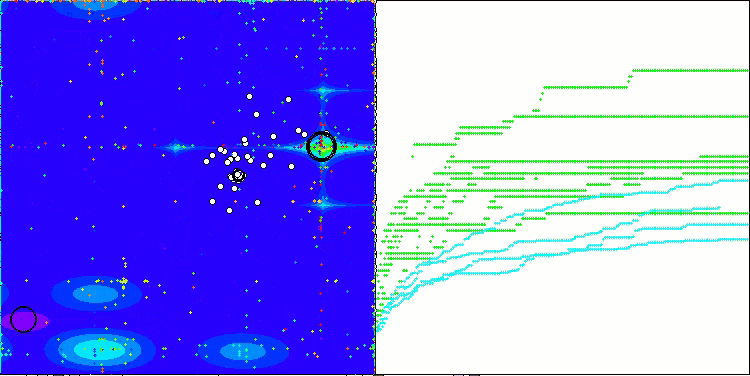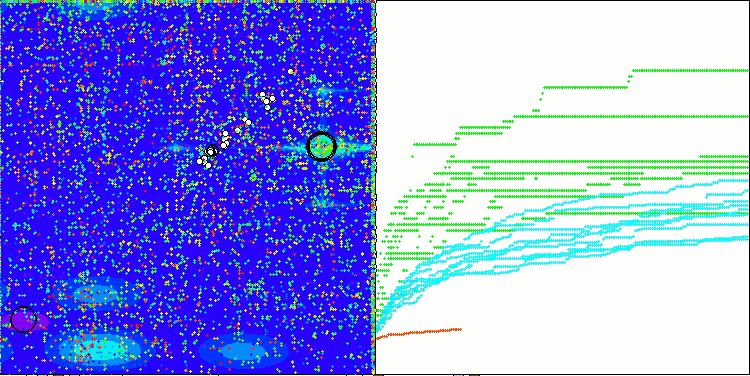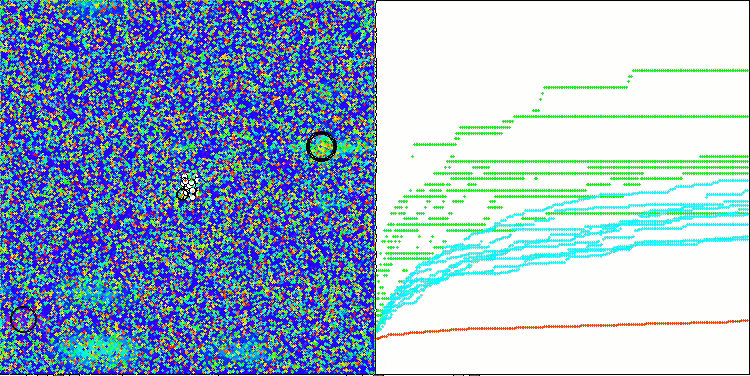
BCOm на тестовой функции Megacity
Алгоритм по итогам тестирования занял стабильное 17 место в общем рейтинге алгоритмов оптимизации.
| № | AO | Description | Hilly | Hilly final | Forest | Forest final | Megacity (discrete) | Megacity final | Final result | % of MAX | ||||||
| 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | ||||||||
| 1 | ANS | across neighbourhood search | 0,94948 | 0,84776 | 0,43857 | 2,23581 | 1,00000 | 0,92334 | 0,39988 | 2,32323 | 0,70923 | 0,63477 | 0,23091 | 1,57491 | 6,134 | 68,15 |
| 2 | CLA | code lock algorithm | 0,95345 | 0,87107 | 0,37590 | 2,20042 | 0,98942 | 0,91709 | 0,31642 | 2,22294 | 0,79692 | 0,69385 | 0,19303 | 1,68380 | 6,107 | 67,86 |
| 3 | AMOm | animal migration ptimization M | 0,90358 | 0,84317 | 0,46284 | 2,20959 | 0,99001 | 0,92436 | 0,46598 | 2,38034 | 0,56769 | 0,59132 | 0,23773 | 1,39675 | 5,987 | 66,52 |
| 4 | (P+O)ES | (P+O) evolution strategies | 0,92256 | 0,88101 | 0,40021 | 2,20379 | 0,97750 | 0,87490 | 0,31945 | 2,17185 | 0,67385 | 0,62985 | 0,18634 | 1,49003 | 5,866 | 65,17 |
| 5 | CTA | comet tail algorithm | 0,95346 | 0,86319 | 0,27770 | 2,09435 | 0,99794 | 0,85740 | 0,33949 | 2,19484 | 0,88769 | 0,56431 | 0,10512 | 1,55712 | 5,846 | 64,96 |
| 6 | SDSm | stochastic diffusion search M | 0,93066 | 0,85445 | 0,39476 | 2,17988 | 0,99983 | 0,89244 | 0,19619 | 2,08846 | 0,72333 | 0,61100 | 0,10670 | 1,44103 | 5,709 | 63,44 |
| 7 | ESG | evolution of social groups | 0,99906 | 0,79654 | 0,35056 | 2,14616 | 1,00000 | 0,82863 | 0,13102 | 1,95965 | 0,82333 | 0,55300 | 0,04725 | 1,42358 | 5,529 | 61,44 |
| 8 | SIA | simulated isotropic annealing | 0,95784 | 0,84264 | 0,41465 | 2,21513 | 0,98239 | 0,79586 | 0,20507 | 1,98332 | 0,68667 | 0,49300 | 0,09053 | 1,27020 | 5,469 | 60,76 |
| 9 | ACS | artificial cooperative search | 0,75547 | 0,74744 | 0,30407 | 1,80698 | 1,00000 | 0,88861 | 0,22413 | 2,11274 | 0,69077 | 0,48185 | 0,13322 | 1,30583 | 5,226 | 58,06 |
| 10 | ASO | anarchy society optimization | 0,84872 | 0,74646 | 0,31465 | 1,90983 | 0,96148 | 0,79150 | 0,23803 | 1,99101 | 0,57077 | 0,54062 | 0,16614 | 1,27752 | 5,178 | 57,54 |
| 11 | TSEA | turtle shell evolution algorithm | 0,96798 | 0,64480 | 0,29672 | 1,90949 | 0,99449 | 0,61981 | 0,22708 | 1,84139 | 0,69077 | 0,42646 | 0,13598 | 1,25322 | 5,004 | 55,60 |
| 12 | DE | differential evolution | 0,95044 | 0,61674 | 0,30308 | 1,87026 | 0,95317 | 0,78896 | 0,16652 | 1,90865 | 0,78667 | 0,36033 | 0,02953 | 1,17653 | 4,955 | 55,06 |
| 13 | CRO | chemical reaction optimisation | 0,94629 | 0,66112 | 0,29853 | 1,90593 | 0,87906 | 0,58422 | 0,21146 | 1,67473 | 0,75846 | 0,42646 | 0,12686 | 1,31178 | 4,892 | 54,36 |
| 14 | BSA | bird swarm algorithm | 0,89306 | 0,64900 | 0,26250 | 1,80455 | 0,92420 | 0,71121 | 0,24939 | 1,88479 | 0,69385 | 0,32615 | 0,10012 | 1,12012 | 4,809 | 53,44 |
| 15 | HS | harmony search | 0,86509 | 0,68782 | 0,32527 | 1,87818 | 0,99999 | 0,68002 | 0,09590 | 1,77592 | 0,62000 | 0,42267 | 0,05458 | 1,09725 | 4,751 | 52,79 |
| 16 | SSG | saplings sowing and growing | 0,77839 | 0,64925 | 0,39543 | 1,82308 | 0,85973 | 0,62467 | 0,17429 | 1,65869 | 0,64667 | 0,44133 | 0,10598 | 1,19398 | 4,676 | 51,95 |
| 17 | BCOm | bacterial chemotaxis optimization M | 0,75953 | 0,62268 | 0,31483 | 1,69704 | 0,89378 | 0,61339 | 0,22542 | 1,73259 | 0,65385 | 0,42092 | 0,14435 | 1,21912 | 4,649 | 51,65 |
| 18 | (PO)ES | (PO) evolution strategies | 0,79025 | 0,62647 | 0,42935 | 1,84606 | 0,87616 | 0,60943 | 0,19591 | 1,68151 | 0,59000 | 0,37933 | 0,11322 | 1,08255 | 4,610 | 51,22 |
| 19 | TSm | tabu search M | 0,87795 | 0,61431 | 0,29104 | 1,78330 | 0,92885 | 0,51844 | 0,19054 | 1,63783 | 0,61077 | 0,38215 | 0,12157 | 1,11449 | 4,536 | 50,40 |
| 20 | BSO | brain storm optimization | 0,93736 | 0,57616 | 0,29688 | 1,81041 | 0,93131 | 0,55866 | 0,23537 | 1,72534 | 0,55231 | 0,29077 | 0,11914 | 0,96222 | 4,498 | 49,98 |
| 21 | WOAm | wale optimization algorithm M | 0,84521 | 0,56298 | 0,26263 | 1,67081 | 0,93100 | 0,52278 | 0,16365 | 1,61743 | 0,66308 | 0,41138 | 0,11357 | 1,18803 | 4,476 | 49,74 |
| 22 | AEFA | artificial electric field algorithm | 0,87700 | 0,61753 | 0,25235 | 1,74688 | 0,92729 | 0,72698 | 0,18064 | 1,83490 | 0,66615 | 0,11631 | 0,09508 | 0,87754 | 4,459 | 49,55 |
| 23 | ACOm | ant colony optimization M | 0,88190 | 0,66127 | 0,30377 | 1,84693 | 0,85873 | 0,58680 | 0,15051 | 1,59604 | 0,59667 | 0,37333 | 0,02472 | 0,99472 | 4,438 | 49,31 |
| 24 | BFO-GA | bacterial foraging optimization - ga | 0,89150 | 0,55111 | 0,31529 | 1,75790 | 0,96982 | 0,39612 | 0,06305 | 1,42899 | 0,72667 | 0,27500 | 0,03525 | 1,03692 | 4,224 | 46,93 |
| 25 | ABHA | artificial bee hive algorithm | 0,84131 | 0,54227 | 0,26304 | 1,64663 | 0,87858 | 0,47779 | 0,17181 | 1,52818 | 0,50923 | 0,33877 | 0,10397 | 0,95197 | 4,127 | 45,85 |
| 26 | ASBO | adaptive social behavior optimization | 0,76331 | 0,49253 | 0,32619 | 1,58202 | 0,79546 | 0,40035 | 0,26097 | 1,45677 | 0,26462 | 0,17169 | 0,18200 | 0,61831 | 3,657 | 40,63 |
| 27 | MEC | mind evolutionary computation | 0,69533 | 0,53376 | 0,32661 | 1,55569 | 0,72464 | 0,33036 | 0,07198 | 1,12698 | 0,52500 | 0,22000 | 0,04198 | 0,78698 | 3,470 | 38,55 |
| 28 | IWO | invasive weed optimization | 0,72679 | 0,52256 | 0,33123 | 1,58058 | 0,70756 | 0,33955 | 0,07484 | 1,12196 | 0,42333 | 0,23067 | 0,04617 | 0,70017 | 3,403 | 37,81 |
| 29 | Micro-AIS | micro artificial immune system | 0,79547 | 0,51922 | 0,30861 | 1,62330 | 0,72956 | 0,36879 | 0,09398 | 1,19233 | 0,37667 | 0,15867 | 0,02802 | 0,56335 | 3,379 | 37,54 |
| 30 | COAm | cuckoo optimization algorithm M | 0,75820 | 0,48652 | 0,31369 | 1,55841 | 0,74054 | 0,28051 | 0,05599 | 1,07704 | 0,50500 | 0,17467 | 0,03380 | 0,71347 | 3,349 | 37,21 |
| 31 | SDOm | spiral dynamics optimization M | 0,74601 | 0,44623 | 0,29687 | 1,48912 | 0,70204 | 0,34678 | 0,10944 | 1,15826 | 0,42833 | 0,16767 | 0,03663 | 0,63263 | 3,280 | 36,44 |
| 32 | NMm | Nelder-Mead method M | 0,73807 | 0,50598 | 0,31342 | 1,55747 | 0,63674 | 0,28302 | 0,08221 | 1,00197 | 0,44667 | 0,18667 | 0,04028 | 0,67362 | 3,233 | 35,92 |
| 33 | FAm | firefly algorithm M | 0,58634 | 0,47228 | 0,32276 | 1,38138 | 0,68467 | 0,37439 | 0,10908 | 1,16814 | 0,28667 | 0,16467 | 0,04722 | 0,49855 | 3,048 | 33,87 |
| 34 | GSA | gravitational search algorithm | 0,64757 | 0,49197 | 0,30062 | 1,44016 | 0,53962 | 0,36353 | 0,09945 | 1,00260 | 0,32667 | 0,12200 | 0,01917 | 0,46783 | 2,911 | 32,34 |
| 35 | BFO | bacterial foraging optimization | 0,61171 | 0,43270 | 0,31318 | 1,35759 | 0,54410 | 0,21511 | 0,05676 | 0,81597 | 0,42167 | 0,13800 | 0,03195 | 0,59162 | 2,765 | 30,72 |
| 36 | ABC | artificial bee colony | 0,63377 | 0,42402 | 0,30892 | 1,36671 | 0,55103 | 0,21874 | 0,05623 | 0,82600 | 0,34000 | 0,14200 | 0,03102 | 0,51302 | 2,706 | 30,06 |
| 37 | BA | bat algorithm | 0,59761 | 0,45911 | 0,35242 | 1,40915 | 0,40321 | 0,19313 | 0,07175 | 0,66810 | 0,21000 | 0,10100 | 0,03517 | 0,34617 | 2,423 | 26,93 |
| 38 | AAA | algae adaptive algorithm | 0,50007 | 0,32040 | 0,25525 | 1,07572 | 0,37021 | 0,22284 | 0,16785 | 0,76089 | 0,27846 | 0,14800 | 0,09755 | 0,52402 | 2,361 | 26,23 |
| 39 | SA | simulated annealing | 0,55787 | 0,42177 | 0,31549 | 1,29513 | 0,34998 | 0,15259 | 0,05023 | 0,55280 | 0,31167 | 0,10033 | 0,02883 | 0,44083 | 2,289 | 25,43 |
| 40 | IWDm | intelligent water drops M | 0,54501 | 0,37897 | 0,30124 | 1,22522 | 0,46104 | 0,14704 | 0,04369 | 0,65177 | 0,25833 | 0,09700 | 0,02308 | 0,37842 | 2,255 | 25,06 |
| 41 | PSO | particle swarm optimisation | 0,59726 | 0,36923 | 0,29928 | 1,26577 | 0,37237 | 0,16324 | 0,07010 | 0,60572 | 0,25667 | 0,08000 | 0,02157 | 0,35823 | 2,230 | 24,77 |
| 42 | Boids | boids algorithm | 0,43340 | 0,30581 | 0,25425 | 0,99346 | 0,35718 | 0,20160 | 0,15708 | 0,71586 | 0,27846 | 0,14277 | 0,09834 | 0,51957 | 2,229 | 24,77 |
| 43 | MA | monkey algorithm | 0,59107 | 0,42681 | 0,31816 | 1,33604 | 0,31138 | 0,14069 | 0,06612 | 0,51819 | 0,22833 | 0,08567 | 0,02790 | 0,34190 | 2,196 | 24,40 |
| 44 | SFL | shuffled frog-leaping | 0,53925 | 0,35816 | 0,29809 | 1,19551 | 0,37141 | 0,11427 | 0,04051 | 0,52618 | 0,27167 | 0,08667 | 0,02402 | 0,38235 | 2,104 | 23,38 |
| 45 | FSS | fish school search | 0,55669 | 0,39992 | 0,31172 | 1,26833 | 0,31009 | 0,11889 | 0,04569 | 0,47467 | 0,21167 | 0,07633 | 0,02488 | 0,31288 | 2,056 | 22,84 |
Выводы
Вашему вниманию были представлены версии алгоритма BCO оригинальная и BCOm модифицированная. Оригинальная версия, воссозданная по информации из открытых источников, оказалась перенагруженнной тяжелыми тригонометрическими вычислениями и со слабыми поисковыми свойствами, а также имела математические "ляпы". Необходимо было переосмыслить весь алгоритм и провести глубокий анализ поисковой стратегии и создать новую модифицированную версию. Изменения в логике алгоритма привели к более простым вычислениям на каждом шаге оптимизации, что положительно сказалось на скорости выполнения кода. Новый алгоритм также по-другому балансирует между исследованием нового пространства решений и эксплуатацией уже найденных хороших решений.
Несмотря на то, что обновленный подход немного противоречит естественному поведению бактерий, он демонстрирует высокую эффективность в практическом применении. Визуализация работы алгоритма показала его способность к глубокому исследованию значимых участков гиперпространства, что также свидетельствует об его улучшенных исследовательских способностях. В итоге, новая версия алгоритма оказалась более мощной и эффективной в сравнении с оригиналом.
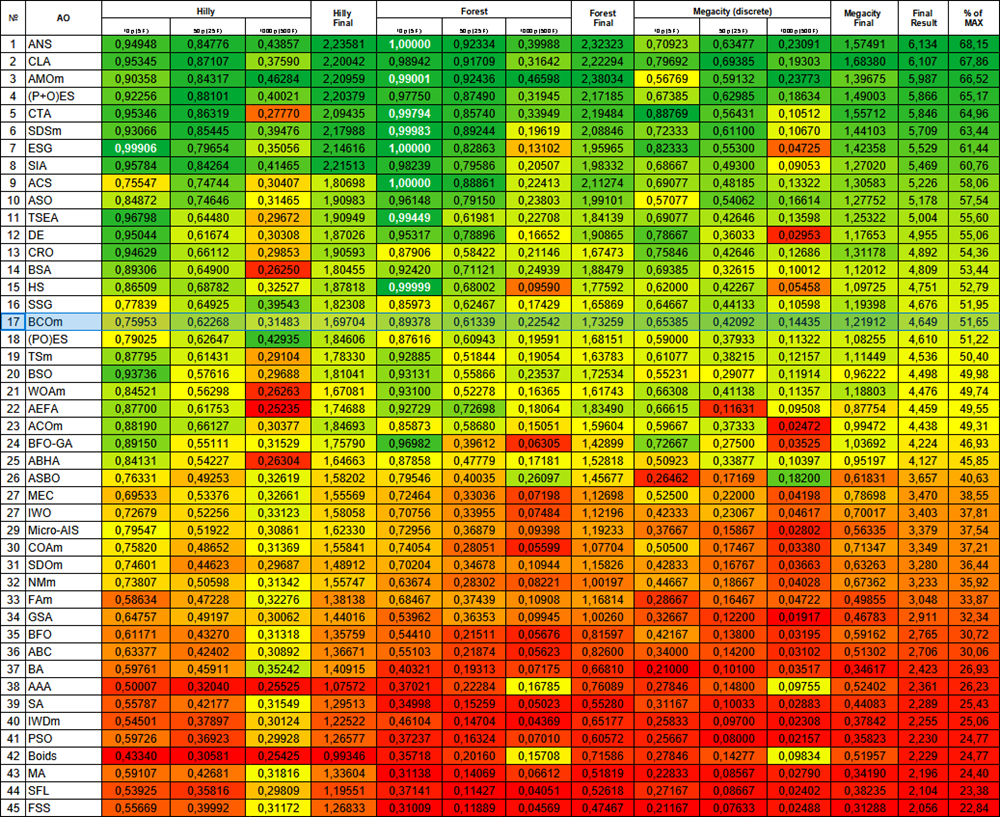
Рисунок 1. Цветовая градация алгоритмов по соответствующим тестам. Белым цветом подсвечены результаты больше или равные 0.99
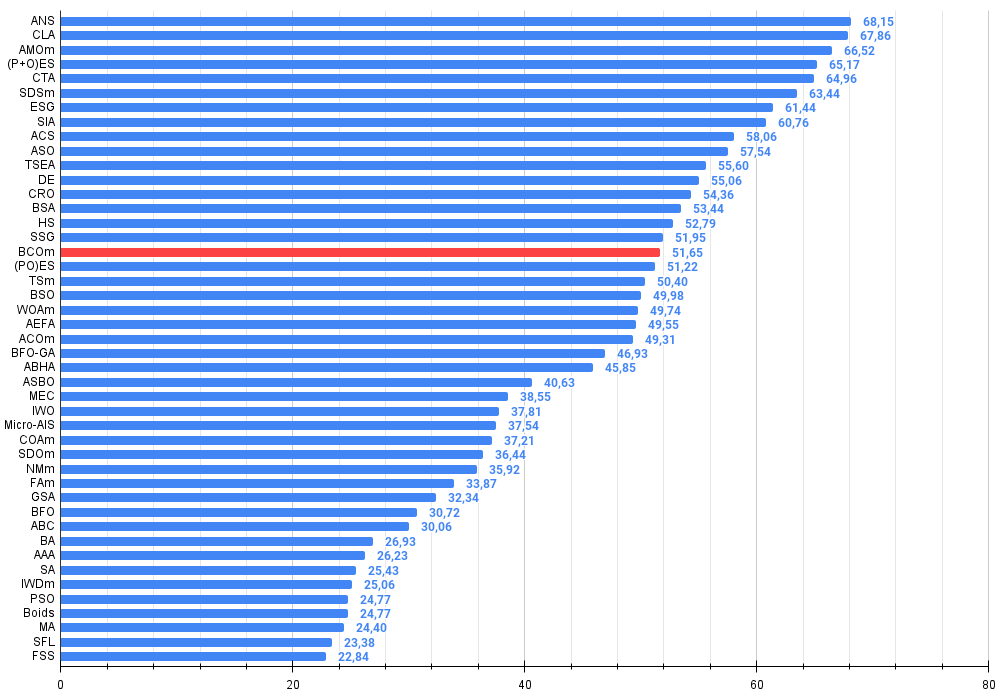
Рисунок 2. Гистограмма результатов тестирования алгоритмов (по шкале от 0 до 100, чем больше, тем лучше,
где 100 - максимально возможный теоретический результат, в архиве скрипт для расчета рейтинговой таблицы)
Плюсы и минусы алгоритма BCOm:
Плюсы:
- Быстрый.
- Самоадаптируемый.
- Хорошая масштабируемость.
- Всего лишь один внешний параметр.
Минусы:
- Высокий разброс результатов на функциях малой размерности.
К статье прикреплён архив с актуальными версиями кодов алгоритмов. Автор статьи не несёт ответственности за абсолютную точность в описании канонических алгоритмов, во многие из них были внесены изменения для улучшения поисковых возможностей. Выводы и суждения, представленные в статьях, основываются на результатах проведённых экспериментов.
Предупреждение: все права на данные материалы принадлежат MetaQuotes Ltd. Полная или частичная перепечатка запрещена.
Данная статья написана пользователем сайта и отражает его личную точку зрения. Компания MetaQuotes Ltd не несет ответственности за достоверность представленной информации, а также за возможные последствия использования описанных решений, стратегий или рекомендаций.
 Разработка системы репликации (Часть 46): Проект Chart Trade (V)
Разработка системы репликации (Часть 46): Проект Chart Trade (V)
 Нейросети в трейдинге: Иерархическое обучение признаков облака точек
Нейросети в трейдинге: Иерархическое обучение признаков облака точек
 Создаем и оптимизируем торговую систему на основе волатильности с индикатором Чайкина
Создаем и оптимизируем торговую систему на основе волатильности с индикатором Чайкина
 Построение модели для ограничения диапазона сигналов по тренду (Часть 1): Для советников и технических индикаторов
Построение модели для ограничения диапазона сигналов по тренду (Часть 1): Для советников и технических индикаторов
- Бесплатные приложения для трейдинга
- 8 000+ сигналов для копирования
- Экономические новости для анализа финансовых рынков
Вы принимаете политику сайта и условия использования