
化学反応最適化(CRO)アルゴリズム(第2回):組み立てと結果
内容
1. はじめに
記事の第2部では、化学反応最適化(CRO)の魅力的な世界をさらに詳しく見ていきます。「分子」と「素反応」の概念に基づいて、基礎となるアルゴリズムの原理を理解し、これらの概念が複雑な最適化問題を解決するためにどのように適用されるかを検討しました。また、CRO内のエネルギー保存の重要なポイントと、分解、合成、分子内および分子間の非効率な衝突など、探索空間を探索して最適解を見つける上で重要な役割を果たすアルゴリズム機能についても学びました。
化学反応最適化アルゴリズムの化学演算子の基本概念と動作原理について説明したので、次はアルゴリズムの一般的な組み立てと実際の応用に移ります。ここでは、さまざまなテスト関数に対するアルゴリズムの結果に焦点を当て、現実世界の問題を解決する際の効率性と可能性を分析します。パフォーマンス、収束、大域最適値を見つける能力を調べて、その適用性を評価するとともに、CROアルゴリズムの結果を他の最適化方法と比較して、その長所と短所を特定します。
2. アルゴリズムの実装
標準のInit、Moving、Revision関数を含む、すべてのアルゴリズムですでに使い慣れているテンプレートを使用して、アルゴリズムコードの記述を続けましょう。すでに構造体を宣言し、化学反応の主な演算子を実装しているので、すべてのコンポーネントを結び付けるアルゴリズムクラスの記述に進みましょう。
アルゴリズムのアセンブリを実装するには、疑似コードが必要になります。
revisionフラグがリセットされている場合 - 初期化:
0からpopSizeまでの各分子iについて:
0からcoordsまでの各座標cについて:
rangeMinからrangeMaxまでの分子の構造体のランダムな値を生成する
構造体の値を指定された範囲に制限する
構造体を配列aに保存する
終了
運動エネルギーの計算:
minKE = doubleの最大値
0からpopSizeまでの各分子iについて:
分子の適応度がminKE未満の場合:
minKE = 分子の適応度
0からpopSizeまでの各分子iについて:
分子の運動エネルギーKEを、minKEからfB(大域最適解)までの範囲から0.0から1.0までの範囲へのスケーリング適応度として計算
molCNT = 0
まだ止まらない
乱数がmoleCollより小さい場合:
ランダムに2つの分子M1とM2を選択する
両分子のKEがbeta以上の場合:
分子M1とM2の合成を実行する
その他の場合:
M1とM2の間で分子間無効衝突を実行する
その他の場合:
ランダムな分子Mを選択する
分子のNumHitがalphaを超える場合:
分子Mの分解を実行する
その他の場合:
分子Mの衝突を実行する
Mfilialからaに分子構造体ををコピーする
集団aの個体の適応度を計算する
分子構造体をaからMfilialにコピーする
初期化:ind = -1
0からpopSizeまでの各分子iについて:
分子の適応度がfBを超える場合:
fB = 分子の適応度
ind = i
indが-1と等しくない場合:
分子の構造体をcBにコピーする
分子更新:
revisionがない場合:
0からpopSizeまでの各分子iについて:
Mparentの分子の適合性を更新する
revisionをtrueに設定する
終了
0からpopSizeまでの各分子iについて:
Mfilialの分子の適合性を更新する
分子の種類(合成、分子間無効衝突、分解、衝突)に応じて:
適切な作業後処置をおこなう
記事の最初の部分で説明した疑似コードと化学演算子ができたので、コードでCROアルゴリズムの実装を構築し始めることができます。
C_AO基本クラスの継承であり、化学反応最適化(CRO)アルゴリズムの実装であるC_AO_CROクラスを宣言します。
1. publicフィールド
- popSize:母集団サイズ
- moleColl、alpha、beta、molecPerturb:アルゴリズムパラメータ
- params:アルゴリズムパラメータを格納するための配列
- Mparent[]、Mfilial[]:分子を表すS_CRO_Agent構造体のオブジェクト
2. オプション
- C_AO_CRO():クラスフィールドを初期化するクラスコンストラクタ
- SetParams():アルゴリズムパラメータを設定するメソッド
- Init():アルゴリズムを初期化するメソッド(最小検索範囲と最大検索範囲、検索ステップ、エポック数を受ける)
- Moving()およびRevision():アルゴリズムの基本的な操作を実装するメソッド
3. privateフィールドとメソッド
- Synthesis()、InterMolInefColl()、Decomposition()、InefCollision():さまざまな種類の反応を実装するメソッド
- PostSynthesis()、PostInterMolInefColl()、PostDecomposition()、PostInefCollision():対応する反応の後にアクションを実行するメソッド
- N():分子構造のコンポーネント(座標)を変更するメソッド
このクラスは、化学反応最適化(CRO)アルゴリズムの完全な実装であり、それに必要なすべてのデータとメソッドが含まれています。
//—————————————————————————————————————————————————————————————————————————————— class C_AO_CRO : public C_AO { public: //-------------------------------------------------------------------- ~C_AO_CRO () { } C_AO_CRO () { ao_name = "CRO"; ao_desc = "Chemical Reaction Optimisation"; ao_link = "https://www.mql5.com/ja/articles/15041"; popSize = 50; //population size moleColl = 0.9; alpha = 200; beta = 0.01; molecPerturb = 0.5; ArrayResize (params, 5); params [0].name = "popSize"; params [0].val = popSize; params [1].name = "moleColl"; params [1].val = moleColl; params [2].name = "alpha"; params [2].val = alpha; params [3].name = "beta"; params [3].val = beta; params [4].name = "molecPerturb"; params [4].val = molecPerturb; } void SetParams () { popSize = (int)params [0].val; moleColl = params [1].val; alpha = (int)params [2].val; beta = params [3].val; molecPerturb = params [4].val; } bool Init (const double &rangeMinP [], //minimum search range const double &rangeMaxP [], //maximum search range const double &rangeStepP [], //step search const int epochsP = 0); //number of epochs void Moving (); void Revision (); S_CRO_Agent Mparent []; S_CRO_Agent Mfilial []; //---------------------------------------------------------------------------- double moleColl; int alpha; double beta; double molecPerturb; private: //------------------------------------------------------------------- bool Synthesis (int index1, int index2, int &molCNT); bool InterMolInefColl (int index1, int index2, int &molCNT); bool Decomposition (int index, int &molCNT); bool InefCollision (int index, int &molCNT); void PostSynthesis (S_CRO_Agent &mol); void PostInterMolInefColl (S_CRO_Agent &mol); void PostDecomposition (S_CRO_Agent &mol); void PostInefCollision (S_CRO_Agent &mol); void N (double &coord, int coordPos); }; //——————————————————————————————————————————————————————————————————————————————
C_AO_CROクラスのInitメソッドは、渡されたパラメータに基づいてクラス変数を初期化するために使用されます。このメソッドでは以下をおこないます。
1. このメソッドは、最小および最大の検索範囲と検索ステップを取得するStandardInit関数を呼び出します。StandardInitがfalseを返す場合、Initメソッドもfalseを返して作業を完了します。
2. 次に、このメソッドは、Mparent配列とMfilial配列のサイズを、母集団サイズを表すpopSizeに変更します。
3. 次に、Mparent配列とMfilial配列の各要素に対して、coordsパラメータを使用してInitメソッドが呼び出されます。このメソッドは、集団内の各エージェントのフィールドを初期化します。
4.最後に、メソッドは初期化が正常に完了したことを示すtrueを返します。
このメソッドは、指定されたパラメータを使用して化学反応最適化(CRO)アルゴリズムの初期設定を実行し、最適化を実行する準備をします。
//—————————————————————————————————————————————————————————————————————————————— bool C_AO_CRO::Init (const double &rangeMinP [], //minimum search range const double &rangeMaxP [], //maximum search range const double &rangeStepP [], //step search const int epochsP = 0) //number of epochs { if (!StandardInit (rangeMinP, rangeMaxP, rangeStepP)) return false; //---------------------------------------------------------------------------- ArrayResize (Mparent, popSize); ArrayResize (Mfilial, popSize); for (int i = 0; i < popSize; i++) { Mparent [i].Init (coords); Mfilial [i].Init (coords); } return true; } //——————————————————————————————————————————————————————————————————————————————
C_AO_CROクラスのMovingメソッドは、分子の構造を変更し、最適化中に分子を移動する化学演算子を呼び出すために使用されます。このメソッドでは次をおこないます。
1. revisionがfalseに等しい場合、Mparent集団内の各分子について、構造体はrangeMinからrangeMaxまでの指定された範囲のランダムな値で初期化されます。これらの値は配列aにコピーされます。
2. revisionがfalseと等しくない場合、Mparent集団内のすべての分子の中で関数fの最小値が計算されます。次に、各分子のKE値は、その関数値f、親分子の集団による最小値f、および0.0から1.0の範囲の大域最適解fBのスケーリングに基づいて計算されます。
3. 次に、化学演算子の1つがfalseを返す(これは、娘分子集団に娘分子のためのスペースがなくなったことを意味する)まで次のことが起こります。
- 乱数がmoleCollより小さい場合、2つのランダム分子M1とM2が選択されます。両方の分子のKEがbeta以上の場合、合成が実行されます(つまり、合成はパラメータで指定された相対的な適合値以上の分子に対して実行されます。この目的のために、分子の適合値は事前に0.0から1.0の範囲にスケーリングされています)。そうしないと、分子間の無効な衝突が発生します。
- 乱数がmoleColl以上の場合、ランダムに1つの分子Mが選択されます。分子のNumHitがalphaを超える場合(分子がアルゴリズムパラメータで指定されたよりも多くの衝突を経験し、分子が「分解」した場合)、分解が実行されます。そうしないと衝突が発生します。
4.メソッドの最後に、Mfilial内のすべての分子の構造がa母集団配列にコピーされます。
このメソッドは、システムの現在の状態と指定されたパラメータに従って、化学反応最適化(CRO)アルゴリズムの分子構造を更新する役割を担います。この方法は、合成、分子間の非効率な衝突、分解、衝突などのCROアルゴリズムの基本的な操作を実装します。
//—————————————————————————————————————————————————————————————————————————————— void C_AO_CRO::Moving () { //---------------------------------------------------------------------------- if (!revision) { for (int i = 0; i < popSize; i++) { for (int c = 0; c < coords; c++) { Mparent [i].structure [c] = u.RNDfromCI (rangeMin [c], rangeMax [c]); // Random structure in the range from rangeMin to rangeMax Mparent [i].structure [c] = u.SeInDiSp (Mparent [i].structure [c], rangeMin [c], rangeMax [c], rangeStep [c]); a [i].c [c] = Mparent [i].structure [c]; } } return; } //---------------------------------------------------------------------------- double minKE = DBL_MAX; for (int i = 0; i < popSize; i++) { if (Mparent [i].f < minKE) minKE = Mparent [i].f; } for (int i = 0; i < popSize; i++) { Mparent [i].KE = u.Scale (Mparent [i].f, minKE, fB, 0.0, 1.0); } //---------------------------------------------------------------------------- int molCNT = 0; while (!IsStopped ()) { if (u.RNDprobab () < moleColl) { // Select two random molecules M1 and M2 int index1 = u.RNDminusOne (popSize); int index2 = u.RNDminusOne (popSize); // If KE ≤ β: if (Mparent [index1].KE >= beta && Mparent [index2].KE >= beta) { // Perform Synthesis if (!Synthesis (index1, index2, molCNT)) break; } else { // Perform Intermolecular Inefficient Collision if (!InterMolInefColl (index1, index2, molCNT)) break; } } else { // Select a random molecule M int index = u.RNDminusOne (popSize); // If NumHit > α: if (Mparent [index].NumHit > alpha) { // Perform Decomposition if (!Decomposition (index, molCNT)) break; } else { // Perform Collision if (!InefCollision (index, molCNT)) break; } } } for (int i = 0; i < popSize; i++) { ArrayCopy (a [i].c, Mfilial [i].structure); } } //——————————————————————————————————————————————————————————————————————————————
C_AO_CROクラスのRevisionメソッドは、化学ポスト演算子を実行して、大域最適解を更新し、親集団内の分子の状態を更新するために使用されます。このメソッドによって実行されるアクションは以下の通りです。
1. 大域解を更新する:forループですべての分子を反復処理します。現在の分子の関数fの値が現在の最適なfB値を超える場合、fBが更新され、現在の分子の座標の配列が配列cBにコピーされます。
2. revisionがfalseに等しい場合、Mparent集団内の各分子について、f値はa配列のf値と等しく設定されます。次に、revisionがtrueに設定され、メソッドが終了します。この段階では、親分子の適応度値を取得して、後続のエポックで運動エネルギーに依存する化学演算子(適応度関数の値を0.0~1.0の範囲に正規化)を取得することが重要です。
3. revisionがfalseでない場合、Mfilial集団内の各分子に対して、fはa配列のfと等しく設定されます。次に、分子のrType反応タイプ(分子が参加した反応)に応じて、適切なPostSynthesis、PostInterMolInefColl、PostDecomposition、PostInefCollisionメソッドが呼び出されます。
//—————————————————————————————————————————————————————————————————————————————— void C_AO_CRO::Revision () { //---------------------------------------------------------------------------- int ind = -1; for (int i = 0; i < popSize; i++) { if (a [i].f > fB) { fB = a [i].f; ind = i; } } if (ind != -1) ArrayCopy (cB, a [ind].c, 0, 0, WHOLE_ARRAY); //---------------------------------------------------------------------------- if (!revision) { for (int i = 0; i < popSize; i++) { for (int c = 0; c < coords; c++) { Mparent [i].f = a [i].f; } } revision = true; return; } //---------------------------------------------------------------------------- for (int i = 0; i < popSize; i++) { for (int c = 0; c < coords; c++) { Mfilial [i].f = a [i].f; } switch (Mfilial [i].rType) { case synthesis: PostSynthesis (Mfilial [i]); break; case interMolecularInefColl: PostInterMolInefColl (Mfilial [i]); break; case decomposition: PostDecomposition (Mfilial [i]); break; case inefCollision: PostInefCollision (Mfilial [i]); break; } } } //——————————————————————————————————————————————————————————————————————————————
3. テスト結果
CROアルゴリズムは、Hilly、Forest、Megacity関数でテストされました。それぞれのケースでは、各ランドスケープタイプ(5、25、500関数)に対して10回の関数実行が実行され、最適化結果が得られました。
CRO|Chemical Reaction Optimisation|50.0|0.9|200.0|0.01|0.5|
=============================
5 Hilly's; Func runs:10000; result:0.9462894520167225
25 Hilly's; Func runs:10000; result:0.6611186250435438
500 Hilly's; Func runs:10000; result:0.2985263035668822
=============================
5 Forest's; Func runs:10000; result:0.8790568514481787
25 Forest's; Func runs:10000; result:0.584216839762206
500 Forest's; Func runs:10000; result:0.2114595696419046
=============================
5 Megacity's; Func runs:10000; result:0.7584615384615384
25 Megacity's; Func runs:10000; result:0.4264615384615384
500 Megacity's; Func runs:10000; result:0.12686153846153955
=============================
All score:4.89245 (54.36%)
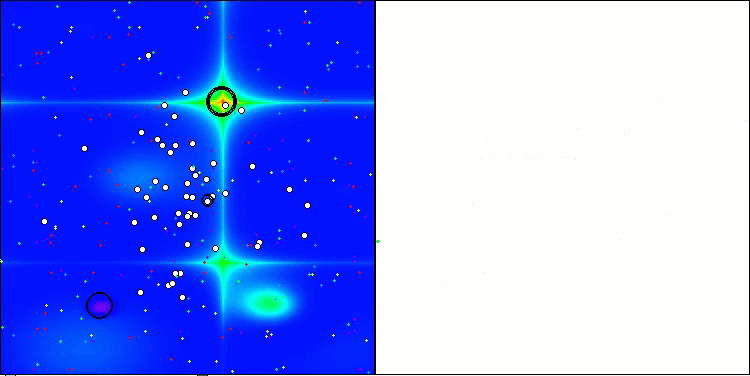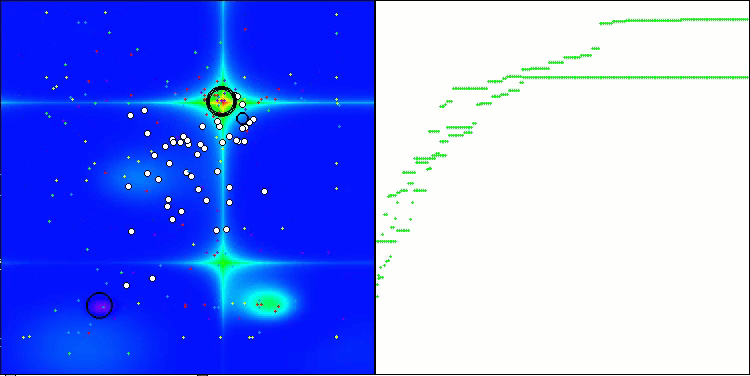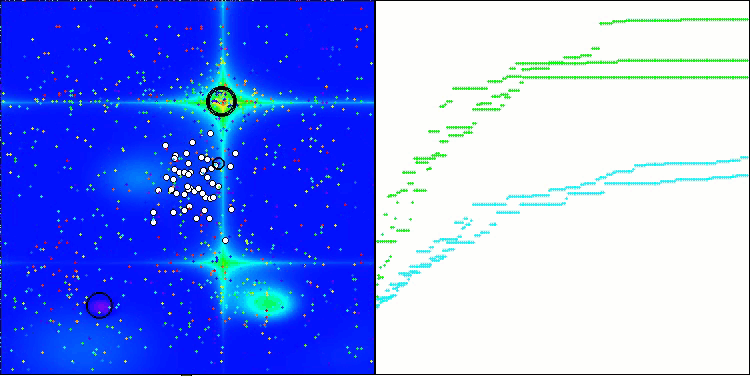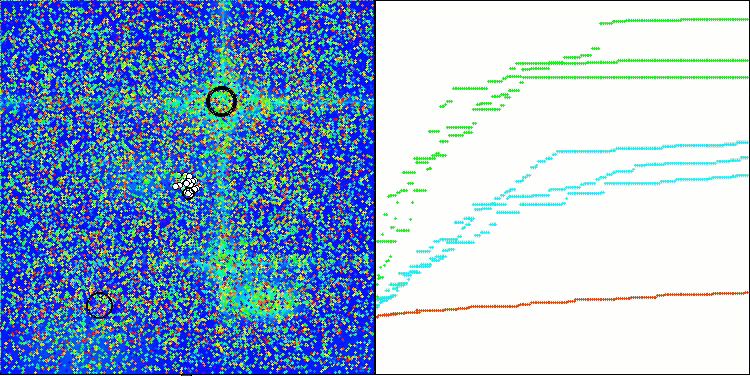
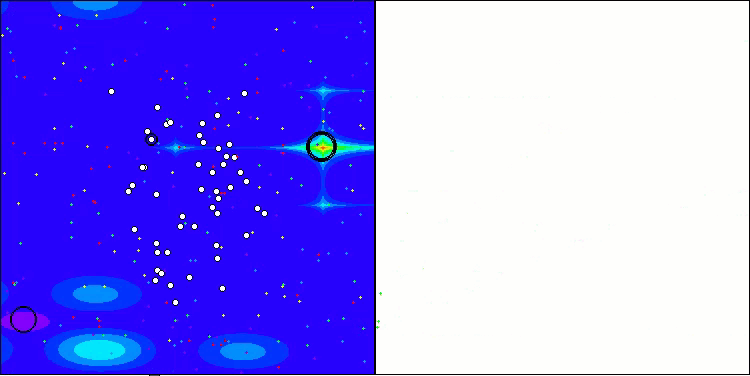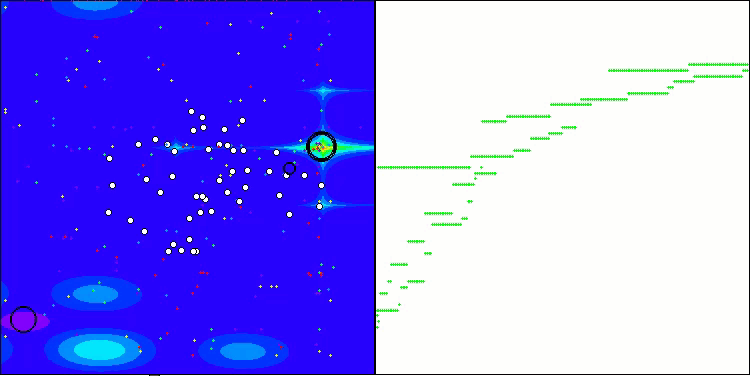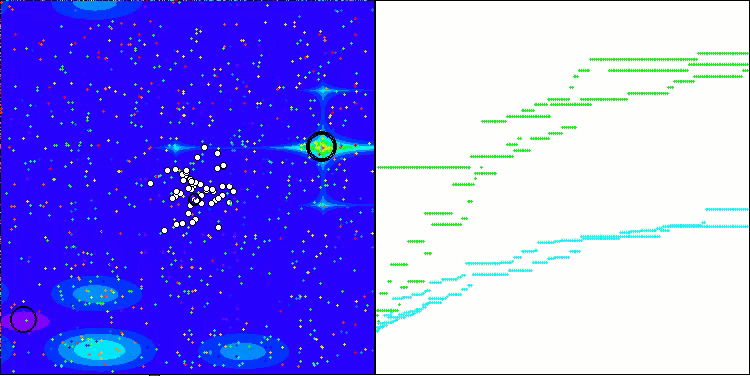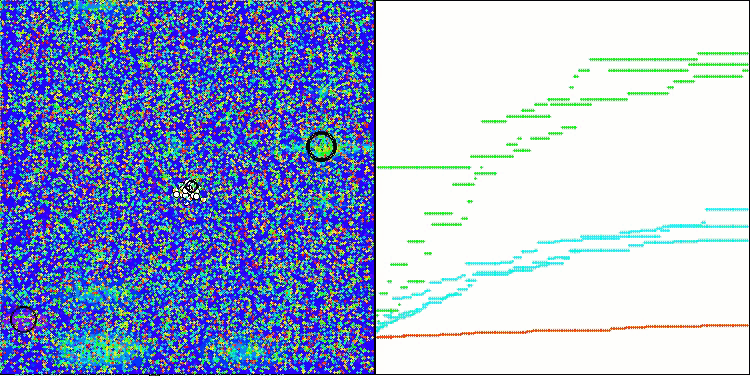
CROアルゴリズムを使用したテストベンチ操作の視覚化により、興味深いアルゴリズムの機能が示されます。CROは、収束グラフの長い平坦なセクションからもわかるように、時々行き詰まることがありますが、それでも全体的には良好な結果を示しています。
CRO動作の注目すべき点の1つは、検索領域内の「分子」の動きです。一見すると、この動きは混沌としていてブラウン運動に似ています。しかし、外部のランダム性にもかかわらず、「分子」は大域最適値ゾーンを見つけることができます。これは、化学反応の原理を使用して最適化問題を解決するCROアルゴリズムの複雑で洗練された性質を示しています。
全体として、CROアルゴリズムは、いくつかの課題はあるものの、さまざまなタスクを処理できる強力な最適化ツールです。そのユニークな特性と大域最適値を見つける能力により、最適化の分野で貴重なツールとなります。
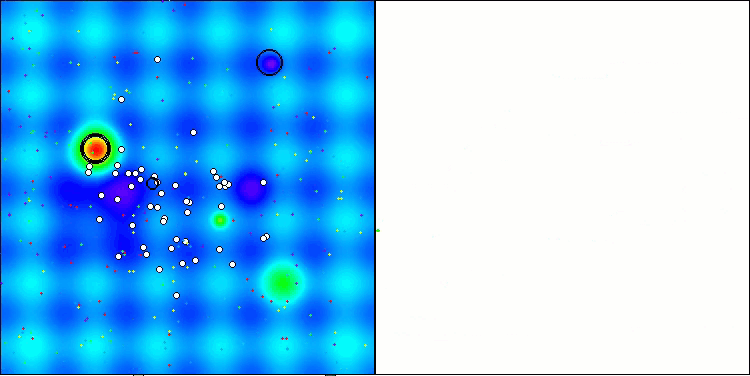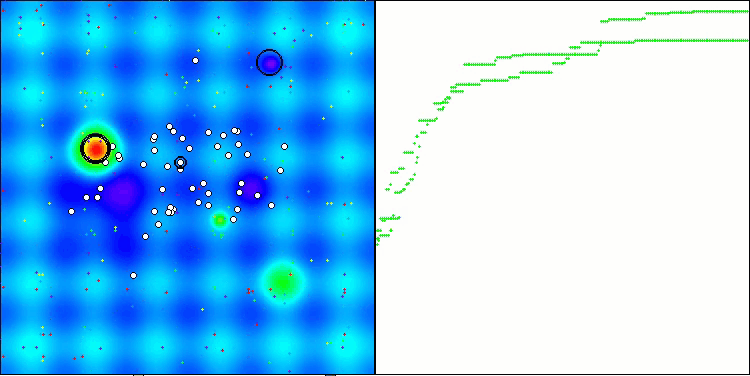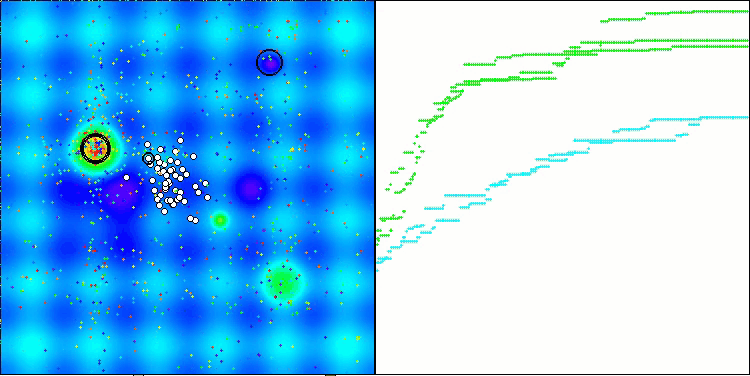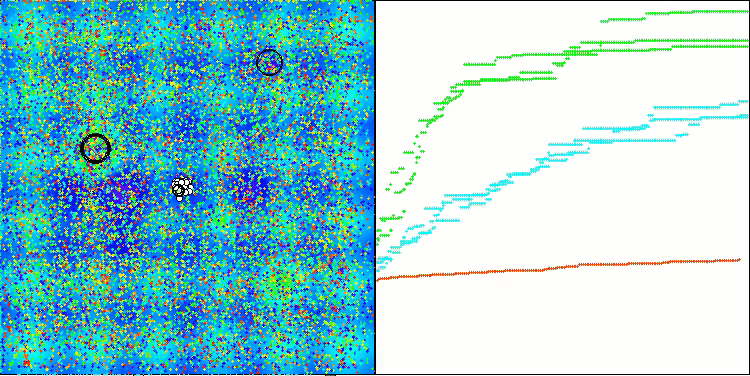
Hillyテスト関数のCRO
Forestテスト関数のCRO
Megacityテスト関数のCRO
| # | AO | 詳細 | Hilly | Hilly最終 | Forest | Forest最終 | Megacity(離散) | Megacity最終 | 最終結果 | MAXの% | ||||||
| 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | ||||||||
| 1 | BGA | バイナリ遺伝的アルゴリズム | 0.99989 | 0.99518 | 0.42835 | 2.42341 | 0.96153 | 0.96181 | 0.32027 | 2.24360 | 0.91385 | 0.95908 | 0.24220 | 2.11512 | 6.782 | 75.36 |
| 2 | CLA | コードロックアルゴリズム | 0.95345 | 0.87107 | 0.37590 | 2.20042 | 0.98942 | 0.91709 | 0.31642 | 2.22294 | 0.79692 | 0.69385 | 0.19303 | 1.68380 | 6.107 | 67.86 |
| 3 | (P+O)ES | (P+O)進化戦略 | 0.92256 | 0.88101 | 0.40021 | 2.20379 | 0.97750 | 0.87490 | 0.31945 | 2.17185 | 0.67385 | 0.62985 | 0.18634 | 1.49003 | 5.866 | 65.17 |
| 4 | CTA | 彗尾アルゴリズム | 0.95346 | 0.86319 | 0.27770 | 2.09435 | 0.99794 | 0.85740 | 0.33949 | 2.19484 | 0.88769 | 0.56431 | 0.10512 | 1.55712 | 5.846 | 64.96 |
| 5 | SDSm | 確率的拡散探索M | 0.93066 | 0.85445 | 0.39476 | 2.17988 | 0.99983 | 0.89244 | 0.19619 | 2.08846 | 0.72333 | 0.61100 | 0.10670 | 1.44103 | 5.709 | 63.44 |
| 6 | ESG | 社会集団の進化 | 0.99906 | 0.79654 | 0.35056 | 2.14616 | 1.00000 | 0.82863 | 0.13102 | 1.95965 | 0.82333 | 0.55300 | 0.04725 | 1.42358 | 5.529 | 61.44 |
| 7 | SIA | 等方的焼きなまし | 0.95784 | 0.84264 | 0.41465 | 2.21513 | 0.98239 | 0.79586 | 0.20507 | 1.98332 | 0.68667 | 0.49300 | 0.09053 | 1.27020 | 5.469 | 60.76 |
| 8 | ACS | 人工協調探索 | 0.75547 | 0.74744 | 0.30407 | 1.80698 | 1.00000 | 0.88861 | 0.22413 | 2.11274 | 0.69077 | 0.48185 | 0.13322 | 1.30583 | 5.226 | 58.06 |
| 9 | TSEA | 亀甲進化アルゴリズム | 0.96798 | 0.64480 | 0.29672 | 1.90949 | 0.99449 | 0.61981 | 0.22708 | 1.84139 | 0.69077 | 0.42646 | 0.13598 | 1.25322 | 5.004 | 55.60 |
| 10 | DE | 差分進化 | 0.95044 | 0.61674 | 0.30308 | 1.87026 | 0.95317 | 0.78896 | 0.16652 | 1.90865 | 0.78667 | 0.36033 | 0.02953 | 1.17653 | 4.955 | 55.06 |
| 11 | CRO | 化学反応の最適化 | 0.94629 | 0.66112 | 0.29853 | 1.90593 | 0.87906 | 0.58422 | 0.21146 | 1.67473 | 0.75846 | 0.42646 | 0.12686 | 1.31178 | 4.892 | 54.36 |
| 12 | BSA | 鳥群アルゴリズム | 0.89306 | 0.64900 | 0.26250 | 1.80455 | 0.92420 | 0.71121 | 0.24939 | 1.88479 | 0.69385 | 0.32615 | 0.10012 | 1.12012 | 4.809 | 53.44 |
| 13 | HS | ハーモニー検索 | 0.86509 | 0.68782 | 0.32527 | 1.87818 | 0.99999 | 0.68002 | 0.09590 | 1.77592 | 0.62000 | 0.42267 | 0.05458 | 1.09725 | 4.751 | 52.79 |
| 14 | SSG | 苗木の播種と育成 | 0.77839 | 0.64925 | 0.39543 | 1.82308 | 0.85973 | 0.62467 | 0.17429 | 1.65869 | 0.64667 | 0.44133 | 0.10598 | 1.19398 | 4.676 | 51.95 |
| 15 | (PO)ES | (PO)進化戦略 | 0.79025 | 0.62647 | 0.42935 | 1.84606 | 0.87616 | 0.60943 | 0.19591 | 1.68151 | 0.59000 | 0.37933 | 0.11322 | 1.08255 | 4.610 | 51.22 |
| 16 | BSO | ブレインストーム最適化 | 0.93736 | 0.57616 | 0.29688 | 1.81041 | 0.93131 | 0.55866 | 0.23537 | 1.72534 | 0.55231 | 0.29077 | 0.11914 | 0.96222 | 4.498 | 49.98 |
| 17 | WOAm | 鯨最適化アルゴリズムM | 0.84521 | 0.56298 | 0.26263 | 1.67081 | 0.93100 | 0.52278 | 0.16365 | 1.61743 | 0.66308 | 0.41138 | 0.11357 | 1.18803 | 4.476 | 49.74 |
| 18 | ACOm | 蟻コロニー最適化M | 0.88190 | 0.66127 | 0.30377 | 1.84693 | 0.85873 | 0.58680 | 0.15051 | 1.59604 | 0.59667 | 0.37333 | 0.02472 | 0.99472 | 4.438 | 49.31 |
| 19 | BFO-GA | 細菌採食の最適化:Ga | 0.89150 | 0.55111 | 0.31529 | 1.75790 | 0.96982 | 0.39612 | 0.06305 | 1.42899 | 0.72667 | 0.27500 | 0.03525 | 1.03692 | 4.224 | 46.93 |
| 20 | MEC | Mind Evolutionary Computation | 0.69533 | 0.53376 | 0.32661 | 1.55569 | 0.72464 | 0.33036 | 0.07198 | 1.12698 | 0.52500 | 0.22000 | 0.04198 | 0.78698 | 3.470 | 38.55 |
| 21 | IWO | 侵入雑草最適化 | 0.72679 | 0.52256 | 0.33123 | 1.58058 | 0.70756 | 0.33955 | 0.07484 | 1.12196 | 0.42333 | 0.23067 | 0.04617 | 0.70017 | 3.403 | 37.81 |
| 22 | Micro-AIS | 微小人工免疫系 | 0.79547 | 0.51922 | 0.30861 | 1.62330 | 0.72956 | 0.36879 | 0.09398 | 1.19233 | 0.37667 | 0.15867 | 0.02802 | 0.56335 | 3.379 | 37.54 |
| 23 | COAm | カッコウ最適化アルゴリズムM | 0.75820 | 0.48652 | 0.31369 | 1.55841 | 0.74054 | 0.28051 | 0.05599 | 1.07704 | 0.50500 | 0.17467 | 0.03380 | 0.71347 | 3.349 | 37.21 |
| 24 | SDOm | 螺旋ダイナミクス最適化M | 0.74601 | 0.44623 | 0.29687 | 1.48912 | 0.70204 | 0.34678 | 0.10944 | 1.15826 | 0.42833 | 0.16767 | 0.03663 | 0.63263 | 3.280 | 36.44 |
| 25 | NMm | ネルダー=ミード法M | 0.73807 | 0.50598 | 0.31342 | 1.55747 | 0.63674 | 0.28302 | 0.08221 | 1.00197 | 0.44667 | 0.18667 | 0.04028 | 0.67362 | 3.233 | 35.92 |
| 26 | FAm | ホタルアルゴリズムM | 0.58634 | 0.47228 | 0.32276 | 1.38138 | 0.68467 | 0.37439 | 0.10908 | 1.16814 | 0.28667 | 0.16467 | 0.04722 | 0.49855 | 3.048 | 33.87 |
| 27 | GSA | 重力探索法 | 0.64757 | 0.49197 | 0.30062 | 1.44016 | 0.53962 | 0.36353 | 0.09945 | 1.00260 | 0.32667 | 0.12200 | 0.01917 | 0.46783 | 2.911 | 32.34 |
| 28 | BFO | 細菌採餌最適化 | 0.61171 | 0.43270 | 0.31318 | 1.35759 | 0.54410 | 0.21511 | 0.05676 | 0.81597 | 0.42167 | 0.13800 | 0.03195 | 0.59162 | 2.765 | 30.72 |
| 29 | ABC | 人工蜂コロニー | 0.63377 | 0.42402 | 0.30892 | 1.36671 | 0.55103 | 0.21874 | 0.05623 | 0.82600 | 0.34000 | 0.14200 | 0.03102 | 0.51302 | 2.706 | 30.06 |
| 30 | BA | コウモリアルゴリズム | 0.59761 | 0.45911 | 0.35242 | 1.40915 | 0.40321 | 0.19313 | 0.07175 | 0.66810 | 0.21000 | 0.10100 | 0.03517 | 0.34617 | 2.423 | 26.93 |
| 31 | SA | 焼きなまし | 0.55787 | 0.42177 | 0.31549 | 1.29513 | 0.34998 | 0.15259 | 0.05023 | 0.55280 | 0.31167 | 0.10033 | 0.02883 | 0.44083 | 2.289 | 25.43 |
| 32 | IWDm | intelligent water drops M | 0.54501 | 0.37897 | 0.30124 | 1.22522 | 0.46104 | 0.14704 | 0.04369 | 0.65177 | 0.25833 | 0.09700 | 0.02308 | 0.37842 | 2.255 | 25.06 |
| 33 | PSO | 粒子群最適化 | 0.59726 | 0.36923 | 0.29928 | 1.26577 | 0.37237 | 0.16324 | 0.07010 | 0.60572 | 0.25667 | 0.08000 | 0.02157 | 0.35823 | 2.230 | 24.77 |
| 34 | ボイド | ボイドアルゴリズム | 0.43340 | 0.30581 | 0.25425 | 0.99346 | 0.35718 | 0.20160 | 0.15708 | 0.71586 | 0.27846 | 0.14277 | 0.09834 | 0.51957 | 2.229 | 24.77 |
| 35 | MA | モンキーアルゴリズム | 0.59107 | 0.42681 | 0.31816 | 1.33604 | 0.31138 | 0.14069 | 0.06612 | 0.51819 | 0.22833 | 0.08567 | 0.02790 | 0.34190 | 2.196 | 24.40 |
| 36 | SFL | Shuffled Frog-Leaping | 0.53925 | 0.35816 | 0.29809 | 1.19551 | 0.37141 | 0.11427 | 0.04051 | 0.52618 | 0.27167 | 0.08667 | 0.02402 | 0.38235 | 2.104 | 23.38 |
| 37 | FSS | 魚群検索 | 0.55669 | 0.39992 | 0.31172 | 1.26833 | 0.31009 | 0.11889 | 0.04569 | 0.47467 | 0.21167 | 0.07633 | 0.02488 | 0.31288 | 2.056 | 22.84 |
| 38 | RND | ランダム | 0.52033 | 0.36068 | 0.30133 | 1.18234 | 0.31335 | 0.11787 | 0.04354 | 0.47476 | 0.25333 | 0.07933 | 0.02382 | 0.35648 | 2.014 | 22.37 |
| 39 | GWO | 灰色オオカミオプティマイザ | 0.59169 | 0.36561 | 0.29595 | 1.25326 | 0.24499 | 0.09047 | 0.03612 | 0.37158 | 0.27667 | 0.08567 | 0.02170 | 0.38403 | 2.009 | 22.32 |
| 40 | CSS | 荷電系探索 | 0.44252 | 0.35454 | 0.35201 | 1.14907 | 0.24140 | 0.11345 | 0.06814 | 0.42299 | 0.18333 | 0.06300 | 0.02322 | 0.26955 | 1.842 | 20.46 |
| 41 | EM | 電磁気学的アルゴリズム | 0.46250 | 0.34594 | 0.32285 | 1.13129 | 0.21245 | 0.09783 | 0.10057 | 0.41085 | 0.15667 | 0.06033 | 0.02712 | 0.24412 | 1.786 | 19.85 |
まとめ
提供された表と結果に基づいて、CROアルゴリズムのパフォーマンスについて次の結論を導き出すことができます。
1. CROはHillyテスト関数で優れた結果を示します。パラメータが5つの場合の結果は約0.95、パラメータが25の場合の結果は約0.66、パラメータが500の場合の結果は約0.30でした。これは、CROが滑らかな関数、特にパラメータが少ない関数に効果的であることを示しています。
2.Forestテスト関数でも、CROは良好な結果を示しています。パラメータが5つの場合の結果は約0.88、パラメータが25の場合の結果は約0.58、パラメータが500の場合の結果は約0.21でした。これは、CROが「鋭い」極値を持つ関数にも効果的であるが、最適点を見つけるのが多少難しいことを示唆しています。
3. Megacityテスト関数では、CROは引き続き優れたパフォーマンスを発揮します。パラメータが5つの場合の結果は約0.76、パラメータが25の場合の結果は約0.43、パラメータが500の場合の結果は約0.13でした。これは、CROがこの離散関数に効果的であり、その結果が、表の上位にある他のアルゴリズムと比較しても一様に「緑」であることを示しています。
提供された表に基づくと、CROアルゴリズムは他のアルゴリズムと比較して優れた結果を示しています。特に、Hilly、Forest、Megacity関数では、CROは、特にパラメータが少ない場合に競争力を発揮します。
CROアルゴリズムはランキング表で11位になりました。下の表の色のグラデーション(濃い緑色はより良い結果を示します)に基づくと、CROは一般的に良好で安定したパフォーマンス(安定した均一な色)を示していると言えます。1000個のパラメータを持つHilly関数では、結果はやや弱くなります。
CROアルゴリズムは、有望な最適化アプローチであることが証明されています。私の実装では、多様性を提供し、局所最適値に陥らないようにするために、相互に作用する2つのエージェント集団を使用します。このアルゴリズムの特徴的な機能の1つは、化学反応、分解、合成などに類似した特殊な演算子を使用することです。
一般的に、CROアルゴリズムは、独創性とさまざまな最適化問題で高い結果を達成できる能力を特徴とする有望な最適化手法です。
最適化アルゴリズムの選択は、特定のタスクとパフォーマンス要件に基づいておこなう必要があり、ランキング表がこれに役立ちます。CROアルゴリズムのオリジナルバージョンを、母集団アルゴリズムに採用したC_AOクラスから継承するように作り直すことで、この興味深いアルゴリズムを一般的な最適化問題に適用できるようになります。
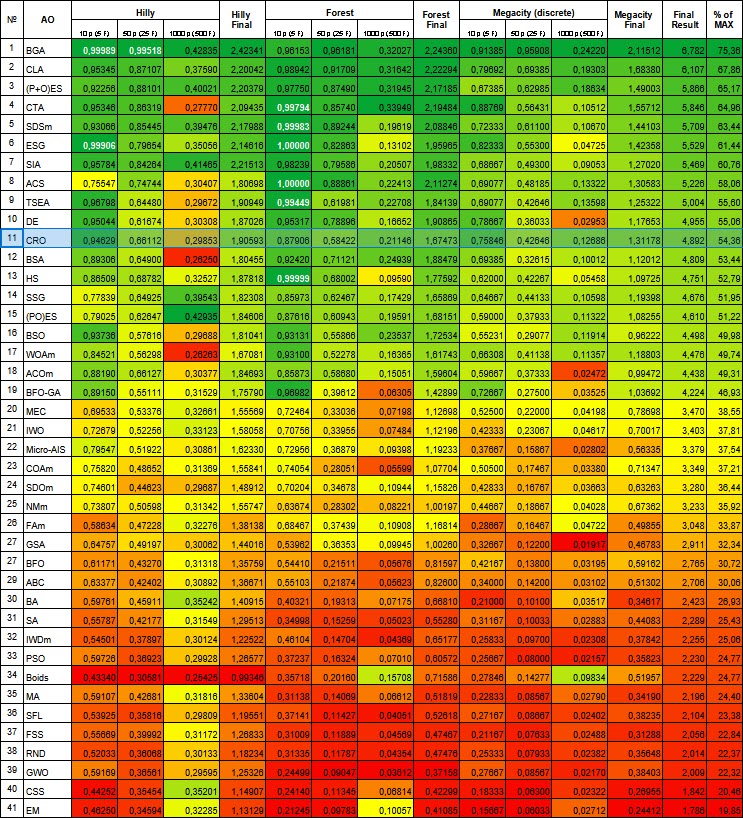
図1:関連テストによるアルゴリズムのカラーグラデーション0.99以上の結果は白で強調表示される
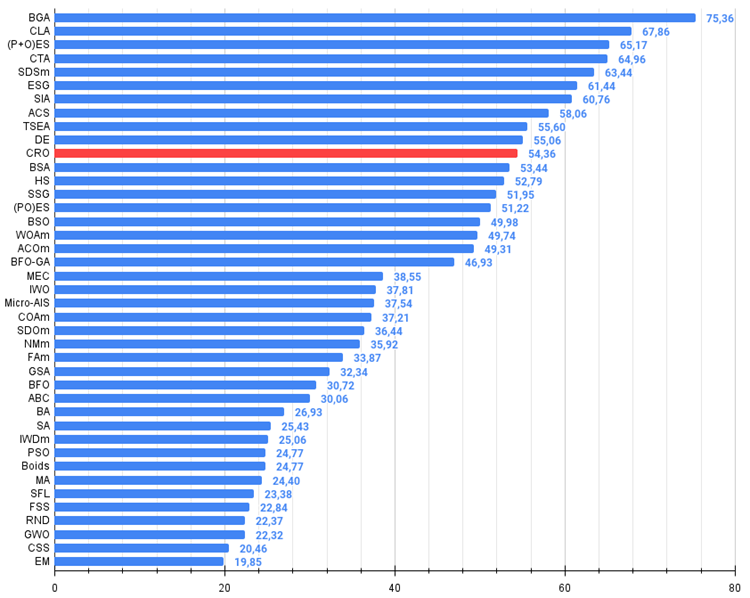
図2:アルゴリズムのテスト結果のヒストグラム(0から100までのスケールで、多ければ多いほど良い、
ここで、100は理論的に可能な最大の結果であり、アーカイブにはレーティング表を計算するスクリプトが含まれている)
CROの一般的な長所と短所
長所:
- 様々な種類の関数に対して収束性が良い
- 複雑なアーキテクチャにもかかわらず、非常に高速
- 優れたスケーラビリティ
短所
- 時々、極値にはまってしまうことがある
この記事には、最新版のアルゴリズムコードを含むアーカイブが添付されています。記事の著者は、正規アルゴリズムの説明の絶対的な正確さについて責任を負いません。検索機能を向上させるために、それらの多くに変更が加えられています。記事に示された結論と判断は、実験結果に基づいています。
MetaQuotes Ltdによってロシア語から翻訳されました。
元の記事: https://www.mql5.com/ru/articles/15080
警告: これらの資料についてのすべての権利はMetaQuotes Ltd.が保有しています。これらの資料の全部または一部の複製や再プリントは禁じられています。
この記事はサイトのユーザーによって執筆されたものであり、著者の個人的な見解を反映しています。MetaQuotes Ltdは、提示された情報の正確性や、記載されているソリューション、戦略、または推奨事項の使用によって生じたいかなる結果についても責任を負いません。
 ニューラルネットワークが簡単に(第95回):Transformerモデルにおけるメモリ消費の削減
ニューラルネットワークが簡単に(第95回):Transformerモデルにおけるメモリ消費の削減
 ニューラルネットワークが簡単に(第94回):入力シーケンスの最適化
ニューラルネットワークが簡単に(第94回):入力シーケンスの最適化
 PythonとMQL5でロボットを開発する(第3回):モデルベース取引アルゴリズムの実装
PythonとMQL5でロボットを開発する(第3回):モデルベース取引アルゴリズムの実装
 ニューラルネットワークの実践:擬似逆行列(I)
ニューラルネットワークの実践:擬似逆行列(I)
- 無料取引アプリ
- 8千を超えるシグナルをコピー
- 金融ニュースで金融マーケットを探索