母集団最適化アルゴリズム:細菌採餌最適化-遺伝的アルゴリズム(BFO-GA)
内容
1.はじめに
BFO-GAハイブリッド最適化アルゴリズムは、採餌最適化(BFO)アルゴリズムと遺伝的アルゴリズム(GA)という2つの異なる最適化アルゴリズムを組み合わせたものです。このハイブリッドアルゴリズムは、最適化の効率を向上させ、個々のアルゴリズムの欠点を克服するために作成されました。
BFO (Bacterial Foraging Optimization、細菌採餌最適化)は、細菌の採食行動にヒントを得た最適化アルゴリズムです。BFOは、2002年にRahul K. Kujurによって提唱されました。遷移、拡散、位置更新という3つの主なメカニズムを用いて細菌の動きがモデル化されます。アルゴリズム内の各細菌は最適化問題の解を表し、餌は最適解に相当します。細菌は探索空間を移動し、最適な餌を探します。
遺伝的アルゴリズム(GA)とは、自然淘汰と遺伝学の原理にヒントを得た最適化アルゴリズムです。1970年代にJohn Hollandによって開発されました。GAでは、最適化問題の解を表す個体の集団を扱います。個体は交配(遺伝情報の結合)と突然変異(遺伝情報の無作為な変化)の操作を受けて新しい世代を作成します。数世代後、GAは最適解を見つけようと努力します。
ハイブリッドBFO-GAアルゴリズムは、両アルゴリズムの長所を兼ね備えています。BFOは局所探索に優れ、収束も早いですが、大域探索では奮闘するかもしれません。一方、GAは大域探索能力に優れていますが、収束が遅く、局所最適解から抜け出しにくくなります。ハイブリッドBFO-GAアルゴリズムは、大域的探索にBFOを使用し、局所最適解の精緻化にGAを使用することで、これらの限界を克服しようとするものです。
BFO-GA (Bacterial Foraging Optimization - Genetic Algorithm)ハイブリッドアルゴリズムは、2007年にD. H. Kim、A. Abraham、J.H. Choによって提案されました。このアルゴリズムは、群がる細菌の行動に基づく最適化原理と、選択、交叉、突然変異の遺伝的演算子を組み合わせたものです。
BFO-GAは、細菌アルゴリズムを基本として、遺伝的選択、交叉、突然変異演算子で拡張したものです。このアルゴリズムでは、細菌の群れを利用して最適解を見つけます。
BFO-GAでは、ルーレット法に基づくアルゴリズムを選択演算子として使用します。この方法は、適合度に比例した確率で親細菌を選択します。従って、適合性のより高い細菌は交雑のために選択される可能性が高くなります。
BFO-GAアルゴリズムにおける交叉は、算術交差演算子を用いて実装されます。2つの親菌の遺伝情報を組み合わせ、新しい遺伝子の組み合わせを持つ新しい子孫が作成されます。
BFO-GAにおける突然変異は、非一様ミハルイェビッチ突然変異体(non-uniform Mihaljevic mutator)を用いておこなわれます。この変異体は細菌の遺伝情報を無作為に変化させます。突然変異の不均一性とは、突然変異の確率が様々な要因によって変化することを意味します。
これらの修正演算子は、細菌アルゴリズムの走化性と繁殖の手順が完了した後、このアルゴリズムの排除と分散の手順の前に実行されます。つまり、細菌が最適解に向かって移動し、子孫を残した後、遺伝的演算子を適用して解を多様化し、改善します。
2.アルゴリズム
BFO-GAアルゴリズムは、最適化問題の最適解を見つけるために、群がる細菌の行動のモデリングを使用します。これはパラメータ空間における細菌の動きをシミュレートするもので、それぞれの細菌は問題に対する潜在的な解を表しています。細菌は、主に2つのタイプの動き、すなわち栄養勾配の方向への動きと無作為な方向への動きをすることによって、パラメータ空間内を移動します。
BFO-GAアルゴリズムは以下のステップを使用します。
- 初期化:細菌の初期母集団を作り、それぞれが問題に対する潜在的な解を示します。各細菌は、最適化プロセス中に変更可能な独自のパラメータを持っています。
- 栄養勾配の推定:母集団内の各細菌の適合度関数の値を計算し、最後の2つの値を比較し、この値の差が各細菌が提示した解の質を決定します。
- 勾配の方向への移動:各細菌は食物勾配の方向に移動するため、位置の変化ベクトルが一定で、より最適な解に向かって移動することができます。
- 無作為な方向への移動:細菌の中には、局所最適解にはまらないように無作為な動きをするものもあります。この動きは、過去に移動に失敗した場合の細菌のパラメータの無作為な変化に基づいています。
- 遺伝的演算子:選択、交配、突然変異という遺伝的演算子を細菌の集団に適用します。これにより、パラメータの新しい組み合わせを生み出し、解の質を向上させることができます。
- ステップ2~5を繰り返す:勾配方向への移動、無作為方向への移動、遺伝的演算子の適用といったステップを、最大反復回数に達するか、特定の適合関数値に達するといった停止基準に達するまで繰り返します。
理論的には、ハイブリッドBFO-GAアルゴリズムは、BFOに対して特筆すべき多くの利点を持つはずです。
- 探査と開発:BFO-GAには、細菌の無作為な動きによるパラメータ空間の探索能力と、食物勾配方向の動きによる搾取能力があります。これにより、アルゴリズムは局所最適解を避け、大域最適に収束することで、最適解を見つけることができます。
- 適応力:BFO-GAには、変化する最適化条件に適応する能力があります。アルゴリズムの作動中、細菌のパラメータは変化することができ、それによって細菌は新しい条件に適応し、より最適な解を見つけることができます。
- パラメータのチューニングは不要:他の最適化アルゴリズムと同様、BFO-GAにも、より良い結果を得るために調整可能なパラメータがあります。これには、細菌集団のサイズ、勾配方向への移動のステップと無作為な移動、遺伝的演算子の確率などに関するパラメータが含まれます。パラメータを変えても、結果に大きな影響はありません。

図1:娘菌による親の「遺伝子」の継承
理論的な基礎から実践的な実装に移りましょう。
第一に、ルーレット法(この方法は人工蜂コロニーアルゴリズムで使用されていた)による選択を断念しました。BFO-GAでは、親母集団からの単純無作為サンプリングを用いた実験の方が良い結果が得られました。
第二に、非一様ミハリエヴィッチ突然変異体を冪乗則突然変異に置き換えることにしました(いくつかの情報源では、冪乗則突然変異体の一種とされている)。実際、これはスマート頭足類稿で触れた、冪乗分布の乱数の生成です。
第三に、算術交叉演算子は、一様分布法則に従って無作為に選択された異なる親からの遺伝子の単純な無作為継承に置き換えられます。
細菌を記述するために、以下のフィールドを含むS_Bacteria構造体を記述します。
- c:細菌の座標の配列で、パラメータ空間における細菌の現在の座標を表します。c配列のサイズは、最適化問題の変数数に依存します。
- cLast:新しい位置が計算されるパラメータの空間における、細菌の前の位置を格納するために使用される、細菌の前の座標の配列。
- v:細菌ベクトルの配列で、パラメータ空間における細菌の現在の移動方向を表します。v配列のサイズは、最適化問題の変数数に依存します。
- f:細菌の健康状態(適合度)の現在値で、パラメータ空間における細菌の現在位置の質を決定します。fの値は大きいほど良いです。
- fLast:細菌の位置の以前の品質を追跡するために使用され、栄養勾配を決定する際に適用される細菌の以前の健康値。
- lifeCNT:細菌のライフカウンタは、細菌が現在の位置で過ごした反復回数を追跡します。このフィールドは、細菌が一方向に進むステップ数を制限するために使用されます。
//—————————————————————————————————————————————————————————————————————————————— struct S_Bacteria { double c []; //coordinates double cLast []; //coordinates double v []; //vector double f; //current health double fLast; //previous health double lifeCNT; //life counter }; //——————————————————————————————————————————————————————————————————————————————
BFO-GAアルゴリズムを実装したC_AO_BFO_GAクラスを宣言します。各フィールドとクラスメソッドを見てみましょう。
クラスのフィールド
- b:細菌の配列(配列の各要素は、アルゴリズム内の細菌を表すS_Bacteria型のオブジェクト)
- rangeMax: 最適化問題の各変数の検索範囲の最大値の配列
- rangeMin: 各変数の検索範囲の最小値の配列
- rangeStep: 各変数の検索ステップの配列
- cB: アルゴリズムによって見つかった最良の座標の配列
- fB:最良の座標に対する適合度関数の値
クラスのメソッド
- Init:BFO-GAアルゴリズムのパラメータを初期化します。paramsP(最適化パラメータ数)、opulationSizeP(個体群サイズ)、λ(細菌の移動長)、reproductionP(繁殖確率)、lifeCounterP(ライフカウンタ、ライフ数が終了すると細菌は宙返りをおこないます)、powerMutP(突然変異パワー)です。
- Swimming:パラメータ空間における細菌の移動をおこないます。
- Evaluation:母集団の修正と大域解の更新をおこないます。
privateクラスメソッド
- NewVector:指定された paramInd細菌パラメータ用の新しいベクトルを生成します。
- PowerDistribution:指定されたoutMin、outMax、power各パラメータで、In入力にパワー分布を適用します。
- Sorting:母集団内の細菌を適合度関数の値で降順に並び替えます。
- SeInDiSp:[InMin,InMax]範囲のIn入力をStepで正規化します。
- RNDfromCI:与えられた[min, max]範囲で乱数を生成します。
- Scale:In入力を[InMIN, InMAX]範囲から[OutMIN, OutMAX]範囲にスケーリングします。
//—————————————————————————————————————————————————————————————————————————————— class C_AO_BFO_GA { //---------------------------------------------------------------------------- public: S_Bacteria b []; //bacteria public: double rangeMax []; //maximum search range public: double rangeMin []; //manimum search range public: double rangeStep []; //step search public: double cB []; //best coordinates public: double fB; //FF of the best coordinates public: void Init (const int paramsP, //number of opt. parameters const int populationSizeP, //population size const double lambdaP, //lambda const double reproductionP, //probability of reproduction const int lifeCounterP, //life counter const double powerMutP); //mutation power public: void Swimming (); public: void Evaluation (); //---------------------------------------------------------------------------- private: double NewVector (int paramInd); private: S_Bacteria bT []; //bacteria private: double v []; private: int ind []; private: double val []; private: int populationSize; //population size private: int parameters; //number of optimized parameters private: double lambda; //lambda private: double reproduction; //probability of reproduction private: int lifeCounter; //life counter private: double powerMut; //mutation power private: bool evaluation; private: double PowerDistribution (const double In, const double outMin, const double outMax, const double power); private: void Sorting (); private: double SeInDiSp (double In, double InMin, double InMax, double Step); private: double RNDfromCI (double min, double max); private: double Scale (double In, double InMIN, double InMAX, double OutMIN, double OutMAX, bool revers); }; //——————————————————————————————————————————————————————————————————————————————
Initメソッドはクラスを初期化するために使用されます。このメソッドは、BFO-GAアルゴリズムのパラメータを初期化します。
メソッドの入力
- paramsP:最適化されたパラメータの数
- opulationSizeP:母集団のサイズ
- λP:各ステップでの細菌の移動距離を決定するλパラメータ
- reproductionP:繁殖確率
- lifeCounterP:ライフカウンタ
- powerMutP:突然変異のパワー
メソッド内部では、C_AO_BFO_GAクラスのフィールドが初期値で初期化され、配列のサイズが設定されます。
//—————————————————————————————————————————————————————————————————————————————— void C_AO_BFO_GA::Init (const int paramsP, //number of opt. parameters const int populationSizeP, //population size const double lambdaP, //lambda const double reproductionP, //probability of reproduction const int lifeCounterP, //life counter const double powerMutP) //mutation power { fB = -DBL_MAX; evaluation = false; parameters = paramsP; populationSize = populationSizeP; lambda = lambdaP; reproduction = reproductionP; lifeCounter = lifeCounterP; powerMut = powerMutP; ArrayResize (rangeMax, parameters); ArrayResize (rangeMin, parameters); ArrayResize (rangeStep, parameters); ArrayResize (v, parameters); ArrayResize (ind, populationSize); ArrayResize (val, populationSize); ArrayResize (b, populationSize); ArrayResize (bT, populationSize); for (int i = 0; i < populationSize; i++) { ArrayResize (b [i].c, parameters); ArrayResize (b [i].cLast, parameters); ArrayResize (b [i].v, parameters); b [i].f = -DBL_MAX; b [i].fLast = -DBL_MAX; ArrayResize (bT [i].c, parameters); ArrayResize (bT [i].cLast, parameters); ArrayResize (bT [i].v, parameters); } ArrayResize (cB, parameters); } //——————————————————————————————————————————————————————————————————————————————
細菌の半方向性(hemitaxis)、その複製、選択、特徴の継承、突然変異をおこなうためのSwimmingメソッドを書きます。
void Swimming ()
{
} 最初の反復では、evaluationフラグはfalseに等しいです。探索空間全体に細菌を一様分布で無作為に分布させます。この段階では、現在の健康状態(適応度)と以前の健康状態は不明です。DBL_MAX値を対応する変数に割り当てましょう。また、新しく作った細菌に無作為な動きベクトルを加えます。
細菌の座標が境界を越えていないか確認し、最適化されたパラメータを変更するステップを適用します。移動ベクトルによって、各細菌は一様に前方に泳ぐことができます。
if (!evaluation) { fB = -DBL_MAX; for (int s = 0; s < populationSize; s++) { for (int k = 0; k < parameters; k++) { b [s].c [k] = RNDfromCI (rangeMin [k], rangeMax [k]); b [s].c [k] = SeInDiSp (b [s].c [k], rangeMin [k], rangeMax [k], rangeStep [k]); v [k] = rangeMax [k] - rangeMin [k]; b [s].v [k] = NewVector (k); b [s].f = -DBL_MAX; b [s].fLast = -DBL_MAX; b [s].lifeCNT = 0; } } evaluation = true; return; }
細菌の繁殖(複製)確率は、まず2回目以降の反復で確認されます。もし繁殖の確率が満たされれば、次のことが起こります。
- 元の細菌:選別された細菌母集団の最良の半分は、同じ方向に移動を続けます。そのために、各細菌の移動ベクトルを前の位置の座標に加えます。
- 細菌クローン:母集団の後半分は、異なる親細菌の「遺伝子」(座標)から得られた娘細菌で満たされます。したがって、これらは特徴を継承し、アルゴリズムの組み合わせ能力を発揮します(上の図1を参照)。
- 異なる親から遺伝子を受け継いだクローン菌は、親の運動ベクトルの成分も受け継ぎ、受け継いだ遺伝子は冪乗則分布に従って突然変異を起こします。
このように、クローン菌は異なる親の特徴を受け継ぎます。この場合、遺伝子はその近傍で高い確率で変化します。確率は親遺伝子の値からの距離が遠くなるにつれて減少します。これによって、集団の優秀なメンバーだけが遺伝子を継承することができるため、両親の成功特性を組み合わせることができます。さらに、受け継がれた移動ベクトルの成分によって、娘菌は次の反復で、親ベクトルの最良の成分に従って移動します。
理想的には、これは魚の群れの動きに似ているはずですが、細菌の動きには多くの無作為な要因が影響するため、この例えは必ずしも正しくありません。そのような要因には、適性関数の景観、より幸運な集団のメンバーとの入れ替わり、生命数の制限による移動のベクトルの不可避な変化などがあります。
コロニーの上半分の細菌のライフカウンタは1つ増えますが、クローン化された細菌のライフカウンタはリセットされます。
double r = RNDfromCI (0.0, 1.0); if (r < reproduction) { int st = populationSize / 2; //bacterium original-------------------------------------------------------- for (int s = 0; s < st; s++) { for (int k = 0; k < parameters; k++) { b [s].c [k] = b [s].cLast [k] + b [s].v [k]; b [s].c [k] = SeInDiSp (b [s].c [k], rangeMin [k], rangeMax [k], rangeStep [k]); } b [s].lifeCNT++; } //bacterium clone----------------------------------------------------------- for (int s = st; s < populationSize; s++) { for (int k = 0; k < parameters; k++) { int i = (int)RNDfromCI (0, st); if (i >= st) i = st - 1; b [s].c [k] = b [i].cLast [k]; b [s].c [k] = PowerDistribution (b [s].c [k], rangeMin [k], rangeMax [k], powerMut); b [s].c [k] = SeInDiSp (b [s].c [k], rangeMin [k], rangeMax [k], rangeStep [k]); } b [s].lifeCNT = 0; } }
次に、繁殖確率が満たされない場合は、母集団全体についてループします。
for (int s = 0; s < populationSize; s++)
細菌の寿命が限界に達しているかどうかの確認が最初におこなわれます。生命の限界に達した細菌は宙返りをし、運動のベクトルを変えて新しい方向に動き始めます。ライフカウンタがリセットされます。
細菌が生命限界に達するのを確認した後、限界に達した細菌は突然変異を起こし、次の反復で移動ベクトルを変えて新しい方向に動き始めます。ライフカウンタがリセットされます。
if (b [s].lifeCNT >= lifeCounter) { for (int k = 0; k < parameters; k++) { b [s].v [k] = NewVector (k); b [s].c [k] = PowerDistribution (b [s].cLast [k], rangeMin [k], rangeMax [k], powerMut); b [s].c [k] = SeInDiSp (b [s].c [k], rangeMin [k], rangeMax [k], rangeStep [k]); } b [s].lifeCNT = 0; }
細菌がまだ寿命に達していない場合は、正の走化性、つまり細菌が増加する食物勾配に向かって移動しているかどうかを確認します。前の2つの反復における適合度値が等しい場合、細菌の動きは正しいとみなされます。なぜまったく等しいのでしょうか。次の段階であるEvaluationメソッドでは、細菌の健康状態にポジティブな変化があるかどうかを確認します。変化がポジティブであれば、現在の状態が前の状態に割り当てられます。最後の2つの反復における健康状態が同じであれば、これはより多くの餌を求める細菌の移動方向が正しいことを示しています。こうして細菌は、より多くの餌に向かって進むしかありません。そうしないと、細菌はその場で転がり始めます。ただし、これは問題ではありません。遅かれ早かれ、動けなくなった細菌は新しい状態に変異するか、より幸運な両親の遺伝子を受け継いで移動します。
正しい方向に進む細菌のライフカウンタは徐々に増えていきます。つまり、成功した細菌でさえ、遅かれ早かれ突然変異を起こすということです。これは前のコードブロックで言及されています。
if (b [s].f == b [s].fLast) { for (int k = 0; k < parameters; k++) { b [s].c [k] = b [s].cLast [k] + b [s].v [k]; b [s].c [k] = SeInDiSp (b [s].c [k], rangeMin [k], rangeMax [k], rangeStep [k]); } b [s].lifeCNT++; }
もしある細菌で正の走化性が観察されない場合、そのような細菌は移動のベクトルを変えて宙返りをします。このような細菌のライフカウンタも刻々と変化しています。このような不成功に終わった細菌のライフカウンタの値をもっと集中的に上げるというアイデアが生まれましたが、私はこのアイデアをテストしたことはありません。
for (int k = 0; k < parameters; k++) { b [s].v [k] = NewVector (k); b [s].c [k] = b [s].cLast [k] + b [s].v [k]; b [s].c [k] = SeInDiSp (b [s].c [k], rangeMin [k], rangeMax [k], rangeStep [k]); } b [s].lifeCNT++;
宙返り(移動ベクトルの変更)をおこなうには、paramIndインデックスの最適化されたパラメータに対して実行されるNewVectorメソッドを使用します。
- 乱数rは、RNDfromCI関数を使用して、[-1.0, 1.0]区間で一様分布で生成されます。
- 最適化されたパラメータの値の許容範囲vにλ比とr乱数を乗算することにより、ベクトル成分の新しい値が計算されます。
//—————————————————————————————————————————————————————————————————————————————— double C_AO_BFO_GA::NewVector (int paramInd) { double r = RNDfromCI (-1.0, 1.0); return lambda * v [paramInd] * r; } //——————————————————————————————————————————————————————————————————————————————Evaluationメソッドは、細菌間の競争を整理し、大域解を更新するために使用されます。
メソッドの開始時に、集団内の各細菌の正の走化性がテストされます。適合度関数の現在値b[s].fが以前の値 b[s].fLastよりも大きければ、以前の適合度と座標(細菌はここから移動する)が更新されます。
次に、細菌のfLast値を用いて、適合度関数の値の降順にSortingメソッドを用いて母集団を並び替えます。
この後、大域解が更新されます。
//—————————————————————————————————————————————————————————————————————————————— void C_AO_BFO_GA::Evaluation () { for (int s = 0; s < populationSize; s++) { if (b [s].f > b [s].fLast) { b [s].fLast = b [s].f; ArrayCopy (b [s].cLast, b [s].c, 0, 0, WHOLE_ARRAY); } } Sorting (); if (b [0].fLast > fB) { fB = b [0].fLast; ArrayCopy (cB, b [0].cLast, 0, 0, WHOLE_ARRAY); } } //——————————————————————————————————————————————————————————————————————————————
3.テスト結果
BFO-GAテストスタンドの結果
C_AO_BFO_GA:50;0.01;0.8;50;10.0
=============================
5 Hilly's; Func runs:10000; result:0.8914957961234459
25 Hilly's; Func runs:10000; result:0.5511088047221568
500 Hilly's; Func runs:10000; result:0.31529201642392934
=============================
5 Forest's; Func runs:10000; result:0.9698155977381523
25 Forest's; Func runs:10000; result:0.39612322805995287
500 Forest's; Func runs:10000; result:0.06304955092899359
=============================
5 Megacity's; Func runs:10000; result:0.7266666666666667
25 Megacity's; Func runs:10000; result:0.275
500 Megacity's; Func runs:10000; result:0.035250000000000004
=============================
All score:4.22380 (46.93%)
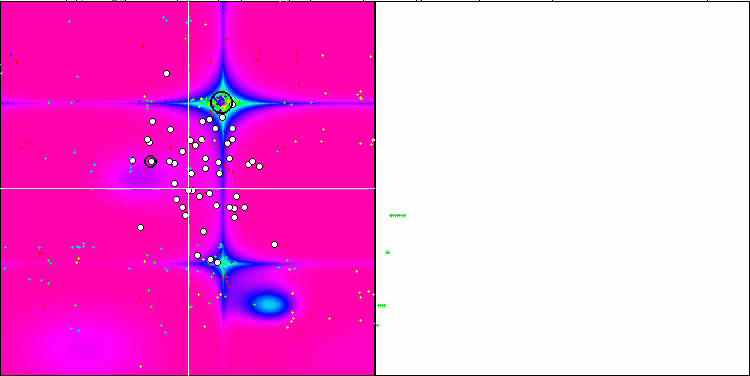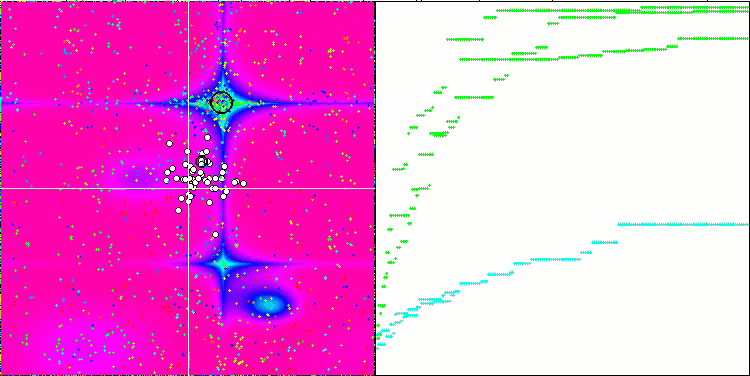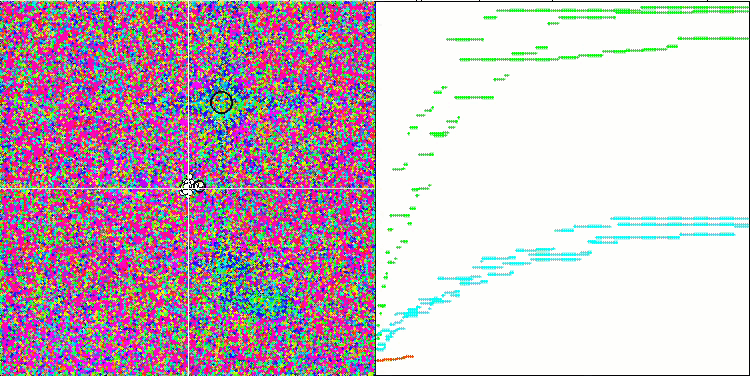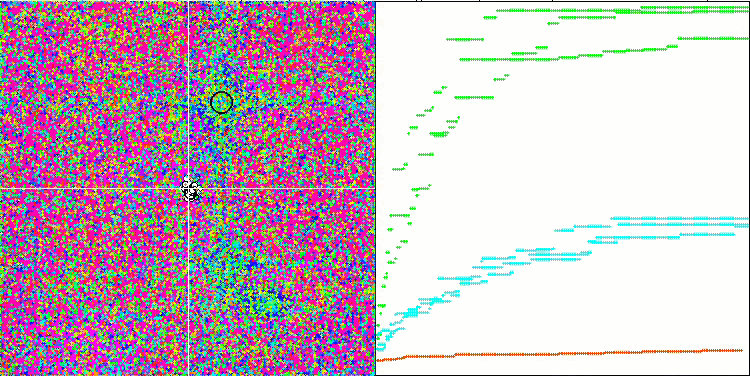
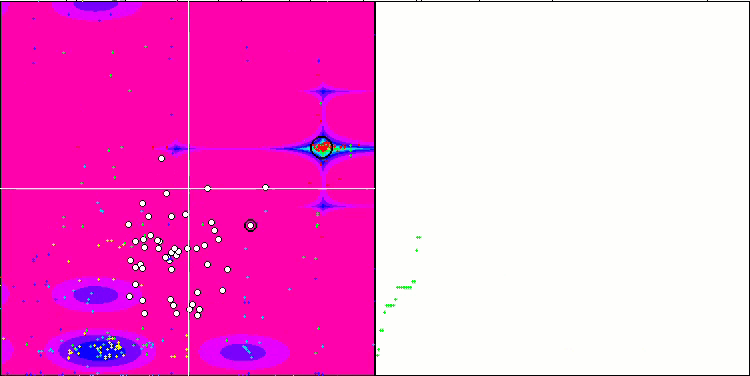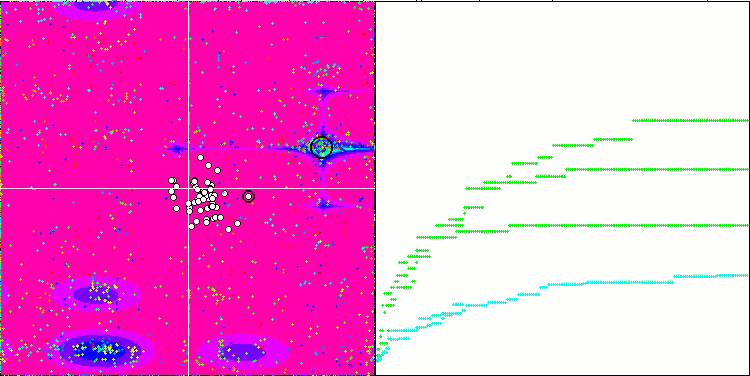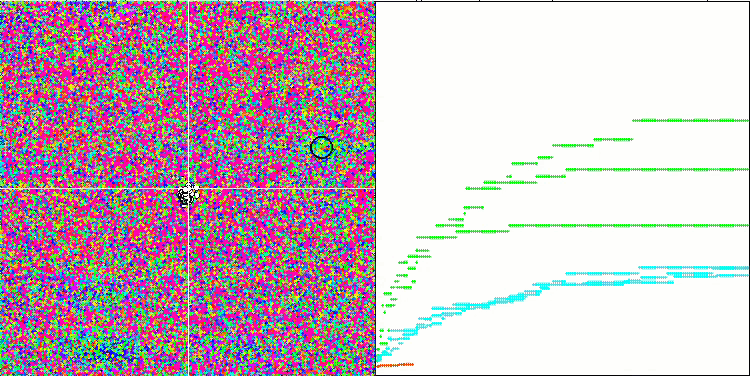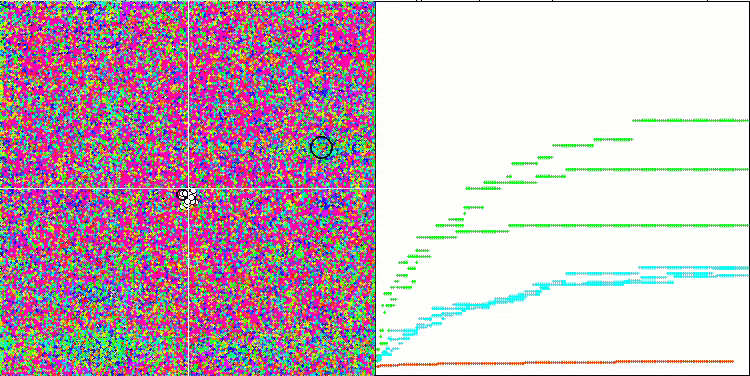
テストスタンドの動作を視覚的に分析すると、BFO-GAアルゴリズムは全域的極値の領域を素早く検出する一方で、局所的極値の研究にはあまり注意を払っていないことがわかります。これは、全域的極値が間違っていることが判明した場合に行き詰まりを引き起こす可能性があります。一般に、このアルゴリズムは、やや遅いものの、かなり確実に収束します。いくつかの「群れ」が観察され、これは良い兆候でありますが、細菌間の完全なコミュニケーションはなく、独立した集団単位として行動しています。
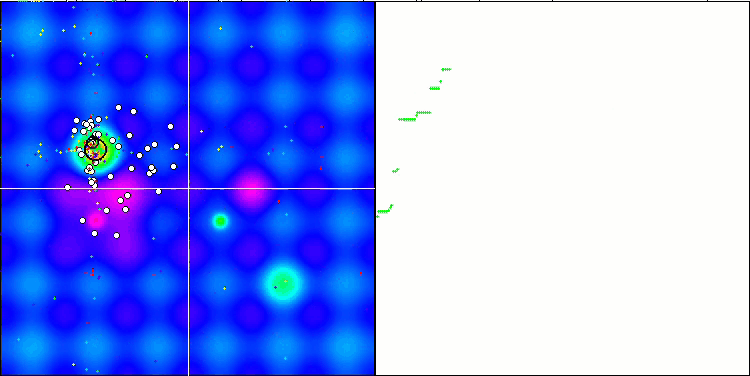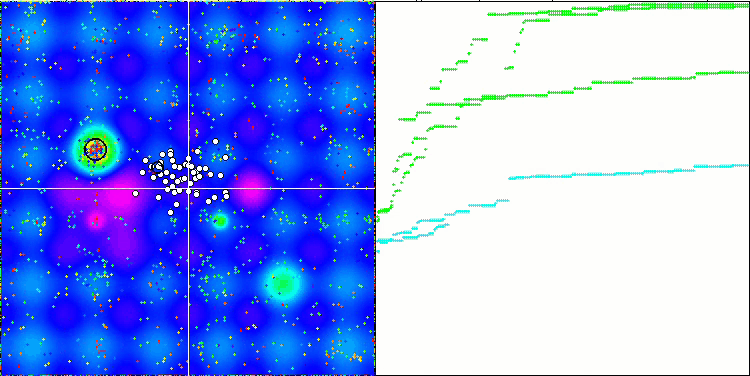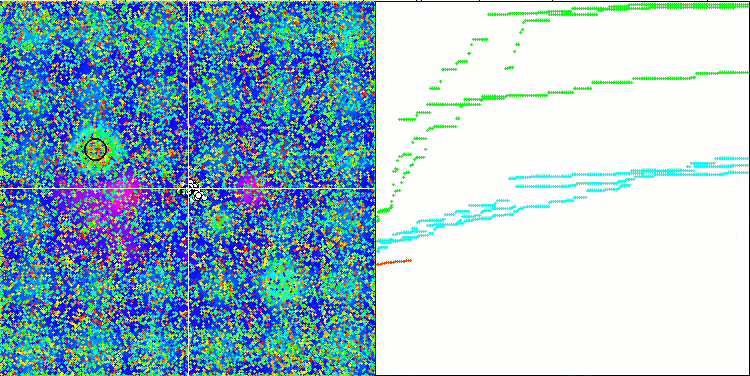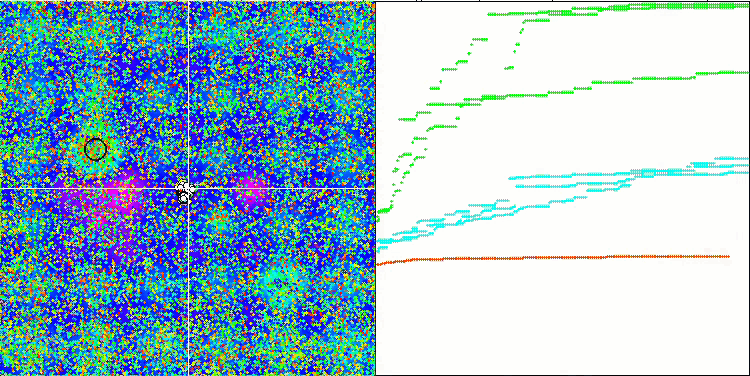
Hillyテスト関数でのBFO-GA
Forestテスト関数のBFO-GA
Megacityテスト関数のBFO-GA
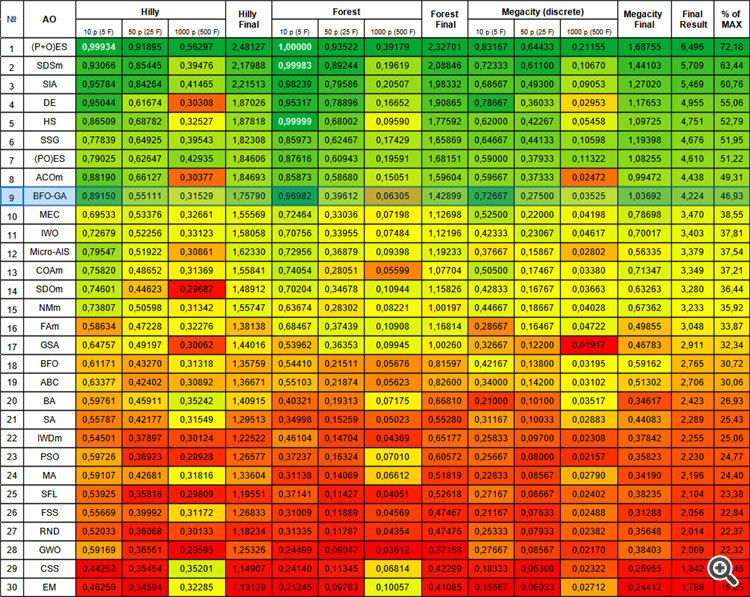
総合評価表では、BFO-GAアルゴリズムが9位を獲得しており、これはかなり高い結果です。これは、元のBFOアルゴリズムに遺伝的アルゴリズム演算子を追加する際におこなった変換が無駄ではなく、適切な結果につながったことを示しています。このアルゴリズムは、変数数の少ないテスト関数において、他のほとんどのアルゴリズムを凌駕する最高の結果を示しました。
| # | AO | 詳細 | Hilly | Hilly最終 | Forest | Forest最終 | Megacity(離散) | Megacity最終 | 最終結果 | MAXの% | ||||||
| 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | ||||||||
| 1 | (P+O)ES | (P+O)進化戦略 | 0.99934 | 0.91895 | 0.56297 | 2.48127 | 1.00000 | 0.93522 | 0.39179 | 2.32701 | 0.83167 | 0.64433 | 0.21155 | 1.68755 | 6.496 | 72.18 |
| 2 | SDSm | 確率的拡散探索M | 0.93066 | 0.85445 | 0.39476 | 2.17988 | 0.99983 | 0.89244 | 0.19619 | 2.08846 | 0.72333 | 0.61100 | 0.10670 | 1.44103 | 5.709 | 63.44 |
| 3 | SIA | 等方的焼きなまし | 0.95784 | 0.84264 | 0.41465 | 2.21513 | 0.98239 | 0.79586 | 0.20507 | 1.98332 | 0.68667 | 0.49300 | 0.09053 | 1.27020 | 5.469 | 60.76 |
| 4 | DE | 差分進化 | 0.95044 | 0.61674 | 0.30308 | 1.87026 | 0.95317 | 0.78896 | 0.16652 | 1.90865 | 0.78667 | 0.36033 | 0.02953 | 1.17653 | 4.955 | 55.06 |
| 5 | HS | ハーモニー検索 | 0.86509 | 0.68782 | 0.32527 | 1.87818 | 0.99999 | 0.68002 | 0.09590 | 1.77592 | 0.62000 | 0.42267 | 0.05458 | 1.09725 | 4.751 | 52.79 |
| 6 | SSG | 苗木の播種と育成 | 0.77839 | 0.64925 | 0.39543 | 1.82308 | 0.85973 | 0.62467 | 0.17429 | 1.65869 | 0.64667 | 0.44133 | 0.10598 | 1.19398 | 4.676 | 51.95 |
| 7 | (PO)ES | (PO)進化戦略 | 0.79025 | 0.62647 | 0.42935 | 1.84606 | 0.87616 | 0.60943 | 0.19591 | 1.68151 | 0.59000 | 0.37933 | 0.11322 | 1.08255 | 4.610 | 51.22 |
| 8 | ACOm | 蟻コロニー最適化M | 0.88190 | 0.66127 | 0.30377 | 1.84693 | 0.85873 | 0.58680 | 0.15051 | 1.59604 | 0.59667 | 0.37333 | 0.02472 | 0.99472 | 4.438 | 49.31 |
| 9 | BFO-GA | 細菌採食の最適化 - Ga | 0.89150 | 0.55111 | 0.31529 | 1.75790 | 0.96982 | 0.39612 | 0.06305 | 1.42899 | 0.72667 | 0.27500 | 0.03525 | 1.03692 | 4.224 | 46.93 |
| 10 | MEC | MindEvolutionaryComputation | 0.69533 | 0.53376 | 0.32661 | 1.55569 | 0.72464 | 0.33036 | 0.07198 | 1.12698 | 0.52500 | 0.22000 | 0.04198 | 0.78698 | 3.470 | 38.55 |
| 11 | IWO | 侵入雑草最適化 | 0.72679 | 0.52256 | 0.33123 | 1.58058 | 0.70756 | 0.33955 | 0.07484 | 1.12196 | 0.42333 | 0.23067 | 0.04617 | 0.70017 | 3.403 | 37.81 |
| 12 | Micro-AIS | 微小人工免疫系 | 0.79547 | 0.51922 | 0.30861 | 1.62330 | 0.72956 | 0.36879 | 0.09398 | 1.19233 | 0.37667 | 0.15867 | 0.02802 | 0.56335 | 3.379 | 37.54 |
| 13 | COAm | カッコウ最適化アルゴリズムM | 0.75820 | 0.48652 | 0.31369 | 1.55841 | 0.74054 | 0.28051 | 0.05599 | 1.07704 | 0.50500 | 0.17467 | 0.03380 | 0.71347 | 3.349 | 37.21 |
| 14 | SDOm | 螺旋ダイナミクス最適化M | 0.74601 | 0.44623 | 0.29687 | 1.48912 | 0.70204 | 0.34678 | 0.10944 | 1.15826 | 0.42833 | 0.16767 | 0.03663 | 0.63263 | 3.280 | 36.44 |
| 15 | NMm | ネルダー=ミード法M | 0.73807 | 0.50598 | 0.31342 | 1.55747 | 0.63674 | 0.28302 | 0.08221 | 1.00197 | 0.44667 | 0.18667 | 0.04028 | 0.67362 | 3.233 | 35.92 |
| 16 | FAm | ホタルアルゴリズムM | 0.58634 | 0.47228 | 0.32276 | 1.38138 | 0.68467 | 0.37439 | 0.10908 | 1.16814 | 0.28667 | 0.16467 | 0.04722 | 0.49855 | 3.048 | 33.87 |
| 17 | GSA | 重力探索法 | 0.64757 | 0.49197 | 0.30062 | 1.44016 | 0.53962 | 0.36353 | 0.09945 | 1.00260 | 0.32667 | 0.12200 | 0.01917 | 0.46783 | 2.911 | 32.34 |
| 18 | BFO | 細菌採餌の最適化 | 0.61171 | 0.43270 | 0.31318 | 1.35759 | 0.54410 | 0.21511 | 0.05676 | 0.81597 | 0.42167 | 0.13800 | 0.03195 | 0.59162 | 2.765 | 30.72 |
| 19 | ABC | 人工蜂コロニー | 0.63377 | 0.42402 | 0.30892 | 1.36671 | 0.55103 | 0.21874 | 0.05623 | 0.82600 | 0.34000 | 0.14200 | 0.03102 | 0.51302 | 2.706 | 30.06 |
| 20 | BA | コウモリアルゴリズム | 0.59761 | 0.45911 | 0.35242 | 1.40915 | 0.40321 | 0.19313 | 0.07175 | 0.66810 | 0.21000 | 0.10100 | 0.03517 | 0.34617 | 2.423 | 26.93 |
| 21 | SA | 焼きなまし | 0.55787 | 0.42177 | 0.31549 | 1.29513 | 0.34998 | 0.15259 | 0.05023 | 0.55280 | 0.31167 | 0.10033 | 0.02883 | 0.44083 | 2.289 | 25.43 |
| 22 | IWDm | インテリジェント水滴M | 0.54501 | 0.37897 | 0.30124 | 1.22522 | 0.46104 | 0.14704 | 0.04369 | 0.65177 | 0.25833 | 0.09700 | 0.02308 | 0.37842 | 2.255 | 25.06 |
| 23 | PSO | 粒子群最適化 | 0.59726 | 0.36923 | 0.29928 | 1.26577 | 0.37237 | 0.16324 | 0.07010 | 0.60572 | 0.25667 | 0.08000 | 0.02157 | 0.35823 | 2.230 | 24.77 |
| 24 | MA | モンキーアルゴリズム | 0.59107 | 0.42681 | 0.31816 | 1.33604 | 0.31138 | 0.14069 | 0.06612 | 0.51819 | 0.22833 | 0.08567 | 0.02790 | 0.34190 | 2.196 | 24.40 |
| 25 | SFL | ShuffledFrog-Leaping | 0.53925 | 0.35816 | 0.29809 | 1.19551 | 0.37141 | 0.11427 | 0.04051 | 0.52618 | 0.27167 | 0.08667 | 0.02402 | 0.38235 | 2.104 | 23.38 |
| 26 | FSS | 魚群検索 | 0.55669 | 0.39992 | 0.31172 | 1.26833 | 0.31009 | 0.11889 | 0.04569 | 0.47467 | 0.21167 | 0.07633 | 0.02488 | 0.31288 | 2.056 | 22.84 |
| 27 | RND | 無作為 | 0.52033 | 0.36068 | 0.30133 | 1.18234 | 0.31335 | 0.11787 | 0.04354 | 0.47476 | 0.25333 | 0.07933 | 0.02382 | 0.35648 | 2.014 | 22.37 |
| 28 | GWO | 灰色オオカミオプティマイザ | 0.59169 | 0.36561 | 0.29595 | 1.25326 | 0.24499 | 0.09047 | 0.03612 | 0.37158 | 0.27667 | 0.08567 | 0.02170 | 0.38403 | 2.009 | 22.32 |
| 29 | CSS | 荷電系探索 | 0.44252 | 0.35454 | 0.35201 | 1.14907 | 0.24140 | 0.11345 | 0.06814 | 0.42299 | 0.18333 | 0.06300 | 0.02322 | 0.26955 | 1.842 | 20.46 |
| 30 | EM | 電磁気学的アルゴリズム | 0.46250 | 0.34594 | 0.32285 | 1.13129 | 0.21245 | 0.09783 | 0.10057 | 0.41085 | 0.15667 | 0.06033 | 0.02712 | 0.24412 | 1.786 | 19.85 |
まとめ
BFO-GAの結果は、元のBFOアルゴリズムと比較して明らかに改善されていることがわかります。これは、遺伝的アルゴリズムの演算子である選択、突然変異、継承の使用によるものです。BFOは以前、局所的極値から抜け出すメカニズムを持っていませんでしたが、この問題は解決されています。このアルゴリズムは、細菌の採餌戦略の構成要素の順序とGAの演算子を組み合わせることで、実験の大きな機会を提供します。まだあまり試したことがありません。
BFOアルゴリズムにおいて、以前細菌の生存数を計算する際にミスがありましたが、このミスは修正されました。BFOの結果はわずかに改善し、ABCの1行上に浮上しました。最適化アルゴリズムを書く際に、検索戦略のコードやロジックの誤りを発見するのは難しいということを記しておきたいです。たとえエラーがあっても、アルゴリズムはほとんど常に検索を続けます。エラーによって常に結果が損なわれるわけではありません。時には、結果が大幅に向上し、「間違い」が戦略の一部となることもあります。最適化アルゴリズムでは、ロジックの大きな変更が必ずしも結果の大幅な改善につながるとは限らず、小さな変更が劇的な改善につながることもあるということは、常に覚えておく価値があります。修正されたBFOバージョンはアーカイブにあります。
図2:関連テストによるアルゴリズムのカラーグラデーション
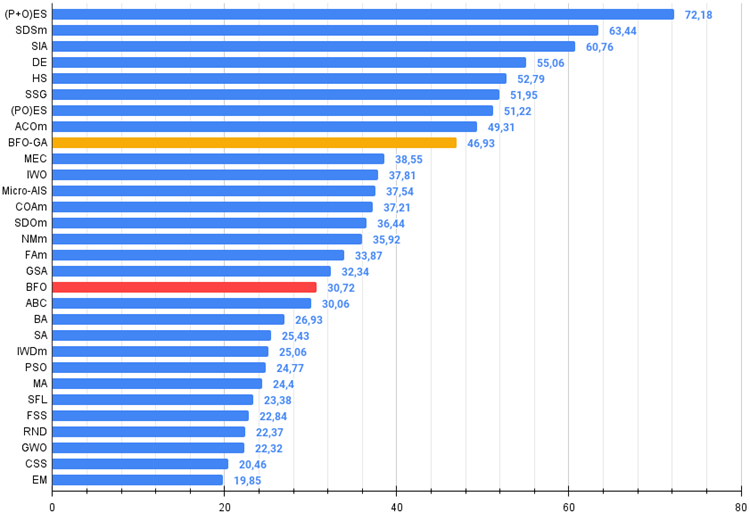
図3:アルゴリズムのテスト結果のヒストグラム(0から100までのスケールで、多ければ多いほど良いです。
ここで、100は理論的に可能な最大の結果です(アーカイブには格付け表を計算するスクリプトがある)。
BFO-GAの長所と短所
- アルゴリズムの動作が高速
- 実装がシンプル
- パラメータ数の少ない関数で収束性が良い
- 探索空間が広いタスクでは収束性が低い
この記事には、過去の記事で説明したアルゴリズムコードの最新版を更新したアーカイブが添付されています。記事の著者は、正規アルゴリズムの説明の絶対的な正確さについて責任を負いません。検索機能を向上させるために、それらの多くに変更が加えられています。記事に示された結論と判断は、実験結果に基づいています。
MetaQuotes Ltdによってロシア語から翻訳されました。
元の記事: https://www.mql5.com/ru/articles/14011
警告: これらの資料についてのすべての権利はMetaQuotes Ltd.が保有しています。これらの資料の全部または一部の複製や再プリントは禁じられています。
この記事はサイトのユーザーによって執筆されたものであり、著者の個人的な見解を反映しています。MetaQuotes Ltdは、提示された情報の正確性や、記載されているソリューション、戦略、または推奨事項の使用によって生じたいかなる結果についても責任を負いません。
 母集団最適化アルゴリズム:進化戦略、(μ,λ)-ESと(μ+λ)-ES
母集団最適化アルゴリズム:進化戦略、(μ,λ)-ESと(μ+λ)-ES
 母集団最適化アルゴリズム:微小人工免疫系(Micro-AIS)
母集団最適化アルゴリズム:微小人工免疫系(Micro-AIS)
 MQL5における修正グリッドヘッジEA(第2部):シンプルなグリッドEAを作る
MQL5における修正グリッドヘッジEA(第2部):シンプルなグリッドEAを作る
- 無料取引アプリ
- 8千を超えるシグナルをコピー
- 金融ニュースで金融マーケットを探索

速度推定の比較図を追加することを提案する。
スピード推定値の比較表を追加することを提案する。
いくつかの "But's "がなければ、これは可能だろう:
1.この図は誤解を招く可能性がある。これはアルゴリズム自身の速度であり、問題解決全体ではないからだ。
2.アルゴリズムコードの実行速度は、テストベンチのコードに対応するカウンターを挿入することで簡単に測定できる。アルゴリズムによっては実行に時間がかかるものもあり、そのようなテストに理想的な条件を作り出すのは難しい。
大丈夫そうだ。