
Algoritmo de optimización de reacciones químicas (CRO) (Parte II): Ensamblaje y resultados
Contenido
1. Introducción
En la segunda parte de nuestro artículo, continuamos nuestra inmersión en el fascinante mundo de la optimización de reacciones químicas (CRO). Basándonos en el concepto de "moléculas" y "reacciones elementales", nos familiarizamos con los principios algorítmicos subyacentes y consideró cómo se aplican estos conceptos para resolver problemas de optimización complejos. También conocimos los puntos clave de la conservación de la energía dentro del CRO y las funciones del algoritmo, como descomposición, síntesis, intramolecular y intermolecular colisiones ineficientes, que desempeñan un papel fundamental a la hora de explorar el espacio de búsqueda y encontrar soluciones óptimas.
Ahora que hemos cubierto los conceptos básicos y los principios de funcionamiento de los operadores químicos del algoritmo de optimización de reacciones químicas, es el momento de pasar al montaje general del algoritmo y a su aplicación práctica. Aquí nos centraremos en los resultados del algoritmo en varias funciones de prueba para analizar su eficacia y potencial en la resolución de problemas del mundo real. Examinaremos su rendimiento, convergencia y capacidad para encontrar óptimos globales, lo que nos permitirá evaluar su aplicabilidad, y también comparar los resultados del algoritmo CRO con otros métodos de optimización, identificando sus ventajas e inconvenientes.
2. Implementación del algoritmo
Sigamos escribiendo el código del algoritmo utilizando la plantilla que ya te es familiar para todos los algoritmos, que incluye las funciones estándar Init, Moving y Revision. Como ya hemos declarado la estructura e implementado los operadores principales de las reacciones químicas, pasemos a escribir la clase algoritmo para unir todos los componentes.
Necesitaremos un pseudocódigo para implementar el ensamblaje del algoritmo:
Si se restablece el indicador de revisión - inicialización:
Para cada molécula i de 0 a popSize:
Para cada coordenada c de 0 a coordenadas
Genera un valor aleatorio para la estructura de una molécula desde rangeMin hasta rangeMax
. Limita el valor de una estructura a un rango determinado
Guarda la estructura en a
arrayExit
Cálculo de la energía cinética:
minKE = valor máximo del double
type Para cada molécula i de 0 a popSize:
Si el ajuste de la molécula es inferior a minKE:
minKE = ajuste de la molécula
Para cada molécula i de 0 a popSize:
Calcular la energía cinética de una molécula KE como escalar elajuste del rango de minKE a fB (mejor solución global) al rango de 0,0a 1,0
.molCNT = 0
Aún no se ha detenido:
Si el número aleatorio es menor que moleColl:
Selecciona dos moléculas al azar M1 y M2
Si KE de ambas moléculas es mayor o igual que β:
Realizar la síntesis de las moléculas M1 y M2
. De lo contrario:
Realizar colisión intermolecular ineficaz entre M1 y M2
. De lo contrario:
Selecciona una molécula aleatoria M
Si NumHit de la molécula supera α:
Realizar la descomposición de la molécula M
De lo contrario:
Realiza una colisión de la molécula M
Copiar estructuras moleculares de Mfilial a a
Calcular la aptitud de los individuos de la población a
.Copiar estructuras moleculares de a a Mfilial
Inicialización: ind = -1
Para cada molécula i de 0 a popSize:
Si el ajuste de la molécula supera fB:
fB = ajuste de la molécula
ind = i
Si ind no es igual a -1:
Copia la estructura de la molécula en cB
Actualización de moléculas:
Si no hay revisión:
Para cada molécula i de 0 a popSize:
Actualiza el ajuste de la molécula Mparent
. Establecer revisión a true
Salir
Para cada molécula i de 0 a popSize:
Actualiza el ajuste de la molécula en Mfilial
. Según el tipo de molécula (síntesis, colisión intermolecular ineficaz, descomposición, colisión):
Realizar el postoperatorio adecuado
Ahora que tenemos el pseudocódigo y los operadores químicos descritos en la primera parte del artículo, podemos empezar a construir la implementación del algoritmo CRO en código.
Declara la clase C_AO_CRO, que es heredera de la clase base C_AO y es una implementación del algoritmo de optimización de reacciones químicas (CRO).
1. Campos públicos:
- popSize - tamaño de la población.
- moleColl, alpha, beta, molecPerturb - parámetros del algoritmo.
- params - matriz para almacenar los parámetros del algoritmo.
- Mparent[], Mfilial[] - objetos de la estructura S_CRO_Agent, que representan moléculas.
2. Las opciones disponibles son:
- C_AO_CRO() - constructor de la clase que inicializa los campos de la clase.
- SetParams() - método para establecer los parámetros del algoritmo.
- Init() - método para inicializar el algoritmo. El método acepta rangos de búsqueda mínimo y máximo, paso de búsqueda y número de épocas.
- Moving() y Revision() - métodos que implementan las operaciones básicas del algoritmo.
3. Campos y métodos privados:
- Synthesis(), InterMolInefColl(), Decomposition(), InefCollision() - métodos que implementan diferentes tipos de reacciones.
- PostSynthesis(), PostInterMolInefColl(), PostDecomposition(), PostInefCollision() - métodos que realizan acciones después de las reacciones correspondientes.
- N() - método para modificar un componente (coordenada) de la estructura de una molécula.
Esta clase es una implementación completa del algoritmo de optimización de reacciones químicas (CRO) y contiene todos los datos y métodos necesarios para ello.
//—————————————————————————————————————————————————————————————————————————————— class C_AO_CRO : public C_AO { public: //-------------------------------------------------------------------- ~C_AO_CRO () { } C_AO_CRO () { ao_name = "CRO"; ao_desc = "Chemical Reaction Optimisation"; ao_link = "https://www.mql5.com/en/articles/15041"; popSize = 50; //population size moleColl = 0.9; alpha = 200; beta = 0.01; molecPerturb = 0.5; ArrayResize (params, 5); params [0].name = "popSize"; params [0].val = popSize; params [1].name = "moleColl"; params [1].val = moleColl; params [2].name = "alpha"; params [2].val = alpha; params [3].name = "beta"; params [3].val = beta; params [4].name = "molecPerturb"; params [4].val = molecPerturb; } void SetParams () { popSize = (int)params [0].val; moleColl = params [1].val; alpha = (int)params [2].val; beta = params [3].val; molecPerturb = params [4].val; } bool Init (const double &rangeMinP [], //minimum search range const double &rangeMaxP [], //maximum search range const double &rangeStepP [], //step search const int epochsP = 0); //number of epochs void Moving (); void Revision (); S_CRO_Agent Mparent []; S_CRO_Agent Mfilial []; //---------------------------------------------------------------------------- double moleColl; int alpha; double beta; double molecPerturb; private: //------------------------------------------------------------------- bool Synthesis (int index1, int index2, int &molCNT); bool InterMolInefColl (int index1, int index2, int &molCNT); bool Decomposition (int index, int &molCNT); bool InefCollision (int index, int &molCNT); void PostSynthesis (S_CRO_Agent &mol); void PostInterMolInefColl (S_CRO_Agent &mol); void PostDecomposition (S_CRO_Agent &mol); void PostInefCollision (S_CRO_Agent &mol); void N (double &coord, int coordPos); }; //——————————————————————————————————————————————————————————————————————————————
El método Init de la clase C_AO_CRO se utiliza para inicializar las variables de la clase basándose en los parámetros pasados. Esto es lo que ocurre con este método:
1. El método llama a la función StandardInit, que toma los rangos de búsqueda mínimo y máximo, así como el paso de búsqueda. Si StandardInit devuelve false, el método Init también devuelve false y completa su trabajo.
2. A continuación, el método redimensiona las matrices Mparent y Mfilial a popSize, que representa el tamaño de la población.
3. A continuación, para cada elemento de las matrices Mparent y Mfilial, se llama al método Init con el parámetro coords. Este método inicializa los campos de cada agente de la población.
4. Al final, el método devuelve true indicando que la inicialización se ha completado con éxito.
Este método realiza la configuración inicial del algoritmo de optimización de reacción química (CRO) con parámetros dados y lo prepara para realizar la optimización.
//—————————————————————————————————————————————————————————————————————————————— bool C_AO_CRO::Init (const double &rangeMinP [], //minimum search range const double &rangeMaxP [], //maximum search range const double &rangeStepP [], //step search const int epochsP = 0) //number of epochs { if (!StandardInit (rangeMinP, rangeMaxP, rangeStepP)) return false; //---------------------------------------------------------------------------- ArrayResize (Mparent, popSize); ArrayResize (Mfilial, popSize); for (int i = 0; i < popSize; i++) { Mparent [i].Init (coords); Mfilial [i].Init (coords); } return true; } //——————————————————————————————————————————————————————————————————————————————
El método Moving de la clase C_AO_CRO se utiliza para llamar a operadores químicos que realizan cambios en las estructuras de las moléculas, moviendo así las moléculas durante la optimización. El método hace lo siguiente:
1. Si revision es igual a false, entonces para cada molécula en la población Mparent, las estructuras se inicializan con valores aleatorios en un rango dado de rangeMin a rangeMax. A continuación, estos valores se copian en la matriz a.
2. Si revision no es igual a false, se calcula el valor mínimo de la función f entre todas las moléculas de la población Mparent. A continuación, se calcula el valor KE para cada molécula a partir del escalado de su valor de función f, valor mínimo f por la población de moléculas progenitoras y la mejor solución global fB en el rango comprendido entre 0,0 y 1,0.
3. A continuación, hasta que uno de los operadores químicos devuelva false (esto significa que la población hija se ha quedado sin espacio para moléculas hijas), ocurre lo siguiente:
- Si el número aleatorio es menor que moleColl, se seleccionan dos moléculas aleatorias M1 y M2. Si KE de ambas moléculas es mayor o igual que beta, entonces se realiza la síntesis (es decir, la síntesis se realiza para moléculas no inferiores al valor de adecuación relativa especificado en los parámetros; fue a estos efectos que los valores de adecuación de las moléculas se escalaron previamente al rango de 0,0 a 1,0). De lo contrario, se produce una colisión intermolecular ineficaz.
- Si el número aleatorio es mayor o igual que moleColl, se selecciona una molécula aleatoria M. Si el NumHit de la molécula supera alpha (si la molécula ha sufrido más colisiones de las especificadas en los parámetros del algoritmo, entonces la molécula «se rompe»), se realiza la descomposición. De lo contrario, se produce una colisión.
4. Al final del método, las estructuras de todas las moléculas de Mfilial se copian en la matriz de población <a.
Este método se encarga de actualizar las estructuras moleculares en el algoritmo de optimización de reacciones químicas (CRO) en función del estado actual del sistema y de los parámetros dados. El método implementa las operaciones básicas del algoritmo CRO, como la síntesis, la colisión intermolecular ineficiente, la descomposición y la colisión.
//—————————————————————————————————————————————————————————————————————————————— void C_AO_CRO::Moving () { //---------------------------------------------------------------------------- if (!revision) { for (int i = 0; i < popSize; i++) { for (int c = 0; c < coords; c++) { Mparent [i].structure [c] = u.RNDfromCI (rangeMin [c], rangeMax [c]); // Random structure in the range from rangeMin to rangeMax Mparent [i].structure [c] = u.SeInDiSp (Mparent [i].structure [c], rangeMin [c], rangeMax [c], rangeStep [c]); a [i].c [c] = Mparent [i].structure [c]; } } return; } //---------------------------------------------------------------------------- double minKE = DBL_MAX; for (int i = 0; i < popSize; i++) { if (Mparent [i].f < minKE) minKE = Mparent [i].f; } for (int i = 0; i < popSize; i++) { Mparent [i].KE = u.Scale (Mparent [i].f, minKE, fB, 0.0, 1.0); } //---------------------------------------------------------------------------- int molCNT = 0; while (!IsStopped ()) { if (u.RNDprobab () < moleColl) { // Select two random molecules M1 and M2 int index1 = u.RNDminusOne (popSize); int index2 = u.RNDminusOne (popSize); // If KE ≤ β: if (Mparent [index1].KE >= beta && Mparent [index2].KE >= beta) { // Perform Synthesis if (!Synthesis (index1, index2, molCNT)) break; } else { // Perform Intermolecular Inefficient Collision if (!InterMolInefColl (index1, index2, molCNT)) break; } } else { // Select a random molecule M int index = u.RNDminusOne (popSize); // If NumHit > α: if (Mparent [index].NumHit > alpha) { // Perform Decomposition if (!Decomposition (index, molCNT)) break; } else { // Perform Collision if (!InefCollision (index, molCNT)) break; } } } for (int i = 0; i < popSize; i++) { ArrayCopy (a [i].c, Mfilial [i].structure); } } //——————————————————————————————————————————————————————————————————————————————
El método Revision de la clase C_AO_CRO se utiliza para actualizar la mejor solución global y actualizar los estados de las moléculas de la población padre realizando post-operaciones químicas. Acciones realizadas por este método:
1. Actualización de la solución global. En el bucle for, el método itera a través de todas las moléculas. Si el valor de la función f de la molécula actual supera el mejor valor fB actual, se actualiza fB y la matriz de coordenadas de la molécula actual se copia en la matriz cB.
2. Si revision es igual a false, entonces para cada molécula de la población Mparent, el valor f se establece igual al valor f de la matriz a. Entonces revision se establece en true y el método sale. En esta etapa, es importante obtener los valores de ajuste de las moléculas progenitoras para que en épocas posteriores, los operadores químicos que dependen de la energía cinética (el valor de la función de ajuste normalizado al rango de 0,0 a 1,0).
3. Si revision no es false, para cada molécula de la población Mfilial se establece la f igual a f de la matriz a. A continuación, dependiendo del tipo de reacción rType de la molécula (reacción en la que participó la molécula), se llama al método PostSynthesis, PostInterMolInefColl, PostDecomposition, PostInefCollision apropiado.
//—————————————————————————————————————————————————————————————————————————————— void C_AO_CRO::Revision () { //---------------------------------------------------------------------------- int ind = -1; for (int i = 0; i < popSize; i++) { if (a [i].f > fB) { fB = a [i].f; ind = i; } } if (ind != -1) ArrayCopy (cB, a [ind].c, 0, 0, WHOLE_ARRAY); //---------------------------------------------------------------------------- if (!revision) { for (int i = 0; i < popSize; i++) { for (int c = 0; c < coords; c++) { Mparent [i].f = a [i].f; } } revision = true; return; } //---------------------------------------------------------------------------- for (int i = 0; i < popSize; i++) { for (int c = 0; c < coords; c++) { Mfilial [i].f = a [i].f; } switch (Mfilial [i].rType) { case synthesis: PostSynthesis (Mfilial [i]); break; case interMolecularInefColl: PostInterMolInefColl (Mfilial [i]); break; case decomposition: PostDecomposition (Mfilial [i]); break; case inefCollision: PostInefCollision (Mfilial [i]); break; } } } //——————————————————————————————————————————————————————————————————————————————
3. Resultados de la prueba
El algoritmo CRO se probó en las funciones Hilly, Forest y Megacity. En cada caso, se realizaron diez ejecuciones de funciones para cada tipo de paisaje (5, 25 y 500 funciones) y se obtuvieron los resultados de la optimización.
CRO|Chemical Reaction Optimisation|50.0|0.9|200.0|0.01|0.5|
=============================
5 Hilly's; Func runs: 10000; result: 0.9462894520167225
25 Hilly's; Func runs: 10000; result: 0.6611186250435438
500 Hilly's; Func runs: 10000; result: 0.2985263035668822
=============================
5 Forest's; Func runs: 10000; result: 0.8790568514481787
25 Forest's; Func runs: 10000; result: 0.584216839762206
500 Forest's; Func runs: 10000; result: 0.2114595696419046
=============================
5 Megacity's; Func runs: 10000; result: 0.7584615384615384
25 Megacity's; Func runs: 10000; result: 0.4264615384615384
500 Megacity's; Func runs: 10000; result: 0.12686153846153955
=============================
All score: 4.89245 (54.36%)
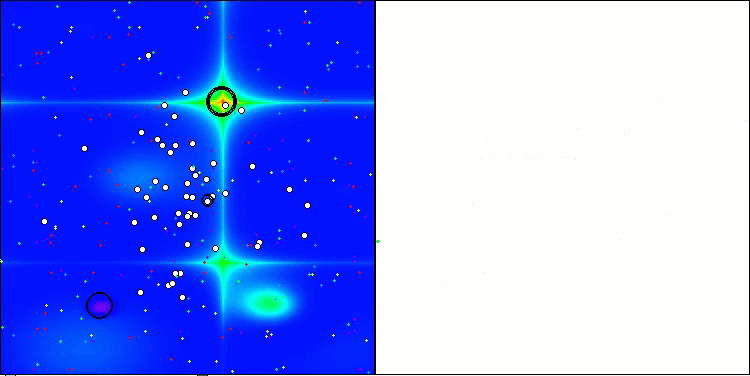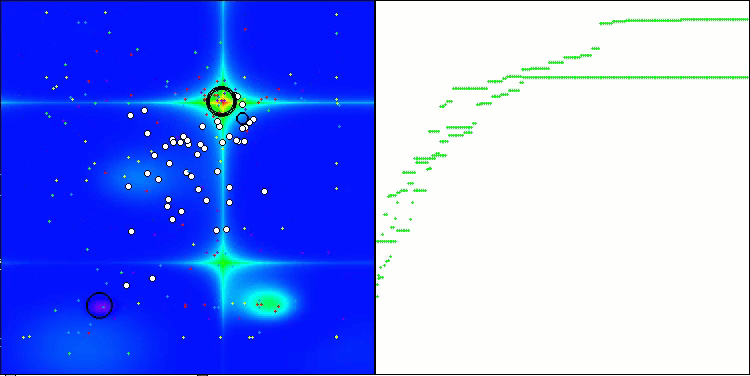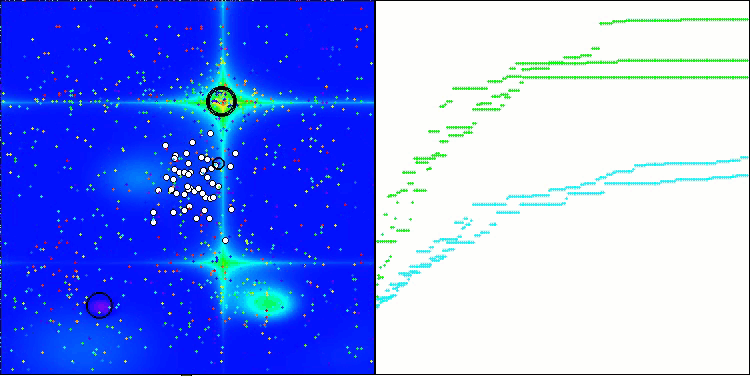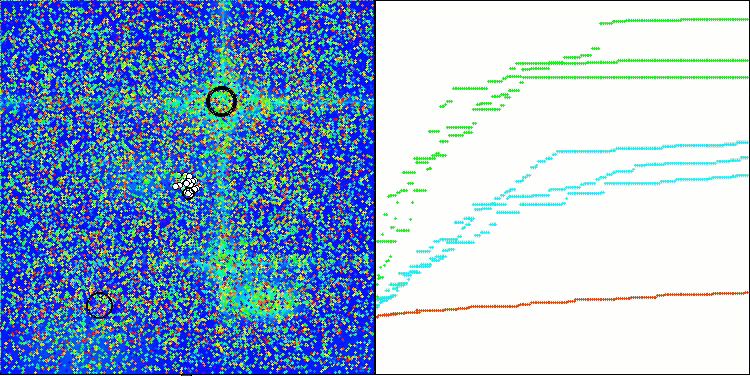
La visualización del funcionamiento del banco de pruebas con el algoritmo CRO demuestra características interesantes del algoritmo. Aunque CRO puede atascarse a veces, como demuestran las largas secciones planas del gráfico de convergencia, sigue mostrando unos resultados generales decentes.
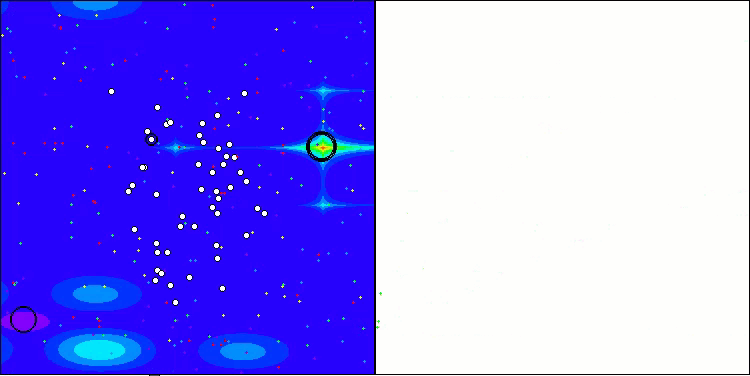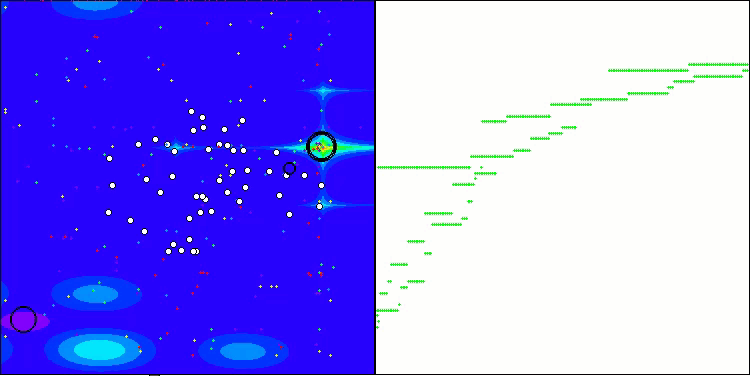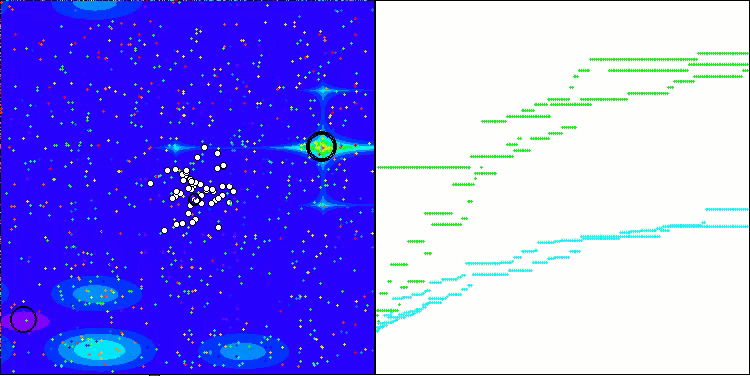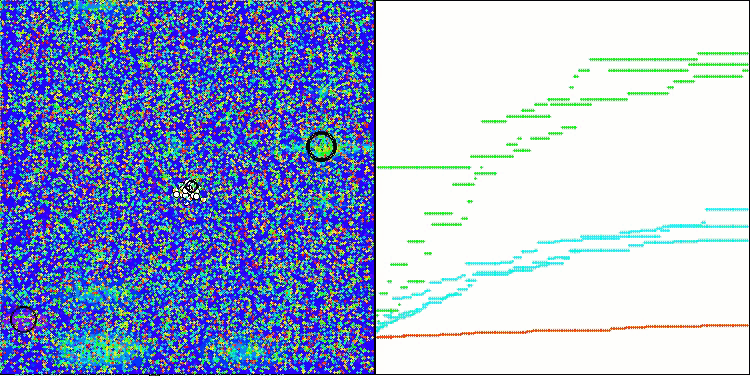
Uno de los aspectos destacables del trabajo de la CRO es el movimiento de «moléculas» en la zona de búsqueda. A primera vista, este movimiento parece caótico y se asemeja al movimiento browniano. Sin embargo, a pesar de la aleatoriedad externa, las «moléculas» consiguen encontrar una zona de óptimo global. Esto demuestra la naturaleza compleja y sofisticada del algoritmo CRO, que utiliza principios de reacciones químicas para resolver problemas de optimización.
En general, el algoritmo CRO es una potente herramienta de optimización que puede manejar una gran variedad de tareas, a pesar de algunas dificultades. Sus propiedades únicas y su capacidad para encontrar óptimos globales lo convierten en una valiosa herramienta en el campo de la optimización.
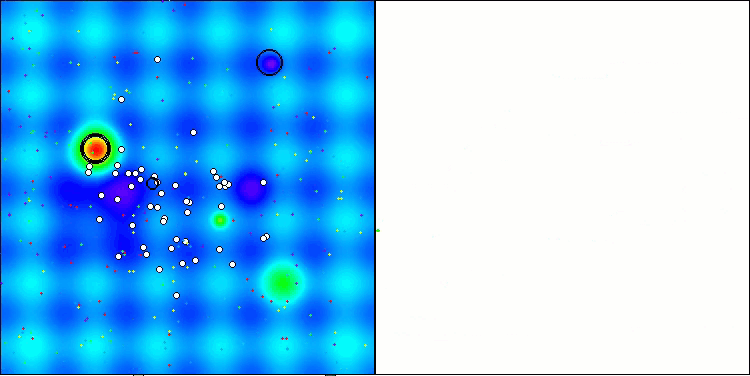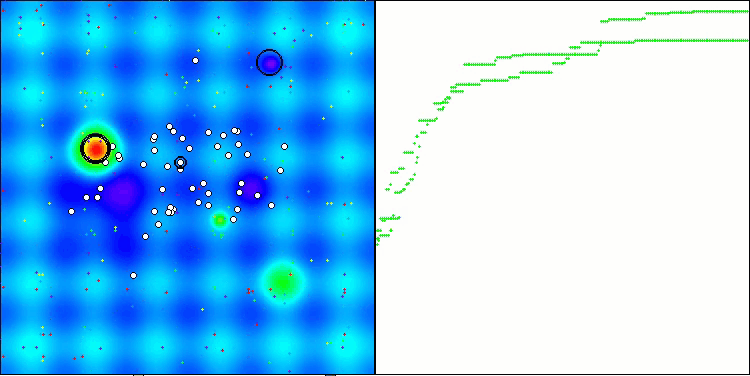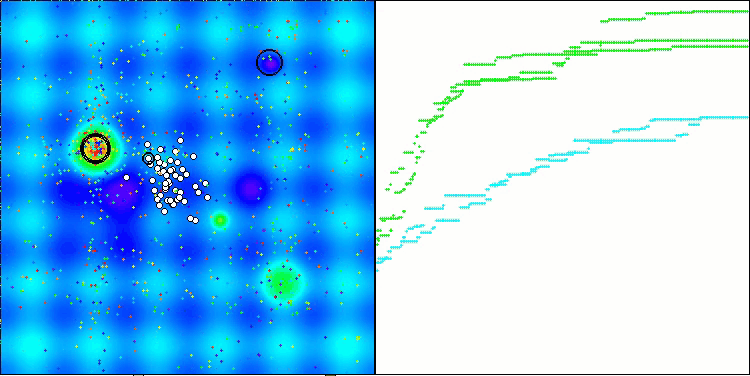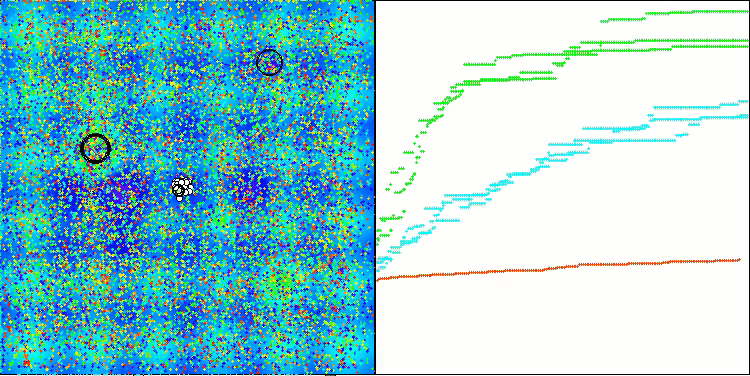
CRO en la función de prueba Hilly.
CRO on the Forest test function
CRO en la función de prueba Megacity.
| # | AO | Description | Hilly | Hilly final | Forest | Forest final | Megacity (discrete) | Megacity final | Final result | % of MAX | ||||||
| 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | ||||||||
| 1 | BGA | algoritmo genético binario | 0.99989 | 0.99518 | 0.42835 | 2.42341 | 0.96153 | 0.96181 | 0.32027 | 2.24360 | 0.91385 | 0.95908 | 0.24220 | 2.11512 | 6.782 | 75.36 |
| 2 | CLA | algoritmo de bloqueo de código | 0.95345 | 0.87107 | 0.37590 | 2.20042 | 0.98942 | 0.91709 | 0.31642 | 2.22294 | 0.79692 | 0.69385 | 0.19303 | 1.68380 | 6.107 | 67.86 |
| 3 | (P+O)ES | Estrategias de evolución (P+O) | 0.92256 | 0.88101 | 0.40021 | 2.20379 | 0.97750 | 0.87490 | 0.31945 | 2.17185 | 0.67385 | 0.62985 | 0.18634 | 1.49003 | 5.866 | 65.17 |
| 4 | CTA | algoritmo de cola de cometa | 0.95346 | 0.86319 | 0.27770 | 2.09435 | 0.99794 | 0.85740 | 0.33949 | 2.19484 | 0.88769 | 0.56431 | 0.10512 | 1.55712 | 5.846 | 64.96 |
| 5 | SDSm | Búsqueda de difusión estocástica M | 0.93066 | 0.85445 | 0.39476 | 2.17988 | 0.99983 | 0.89244 | 0.19619 | 2.08846 | 0.72333 | 0.61100 | 0.10670 | 1.44103 | 5.709 | 63.44 |
| 6 | ESG | evolución de los grupos sociales | 0.99906 | 0.79654 | 0.35056 | 2.14616 | 1.00000 | 0.82863 | 0.13102 | 1.95965 | 0.82333 | 0.55300 | 0.04725 | 1.42358 | 5.529 | 61.44 |
| 7 | SIA | recocido isotrópico simulado | 0.95784 | 0.84264 | 0.41465 | 2.21513 | 0.98239 | 0.79586 | 0.20507 | 1.98332 | 0.68667 | 0.49300 | 0.09053 | 1.27020 | 5.469 | 60.76 |
| 8 | ACS | búsqueda cooperativa artificial | 0.75547 | 0.74744 | 0.30407 | 1.80698 | 1.00000 | 0.88861 | 0.22413 | 2.11274 | 0.69077 | 0.48185 | 0.13322 | 1.30583 | 5.226 | 58.06 |
| 9 | TSEA | Algoritmo de evolución del caparazón de tortuga | 0.96798 | 0.64480 | 0.29672 | 1.90949 | 0.99449 | 0.61981 | 0.22708 | 1.84139 | 0.69077 | 0.42646 | 0.13598 | 1.25322 | 5.004 | 55.60 |
| 10 | DE | evolución diferencial | 0.95044 | 0.61674 | 0.30308 | 1.87026 | 0.95317 | 0.78896 | 0.16652 | 1.90865 | 0.78667 | 0.36033 | 0.02953 | 1.17653 | 4.955 | 55.06 |
| 11 | CRO | optimización de reacciones químicas | 0.94629 | 0.66112 | 0.29853 | 1.90593 | 0.87906 | 0.58422 | 0.21146 | 1.67473 | 0.75846 | 0.42646 | 0.12686 | 1.31178 | 4.892 | 54.36 |
| 12 | BSA | algoritmo de enjambre de pájaros | 0.89306 | 0.64900 | 0.26250 | 1.80455 | 0.92420 | 0.71121 | 0.24939 | 1.88479 | 0.69385 | 0.32615 | 0.10012 | 1.12012 | 4.809 | 53.44 |
| 13 | HS | búsqueda de armonía | 0.86509 | 0.68782 | 0.32527 | 1.87818 | 0.99999 | 0.68002 | 0.09590 | 1.77592 | 0.62000 | 0.42267 | 0.05458 | 1.09725 | 4.751 | 52.79 |
| 14 | SSG | Siembra y cultivo de plantones | 0.77839 | 0.64925 | 0.39543 | 1.82308 | 0.85973 | 0.62467 | 0.17429 | 1.65869 | 0.64667 | 0.44133 | 0.10598 | 1.19398 | 4.676 | 51.95 |
| 15 | (PO)ES | (PO) estrategias de evolución | 0.79025 | 0.62647 | 0.42935 | 1.84606 | 0.87616 | 0.60943 | 0.19591 | 1.68151 | 0.59000 | 0.37933 | 0.11322 | 1.08255 | 4.610 | 51.22 |
| 16 | BSO | Optimización de la lluvia de ideas | 0.93736 | 0.57616 | 0.29688 | 1.81041 | 0.93131 | 0.55866 | 0.23537 | 1.72534 | 0.55231 | 0.29077 | 0.11914 | 0.96222 | 4.498 | 49.98 |
| 17 | WOAm | Algoritmo de optimización de Wale M | 0.84521 | 0.56298 | 0.26263 | 1.67081 | 0.93100 | 0.52278 | 0.16365 | 1.61743 | 0.66308 | 0.41138 | 0.11357 | 1.18803 | 4.476 | 49.74 |
| 18 | ACOm | optimización de colonias de hormigas M | 0.88190 | 0.66127 | 0.30377 | 1.84693 | 0.85873 | 0.58680 | 0.15051 | 1.59604 | 0.59667 | 0.37333 | 0.02472 | 0.99472 | 4.438 | 49.31 |
| 19 | BFO-GA | optimización de la alimentación bacteriana - ga | 0.89150 | 0.55111 | 0.31529 | 1.75790 | 0.96982 | 0.39612 | 0.06305 | 1.42899 | 0.72667 | 0.27500 | 0.03525 | 1.03692 | 4.224 | 46.93 |
| 20 | MEC | computación evolutiva de la mente | 0.69533 | 0.53376 | 0.32661 | 1.55569 | 0.72464 | 0.33036 | 0.07198 | 1.12698 | 0.52500 | 0.22000 | 0.04198 | 0.78698 | 3.470 | 38.55 |
| 21 | IWO | Optimización de malezas invasoras | 0.72679 | 0.52256 | 0.33123 | 1.58058 | 0.70756 | 0.33955 | 0.07484 | 1.12196 | 0.42333 | 0.23067 | 0.04617 | 0.70017 | 3.403 | 37.81 |
| 22 | Micro-AIS | microsistema inmunológico artificial | 0.79547 | 0.51922 | 0.30861 | 1.62330 | 0.72956 | 0.36879 | 0.09398 | 1.19233 | 0.37667 | 0.15867 | 0.02802 | 0.56335 | 3.379 | 37.54 |
| 23 | COAm | Algoritmo de optimización del cuco M | 0.75820 | 0.48652 | 0.31369 | 1.55841 | 0.74054 | 0.28051 | 0.05599 | 1.07704 | 0.50500 | 0.17467 | 0.03380 | 0.71347 | 3.349 | 37.21 |
| 24 | SDOm | Optimización de dinámica espiral M | 0.74601 | 0.44623 | 0.29687 | 1.48912 | 0.70204 | 0.34678 | 0.10944 | 1.15826 | 0.42833 | 0.16767 | 0.03663 | 0.63263 | 3.280 | 36.44 |
| 25 | NMm | Método Nelder-Mead M | 0.73807 | 0.50598 | 0.31342 | 1.55747 | 0.63674 | 0.28302 | 0.08221 | 1.00197 | 0.44667 | 0.18667 | 0.04028 | 0.67362 | 3.233 | 35.92 |
| 26 | FAm | Algoritmo de luciérnaga M | 0.58634 | 0.47228 | 0.32276 | 1.38138 | 0.68467 | 0.37439 | 0.10908 | 1.16814 | 0.28667 | 0.16467 | 0.04722 | 0.49855 | 3.048 | 33.87 |
| 27 | GSA | algoritmo de búsqueda gravitacional | 0.64757 | 0.49197 | 0.30062 | 1.44016 | 0.53962 | 0.36353 | 0.09945 | 1.00260 | 0.32667 | 0.12200 | 0.01917 | 0.46783 | 2.911 | 32.34 |
| 28 | BFO | Optimización de la alimentación bacteriana | 0.61171 | 0.43270 | 0.31318 | 1.35759 | 0.54410 | 0.21511 | 0.05676 | 0.81597 | 0.42167 | 0.13800 | 0.03195 | 0.59162 | 2.765 | 30.72 |
| 29 | ABC | colonia de abejas artificial | 0.63377 | 0.42402 | 0.30892 | 1.36671 | 0.55103 | 0.21874 | 0.05623 | 0.82600 | 0.34000 | 0.14200 | 0.03102 | 0.51302 | 2.706 | 30.06 |
| 30 | BA | algoritmo de murciélago | 0.59761 | 0.45911 | 0.35242 | 1.40915 | 0.40321 | 0.19313 | 0.07175 | 0.66810 | 0.21000 | 0.10100 | 0.03517 | 0.34617 | 2.423 | 26.93 |
| 31 | SA | recocido simulado | 0.55787 | 0.42177 | 0.31549 | 1.29513 | 0.34998 | 0.15259 | 0.05023 | 0.55280 | 0.31167 | 0.10033 | 0.02883 | 0.44083 | 2.289 | 25.43 |
| 32 | IWDm | Gotas de agua inteligentes M | 0.54501 | 0.37897 | 0.30124 | 1.22522 | 0.46104 | 0.14704 | 0.04369 | 0.65177 | 0.25833 | 0.09700 | 0.02308 | 0.37842 | 2.255 | 25.06 |
| 33 | PSO | Optimización del enjambre de partículas | 0.59726 | 0.36923 | 0.29928 | 1.26577 | 0.37237 | 0.16324 | 0.07010 | 0.60572 | 0.25667 | 0.08000 | 0.02157 | 0.35823 | 2.230 | 24.77 |
| 34 | Boids | algoritmo de boids | 0.43340 | 0.30581 | 0.25425 | 0.99346 | 0.35718 | 0.20160 | 0.15708 | 0.71586 | 0.27846 | 0.14277 | 0.09834 | 0.51957 | 2.229 | 24.77 |
| 35 | MA | algoritmo del mono | 0.59107 | 0.42681 | 0.31816 | 1.33604 | 0.31138 | 0.14069 | 0.06612 | 0.51819 | 0.22833 | 0.08567 | 0.02790 | 0.34190 | 2.196 | 24.40 |
| 36 | SFL | rana saltando arrastrada | 0.53925 | 0.35816 | 0.29809 | 1.19551 | 0.37141 | 0.11427 | 0.04051 | 0.52618 | 0.27167 | 0.08667 | 0.02402 | 0.38235 | 2.104 | 23.38 |
| 37 | FSS | Búsqueda de bancos de peces | 0.55669 | 0.39992 | 0.31172 | 1.26833 | 0.31009 | 0.11889 | 0.04569 | 0.47467 | 0.21167 | 0.07633 | 0.02488 | 0.31288 | 2.056 | 22.84 |
| 38 | RND | aleatorio | 0.52033 | 0.36068 | 0.30133 | 1.18234 | 0.31335 | 0.11787 | 0.04354 | 0.47476 | 0.25333 | 0.07933 | 0.02382 | 0.35648 | 2.014 | 22.37 |
| 39 | GWO | Optimizador de lobo gris | 0.59169 | 0.36561 | 0.29595 | 1.25326 | 0.24499 | 0.09047 | 0.03612 | 0.37158 | 0.27667 | 0.08567 | 0.02170 | 0.38403 | 2.009 | 22.32 |
| 40 | CSS | búsqueda de sistema cargado | 0.44252 | 0.35454 | 0.35201 | 1.14907 | 0.24140 | 0.11345 | 0.06814 | 0.42299 | 0.18333 | 0.06300 | 0.02322 | 0.26955 | 1.842 | 20.46 |
| 41 | EM | Algoritmo similar al electromagnetismo | 0.46250 | 0.34594 | 0.32285 | 1.13129 | 0.21245 | 0.09783 | 0.10057 | 0.41085 | 0.15667 | 0.06033 | 0.02712 | 0.24412 | 1.786 | 19.85 |
Resumen
A partir de la tabla y los resultados proporcionados, pueden extraerse las siguientes conclusiones sobre el rendimiento del algoritmo CRO:
1. CRO muestra excelentes resultados en la función de prueba Hilly. Con 5 parámetros, el resultado fue de 0,95, con 25 parámetros, de 0,66, y con 500 parámetros, de 0,30. Esto indica que el CRO es eficaz en funciones suaves, especialmente con menos parámetros.
2. En la función de prueba del bosque, CRO también muestra buenos resultados. Con 5 parámetros, el resultado fue de 0,88, con 25 parámetros, de 0,58, y con 500 parámetros, de 0,21. Esto sugiere que el CRO también es eficaz en funciones con extremos «agudos», pero tiene algunas dificultades para encontrar óptimos puntuales.
3. En la función de prueba Megacity, CRO sigue demostrando un buen rendimiento. Con 5 parámetros, el resultado fue de 0,76, con 25 parámetros, de 0,43, y con 500 parámetros, de 0,13. Esto indica que el CRO es eficaz en esta función discreta, sus resultados son uniformemente «verdes» en comparación con otros algoritmos, incluso los que están más arriba en la tabla.
Según la tabla proporcionada, el algoritmo CRO muestra resultados sólidos en comparación con otros algoritmos. En particular, en las funciones Hilly, Forest y Megacity, CRO demuestra competitividad, especialmente con menos parámetros.
El algoritmo CRO ocupó el puesto 11 en la tabla de clasificación. Basándose en la gradación de color de la tabla siguiente (donde el verde oscuro indica mejores resultados), puede decirse que la CRO muestra en general un rendimiento bueno y estable (coloración estable y uniforme). En la función Hilly con 1000 parámetros, los resultados parecen algo más débiles.
El algoritmo CRO ha demostrado ser un enfoque de optimización prometedor. Utiliza dos poblaciones de agentes (en mi implementación) que interactúan entre sí para proporcionar diversidad y evitar estancarse en óptimos locales. Una de las características distintivas del algoritmo es el uso de operadores especiales similares a reacciones químicas, descomposición, síntesis y otros.
En general, el algoritmo CRO es un método de optimización prometedor que se distingue por su originalidad y su capacidad para obtener resultados elevados en diversos problemas de optimización.
La elección del algoritmo de optimización debe basarse en la tarea específica y los requisitos de rendimiento, y nuestra tabla de clasificación le ayudará a ello. Al reelaborar la versión original del algoritmo CRO para que herede de la clase C_AO que he adoptado para los algoritmos poblacionales, este interesante algoritmo puede aplicarse a problemas de optimización en general.
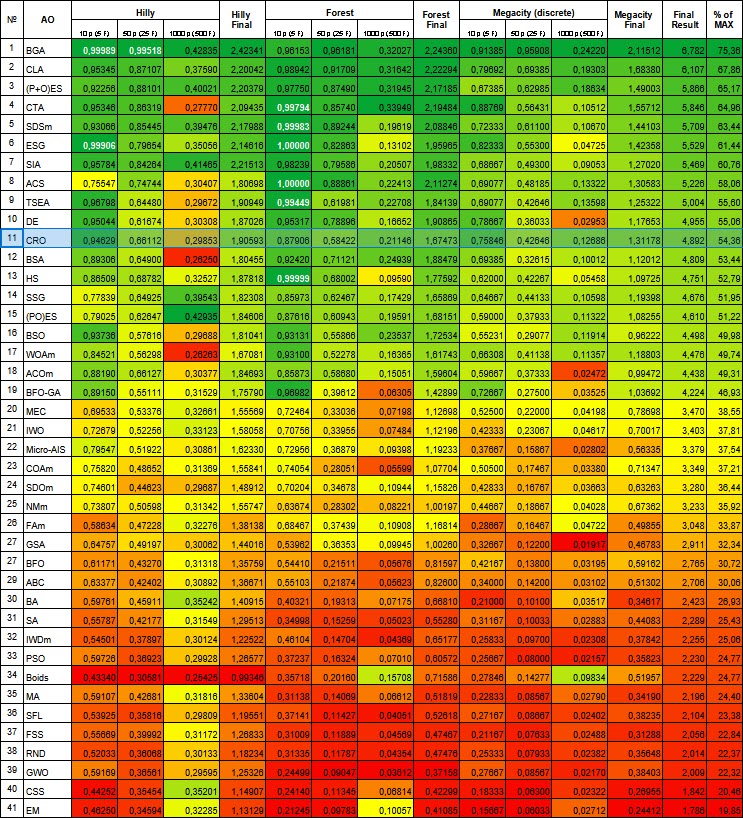
Figura 1. Gradación por colores de los algoritmos según las pruebas pertinentes Los resultados superiores o iguales a 0,99 se resaltan en blanco
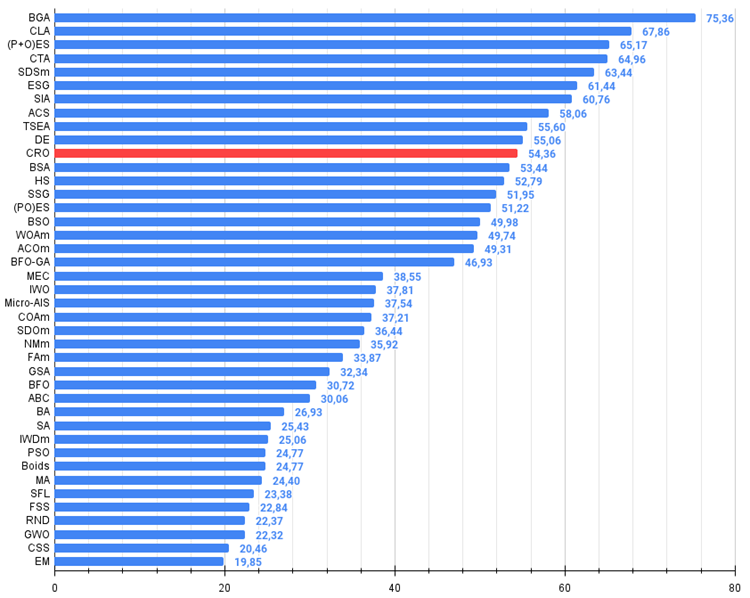
Figura 2. El histograma de los resultados de las pruebas de algoritmos (en una escala de 0 a 100, cuanto más, mejor,
donde 100 es el resultado teórico máximo posible, el archivo contiene un script para calcular la tabla de cotizaciones)
CRO pros y contras generales:
Ventajas:
- Buena convergencia en varios tipos de funciones.
- Muy rápido a pesar de su compleja arquitectura.
- Buena escalabilidad.
Desventajas:
- A veces se queda estancado en los extremos locales.
El artículo va acompañado de un archivo con las versiones actuales de los códigos del algoritmo. El autor del artículo no es responsable de la exactitud absoluta en la descripción de los algoritmos canónicos. Se han realizado cambios en muchos de ellos para mejorar las capacidades de búsqueda. Las conclusiones y juicios presentados en los artículos se basan en los resultados de los experimentos.
Traducción del ruso hecha por MetaQuotes Ltd.
Artículo original: https://www.mql5.com/ru/articles/15080
Advertencia: todos los derechos de estos materiales pertenecen a MetaQuotes Ltd. Queda totalmente prohibido el copiado total o parcial.
Este artículo ha sido escrito por un usuario del sitio web y refleja su punto de vista personal. MetaQuotes Ltd. no se responsabiliza de la exactitud de la información ofrecida, ni de las posibles consecuencias del uso de las soluciones, estrategias o recomendaciones descritas.
 Desarrollamos un asesor experto multidivisa (Parte 17): preparación adicional para el trading real
Desarrollamos un asesor experto multidivisa (Parte 17): preparación adicional para el trading real
 Del básico al intermedio: Variables (II)
Del básico al intermedio: Variables (II)
 Redes neuronales: así de sencillo (Parte 95): Reducción del consumo de memoria en los modelos de transformadores
Redes neuronales: así de sencillo (Parte 95): Reducción del consumo de memoria en los modelos de transformadores
 Del básico al intermedio: Variables (I)
Del básico al intermedio: Variables (I)
- Aplicaciones de trading gratuitas
- 8 000+ señales para copiar
- Noticias económicas para analizar los mercados financieros
Usted acepta la política del sitio web y las condiciones de uso