
化学反応最適化(CRO)アルゴリズム(第1回):最適化におけるプロセス化学
内容
1. はじめに
化学反応最適化(CRO)は、化学変換そのものの本質から着想を得た、刺激的で革新的な手法です。分子のダンスを想像してください。そのひとつひとつの動きや衝突が、複雑な問題の解決において重要な役割を果たします。この手法は、エネルギー保存、分解、合成といった原理を巧みに組み合わせ、柔軟で適応力のある最適化アプローチを生み出します。
CROは、熱力学の第一および第二法則に基づく分子間相互作用の一般原理を活用し、化学反応と最適化を緩やかに結びつけた方法です。このアルゴリズムは、2012年にLamとLiによって提案され、発表されました。
熱力学の第一法則(エネルギー保存の法則)は、「エネルギーは生成も破壊もされない」と述べています。エネルギーは1つの形態から別の形態に変換され、また1つの物体から別の物体へと移動します。CROの文脈では、化学反応系は物質と環境から成り、各粒子は位置エネルギーと運動エネルギーを持っています。
熱力学の第二法則は、システムのエントロピーは増加する傾向があり、エントロピーは無秩序の尺度であり、システムはより多くの自由を渇望していると述べています。位置エネルギーとは、分子の構造に基づいて分子に蓄えられたエネルギーのことです。分子の位置エネルギーが解放されて運動エネルギーに変換されると、システムはより混沌とし、エントロピーが増加します。しかし、この混沌の中でこそ、CROはエネルギーの流れを捉え、最適解へと導く力を発揮します。
たとえば、より高い運動エネルギー(位置エネルギーから変換)を持つ分子がより高速で移動すると、システムはより無秩序になり、エントロピーが増加します。すべての反応系は最終的に、位置エネルギーが最小となる平衡状態に到達しようとします。CROは、この現象を利用して位置エネルギーを運動エネルギーに変換し、分子が環境にエネルギーを徐々に放出することで、最適解を導き出します。
化学反応を経る化学系は通常、不安定で過剰なエネルギーを含み、これを解消して安定化しようとします。このシステムは、化合物を構成する最小単位である分子によって成り立っています。分子は基本的な化学的性質に基づき、さまざまなタイプに分類されます。
化学反応とは、分子同士が衝突し、結合を形成・破壊して構造を変化させる過程です。この複雑な「分子のダンス」の一歩一歩が、エネルギーを最小化し、より安定した生成物を生む一連のサブ反応として展開されます。分子は化学結合にエネルギーを蓄え、その構造のわずかな変化が、より安定した生成物を生む大きな変化をもたらすことがあります。分子構造は化合物の化学的挙動に影響を与え、わずかな構造の違いで化学的特性に大きな違いが生じることがあります。
化学反応は通常、分子間の衝突から始まります。この衝突には、単分子反応(外部物質との衝突)または二分子反応(他の分子との衝突)があります。衝突の効率は、活性化エネルギーや立体効果といった要因によって決まります。このように、分子は絡み合い、離れ、そして新しい形や構造を作り出していきます。
化学反応の多くは、より低いギブスエネルギーを持つ安定した生成物を形成する方向へ進行します。このプロセスでは、中間化合物の形成や遷移状態を経る複数の段階が関与します。これらの過程を理解することは、最適な反応条件を見つけたり、化合物の変化のメカニズムを解明する上で極めて重要です。
さらに、化学合成は、反応物の有効な衝突と無効な衝突の結果として生じる可能性があります。衝突の効率は、活性化エネルギーや分子の立体構造など、さまざまな要因に依存します。分子間でおこなわれる合成は、分子内での合成とは対照的に、より大きな構造変化をもたらします。
CROアルゴリズムは、まるで名手の振付師のように化学の法則を音符として使い、非常に複雑で洗練された最適化ソリューションを作り上げます。では、ここからは複雑な概念から単純な概念まで、すべてをひとつひとつ丁寧に見ていきましょう。
2. 化学演算子の実装
CROの基本単位は「分子」です。集団内の各分子は、「位置エネルギーと運動エネルギー」、「分子構造」などの特定の特性を持っています。基本反応は分子間の相互作用を定義します。これをプログラムで表現する際には、データフィールドが分子の特性を表し、メソッドが基本反応を記述するクラスを定義することが可能です。
CROの主要な概念の1つはエネルギー保存です。この原則により、最適化プロセス全体を通じてシステム内の総エネルギーが一定に保たれます。エネルギー保存は、反応中の分子のさまざまな状態間の遷移を決定し、最適解の探索に影響を与えるエネルギーバランスを維持します。
操作可能なエージェント(分子)、基本反応、エネルギー保存の概念を用いて、CROは複雑な最適化問題を解決するための柔軟で適応的な方法を提供します。演算子のカスタマイズ、集団サイズの動的な変更、および多様な属性の統合が可能であるため、CROはメタヒューリスティックの分野で有望なアプローチとなっています。この独自性と柔軟性により、研究者は最適化の新たな可能性を追求できます。
CROアルゴリズムでは次の演算子が使用されます。
1. 分子間の非効率衝突:この演算子は、2つの分子が衝突しても変わらず存在し、その構造がわずかに変化するプロセスをモデル化します。これにより、アルゴリズムは現在の解の近くで局所的な検索を実行できるようになります。
2. 分解:この演算子は、分子が壁に衝突して2つの新しい分子に分解されるプロセスをモデル化します。これにより、アルゴリズムは解空間の新しい領域を探索できるようになります。
3. 分子内反応:この演算子は、分子が壁に衝突してそのまま残るがその構造がわずかに変化するプロセスをモデル化します。これにより、アルゴリズムは現在の解の近くで局所的な検索を実行できるようになります。
4. 合成:この演算子は、2 つの分子が衝突して結合し、1 つの新しい分子を形成するプロセスをモデル化します。これにより、アルゴリズムは優れたソリューションを組み合わせて、潜在的に優れたソリューションを作成できます。
これらの各演算子はCROアルゴリズムで重要な役割を果たし、検索空間を探索して最適解を見つけることを可能にします。それらのコラボレーションにより、探索(検索空間の新しい領域の探索)と活用(現在の最善のソリューションの改善)のバランスが保たれます。
アルゴリズム内の個々の化学演算子を考えてみましょう(グローバル最小値を検索します)。
アルゴリズム#1:壁面の非効率衝突
1. 入力データ:Mω分子
2. 分子の新しい位置を生成する: ω = N(ω)
3. 新しい位置の位置エネルギーを計算する:PEω = f(ω)
4. 衝突カウンタを増やす:NumHitω = NumHitω + 1
5. 「新しい位置の位置エネルギー + 運動エネルギー」が現在の位置の位置エネルギー以上の場合、以下をおこなう。
6. [KELossRate, 1]の範囲で乱数「a」を生成する
7. 運動エネルギーを更新する:KEω = (PEω − PEω + KEω) × a
8. バッファを更新する:buffer = buffer + (PEω − PEω + KEω) × (1 − a)
9. 現在の位置とエネルギーを維持する
10. 新しい位置の位置エネルギーが最小位置エネルギーより小さい場合、最小値を更新する
11. 条件終了
アルゴリズム#2:分解
1. 入力データ:Mω分子
2. 2つの新しい分子Mω1とMω2を作成する
3. ωから新しい分子ω1とω2の位置を取得する
4. 新しい分子の潜在エネルギーを計算する:PEω1 = f(ω1) およびPEω2 = f(ω2)
5. 「現在の位置の位置エネルギー + 運動エネルギー」が新しい位置の合計の位置エネルギー以上の場合、次をおこなう。
6. 分解エネルギーを計算する:Edec = PEω + KEω − (PEω1 + PEω2)
7. ステップ13に進む
8. その他の場合、以下をおこなう。
9. 範囲[0, 1]内の乱数δ1、δ2を生成する
10. 分解エネルギーを計算する:Edec = PEω + KEω + δ1δ2 × buffer − (PEω1 + PEω2)
11. Edecが0以上の場合、次をおこなう。
12. バッファを更新する
13. 範囲[0, 1]で乱数δ3を生成する
14. 運動エネルギーを新しい分子間に分配する
15. 新しい分子ごとに最小値を維持する
16. 現在の分子を破壊する
17. その他の場合、以下をおこなう。
18. 衝突カウンタを増やす
19.新しい分子を破壊する
20.条件の終了
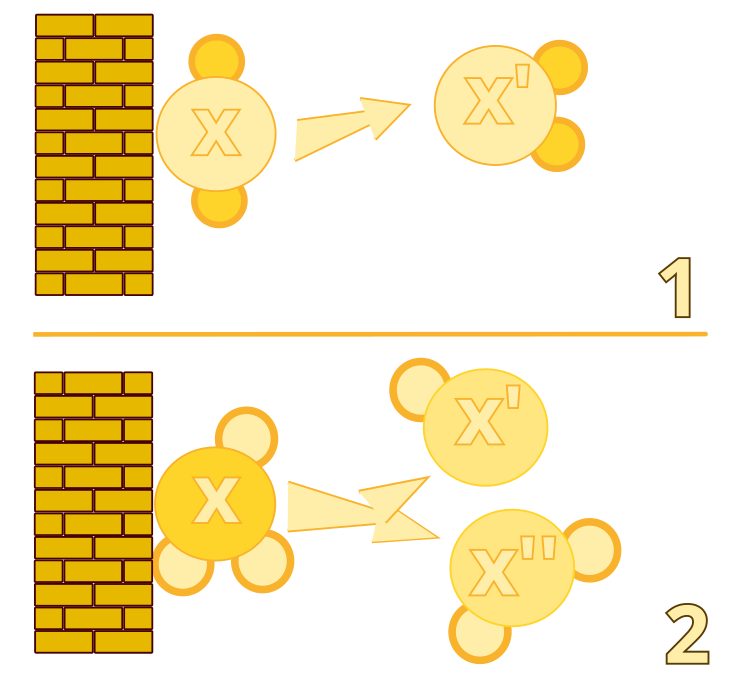
図1:非効率的な壁衝突(アルゴリズム#1)と分解(アルゴリズム#2)
アルゴリズム#3:分子間の非効率衝突
1. 入力データ:Mω1とMω2分子
2. 分子の新しい位置を生成する:ω1 = N(ω1)および ω2 = N(ω2)
3. 新しい位置の位置エネルギーを計算する:PEω1 = f(ω1)およびPEω2 = f(ω2)
4. 衝突カウンタを増やす:NumHitω1 = NumHitω1 + 1およびNumHitω2 = NumHitω2 + 1
5. 分子間相互作用のエネルギーを計算する:Einter = (PEω1 + PEω2 + KEω1 + KEω2) − (PEω1 + PEω2)
6. Einterが0以上の場合、次をおこないます。
7. 範囲[0, 1]で乱数δ4を生成する
8. 運動エネルギーを分子間に分配する:KEω1 = Einter × δ4およびKEω2 = Einter × (1 − δ4)
9. 分子の位置とエネルギーを更新する
10. 新しい位置の位置エネルギーが最小位置エネルギーより小さい場合は、各分子の最小値を更新する
11. 条件の終了
アルゴリズム#4:合成
1. 入力データ:Mω1とMω2分子
2. 新しいMω分子を作成する
3. ω1とω2から新しい分子ωの位置を取得する
4. 新しい分子の位置エネルギーを計算する:PEω = f(ω)
5. 新しい分子の全位置エネルギーと運動エネルギーが元の分子の全位置エネルギーと運動エネルギー以上の場合、次をおこないます。
6. 余剰運動エネルギーを新しい分子に分配する:KEω = (PEω1 + PEω2 + KEω1 + KEω2) − PEω
7. 新しい分子の最小値を更新する
8. 元の分子を破壊する
9. その他の場合、以下をおこなう。
10. ソース分子の衝突カウンタを増やす
11. 新しい分子を破壊する
12. 条件の終了
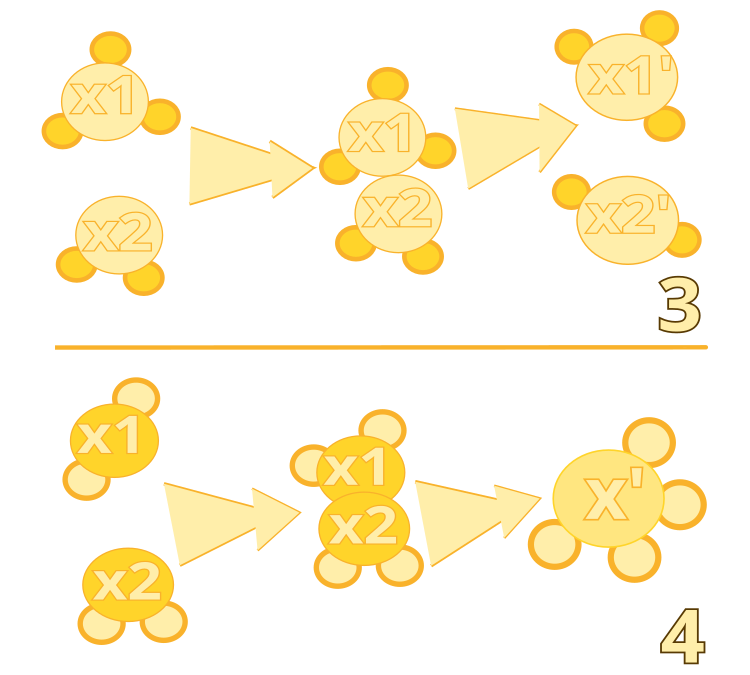
図2:非効率的な分子間衝突(アルゴリズム#3)と合成(アルゴリズム#4)
CROアルゴリズムの提案された説明は、このアプローチに対する著者のビジョンを反映しています。ただし、アルゴリズムのパフォーマンスと検索機能に大きな影響を与える可能性のあるいくつかの重要な点が考慮されていません。
このアルゴリズムには、閉空間でのエネルギーの保存と、あるタイプのエネルギーから別のタイプのエネルギーへの移行が含まれます。ただし、著者は数値的なエネルギー指標とターゲット(適合度)関数の値との対応関係を明確に示していません。適合度の役割は、著者によって位置エネルギーとして定義されており、運動エネルギーは位置エネルギーの減少を補完するメカニズムとして機能します(すべてのエネルギーの合計は理想的には一定であるべきです)。
たとえば、最適化問題の基準が利益率である場合、ターゲット関数の値は、残高を基準とする問題と比較して、小さな範囲で変動します。このような場合、ターゲット関数の値は特定の最適化基準に応じて変化することがわかりますが、著者は、アルゴリズムで計算されたエネルギーと比較することができない定数をアルゴリズム外部のパラメータとして使用しています。
このため、元のCROアルゴリズム(変更なし)は適用できるタスクの範囲がかなり限られており、汎用性に欠けます。本連載では、一般的な最適化問題に適用可能な汎用アルゴリズムのみを検討し、アルゴリズムがその条件を満たさない場合は、通常、それを修正して汎用的な形式に統一します。
次に、元のアルゴリズムでは、適合度関数の計算がロジックの異なる部分で無秩序におこなわれるため、実際のタスクで使用したり、プロジェクトに統合したりする際に問題が発生する可能性があります。この点も修正します。そのためには、化学的な演算子を2つの部分に分割する必要があります。1つ目は演算子そのものであり、2つ目はいわゆる「後処理演算子」です。前者は分子の位置を変更し、後者は分子の適合度を計算した後に必要な操作を実行します。
さらに、元のアルゴリズムでは分子の集団サイズが動的に変化します。新しい分子が生成され、古い分子の一部が破壊されますが、集団サイズを調整するメカニズムがなく、そのサイズは非常に幅広い範囲で変動する可能性があります。実験では、外部パラメータの特定の設定により、集団が初期の50分子から1,000分子以上に増加する可能性があることが示されています。この問題を解決するために、演算子の実行結果として分子を順次集団に追加し、単純なカウンタを使用して親分子のインデックスを親集団に保存する方法を採用しました。これにより、集団サイズを一定に保ちながら、分子を削除する操作を不要にしました。条件が満たされると、娘分子が対応する親分子を単に置き換える仕組みです。
これらの変更により、CROアルゴリズムを一般的な最適化問題の解決に適した形式に改善し、プロジェクトへの統合を容易にすると同時に、化学反応という元々のコンセプトを保持することができました。CROアルゴリズムの詳細を説明します。希望があれば、このアルゴリズムに位置エネルギーと運動エネルギーの計算を実装してみてください。
次にコードに移りましょう。反応の種類を列挙し、反応後に適切な後処理演算子と分子の構造を選択できるようにします。
E_ReactionType列挙体
1. synthesisは、2つの分子が結合して新しい分子を形成するプロセスです。
2. interMolecularInefCollは分子間の非効率衝突であり、分子は衝突しますが、互いに反応しません。
3. decompositionは、複雑な分子がより単純な成分に分解されるプロセスです。
4. inefCollisionは、壁との非効率衝突であり、分子の構造を変化させます。
これは、アルゴリズムのコンテキストにおける分子のモデルを表すS_CRO_Agent構造体を定義します。次の構造体にどのようなフィールドが含まれているかを見てみましょう。
- structure[]:分子構造を表す配列
- NumHit:「ヒット」または分子相互作用の数のカウンタ
- indMolecule_1とindMolecule_2:相互作用する分子のインデックス
- KE:分子の運動エネルギーを表す変数
- f:適応度関数(分子の適応度)
- rType:E_ReactionType型変数、分子が参加する反応のタイプ
Init:フィールドを初期化する構造メソッドArrayResize関数を使用して構造体配列のサイズを変更するために適用されるcoords整数引数を取ります。NumHit、indMolecule_1、indMolecule_2、f、KEはゼロまたは-DBL_MAXを使用して初期化されます。
以下のコードは、CROアルゴリズムの分子の基本データ構造を表し、新しい分子が作成されるときにフィールドを初期化します。
enum E_ReactionType { synthesis, interMolecularInefColl, decomposition, inefCollision }; // Molecule structure struct S_CRO_Agent { double structure []; int NumHit; int indMolecule_1; int indMolecule_2; double KE; double f; E_ReactionType rType; // Initialization method void Init (int coords) { ArrayResize (structure, coords); NumHit = 0; indMolecule_1 = 0; indMolecule_2 = 0; f = -DBL_MAX; KE = -DBL_MAX; } };
C_AO_CROクラスのInefCollisionメソッドは、1つの親分子を移動させることによって新しい分子を作成する非効率衝突です。このメソッドでは以下をおこないます。
1. 親分子のindexとmolCNT分子カウンタへのリンクを受け入れます。molCNTがpopSizeの集団サイズ以上の場合、falseを返して終了します。
2. 衝突の結果として作成される新しい分子index1_のインデックスを決定します。
3. 親分子の構造体を新しい分子の構造体にコピーします。
4. 新しい分子の各座標に対してN関数を実行します。この関数は、古い座標値の近くに新しい座標値を生成します。
5. 新しい座標値をSeInDiSp関数を使用して調整し、rangeMinからrangeMaxまでの指定された範囲内に収まるようにします。
6. 次に、新しい分子のindMolecule_1、rType、NumHitフィールドの値を設定します。indMolecule_1には親分子のインデックスが格納され、rTypeはinefCollisionに設定され、NumHitはリセットされます。
7. 最後に、molCNT分子カウンタが1増加し、trueを返します。
//—————————————————————————————————————————————————————————————————————————————— // Ineffective collision. Obtaining a new molecule by displacing a parent one. bool C_AO_CRO::InefCollision (int index, int &molCNT) { if (molCNT >= popSize) return false; int index1_ = molCNT; ArrayCopy (Mfilial [index1_].structure, Mparent [index].structure); for (int c = 0; c < coords; c++) { N (Mfilial [index1_].structure [c], c); Mfilial [index1_].structure [c] = u.SeInDiSp (Mfilial [index1_].structure [c], rangeMin [c], rangeMax [c], rangeStep [c]); } Mfilial [index1_].indMolecule_1 = index; // save the parent molecule index Mfilial [index1_].rType = inefCollision; Mfilial [index1_].NumHit = 0; molCNT++; return true; } //——————————————————————————————————————————————————————————————————————————————
C_AO_CROクラスのPostInefCollisionメソッドは、化学反応シミュレーションで分子と他の分子との非効率衝突の結果を処理するためのものです。このメソッドでは次をおこないます。
1. ind変数を宣言し、molオブジェクトのindMolecule_1値を使用して初期化します。
2. 条件式は、molオブジェクトのf値がindインデックスを持つ親分子のf値を超えているかどうかを確認します。
3. 条件がtrueの場合、mol構造体をindインデックスを持つ親分子の構造体にコピーします。
4. 親分子のf値をmolのf値を使用して更新します。
5. 親分子のNumHitカウンタをゼロにリセットします。
6. mol.f > Mparent [ind].fがfalseの場合、親分子のNumHitカウンタを1つ増加させます。
一般に、このメソッドでは、非効率衝突の結果に基づいて、親分子の構造と適合関数の値が更新されます。新しいmol構造体が適応度の向上につながる場合、親分子の構造体が置き換えられ、NumHitカウンタがリセットされます。それ以外の場合は、親分子のNumHitカウンタが増加します。
//—————————————————————————————————————————————————————————————————————————————— // Handling the results of an ineffective collision. void C_AO_CRO::PostInefCollision (S_CRO_Agent &mol) { int ind = mol.indMolecule_1; if (mol.f > Mparent [ind].f) { ArrayCopy (Mparent [ind].structure, mol.structure); Mparent [ind].f = mol.f; Mparent [ind].NumHit = 0; } else { Mparent [ind].NumHit++; } } //——————————————————————————————————————————————————————————————————————————————
C_AO_CROクラスのDecompositionメソッドは、1 つの親分子を分解して2つの新しい分子を作成する分解プロセスです。このメソッドでは以下をおこないます。
1. 親分子のindexとmolCNT分子カウンタへのリンクを受け入れます。molCNTがpopSize - 1の集団サイズ以上の場合、falseを返して終了します。
2. 次に、分解の結果として作成される2つの新しい分子index1_とindex2_のインデックスを決定します。
3. 親分子の構造体を新しい分子の構造体にコピーします。
4. 新しい分子の各座標に対してN関数を実行します。この関数は、古い座標値の近くに新しい座標値を生成します。これを、最初の娘分子のindex1_の座標の前半と、2番目の娘分子のindex2_の座標の後半に対してそれぞれ別々に実行します。
5. 新しい座標値をSeInDiSp関数を使用して調整し、rangeMinからrangeMaxまでの指定された範囲内に収まるようにします。
6. 次に、新しい分子のindMolecule_1、indMolecule_2、rType、NumHitフィールドの値を設定します。indMolecule_1とindMolecule_2でそれぞれ親分子と姉妹分子のインデックスを保存し、rTypeをdecompositionに設定し、NumHitをゼロに設定します。
7. メソッドの最後に、molCNT分子カウンタを2増加し、trueを返します。
//—————————————————————————————————————————————————————————————————————————————— // Decomposition. Obtaining two new molecules by decomposing a parent one. bool C_AO_CRO::Decomposition (int index, int &molCNT) { if (molCNT >= popSize - 1) return false; // Creating two new molecules M_ω'_1 and M_ω'_2 from M_ω int index1_ = molCNT; int index2_ = molCNT + 1; ArrayCopy (Mfilial [index1_].structure, Mparent [index].structure); ArrayCopy (Mfilial [index2_].structure, Mparent [index].structure); for (int c = 0; c < coords / 2; c++) { N (Mfilial [index1_].structure [c], c); Mfilial [index1_].structure [c] = u.SeInDiSp (Mfilial [index1_].structure [c], rangeMin [c], rangeMax [c], rangeStep [c]); } for (int c = coords / 2; c < coords; c++) { N (Mfilial [index2_].structure [c], c); Mfilial [index2_].structure [c] = u.SeInDiSp (Mfilial [index2_].structure [c], rangeMin [c], rangeMax [c], rangeStep [c]); } Mfilial [index1_].indMolecule_1 = index; // save the parent molecule index Mfilial [index1_].indMolecule_2 = index2_; // save the index of the second daughter molecule Mfilial [index1_].rType = decomposition; Mfilial [index1_].NumHit = 0; Mfilial [index2_].indMolecule_1 = index1_; // save the index of the first daughter molecule Mfilial [index2_].indMolecule_2 = -1; // mark the molecule so we do not handle it twice Mfilial [index2_].rType = decomposition; Mfilial [index2_].NumHit = 0; molCNT += 2; return true; } //——————————————————————————————————————————————————————————————————————————————
C_AO_CROクラスのPostDecompositionメソッドは、分子分解の結果を処理します。このメソッドでは次をおこないます。
1. 分解の結果として得られたmol分子への参照を受け入れます。分子のindMolecule_2が-1に等しい場合(分子がすでに処理されていることを意味する)、終了します。
2. 次に、親分子のindインデックスを、2つの「娘」分子index1_とindex2_のインデックスとともに抽出します。
3. 次に、最初の「娘」分子のf適合関数値が、2番目の「娘」分子と親分子のf関数値を超えているかどうかを確認します。この場合、最初の「娘」分子が親分子に置き換わります。置き換えられた分子のNumHit値をリセットし、flagを設定します。
4. flagがまだfalseの場合、2番目の「娘」分子に対して同様のチェックを実行します。
5. すべてのチェック後もflagがfalseの場合、親分子のNumHitを1増加させます。
PostDecompositionメソッドは、CROアルゴリズムでの分解後に親分子の状態を更新する役割を担います。
//—————————————————————————————————————————————————————————————————————————————— // Handling decomposition results. void C_AO_CRO::PostDecomposition (S_CRO_Agent &mol) { if (mol.indMolecule_2 == -1) return; int ind = mol.indMolecule_1; int index2_ = mol.indMolecule_2; int index1_ = Mfilial [index2_].indMolecule_1; bool flag = false; if (Mfilial [index1_].f > Mfilial [index2_].f && Mfilial [index1_].f > Mparent [ind].f) { ArrayCopy (Mparent [ind].structure, Mfilial [index1_].structure); Mparent [ind].f = Mfilial [index1_].f; Mparent [ind].NumHit = 0; flag = true; } if (!flag) { if (Mfilial [index2_].f > Mfilial [index1_].f && Mfilial [index2_].f > Mparent [ind].f) { ArrayCopy (Mparent [ind].structure, Mfilial [index2_].structure); Mparent [ind].f = Mfilial [index2_].f; Mparent [ind].NumHit = 0; flag = true; } } if (!flag) { Mparent [ind].NumHit++; } } //——————————————————————————————————————————————————————————————————————————————
C_AO_CROクラスのInterMolInefCollメソッドは、2つの親分子を変更して2つの新しい分子を作成する非効率衝突です。このメソッドでは以下をおこないます。
1. このメソッドは、2つの親分子のインデックス(index1とindex2)と、molCNT分子カウンタへのリンクを受け入れます。molCNTがpopSize - 1の集団サイズ以上の場合、falseを返して終了します。
2. 次に、このメソッドは、衝突の結果として作成される2つの新しい娘分子index1_とindex2_のインデックスを決定します。
3. 親分子の構造体を新しい分子の構造体にコピーします。
4. 新しい分子の各座標に対してN関数を実行します。この関数は、古い座標値の近くに新しい座標値を生成します。
5. 次に、SeInDiSp 関数を使用して、新しい座標値が指定されたrangeMinからrangeMaxの範囲内に収まるように調整します。
6. 次に、新しい分子のindMolecule_1、indMolecule_2、rTypeおよびNumHitフィールドの値を設定します。indMolecule_1とindMolecule_2は親分子のインデックスを保持し、rTypeはinterMolecularInefCollに設定され、NumHitはゼロに設定されます。
7. メソッドの最後に、molCNT分子カウンタを2増加し、trueを返します。
//—————————————————————————————————————————————————————————————————————————————— // Intermolecular ineffective collision. Obtaining two new molecules by changing two parent ones bool C_AO_CRO::InterMolInefColl (int index1, int index2, int &molCNT) { if (molCNT >= popSize - 1) return false; int index1_ = molCNT; int index2_ = molCNT + 1; // Obtaining molecules ArrayCopy (Mfilial [index1_].structure, Mparent [index1].structure); ArrayCopy (Mfilial [index2_].structure, Mparent [index2].structure); // Generating new molecules ω'_1 = N(ω1) and ω'_2 = N(ω2) in the vicinity of ω1 and ω2 for (int c = 0; c < coords; c++) { N (Mfilial [index1_].structure [c], c); N (Mfilial [index2_].structure [c], c); } for (int c = 0; c < coords; c++) { Mfilial [index1_].structure [c] = u.SeInDiSp (Mfilial [index1_].structure [c], rangeMin [c], rangeMax [c], rangeStep [c]); Mfilial [index2_].structure [c] = u.SeInDiSp (Mfilial [index2_].structure [c], rangeMin [c], rangeMax [c], rangeStep [c]); } Mfilial [index1_].indMolecule_1 = index1; // save the index of the first parent molecule Mfilial [index1_].indMolecule_2 = index2_; // save the index of the second daughter molecule Mfilial [index1_].rType = interMolecularInefColl; Mfilial [index1_].NumHit = 0; Mfilial [index2_].indMolecule_1 = index2; // save the index of the second parent molecule Mfilial [index2_].indMolecule_2 = -1; // mark the molecule so we do not handle it twice Mfilial [index2_].rType = interMolecularInefColl; Mfilial [index2_].NumHit = 0; molCNT += 2; return true; } //——————————————————————————————————————————————————————————————————————————————
C_AO_CROクラスのPostInterMolInefCollメソッドは、分子間の非効率衝突の結果を処理します。このメソッドの目的は次のとおりです。
1. 衝突の結果として得られたmol分子への参照を受け入れます。分子のindMolecule_2が-1に等しい場合(分子がすでに処理されていることを意味する)、終了します。
2. 次に、2 つの親分子のind1インデックスと ind2インデックスを抽出します。
3. 次に、新しい分子とその「姉妹」分子の適応度のf合計が、両方の親分子の適応度のf合計を超えるかどうかを確認します。これがtrueであれば、新しい分子が親分子を置き換えます。置き換えられた分子のNumHit値をリセットします。
4. それ以外の場合、親分子のNumHit値を1増加させます。
PostInterMolInefCollメソッドは、CROアルゴリズムにおける分子間の非効率衝突後に親分子の状態を更新する役割を担います。
//—————————————————————————————————————————————————————————————————————————————— // Handling the results of an intermolecular ineffective collision. void C_AO_CRO::PostInterMolInefColl (S_CRO_Agent &mol) { if (mol.indMolecule_2 == -1) return; int ind1 = mol.indMolecule_1; int ind2 = Mfilial [mol.indMolecule_2].indMolecule_1; Mparent [ind1].NumHit++; Mparent [ind2].NumHit++; if (mol.f + Mfilial [mol.indMolecule_2].f > Mparent [ind1].f + Mparent [ind2].f) { ArrayCopy (Mparent [ind1].structure, mol.structure); Mparent [ind1].f = mol.f; ArrayCopy (Mparent [ind2].structure, Mfilial [mol.indMolecule_2].structure); Mparent [ind2].f = Mfilial [mol.indMolecule_2].f; } } //——————————————————————————————————————————————————————————————————————————————
アルゴリズムの最後の化学反応演算子(C_AO_CROクラスのSynthesisメソッド)は、2つの親分子を融合して新しい分子を作成する合成プロセスです。
1. このメソッドは、2つの親分子のインデックス(index1とindex2)と、molCNT分子カウンタへのリンクを受け入れます。molCNTがpopSizeの集団サイズ以上の場合、falseを返して終了します。
2. ループでは、新しい分子の各座標に対して次の処理をおこないます。乱数が0.5未満の場合、座標は最初の親分子Mparent[index1].structure[i]の対応する座標の値を受け取ります。それ以外の場合は、2番目の親分子Mparent[index2].structure[i]の対応する座標の値を受け取ります。
3. 次に、新しい分子のindMolecule_1、indMolecule_2、rTypeおよびNumHitフィールドの値を設定します。indMolecule_1とindMolecule_2は親分子のインデックスを保持し、rTypeはsynthesisに設定され、NumHitはゼロに設定されます。
4. 最後に、molCNT分子カウンタを増加させ、trueを返します。
//—————————————————————————————————————————————————————————————————————————————— // Synthesis. Obtaining a new molecule by fusing two parent ones bool C_AO_CRO::Synthesis (int index1, int index2, int &molCNT) { if (molCNT >= popSize) return false; // Create a new M_ω' molecule from M_ω1 and M_ω2 for (int i = 0; i < coords; i++) { if (u.RNDprobab () < 0.5) Mfilial [molCNT].structure [i] = Mparent [index1].structure [i]; else Mfilial [molCNT].structure [i] = Mparent [index2].structure [i]; } Mfilial [molCNT].indMolecule_1 = index1; // save the index of the first parent molecule Mfilial [molCNT].indMolecule_2 = index2; // save the index of the second parent molecule Mfilial [molCNT].rType = synthesis; Mfilial [molCNT].NumHit = 0; molCNT++; return true; } //——————————————————————————————————————————————————————————————————————————————
C_AO_CROクラスのPostSynthesisメソッドは合成結果を処理します。その中では以下が起こります。
1. 合成の結果として得られたmol分子への参照を受け入れます。次に、2 つの親分子のind1インデックスとind2インデックスを抽出します。
2. 次に、新しい分子のf適応度値が両方の親分子のf適応度値を超えているかどうかを確認します。この場合、新しい分子は、f値がより小さい親分子の1つを置き換えます。置き換えられた分子のNumHit値をゼロに設定します。
3. それ以外の場合、親分子のNumHit値を1増加させます。
PostSynthesisメソッドは、CROアルゴリズムの合成プロセス後に親分子の状態を更新する役割を担います。
//—————————————————————————————————————————————————————————————————————————————— // Handling synthesis results. void C_AO_CRO::PostSynthesis (S_CRO_Agent &mol) { int ind1 = mol.indMolecule_1; int ind2 = mol.indMolecule_2; if (mol.f > Mparent [ind1].f && mol.f > Mparent [ind2].f) { if (Mparent [ind1].f < Mparent [ind2].f) { ArrayCopy (Mparent [ind1].structure, mol.structure); Mparent [ind1].f = mol.f; Mparent [ind1].NumHit = 0; } else { ArrayCopy (Mparent [ind2].structure, mol.structure); Mparent [ind2].f = mol.f; Mparent [ind2].NumHit = 0; } } else { Mparent [ind1].NumHit++; Mparent [ind2].NumHit++; } } //——————————————————————————————————————————————————————————————————————————————
最後に、化学反応演算子で分子構造を変更するために使用されるC_AO_CROクラスのNメソッドについて説明します。このメソッドは、指定された範囲内で分子座標の新しい値を生成するために使用されます。
1. このメソッドは、参照として変更する必要があるcoord分子座標と、構造内のcoordPos座標位置を受け入れます。
2. 次に、範囲の最大値と最小値の差であるdist距離を計算し、これに現在の座標値の付近の値の広がりを示すmolecPerturbパラメータを掛けます。
3. 次に、新しい座標の最小値(min)と最大値(max)を定義します。これらの値は、古い座標値にdist距離を加算または減算したものに等しくなりますが、rangeMin[coordPos]からrangeMax[coordPos]までの指定された範囲を超えることはできません。
4. 最後に、座標の古い値、標準偏差の最小値と最大値を8に等しくするu.GaussDistributionガウス分布関数を使用して、新しい座標値を生成します。
//—————————————————————————————————————————————————————————————————————————————— void C_AO_CRO::N (double &coord, int coordPos) { double dist = (rangeMax [coordPos] - rangeMin [coordPos]) * molecPerturb; double min = coord - dist; if (min < rangeMin [coordPos]) min = rangeMin [coordPos]; double max = coord + dist; if (max > rangeMax [coordPos]) max = rangeMax [coordPos]; coord = u.GaussDistribution (coord, min, max, 8); } //——————————————————————————————————————————————————————————————————————————————
最適化プロセスにおける機能と役割の観点から、すべての化学演算子を詳細に分析しました。この分析により、化学反応モデリング(CRO)における分子のダイナミクスを規定するメカニズムについて、深い洞察を得ることができました。
次回の記事では、アルゴリズムを構築し、それをテスト関数に基づいて検証します。これまで研究した演算子を組み合わせて構成し、実用的なタスクに対応可能な本格的なCROアルゴリズムを作成します。その後、多様なテスト関数を用いた一連の実験を実施し、このアプローチの効率性と堅牢性を評価します。
実験の結果に基づき、アルゴリズムの強みと弱点を明らかにし、さらに改善すべき方向性について結論を導きます。これにより、私たちの手法を改良するだけでなく、化学プロセスの最適化やモデリング分野で研究を進める他の研究者に対して、有益な提言を提供できるようになります。
したがって、次回の記事は、理論から実践への移行を象徴する重要なステップとなります。複雑な最適化問題を解決し、最良の結果を達成するために、アルゴリズムのテストと改良をおこなう決定的な機会となるでしょう。
MetaQuotes Ltdによってロシア語から翻訳されました。
元の記事: https://www.mql5.com/ru/articles/15041
警告: これらの資料についてのすべての権利はMetaQuotes Ltd.が保有しています。これらの資料の全部または一部の複製や再プリントは禁じられています。
この記事はサイトのユーザーによって執筆されたものであり、著者の個人的な見解を反映しています。MetaQuotes Ltdは、提示された情報の正確性や、記載されているソリューション、戦略、または推奨事項の使用によって生じたいかなる結果についても責任を負いません。
 データサイエンスとML(第30回):株式市場を予測するパワーカップル、畳み込みニューラルネットワーク(CNN)と再帰型ニューラルネットワーク(RNN)
データサイエンスとML(第30回):株式市場を予測するパワーカップル、畳み込みニューラルネットワーク(CNN)と再帰型ニューラルネットワーク(RNN)
 Candlestick Trend Constraintモデルの構築(第9回):マルチ戦略エキスパートアドバイザー(I)
Candlestick Trend Constraintモデルの構築(第9回):マルチ戦略エキスパートアドバイザー(I)
 MetaTraderとGoogleスプレッドシートを使用して取引ジャーナルを作成する方法
MetaTraderとGoogleスプレッドシートを使用して取引ジャーナルを作成する方法
 ニューラルネットワークが簡単に(第93回):周波数領域と時間領域における適応予測(最終回)
ニューラルネットワークが簡単に(第93回):周波数領域と時間領域における適応予測(最終回)
- 無料取引アプリ
- 8千を超えるシグナルをコピー
- 金融ニュースで金融マーケットを探索
取 引における実践的な アプリケーションについてです。 読んで、 勉強して、 そして 実践に応用してください。 成功を祈っています 取引において。
記事
最適化アルゴリズムを使ってEAのパラメータをオンザフライで設定する
アンドレイ・ディク, 2024.06.07 14:30
この記事では、最適化アルゴリズムを使ってオンザフライで最適なEAパラメータを見つけることの実践的な側面と、取引操作とEAロジックの仮想化について説明します。この記事は、最適化アルゴリズムをEAに実装するためのインストラクションとして使用できます。ここでは 記事 について 実践的な 取引における の応用。 読む、 勉強する と 実践に応用する。 私は あなた 成功を祈る 取引において。
アンドレイ、私はあなたの視点が好きだ! 偉大なイノベーションは研究から生まれる。それは必ずしもお金の問題ではないし、私はあなたのコミュニティへの貢献に感謝している。