
Algoritmo de otimização por reações químicas — Chemical Reaction Optimisation, CRO (Parte II): Montagem e resultados
Conteúdo:
1. Introdução
Na segunda parte de nosso artigo, continuamos a mergulhar no fascinante mundo da otimização por reações químicas (CRO). Com base nos conceitos de “moléculas” e “reações elementares”, apresentamos os princípios subjacentes à operação do algoritmo e analisamos como esses conceitos são aplicados a problemas complexos de otimização. Além disso, discutimos os pontos principais da conservação de energia no CRO e as funções do algoritmo, como: decomposição, síntese, colisões ineficazes intramolecular e intermolecular, que desempenham um papel importante na exploração do espaço de busca e na obtenção de soluções ótimas.
Depois de abordarmos os conceitos e princípios básicos dos operadores químicos do algoritmo de otimização química, é hora de passarmos à montagem geral do algoritmo e à sua aplicação prática. Nesta parte do documento, concentramo-nos nos resultados do algoritmo em diferentes funções de teste para analisar sua eficácia e seu potencial na solução de problemas do mundo real. Examinaremos seu desempenho, convergência e capacidade de encontrar ótimos globais, o que nos permitirá avaliar sua aplicabilidade e comparar os resultados do algoritmo CRO com outros métodos de otimização, identificando suas vantagens e desvantagens.
2.Implementação do algoritmo
Continuaremos a escrever o código do algoritmo seguindo o padrão que você já conhece para todos os algoritmos, o qual inclui funções padrão. "Init", "Moving", "Revision", e como a estrutura já está definida, com os principais operadores químicos de reação já escritos, prosseguiremos com a escrita da classe do algoritmo para unificar todos os componentes em um único todo.
Precisaremos escrever um pseudocódigo para realizar a montagem do algoritmo:
Se a flag de revisão não estiver ativa, inicialização:
Para cada molécula i de 0 até popSize:
Para cada coordenada c de 0 até coords
Gerar um valor aleatório para a estrutura da molécula dentro do intervalo de rangeMin a rangeMax
Restringir o valor da estrutura ao intervalo definido
Armazenar a estrutura na matriz a
Fim
Cálculo da energia cinética:
minKE = valor máximo do tipo double
Para cada molécula i de 0 até popSize:
Se fitness da molécula for menor que minKE:
minKE = fitness da molécula
Para cada molécula i de 0 até popSize:
Calcular a energia cinética da molécula KE como uma escala do fitness do intervalo de minKE até fB (melhor solução global) para o intervalo de 0.0 a 1.0.
molCNT = 0
Enquanto não interrompido:
Se um número aleatório for menor que moleColl:
Selecionar duas moléculas aleatórias M1 e M2
Se KE de ambas as moléculas for maior ou igual a β:
Realizar a síntese das moléculas M1 e M2
Caso contrário:
Executar uma colisão intermolecular ineficaz entre M1 e M2
Caso contrário:
Selecionar uma molécula aleatória M
Se NumHit da molécula for maior que α:
Realizar a decomposição da molécula M
Caso contrário:
Executar uma colisão na molécula M
Copiar as estruturas das moléculas de Mfilial para a
Calcular aptidão para os indivíduos da população a
Copiar as estruturas das moléculas de a para Mfilial
Inicialização: ind = -1
Para cada molécula i de 0 até popSize:
Se aptidão da molécula for maior que fB:
fB = aptidão da molécula
ind = i
Se ind for diferente de -1:
Copiar a estrutura da molécula para cB
Atualização das moléculas:
Se não houver revisão:
Para cada molécula i de 0 até popSize:
Atualizar aptidão da molécula em Mparent
Definir revisão como true
Fim
Para cada molécula i de 0 até popSize:
Atualizar aptidão da molécula em Mfilial
Dependendo do tipo de molécula (síntese, colisão intermolecular ineficaz, decomposição, colisão):
Executar a pós-operação correspondente
Agora que temos o pseudocódigo e os operadores químicos descritos na primeira parte do artigo, podemos começar a montar a implementação do algoritmo CRO em código.
Declararemos a classe "C_AO_CRO", que é um herdeiro da classe base "C_AO" e representa a implementação do algoritmo Chemical Reaction Optimisation (CRO).
1. Campos públicos:
- "popSize" - tamanho da população.
- "moleColl", "alpha", "beta", "molecPerturb" - parâmetros do algoritmo.
- "params" - matriz para armazenar os parâmetros do algoritmo.
- "Mparent[]", "Mfilial[]" - objetos da estrutura "S_CRO_Agent", representando as moléculas.
2. Métodos:
- "C_AO_CRO()" - construtor da classe, inicializando os campos da classe.
- "SetParams()" - método para definir os parâmetros do algoritmo.
- "Init()" - método para inicializar o algoritmo. Recebe os intervalos mínimos e máximos de busca, o passo de busca e o número de épocas.
- "Moving()", "Revision()" - métodos que implementam as principais operações do algoritmo.
3. Campos e métodos privados:
- "Synthesis()", "InterMolInefColl()", "Decomposition()", "InefCollision()" - métodos que implementam os diferentes tipos de reações.
- "PostSynthesis()", "PostInterMolInefColl()", "PostDecomposition()", "PostInefCollision()" - métodos que executam as ações após as respectivas reações.
- "N()" - método para modificar um componente (coordenada) da estrutura da molécula.
Essa classe representa uma implementação completa do algoritmo Chemical Reaction Optimisation (CRO) e contém todos os dados e métodos necessários.
//—————————————————————————————————————————————————————————————————————————————— class C_AO_CRO : public C_AO { public: //-------------------------------------------------------------------- ~C_AO_CRO () { } C_AO_CRO () { ao_name = "CRO"; ao_desc = "Chemical Reaction Optimisation"; ao_link = "https://www.mql5.com/en/articles/15041"; popSize = 50; //population size moleColl = 0.9; alpha = 200; beta = 0.01; molecPerturb = 0.5; ArrayResize (params, 5); params [0].name = "popSize"; params [0].val = popSize; params [1].name = "moleColl"; params [1].val = moleColl; params [2].name = "alpha"; params [2].val = alpha; params [3].name = "beta"; params [3].val = beta; params [4].name = "molecPerturb"; params [4].val = molecPerturb; } void SetParams () { popSize = (int)params [0].val; moleColl = params [1].val; alpha = (int)params [2].val; beta = params [3].val; molecPerturb = params [4].val; } bool Init (const double &rangeMinP [], //minimum search range const double &rangeMaxP [], //maximum search range const double &rangeStepP [], //step search const int epochsP = 0); //number of epochs void Moving (); void Revision (); S_CRO_Agent Mparent []; S_CRO_Agent Mfilial []; //---------------------------------------------------------------------------- double moleColl; int alpha; double beta; double molecPerturb; private: //------------------------------------------------------------------- bool Synthesis (int index1, int index2, int &molCNT); bool InterMolInefColl (int index1, int index2, int &molCNT); bool Decomposition (int index, int &molCNT); bool InefCollision (int index, int &molCNT); void PostSynthesis (S_CRO_Agent &mol); void PostInterMolInefColl (S_CRO_Agent &mol); void PostDecomposition (S_CRO_Agent &mol); void PostInefCollision (S_CRO_Agent &mol); void N (double &coord, int coordPos); }; //——————————————————————————————————————————————————————————————————————————————
O método "Init" da classe "C_AO_CRO" é usado para inicializar as variáveis da classe com base nos parâmetros fornecidos. Veja o que acontece neste método:
1. O método chama a função "StandardInit", que recebe os intervalos mínimos e máximos de busca, assim como o passo de busca. Se "StandardInit" retornar "false", então o método "Init" também retorna "false" e encerra sua execução.
2. Em seguida, o método redimensiona os arrays "Mparent" e "Mfilial" para o tamanho "popSize", que representa o tamanho da população.
3. Depois, para cada elemento nos arrays "Mparent" e "Mfilial", o método "Init" é chamado com o parâmetro "coords". Este método inicializa os campos de cada agente na população.
4. No final, o método retorna "true", indicando a conclusão bem-sucedida da inicialização.
Este método executa a configuração inicial do algoritmo Chemical Reaction Optimisation (CRO) com os parâmetros fornecidos, preparando-o para a execução da otimização.
//—————————————————————————————————————————————————————————————————————————————— bool C_AO_CRO::Init (const double &rangeMinP [], //minimum search range const double &rangeMaxP [], //maximum search range const double &rangeStepP [], //step search const int epochsP = 0) //number of epochs { if (!StandardInit (rangeMinP, rangeMaxP, rangeStepP)) return false; //---------------------------------------------------------------------------- ArrayResize (Mparent, popSize); ArrayResize (Mfilial, popSize); for (int i = 0; i < popSize; i++) { Mparent [i].Init (coords); Mfilial [i].Init (coords); } return true; } //——————————————————————————————————————————————————————————————————————————————
O método "Moving" da classe "C_AO_CRO" é usado para chamar os operadores químicos, que alteram as estruturas das moléculas, deslocando-as durante o processo de otimização. O método realiza as seguintes ações:
1. Se "revision" for igual a "false", então, para cada molécula na população "Mparent", as estruturas são inicializadas com valores aleatórios dentro do intervalo especificado de "rangeMin" a "rangeMax". Esses valores são então copiados para o array "a".
2. Se "revision" não for igual a "false", o menor valor da função "f" é calculado entre todas as moléculas na população "Mparent". Depois, para cada molécula, o valor "KE" é calculado com base no escalonamento do valor da função "f", o valor mínimo "f" da população de moléculas parentais e a melhor solução global fB para o intervalo de 0.0 a 1.0.
3. Então, enquanto um dos operadores químicos não retornar false (isso significa que não há mais espaço na população filha para moléculas filhas), o seguinte ocorre:
- Se um número aleatório for menor que "moleColl", duas moléculas aleatórias "M1" e "M2" são selecionadas. Se "KE" de ambas as moléculas for maior ou igual a "beta", ocorre a síntese (ou seja, a síntese é realizada para moléculas que não estão abaixo do valor relativo de adaptabilidade definido nos parâmetros; para isso, o escalonamento de adaptabilidade foi realizado previamente para o intervalo de 0.0 a 1.0). Caso contrário, ocorre uma colisão intermolecular ineficaz.
- Se o número aleatório for maior ou igual a "moleColl", uma molécula aleatória "M" é selecionada. Se "NumHit" dessa molécula for maior que "alpha" (se a molécula tiver sofrido mais colisões do que o limite definido nos parâmetros do algoritmo, ela "se decompõe"), a decomposição é realizada. Caso contrário, ocorre uma colisão.
4. No final do método, as estruturas de todas as moléculas em "Mfilial" são copiadas para o array de população "a".
Este método é responsável por atualizar as estruturas das moléculas no algoritmo Chemical Reaction Optimisation (CRO) de acordo com o estado atual do sistema e os parâmetros fornecidos. O método implementa as operações principais do algoritmo CRO, como síntese, colisão intermolecular ineficaz, decomposição e colisão.
//—————————————————————————————————————————————————————————————————————————————— void C_AO_CRO::Moving () { //---------------------------------------------------------------------------- if (!revision) { for (int i = 0; i < popSize; i++) { for (int c = 0; c < coords; c++) { Mparent [i].structure [c] = u.RNDfromCI (rangeMin [c], rangeMax [c]); // Random structure in the range from rangeMin to rangeMax Mparent [i].structure [c] = u.SeInDiSp (Mparent [i].structure [c], rangeMin [c], rangeMax [c], rangeStep [c]); a [i].c [c] = Mparent [i].structure [c]; } } return; } //---------------------------------------------------------------------------- double minKE = DBL_MAX; for (int i = 0; i < popSize; i++) { if (Mparent [i].f < minKE) minKE = Mparent [i].f; } for (int i = 0; i < popSize; i++) { Mparent [i].KE = u.Scale (Mparent [i].f, minKE, fB, 0.0, 1.0); } //---------------------------------------------------------------------------- int molCNT = 0; while (!IsStopped ()) { if (u.RNDprobab () < moleColl) { // Select two random molecules M1 and M2 int index1 = u.RNDminusOne (popSize); int index2 = u.RNDminusOne (popSize); // If KE ≤ β: if (Mparent [index1].KE >= beta && Mparent [index2].KE >= beta) { // Perform Synthesis if (!Synthesis (index1, index2, molCNT)) break; } else { // Perform Intermolecular Inefficient Collision if (!InterMolInefColl (index1, index2, molCNT)) break; } } else { // Select a random molecule M int index = u.RNDminusOne (popSize); // If NumHit > α: if (Mparent [index].NumHit > alpha) { // Perform Decomposition if (!Decomposition (index, molCNT)) break; } else { // Perform Collision if (!InefCollision (index, molCNT)) break; } } } for (int i = 0; i < popSize; i++) { ArrayCopy (a [i].c, Mfilial [i].structure); } } //——————————————————————————————————————————————————————————————————————————————
O método "Revision" da classe "C_AO_CRO" é utilizado para atualizar a melhor solução global e os estados das moléculas na população parental através da execução dos operadores químicos pós-reação. As ações realizadas por este método:
1. Atualização da melhor solução global. No laço "for", o método percorre todas as moléculas. Se o valor da função "f" da molécula atual for superior ao valor atual "fB", então "fB" é atualizado, e o array de coordenadas da molécula atual é copiado para o array "cB".
2. Se "revision" for igual a "false", então, para cada molécula na população "Mparent", o valor "f" é ajustado para ser igual ao valor "f" do array "a". Em seguida, "revision" é definido como "true" e o método finaliza sua execução. Nesta etapa, é importante obter os valores de adaptabilidade das moléculas parentais, para que nas épocas subsequentes possam ser usados os operadores químicos que dependem da energia cinética (o valor da função de aptidão normalizado para o intervalo de 0.0 a 1.0).
3. Se "revision" não for igual a "false", então, para cada molécula na população "Mfilial", o valor "f" é ajustado para ser igual ao valor "f" do array "a". Depois, dependendo do tipo de reação "rType" em que a molécula esteve envolvida, o método correspondente "PostSynthesis", "PostInterMolInefColl", "PostDecomposition", ou "PostInefCollision" é chamado.
//—————————————————————————————————————————————————————————————————————————————— void C_AO_CRO::Revision () { //---------------------------------------------------------------------------- int ind = -1; for (int i = 0; i < popSize; i++) { if (a [i].f > fB) { fB = a [i].f; ind = i; } } if (ind != -1) ArrayCopy (cB, a [ind].c, 0, 0, WHOLE_ARRAY); //---------------------------------------------------------------------------- if (!revision) { for (int i = 0; i < popSize; i++) { for (int c = 0; c < coords; c++) { Mparent [i].f = a [i].f; } } revision = true; return; } //---------------------------------------------------------------------------- for (int i = 0; i < popSize; i++) { for (int c = 0; c < coords; c++) { Mfilial [i].f = a [i].f; } switch (Mfilial [i].rType) { case synthesis: PostSynthesis (Mfilial [i]); break; case interMolecularInefColl: PostInterMolInefColl (Mfilial [i]); break; case decomposition: PostDecomposition (Mfilial [i]); break; case inefCollision: PostInefCollision (Mfilial [i]); break; } } } //——————————————————————————————————————————————————————————————————————————————
3. Resultados dos testes
O algoritmo CRO (Chemical Reaction Optimisation) foi testado em funções de teste Hilly, Forest e Megacity. Em cada caso, foram realizadas dez execuções das funções para cada tipo de paisagem (5, 25 e 500 funções), e os resultados de otimização foram obtidos.
CRO|Chemical Reaction Optimisation|50.0|0.9|200.0|0.01|0.5|
=============================
5 Hilly's; Func runs: 10000; result: 0.9462894520167225
25 Hilly's; Func runs: 10000; result: 0.6611186250435438
500 Hilly's; Func runs: 10000; result: 0.2985263035668822
=============================
5 Forest's; Func runs: 10000; result: 0.8790568514481787
25 Forest's; Func runs: 10000; result: 0.584216839762206
500 Forest's; Func runs: 10000; result: 0.2114595696419046
=============================
5 Megacity's; Func runs: 10000; result: 0.7584615384615384
25 Megacity's; Func runs: 10000; result: 0.4264615384615384
500 Megacity's; Func runs: 10000; result: 0.12686153846153955
=============================
All score: 4.89245 (54.36%)
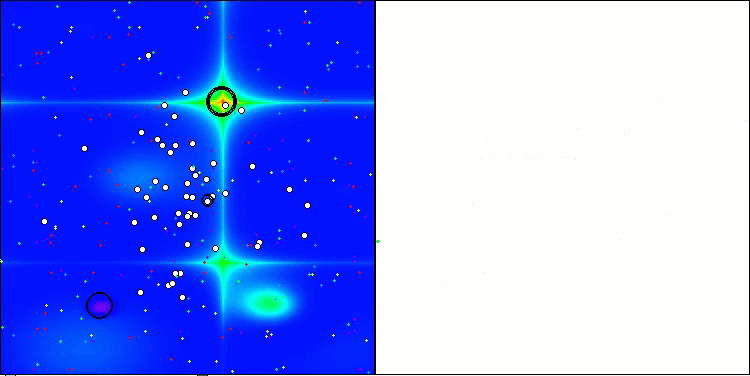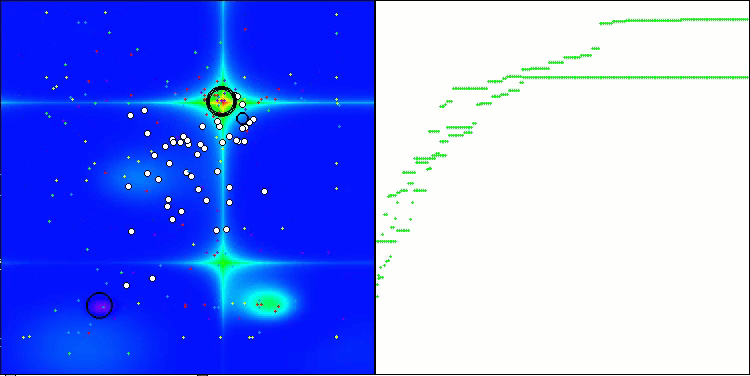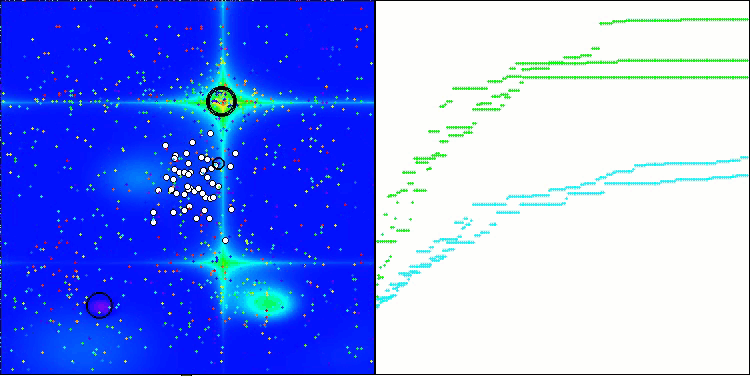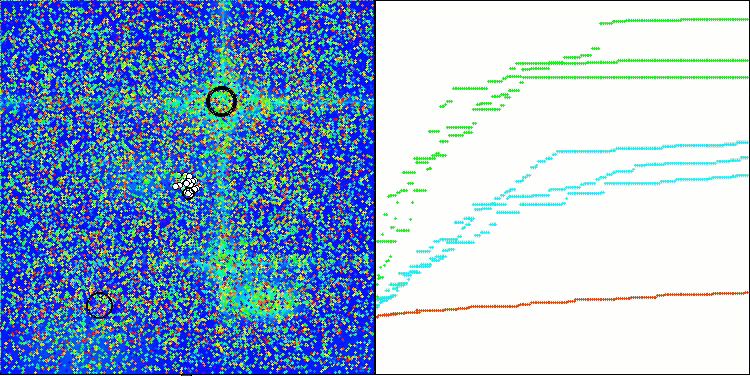
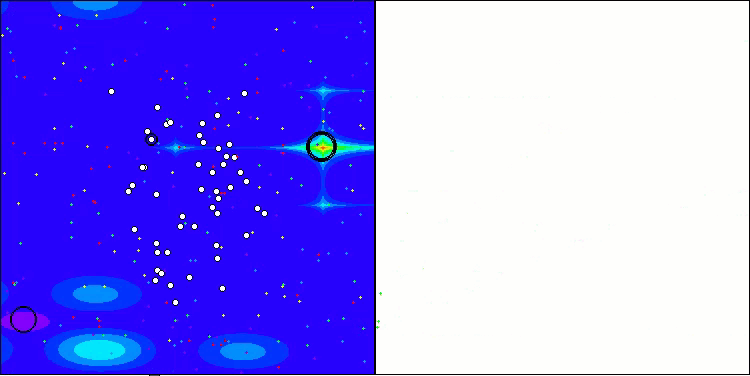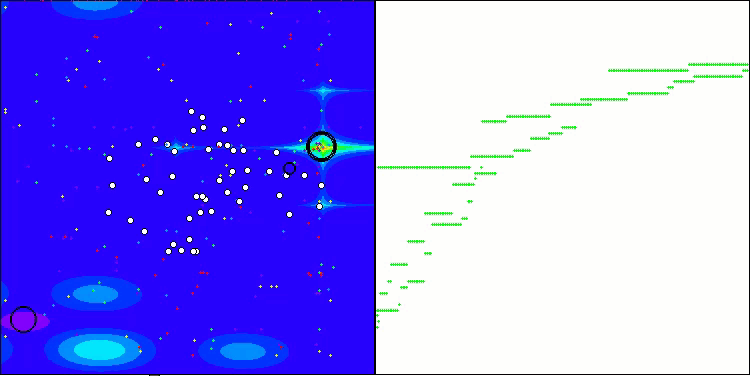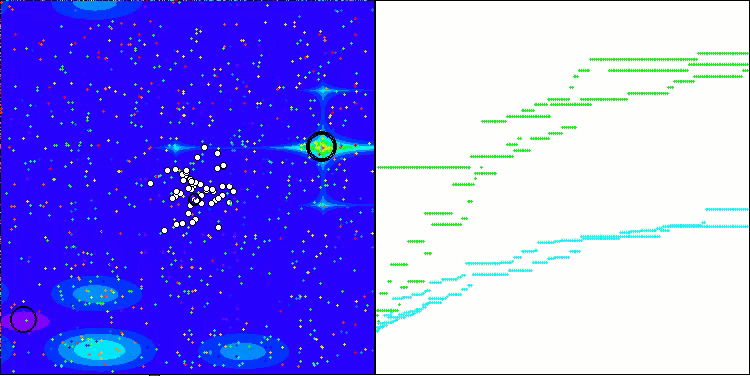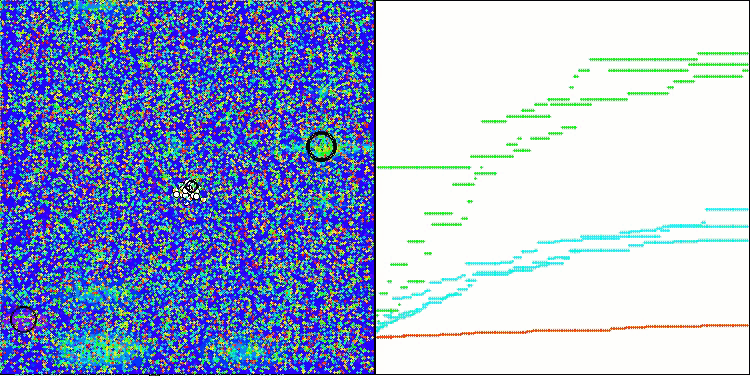
A visualização do funcionamento da bancada de testes com o algoritmo CRO demonstra características interessantes desse algoritmo. Apesar de o CRO, em alguns casos, poder ficar preso, como evidenciado por longas seções planas nos gráficos de progressão, ele ainda apresenta resultados gerais satisfatórios.
Um dos aspectos notáveis do funcionamento do CRO é o movimento das "moléculas" na área de busca. À primeira vista, esse movimento parece caótico e lembra o movimento browniano. No entanto, apesar da aparente aleatoriedade, as "moléculas" conseguem encontrar a zona de ótimo global. Isso evidencia a natureza complexa e engenhosa do algoritmo CRO, que utiliza princípios de reações químicas para resolver problemas de otimização.
No geral, o algoritmo CRO é uma ferramenta poderosa de otimização, capaz de lidar com diversas tarefas, apesar de algumas dificuldades. Suas propriedades únicas e a capacidade de encontrar ótimos globais fazem dele um recurso valioso na área de otimização.
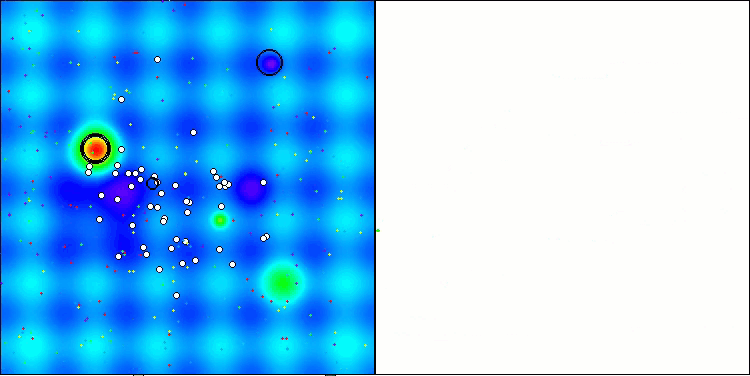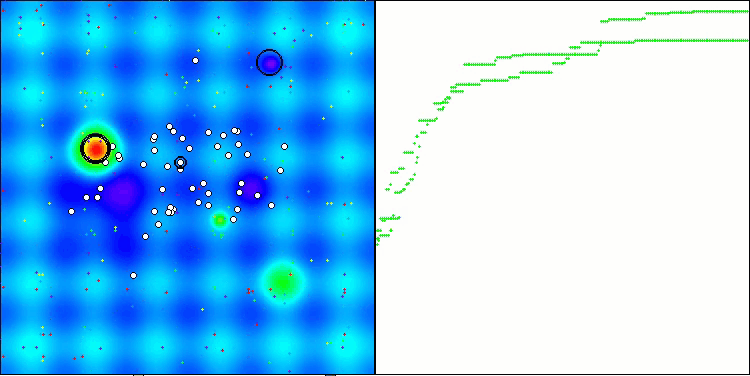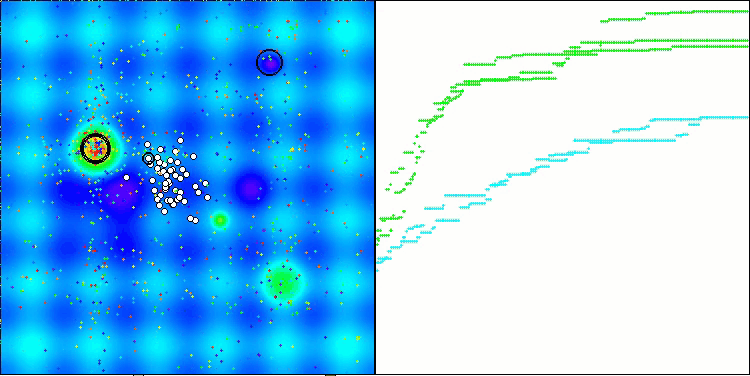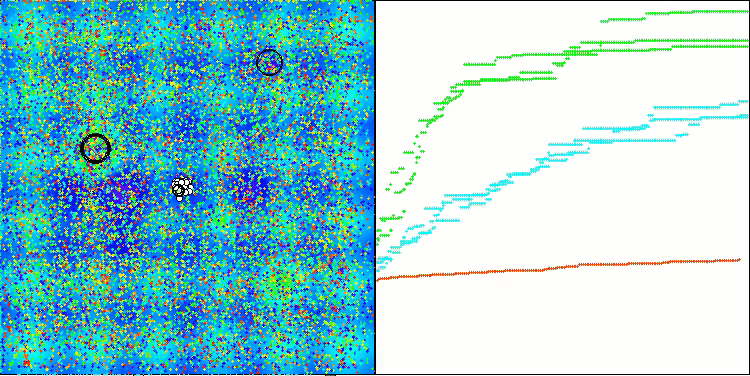
CRO na função de teste Hilly.
CRO na função de teste Forest.
CRO na função de teste Megacity.
| № | AO | Description | Hilly | Hilly final | Forest | Forest final | Megacity (discrete) | Megacity final | Final result | % of MAX | ||||||
| 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | ||||||||
| 1 | BGA | binary genetic algorithm | 0,99989 | 0,99518 | 0,42835 | 2,42341 | 0,96153 | 0,96181 | 0,32027 | 2,24360 | 0,91385 | 0,95908 | 0,24220 | 2,11512 | 6,782 | 75,36 |
| 2 | CLA | code lock algorithm | 0,95345 | 0,87107 | 0,37590 | 2,20042 | 0,98942 | 0,91709 | 0,31642 | 2,22294 | 0,79692 | 0,69385 | 0,19303 | 1,68380 | 6,107 | 67,86 |
| 3 | (P+O)ES | (P+O) evolution strategies | 0,92256 | 0,88101 | 0,40021 | 2,20379 | 0,97750 | 0,87490 | 0,31945 | 2,17185 | 0,67385 | 0,62985 | 0,18634 | 1,49003 | 5,866 | 65,17 |
| 4 | CTA | comet tail algorithm | 0,95346 | 0,86319 | 0,27770 | 2,09435 | 0,99794 | 0,85740 | 0,33949 | 2,19484 | 0,88769 | 0,56431 | 0,10512 | 1,55712 | 5,846 | 64,96 |
| 5 | SDSm | stochastic diffusion search M | 0,93066 | 0,85445 | 0,39476 | 2,17988 | 0,99983 | 0,89244 | 0,19619 | 2,08846 | 0,72333 | 0,61100 | 0,10670 | 1,44103 | 5,709 | 63,44 |
| 6 | ESG | evolution of social groups | 0,99906 | 0,79654 | 0,35056 | 2,14616 | 1,00000 | 0,82863 | 0,13102 | 1,95965 | 0,82333 | 0,55300 | 0,04725 | 1,42358 | 5,529 | 61,44 |
| 7 | SIA | simulated isotropic annealing | 0,95784 | 0,84264 | 0,41465 | 2,21513 | 0,98239 | 0,79586 | 0,20507 | 1,98332 | 0,68667 | 0,49300 | 0,09053 | 1,27020 | 5,469 | 60,76 |
| 8 | ACS | artificial cooperative search | 0,75547 | 0,74744 | 0,30407 | 1,80698 | 1,00000 | 0,88861 | 0,22413 | 2,11274 | 0,69077 | 0,48185 | 0,13322 | 1,30583 | 5,226 | 58,06 |
| 9 | TSEA | turtle shell evolution algorithm | 0,96798 | 0,64480 | 0,29672 | 1,90949 | 0,99449 | 0,61981 | 0,22708 | 1,84139 | 0,69077 | 0,42646 | 0,13598 | 1,25322 | 5,004 | 55,60 |
| 10 | DE | differential evolution | 0,95044 | 0,61674 | 0,30308 | 1,87026 | 0,95317 | 0,78896 | 0,16652 | 1,90865 | 0,78667 | 0,36033 | 0,02953 | 1,17653 | 4,955 | 55,06 |
| 11 | CRO | chemical reaction optimisation | 0,94629 | 0,66112 | 0,29853 | 1,90593 | 0,87906 | 0,58422 | 0,21146 | 1,67473 | 0,75846 | 0,42646 | 0,12686 | 1,31178 | 4,892 | 54,36 |
| 12 | BSA | bird swarm algorithm | 0,89306 | 0,64900 | 0,26250 | 1,80455 | 0,92420 | 0,71121 | 0,24939 | 1,88479 | 0,69385 | 0,32615 | 0,10012 | 1,12012 | 4,809 | 53,44 |
| 13 | HS | harmony search | 0,86509 | 0,68782 | 0,32527 | 1,87818 | 0,99999 | 0,68002 | 0,09590 | 1,77592 | 0,62000 | 0,42267 | 0,05458 | 1,09725 | 4,751 | 52,79 |
| 14 | SSG | saplings sowing and growing | 0,77839 | 0,64925 | 0,39543 | 1,82308 | 0,85973 | 0,62467 | 0,17429 | 1,65869 | 0,64667 | 0,44133 | 0,10598 | 1,19398 | 4,676 | 51,95 |
| 15 | (PO)ES | (PO) evolution strategies | 0,79025 | 0,62647 | 0,42935 | 1,84606 | 0,87616 | 0,60943 | 0,19591 | 1,68151 | 0,59000 | 0,37933 | 0,11322 | 1,08255 | 4,610 | 51,22 |
| 16 | BSO | brain storm optimization | 0,93736 | 0,57616 | 0,29688 | 1,81041 | 0,93131 | 0,55866 | 0,23537 | 1,72534 | 0,55231 | 0,29077 | 0,11914 | 0,96222 | 4,498 | 49,98 |
| 17 | WOAm | wale optimization algorithm M | 0,84521 | 0,56298 | 0,26263 | 1,67081 | 0,93100 | 0,52278 | 0,16365 | 1,61743 | 0,66308 | 0,41138 | 0,11357 | 1,18803 | 4,476 | 49,74 |
| 18 | ACOm | ant colony optimization M | 0,88190 | 0,66127 | 0,30377 | 1,84693 | 0,85873 | 0,58680 | 0,15051 | 1,59604 | 0,59667 | 0,37333 | 0,02472 | 0,99472 | 4,438 | 49,31 |
| 19 | BFO-GA | bacterial foraging optimization - ga | 0,89150 | 0,55111 | 0,31529 | 1,75790 | 0,96982 | 0,39612 | 0,06305 | 1,42899 | 0,72667 | 0,27500 | 0,03525 | 1,03692 | 4,224 | 46,93 |
| 20 | MEC | mind evolutionary computation | 0,69533 | 0,53376 | 0,32661 | 1,55569 | 0,72464 | 0,33036 | 0,07198 | 1,12698 | 0,52500 | 0,22000 | 0,04198 | 0,78698 | 3,470 | 38,55 |
| 21 | IWO | invasive weed optimization | 0,72679 | 0,52256 | 0,33123 | 1,58058 | 0,70756 | 0,33955 | 0,07484 | 1,12196 | 0,42333 | 0,23067 | 0,04617 | 0,70017 | 3,403 | 37,81 |
| 22 | Micro-AIS | micro artificial immune system | 0,79547 | 0,51922 | 0,30861 | 1,62330 | 0,72956 | 0,36879 | 0,09398 | 1,19233 | 0,37667 | 0,15867 | 0,02802 | 0,56335 | 3,379 | 37,54 |
| 23 | COAm | cuckoo optimization algorithm M | 0,75820 | 0,48652 | 0,31369 | 1,55841 | 0,74054 | 0,28051 | 0,05599 | 1,07704 | 0,50500 | 0,17467 | 0,03380 | 0,71347 | 3,349 | 37,21 |
| 24 | SDOm | spiral dynamics optimization M | 0,74601 | 0,44623 | 0,29687 | 1,48912 | 0,70204 | 0,34678 | 0,10944 | 1,15826 | 0,42833 | 0,16767 | 0,03663 | 0,63263 | 3,280 | 36,44 |
| 25 | NMm | Nelder-Mead method M | 0,73807 | 0,50598 | 0,31342 | 1,55747 | 0,63674 | 0,28302 | 0,08221 | 1,00197 | 0,44667 | 0,18667 | 0,04028 | 0,67362 | 3,233 | 35,92 |
| 26 | FAm | firefly algorithm M | 0,58634 | 0,47228 | 0,32276 | 1,38138 | 0,68467 | 0,37439 | 0,10908 | 1,16814 | 0,28667 | 0,16467 | 0,04722 | 0,49855 | 3,048 | 33,87 |
| 27 | GSA | gravitational search algorithm | 0,64757 | 0,49197 | 0,30062 | 1,44016 | 0,53962 | 0,36353 | 0,09945 | 1,00260 | 0,32667 | 0,12200 | 0,01917 | 0,46783 | 2,911 | 32,34 |
| 28 | BFO | bacterial foraging optimization | 0,61171 | 0,43270 | 0,31318 | 1,35759 | 0,54410 | 0,21511 | 0,05676 | 0,81597 | 0,42167 | 0,13800 | 0,03195 | 0,59162 | 2,765 | 30,72 |
| 29 | ABC | artificial bee colony | 0,63377 | 0,42402 | 0,30892 | 1,36671 | 0,55103 | 0,21874 | 0,05623 | 0,82600 | 0,34000 | 0,14200 | 0,03102 | 0,51302 | 2,706 | 30,06 |
| 30 | BA | bat algorithm | 0,59761 | 0,45911 | 0,35242 | 1,40915 | 0,40321 | 0,19313 | 0,07175 | 0,66810 | 0,21000 | 0,10100 | 0,03517 | 0,34617 | 2,423 | 26,93 |
| 31 | SA | simulated annealing | 0,55787 | 0,42177 | 0,31549 | 1,29513 | 0,34998 | 0,15259 | 0,05023 | 0,55280 | 0,31167 | 0,10033 | 0,02883 | 0,44083 | 2,289 | 25,43 |
| 32 | IWDm | intelligent water drops M | 0,54501 | 0,37897 | 0,30124 | 1,22522 | 0,46104 | 0,14704 | 0,04369 | 0,65177 | 0,25833 | 0,09700 | 0,02308 | 0,37842 | 2,255 | 25,06 |
| 33 | PSO | particle swarm optimisation | 0,59726 | 0,36923 | 0,29928 | 1,26577 | 0,37237 | 0,16324 | 0,07010 | 0,60572 | 0,25667 | 0,08000 | 0,02157 | 0,35823 | 2,230 | 24,77 |
| 34 | Boids | boids algorithm | 0,43340 | 0,30581 | 0,25425 | 0,99346 | 0,35718 | 0,20160 | 0,15708 | 0,71586 | 0,27846 | 0,14277 | 0,09834 | 0,51957 | 2,229 | 24,77 |
| 35 | MA | monkey algorithm | 0,59107 | 0,42681 | 0,31816 | 1,33604 | 0,31138 | 0,14069 | 0,06612 | 0,51819 | 0,22833 | 0,08567 | 0,02790 | 0,34190 | 2,196 | 24,40 |
| 36 | SFL | shuffled frog-leaping | 0,53925 | 0,35816 | 0,29809 | 1,19551 | 0,37141 | 0,11427 | 0,04051 | 0,52618 | 0,27167 | 0,08667 | 0,02402 | 0,38235 | 2,104 | 23,38 |
| 37 | FSS | fish school search | 0,55669 | 0,39992 | 0,31172 | 1,26833 | 0,31009 | 0,11889 | 0,04569 | 0,47467 | 0,21167 | 0,07633 | 0,02488 | 0,31288 | 2,056 | 22,84 |
| 38 | RND | random | 0,52033 | 0,36068 | 0,30133 | 1,18234 | 0,31335 | 0,11787 | 0,04354 | 0,47476 | 0,25333 | 0,07933 | 0,02382 | 0,35648 | 2,014 | 22,37 |
| 39 | GWO | grey wolf optimizer | 0,59169 | 0,36561 | 0,29595 | 1,25326 | 0,24499 | 0,09047 | 0,03612 | 0,37158 | 0,27667 | 0,08567 | 0,02170 | 0,38403 | 2,009 | 22,32 |
| 40 | CSS | charged system search | 0,44252 | 0,35454 | 0,35201 | 1,14907 | 0,24140 | 0,11345 | 0,06814 | 0,42299 | 0,18333 | 0,06300 | 0,02322 | 0,26955 | 1,842 | 20,46 |
| 41 | EM | electroMagnetism-like algorithm | 0,46250 | 0,34594 | 0,32285 | 1,13129 | 0,21245 | 0,09783 | 0,10057 | 0,41085 | 0,15667 | 0,06033 | 0,02712 | 0,24412 | 1,786 | 19,85 |
Conclusões
Com base na tabela fornecida e nos resultados, é possível tirar as seguintes conclusões sobre o desempenho do algoritmo Chemical Reaction Optimization (CRO):
1. O CRO apresenta resultados excelentes na função de teste Hilly. Com 5 parâmetros, o resultado foi cerca de 0,95, com 25 parâmetros - aproximadamente 0,66, e com 500 parâmetros - cerca de 0,30. Isso indica que o CRO é eficaz em funções suaves, especialmente com um número menor de parâmetros.
2. O CRO também apresenta bom desempenho na função de teste Forest. Com 5 parâmetros, o resultado foi em torno de 0,88, com 25 parâmetros - aproximadamente 0,58, e com 500 parâmetros - cerca de 0,21. Isso sugere que o CRO também é eficiente em funções com extremos "agudos", embora encontre algumas dificuldades em localizar ótimos pontuais.
3. Na função de teste Megacity, o CRO continua apresentando bom desempenho. Com 5 parâmetros, o resultado foi em torno de 0,76, com 25 parâmetros - cerca de 0,43, e com 500 parâmetros - aproximadamente 0,13. Isso mostra que o CRO é eficaz nessa função discreta, com resultados uniformemente "verdes" em comparação com outros algoritmos, mesmo aqueles que ocupam posições mais altas na tabela.
Com base na tabela apresentada, o algoritmo Chemical Reaction Optimization (CRO) demonstra resultados sólidos em comparação com outros métodos de otimização. Particularmente, nas funções Hilly, Forest e Megacity, o CRO se mostra competitivo, especialmente quando o número de parâmetros é menor.
O CRO ficou em 11º lugar no ranking. Com base na gradação de cores da tabela abaixo (onde o verde escuro indica os melhores resultados), pode-se afirmar que o CRO, de modo geral, apresenta um desempenho bom e estável (com coloração uniforme e consistente). Apenas na função "Hilly" com 1000 parâmetros, os resultados parecem um pouco mais fracos.
O algoritmo Chemical Reaction Optimization (CRO) mostrou ser uma abordagem promissora para otimização. Ele utiliza duas populações de agentes (na minha implementação) que interagem entre si para garantir diversidade e evitar que fiquem presos em ótimos locais. Uma das características distintivas do algoritmo é o uso de operadores especiais, semelhantes às reações químicas, decomposição, síntese e outros.
No geral, o algoritmo CRO se apresenta como um método de otimização promissor, destacando-se por sua originalidade e capacidade de alcançar resultados elevados em diversas tarefas de otimização.
É importante destacar que a escolha do algoritmo de otimização deve ser baseada na tarefa específica e nos requisitos de desempenho, e nossa tabela de classificação pode ajudar nesse processo. Graças à adaptação da implementação original do algoritmo CRO para herdar da classe C_AO, que adotamos para algoritmos populacionais, esse interessante algoritmo pode ser aplicado de forma geral a problemas de otimização.
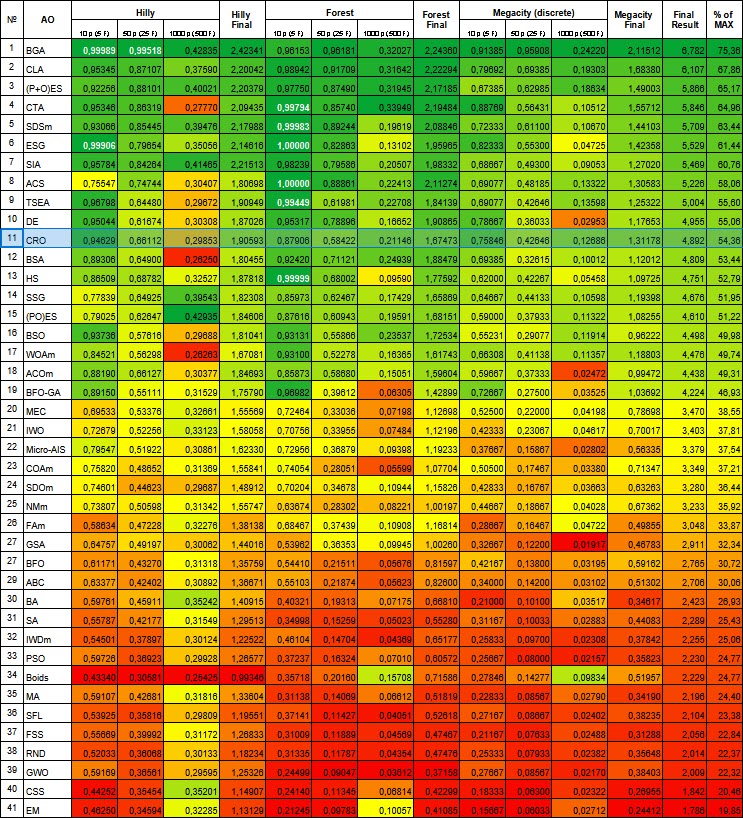
Figura 1. Graduação de cores dos algoritmos nos respectivos testes. Os resultados maiores ou iguais a 0.99 são destacados em branco.
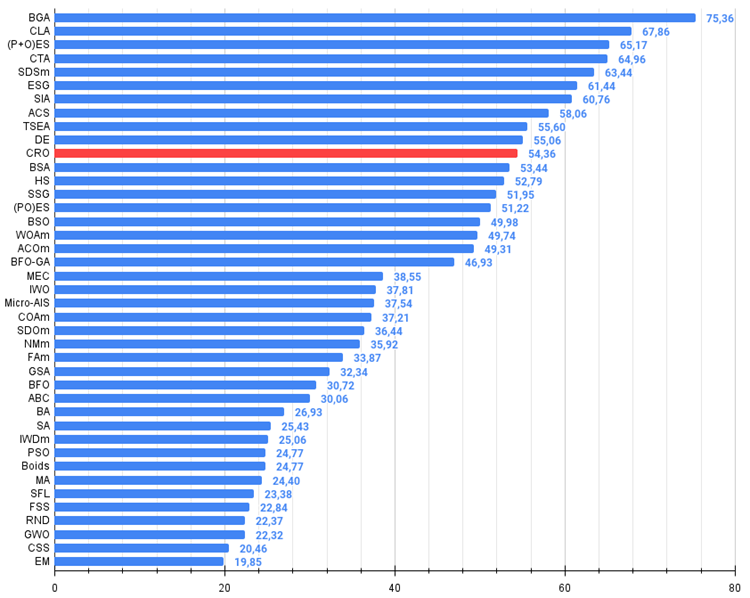
Figura 2. Histograma dos resultados dos testes dos algoritmos (em uma escala de 0 a 100, onde valores mais altos são melhores,
onde 100 é o resultado teórico máximo possível; o script para cálculo da tabela de classificação está incluído no arquivo anexado)
Prós e contras gerais do algoritmo de otimização por reações químicas (CRO):
Prós:
- Boa convergência em vários tipos de funções.
- Muito rápido, apesar da arquitetura complexa.
- Boa escalabilidade.
Contras:
- Às vezes, fica preso em extremos locais.
Um arquivo com as versões atualizadas dos códigos dos algoritmos está anexado ao artigo. O autor do artigo não se responsabiliza pela precisão absoluta na descrição dos algoritmos canônicos, uma vez que muitas alterações foram feitas para melhorar as capacidades de busca. As conclusões e avaliações apresentadas no artigo são baseadas nos resultados dos experimentos realizados.
Traduzido do russo pela MetaQuotes Ltd.
Artigo original: https://www.mql5.com/ru/articles/15080
Aviso: Todos os direitos sobre esses materiais pertencem à MetaQuotes Ltd. É proibida a reimpressão total ou parcial.
Esse artigo foi escrito por um usuário do site e reflete seu ponto de vista pessoal. A MetaQuotes Ltd. não se responsabiliza pela precisão das informações apresentadas nem pelas possíveis consequências decorrentes do uso das soluções, estratégias ou recomendações descritas.
 Redes neurais de maneira fácil (Parte 95): Redução do consumo de memória em modelos Transformer
Redes neurais de maneira fácil (Parte 95): Redução do consumo de memória em modelos Transformer
 Algoritmo de otimização por reações químicas — Chemical Reaction Optimization, CRO (Parte I): A química dos processos na otimização
Algoritmo de otimização por reações químicas — Chemical Reaction Optimization, CRO (Parte I): A química dos processos na otimização
 Desenvolvimento de robô em Python e MQL5 (Parte 3): Implementação do algoritmo de negociação baseado em modelo
Desenvolvimento de robô em Python e MQL5 (Parte 3): Implementação do algoritmo de negociação baseado em modelo
 Desenvolvendo um sistema de Replay (Parte 74): Um novo Chart Trade (I)
Desenvolvendo um sistema de Replay (Parte 74): Um novo Chart Trade (I)
- Aplicativos de negociação gratuitos
- 8 000+ sinais para cópia
- Notícias econômicas para análise dos mercados financeiros
Você concorda com a política do site e com os termos de uso