Популяционные алгоритмы оптимизации: Бинарный генетический алгоритм (Binary Genetic Algorithm, BGA). Часть II
Содержание:
1. Введение
2. Описание алгоритма
3. Результаты тестов
1. Введение
В предыдущей статье мы познакомились со всеми важными основными понятиями и методами, используемых не только в генетических алгоритмах, но, так или иначе, в любых популяционных алгоритмах оптимизации. Теперь, основываясь на этих знаниях, мы можем подробно рассмотреть основной предмет текущего "двухтомника" - бинарный генетический алгоритм (BGA). Начиная с общей информации перейдем к коду этой, во всех смыслах, замечательной стратегии поиска, и в завершении - к результатам тестов.
Генетический алгоритм (GA) относится к эволюционным алгоритмам, включающим в себя различные подходы, такие как генетические алгоритмы, генетическое программирование, эволюционные стратегии и другие. Они основаны на идеях эволюции и наследственности, где решения представляются в виде популяции, а генетические операторы, такие как скрещивание и мутация, применяются для создания новых поколений решений.
Генетические алгоритмы используют принципы естественного отбора и генетики для решения оптимизационных задач. Рассматриваемый в статье бинарный генетический алгоритм (BGA) появился первым среди всех видов генетических алгоритмов. Таким образом, BGA относится к классу эволюционных алгоритмов и является конкретным вариантом генетического алгоритма, который использует бинарное представление данных.
Работы над созданием генетических алгоритмов начались ещё в середине 20-го века. Одним из основоположников является Джон Холланд, который в 1975 году опубликовал книгу "Адаптивные системы искусственного интеллекта" (Adaptation in Natural and Artificial Systems), где впервые представил генетический алгоритм как целое направление подхода к решениям задач оптимизации.
Разработка генетического бинарного алгоритма была вдохновлена несколькими факторами и идеями. Основные из них:
- Естественный отбор и принципы эволюции: BGA основан на принципах естественного отбора и эволюции, которые были предложены Чарльзом Дарвином. Идея заключается в том, что в популяции существует разнообразие решений, и те, которые лучше приспособлены к среде, имеют больше шансов выжить и передать свои характеристики следующему поколению.
- Генетика и наследственность: BGA также использует концепции генетики, такие как ген, хромосома и наследственность. Решения в BGA представлены в виде бинарных строк, где отдельные группы битов представляют конкретные гены, а ген в свою очередь, представляет оптимизируемый параметр. Генетические операторы, такие как скрещивание и мутация, применяются к бинарным строкам для создания новых поколений решений.
В целом, разработка BGA была результатом сочетания идей из области эволюционных алгоритмов, генетики и оптимизации. Он был создан для решения оптимизационных задач, используя принципы естественного отбора и генетики, а его развитие продолжается и в настоящее время, создано огромное количество вариантов GA, а так же широкое использование идей и подходов в генетических алгоритмах в составе гибридных, в том числе и очень сложных, комплексных алгоритмов.
Генетический бинарный алгоритм, BGA, использует бинарное представление данных. Это значит, что каждый индивид (решение) представляется в виде строки из битов (0 и 1). Генетические операторы, такие как скрещивание и мутация, применяются к битовым строкам для создания новых поколений решений.
Интересный факт: генетические алгоритмы, включая BGA, могут использоваться для создания и улучшения искусственного интеллекта. Например, они могут применяться для эволюции нейронных сетей, позволяя создать более эффективные модели машинного обучения.
Генетические алгоритмы в целом и BGA в частности, представляют собой мощный инструмент для решения сложных оптимизационных задач, где традиционные методы могут быть недостаточно эффективными из-за отсутствия аналитического решения. В MetaTrader 5 используется бинарный ГА и поэтому вдвойне интересно изучить принципы работы этого замечательного алгоритма.
2. Описание алгоритма
Бинарный генетический алгоритм включает в себя следующие шаги:
- Инициализация популяции: создать начальную популяцию, состоящую из хромосом со случайными бинарными значениями.
- Оценка приспособленности: оценить приспособленность каждой особи (хромосомы) в дочерней популяции.
- Селекция: выбрать родителей для создания потомства методом рулетки.
- Кроссовер: разбить родительские хромосомы на участки и создать новые дочерние хромосомы с участками обоих родителей.
- Инверсия: разбить хромосому дочерней особи в случайно выбранной точке и поменять местами полученные участки.
- Мутация: случайным образом изменить биты в генах потомства с заданной вероятностью мутации.
- Оценка приспособленности потомства: оценить приспособленность каждого нового потомка.
- Формирование новой популяции: поместить популяцию потомков в конец общей популяции и выполнить сортировку по значению приспособленности.
- Критерий остановки: повторять процесс с п.3 до достижения критерия остановки.
Для обеспечения работы BGA только с положительными числами, для упрощения операций с бинарными строками и повышения скорости вычислений, будем использовать расстояние между "max" и "min" оптимизируемых параметров. Полученные таким образом положительные значения расстояний представим в виде бинарного кода Грея разместим последовательно в один общий массив символов хромосомы так, как представлено на рисунке 1. Нужно отметить, что точки разрыва хромосом при выполнении метода кроссовера располагаются в случайных местах хромосомы и необязательно в точках стыковки генов, точки разрыва могут быть и внутри пространства гена.
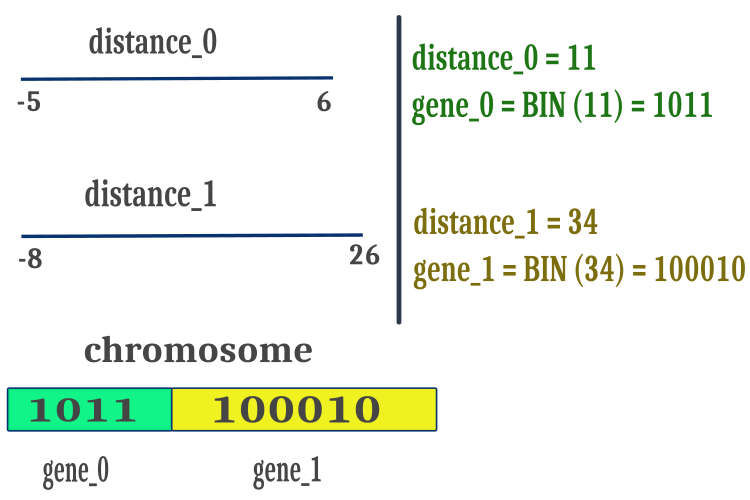
Рисунок 1. Размещение признаков особи (оптимизируемых параметров) в хромосоме.
Внешних параметров у генетического алгоритма существует значительное количество, поэтому целесообразно рассмотреть их более подробно. По умолчанию параметры практически полностью соответствуют рекомендациям многих авторов научных публикаций. Они были проверены мной и подобраны таким образом, чтобы обеспечить максимальную эффективность в большинстве типов задач. Однако, отклонение от этих параметров в любом направлении может привести к достижению 100% сходимости на тестах с 10 или даже 50 оптимизируемыми параметрами на отдельных функциях, но в то же время значительно снизить эффективность на других задачах. Поэтому параметры по умолчанию являются оптимальными для использования в большинстве практически значимых случаев.
- Population_P (размер популяции = 50): параметр определяет количество дочерних особей в каждом поколении популяции. Такой размер популяции используется в большинстве алгоритмов, рассматриваемых в тестах, и обеспечивает баланс между разнообразием решений и скоростью сходимости.
- ParentPopulation_P (размер родительской популяции = 50): параметр определяет количество родительских особей, выбранных для размножения и создания следующего поколения. Уменьшение значения этого параметра улучшает сходимость на гладких функциях (увеличивается точность), увеличение значения - на дискретных (повышается разнообразие генетического материала).
- CrossoverProbab_P (вероятность скрещивания = 1.0): параметр определяет вероятность выполнения операции скрещивания между двумя выбранными родительскими особями. Высокая вероятность скрещивания увеличивает комбинаторные возможности алгоритма, уменьшение значения - увеличивает скорость сходимости, но повышает вероятность застревания.
- CrossoverPoints_P (количество точек скрещивания = 3): параметр определяет количество точек, в которых происходит скрещивание между двумя родительскими хромосомами. Увеличение точек приводит к более интенсивному перемешиванию параметров между собой и в пределе сводится к случайному поведению алгоритма.
- MutationProbab_P (вероятность мутации = 0.001): параметр определяет вероятность мутации каждого бита в генотипе хромосомы. Мутация позволяет вносить случайные изменения в генетический материал и добавляет новые решения в популяцию. Низкая вероятность увеличивает скорость сходимости алгоритма, но уменьшает разнообразие, а слишком высокая вероятность приводит к потере полезной информации.
- InversionProbab_P (вероятность инверсии = 0.7): параметр определяет вероятность выполнения операции инверсии в хромосоме. Высокая вероятность инверсии увеличивает разнообразие генетического материала, но слишком высокая вероятность приводит к случайному поведению алгоритма.
- DoubleDigitsInChromo_P (количество десятичных знаков в гене): параметр определяет количество разрядов после запятой вещественного числа (оптимизируемого параметра), представленного в. бинарном виде в хромосоме. Увеличение значения приводит к увеличению сложности вычислений и увеличении времени оптимизации (без применения бинарного вида напрямую в решении задачи не имеет смысла устанавливать больше 16 - на выходе при конвертации в double разряды будут потеряны).
Переходим к рассмотрению кода.
При инициализации агентов необходимо определить количество битов в бинарном представлении оптимизируемого параметра. Допустим, нам требуется рассмотреть пять знаков после запятой, что соответствует определенной длине кода Грея. Однако в этом коде может быть закодировано число, превышающее требуемое. Поэтому нам необходимо определить максимальное вещественное число, которое может быть закодировано в бинарном виде. В дальнейшем мы сможем масштабировать закодированное число до требуемого диапазона оптимизируемого параметра на выходе. Для дробной части вещественного числа мы используем заданное количество разрядов (указанное в параметрах), а для целой части - столько сколько требуется в бинарном виде.
Например, если входной параметр функции "digitsInGrayCode" равно 3, то функция вернет максимальное значение типа "ulong" с использованием кода Грея для 3 битов, то есть 7 (111 в двоичной системе).
Для достижения этой цели - определения максимально возможного числа, которое может быть закодировано заданным количеством битов для дробной и целой части вещественного числа, мы будем использовать функцию "GetMaxDecimalFromGray".
//—————————————————————————————————————————————————————————————————————————————— //Calculation of the maximum possible ulong number using the Gray code for a given number of bits ulong GetMaxDecimalFromGray (int digitsInGrayCode) { ulong maxValue = 1; for (int i = 1; i < digitsInGrayCode; i++) { maxValue <<= 1; maxValue |= 1; } return maxValue; } //——————————————————————————————————————————————————————————————————————————————
Для представления каждого гена в хромосоме (положение гена в хромосоме) нам послужит структура "S_BinaryGene", которая содержит поля и методы для работы с генами в двоичном формате:
- "gene": массив символов, представляющий ген.
- "integPart": массив символов целой части гена.
- "fractPart": массив символов дробной части гена.
- "integGrayDigits": количество цифр в коде Грея для целой части гена.
- "fractGrayDigits": количество цифр в коде Грея для дробной части гена.
- "length": общая длина гена.
- "minDoubleRange": минимальное значение диапазона вещественных чисел.
- "maxDoubleRange": максимальное значение диапазона вещественных чисел.
- "maxDoubleNumber": максимальное вещественное число, которое может быть представлено с использованием гена.
- "doubleFract": значение для преобразования дробной части гена в вещественное число.
Методы структуры:
- "Init": инициализирует поля структуры. Принимает минимальное и максимальное значения диапазона вещественных чисел, а также количество цифр в дробной части гена. Внутри метода вычисляются значения для максимального вещественного числа, кодирующих частей гена с использованием кода Грея.
- "ToDouble": преобразует ген в вещественное число. Принимает массив символов кода Грея "chromo" (хромосома) и индекс начала гена "indChr". Метод производит чтение участка хромосомы и преобразует считанный ген в десятичное значение, а затем масштабирует его в заданный диапазон вещественных чисел.
- "Scale": выполняет масштабирование входного значения "In" из диапазона "InMIN" и "InMAX" в выходной диапазон "OutMIN" и "OutMAX". Это вспомогательный метод, который используется в "ToDouble".
//—————————————————————————————————————————————————————————————————————————————— struct S_BinaryGene { char gene []; char integPart []; char fractPart []; uint integGrayDigits; uint fractGrayDigits; uint length; double minDoubleRange; double maxDoubleRange; double maxDoubleNumber; double doubleFract; //---------------------------------------------------------------------------- void Init (double min, double max, int doubleDigitsInChromo) { doubleFract = pow (0.1, doubleDigitsInChromo); minDoubleRange = min; maxDoubleRange = max - min; ulong decInfr = 0; for (int i = 0; i < doubleDigitsInChromo; i++) { decInfr += 9 * (ulong)pow (10, i); } //---------------------------------------- DecimalToGray (decInfr, fractPart); ulong maxDecInFr = GetMaxDecimalFromGray (ArraySize (fractPart)); double maxDoubFr = maxDecInFr * doubleFract; //---------------------------------------- DecimalToGray ((ulong)maxDoubleRange, integPart); ulong maxDecInInteg = GetMaxDecimalFromGray (ArraySize (integPart)); double maxDoubInteg = (double)maxDecInInteg + maxDoubFr; maxDoubleNumber = maxDoubInteg; ArrayResize (gene, 0, 1000); integGrayDigits = ArraySize (integPart); fractGrayDigits = ArraySize (fractPart); length = integGrayDigits + fractGrayDigits; ArrayCopy (gene, integPart, 0, 0, WHOLE_ARRAY); ArrayCopy (gene, fractPart, ArraySize (gene), 0, WHOLE_ARRAY); } //---------------------------------------------------------------------------- double ToDouble (const char &chromo [], const int indChr) { double d; if(integGrayDigits > 0)d = (double)GrayToDecimal(chromo, indChr, indChr + integGrayDigits - 1); else d = 0.0; d +=(double)GrayToDecimal(chromo, indChr + integGrayDigits, indChr + integGrayDigits + fractGrayDigits - 1) * doubleFract; return minDoubleRange + Scale(d, 0.0, maxDoubleNumber, 0.0, maxDoubleRange); } //---------------------------------------------------------------------------- double Scale (double In, double InMIN, double InMAX, double OutMIN, double OutMAX) { if (OutMIN == OutMAX) return (OutMIN); if (InMIN == InMAX) return (double((OutMIN + OutMAX) / 2.0)); else { if (In < InMIN) return OutMIN; if (In > InMAX) return OutMAX; return (((In - InMIN) * (OutMAX - OutMIN) / (InMAX - InMIN)) + OutMIN); } } }; //——————————————————————————————————————————————————————————————————————————————
Для описания агента нам потребуется структура "S_Agent", которая представляет агента и содержит в себе помимо хромосомы следующие данные:
- "c": массив значений координат агента в виде вещественного числа.
- "f": значение приспособленности агента.
- "genes": массив структур "S_BinaryGene", описывающих положение каждого гена в хромосоме и правила конвертирования бинарного кода в вещественное число.
- "chromosome": массив символов хромосомы агента.
- "calculated": логическое значение, указывающее, были ли вычислены значения агента (поле присутствует, но в коде не задействовано).
Методы структуры:
- "Init": инициализирует поля структуры. Принимает количество координат "coords", массивы "min" и "max" с минимальными и максимальными значениями для каждого оптимизируемого параметра, а также "doubleDigitsInChromo" - количество разрядов вещественного числа в дробной части гена. Метод создает и инициализирует гены для каждой координаты, а также устанавливает начальное значение приспособленности и флаг "calculated".
- "ExtractGenes": извлекает значения генов из хромосомы и сохраняет их в массив "c", использует метод "ToDouble" из структуры "S_BinaryGene" для преобразования генов из хромосомы в вещественные числа.
//—————————————————————————————————————————————————————————————————————————————— struct S_Agent { void Init (const int coords, const double &min [], const double &max [], int doubleDigitsInChromo) { ArrayResize(c, coords); f = -DBL_MAX; ArrayResize(genes, coords); ArrayResize(chromosome, 0, 1000); for(int i = 0; i < coords; i++) { genes [i].Init(min [i], max [i], doubleDigitsInChromo); ArrayCopy(chromosome, genes [i].gene, ArraySize(chromosome), 0, WHOLE_ARRAY); } calculated = false; } void ExtractGenes () { uint pos = 0; for (int i = 0; i < ArraySize (genes); i++) { c [i] = genes [i].ToDouble (chromosome, pos); pos += genes [i].length; } } double c []; //coordinates double f; //fitness S_BinaryGene genes []; char chromosome []; bool calculated; }; //——————————————————————————————————————————————————————————————————————————————
Далее код представляет определение структуры "S_Roulette", которая представляет сегмент рулетки.
Поля структуры:
- "start": значение начальной точки сегмента рулетки.
- "end": значение конечной точки сегмента рулетки.
//—————————————————————————————————————————————————————————————————————————————— struct S_Roulette { double start; double end; }; //——————————————————————————————————————————————————————————————————————————————
Обьявим класс "C_AO_BGA", реализующий генетический алгоритм:
- "cB": массив значений лучших координат.
- "fB": значение приспособленности лучших координат.
- "a": массив структур "S_Agent", представляющих агентов.
- "rangeMax": массив максимальных значений поискового диапазона.
- "rangeMin": массив минимальных значений поискового диапазона.
- "rangeStep": массив значений, представляющий шаг поиска.
Методы класса:
- "Init": инициализирует поля класса. Принимает параметры: количество координат "coordsP", размер популяции "popSizeP", размер родительской популяции "parentPopSizeP", вероятность скрещивания "crossoverProbabP", количество точек скрещивания "crossoverPointsP", вероятность мутации "mutationProbabP", вероятность инверсии "inversionProbabP", количество десятичных знаков в гене "doubleDigitsInChromoP". Метод инициализирует внутренние переменные и массивы, необходимые для работы генетического алгоритма.
- "Moving": основной метод генетического алгоритма, выполняет операции с популяцией, такие как скрещивание, мутация, инверсия и оценку приспособленности.
- "Revision": метод выполняет ревизию популяции, сортируя агентов и выбирая лучших.
Приватные поля и методы класса используются для внутренней реализации генетического алгоритма, включая операции с рулеткой, масштабирование значений, сортировку и другие операции.
//—————————————————————————————————————————————————————————————————————————————— class C_AO_BGA { //---------------------------------------------------------------------------- public: double cB []; //best coordinates public: double fB; //FF of the best coordinates public: S_Agent a []; //agent public: double rangeMax []; //maximum search range public: double rangeMin []; //manimum search range public: double rangeStep []; //step search public: void Init (const int coordsP, //coordinates number const int popSizeP, //population size const int parentPopSizeP, //parent population size const double crossoverProbabP, //crossover probability const int crossoverPointsP, //crossover points const double mutationProbabP, //mutation probability const double inversionProbabP, //inversion probability const int doubleDigitsInChromoP);//number of decimal places in the gene public: void Moving (); public: void Revision (); //---------------------------------------------------------------------------- private: int coords; //coordinates number private: int popSize; //population size private: int parentPopSize; //parent population size private: double crossoverProbab; //crossover probability private: int crossoverPoints; //crossover points private: double mutationProbab; //mutation probability private: double inversionProbab; //inversion probability private: int doubleDigitsInChromo; //number of decimal places in the gene private: bool revision; private: S_Agent parents []; //parents private: int ind []; //temporary array for sorting the population private: double val []; //temporary array for sorting the population private: S_Agent pTemp []; //temporary array for sorting the population private: char tempChrome []; //temporary chromosome for inversion surgery private: uint lengthChrome; //length of the chromosome (the length of the string of characters according to the Gray code) private: int pCount; //indices of chromosome break points private: uint poRND []; //temporal indices of chromosome break points private: uint points []; //final indices of chromosome break points private: S_Roulette roulette []; //roulette private: void PreCalcRoulette (); private: int SpinRoulette (); private: double SeInDiSp (double In, double InMin, double InMax, double Step); private: double RNDfromCI (double min, double max); private: void Sorting (S_Agent &p [], int size); private: double Scale (double In, double InMIN, double InMAX, double OutMIN, double OutMAX); }; //——————————————————————————————————————————————————————————————————————————————
Далее код представляет реализацию метода "Init" класса "C_AO_BGA". Этот метод инициализирует поля класса и массивы, необходимые для работы генетического алгоритма.
Входные параметры метода:
- "coordsP": количество координат.
- "popSizeP": размер популяции.
- "parentPopSizeP": размер родительской популяции.
- "crossoverProbabP": вероятность скрещивания.
- "crossoverPointsP": количество точек скрещивания.
- "mutationProbabP": вероятность мутации.
- "inversionProbabP": вероятность инверсии.
- "doubleDigitsInChromoP": количество десятичных знаков в гене.
Метод "Init" используется для инициализации полей класса и массивов перед выполнением генетического алгоритма. Он устанавливает значения полей класса, проверяет и корректирует значения некоторых параметров, а также изменяет размеры массивов, выделяя память для них.
//—————————————————————————————————————————————————————————————————————————————— void C_AO_BGA::Init (const int coordsP, //coordinates number const int popSizeP, //population size const int parentPopSizeP, //parent population size const double crossoverProbabP, //crossover probability const int crossoverPointsP, //crossover points const double mutationProbabP, //mutation probability const double inversionProbabP, //inversion probability const int doubleDigitsInChromoP) //number of decimal places in the gene { MathSrand ((int)GetMicrosecondCount ()); // reset of the generator fB = -DBL_MAX; revision = false; coords = coordsP; popSize = popSizeP; parentPopSize = parentPopSizeP; crossoverProbab = crossoverProbabP; crossoverPoints = crossoverPointsP; pCount = crossoverPointsP; mutationProbab = mutationProbabP; inversionProbab = inversionProbabP; doubleDigitsInChromo = doubleDigitsInChromoP; if (crossoverPoints < 1) crossoverPoints = 1; if (pCount < 1) pCount = 1; ArrayResize (poRND, pCount); ArrayResize (points, pCount + 2); ArrayResize (ind, parentPopSize + popSize); ArrayResize (val, parentPopSize + popSize); ArrayResize (pTemp, parentPopSize + popSize); ArrayResize (a, popSize); ArrayResize (parents, parentPopSize + popSize); ArrayResize (roulette, parentPopSize); ArrayResize (rangeMax, coords); ArrayResize (rangeMin, coords); ArrayResize (rangeStep, coords); ArrayResize (cB, coords); } //——————————————————————————————————————————————————————————————————————————————
Все основные операции по работе с генетическим материалом агентов выполняются методом "Moving" класса "C_AO_BGA". Метод "Moving" выполняет шаг генетического алгоритма, включая выборку родительских особей, скрещивание, инверсию и мутацию хромосом, а также применение операций к генам и координатам особей.
Метод логически разделен на несколько частей:
1. "if (!revision)" - если "revision" равно "false", выполняется инициализация особей популяции:
- Вызывается метод "Init" для инициализации особи с заданными параметрами.
- Генерируется случайная хромосома путем заполнения каждого гена случайным значением 0 или 1.
- Вызывается метод "ExtractGenes" для извлечения генов из хромосомы.
- Координаты особи "c" приводятся к диапазону с помощью функции "SeInDiSp".
- Значение приспособленности кадой особи "f" устанавливается в "-DBL_MAX".
- "lengthChrome = ArraySize (a [0].chromosome)" - определяется длина хромосомы особи (у всех особей одинаковая длина).
- "ArrayResize (tempChrome, lengthChrome)" - изменяется размер временного массива "tempChrome" на "lengthChrome".
2. Для каждой особи в популяции:
- Выполняется предварительный расчет рулетки выбора родительских особей с помощью метода "PreCalcRoulette".
- Выполняется выбор родительской особи с помощью метода "SpinRoulette".
- Копируется хромосома выбранной родительской особи в хромосому текущей особи.
- Выполняется операция скрещивания с вероятностью "crossoverProbab".
- Выбирается вторая родительская особь.
- Определяются точки разрыва хромосомы.
- Выполняется скрещивание хромосом родительских особей.
- Выполняется операция инверсии с вероятностью "inversionProbab".
- Определяется случайная точка разрыва хромосомы.
- Части хромосомы меняются местами.
- Выполняется операция мутации для каждого гена хромосомы с вероятностью "mutationProbab".
- Вызывается метод "ExtractGenes" для извлечения генов из хромосомы.
- Координаты особи "c" приводятся к диапазону с помощью функции "SeInDiSp".
//—————————————————————————————————————————————————————————————————————————————— void C_AO_BGA::Moving () { //---------------------------------------------------------------------------- if (!revision) { for (int i = 0; i < popSize; i++) { a [i].Init (coords, rangeMin, rangeMax, doubleDigitsInChromo); int r = 0; for (int len = 0; len < ArraySize (a [i].chromosome); len++) { r = MathRand (); //[0,32767] if (r > 16384) a [i].chromosome [len] = 1; else a [i].chromosome [len] = 0; } a [i].ExtractGenes (); for (int c = 0; c < coords; c++) a [i].c [c] = SeInDiSp (a [i].c [c], rangeMin [c], rangeMax [c], rangeStep [c]); a [i].f = -DBL_MAX; a [i].calculated = true; } lengthChrome = ArraySize (a [0].chromosome); ArrayResize (tempChrome, lengthChrome); for (int i = 0; i < parentPopSize + popSize; i++) { parents [i].Init (coords, rangeMin, rangeMax, doubleDigitsInChromo); parents [i].f = -DBL_MAX; } revision = true; return; } //---------------------------------------------------------------------------- int pos = 0; double r = 0; uint p1 = 0; uint p2 = 0; uint p3 = 0; uint temp = 0; for (int i = 0; i < popSize; i++) { PreCalcRoulette (); //selection, select and copy the parent to the child------------------------ pos = SpinRoulette (); ArrayCopy (a [i].chromosome, parents [pos].chromosome, 0, 0, WHOLE_ARRAY); //crossover----------------------------------------------------------------- r = RNDfromCI (0.0, 1.0); if (r < crossoverProbab) { //choose a second parent to breed with------------------------------------ pos = SpinRoulette (); //determination of chromosome break points-------------------------------- for (int p = 0; p < pCount; p++) { poRND [p] = (int)RNDfromCI (0.0, lengthChrome); if (poRND [p] >= lengthChrome) poRND [p] = lengthChrome - 1; } ArraySort (poRND); ArrayCopy (points, poRND, 1, 0, WHOLE_ARRAY); points [0] = 0; points [pCount + 1] = lengthChrome - 1; r = RNDfromCI (0.0, 1.0); int startPoint = r > 0.5 ? 0 : 1; for (int p = startPoint; p < pCount + 2; p += 2) { if (p < pCount + 1) { for (uint len = points [p]; len < points [p + 1]; len++) a [i].chromosome [len] = parents [pos].chromosome [len]; } } } //perform an inversion------------------------------------------------------ //(break the chromosome, swap the received parts, connect them together) r = RNDfromCI (0.0, 1.0); if (r < inversionProbab) { p1 = (int)RNDfromCI (0.0, lengthChrome); if (p1 >= lengthChrome) p1 = lengthChrome - 1; //copying the second part to the beginning of the temporary array for (uint len = p1; len < lengthChrome; len++) tempChrome [len - p1] = a [i].chromosome [len]; //copying the first part to the end of the temporary array for (uint len = 0; len < p1; len++) tempChrome [lengthChrome - p1 + len] = a [i].chromosome [len]; //copying a temporary array back for (uint len = 0; len < lengthChrome; len++) a [i].chromosome [len] = tempChrome [len]; } //выполнить мутацию--------------------------------------------------------- for (uint len = 0; len < lengthChrome; len++) { r = RNDfromCI (0.0, 1.0); if (r < mutationProbab) a [i].chromosome [len] = a [i].chromosome [len] == 1 ? 0 : 1; } a [i].ExtractGenes (); for (int c = 0; c < coords; c++) a [i].c [c] = SeInDiSp (a [i].c [c], rangeMin [c], rangeMax [c], rangeStep [c]); a [i].calculated = true; } } //——————————————————————————————————————————————————————————————————————————————
Метод "Revision" выполняет ревизию популяции, сортирует особи по значению их функции приспособленности и обновляет приспособленность лучшего решения "fB" и координаты лучшего решения "cB". Перед сортировкой популяции происходит копирование дочерней популяции в конец общей популяции.
//—————————————————————————————————————————————————————————————————————————————— void C_AO_BGA::Revision () { //---------------------------------------------------------------------------- for (int i = parentPopSize; i < parentPopSize + popSize; i++) { parents [i] = a [i - parentPopSize]; } Sorting (parents, parentPopSize + popSize); if (parents [0].f > fB) { fB = parents [0].f; ArrayCopy (cB, parents [0].c, 0, 0, WHOLE_ARRAY); } } //——————————————————————————————————————————————————————————————————————————————
Код предварительной разметки рулетки представляет метод "PreCalcRoulette". Этот метод предварительно вычисляет значения диапазонов для рулетки выбора особей на основе их функции приспособленности.
//—————————————————————————————————————————————————————————————————————————————— void C_AO_BGA::PreCalcRoulette () { roulette [0].start = parents [0].f; roulette [0].end = roulette [0].start + (parents [0].f - parents [parentPopSize - 1].f); for (int s = 1; s < parentPopSize; s++) { if (s != parentPopSize - 1) { roulette [s].start = roulette [s - 1].end; roulette [s].end = roulette [s].start + (parents [s].f - parents [parentPopSize - 1].f); } else { roulette [s].start = roulette [s - 1].end; roulette [s].end = roulette [s].start + (parents [s - 1].f - parents [s].f) * 0.1; } } } //——————————————————————————————————————————————————————————————————————————————
Обеспечение исполнения вероятности выбора позиции родительской особи выполняет метод "SpinRoulette".
//—————————————————————————————————————————————————————————————————————————————— int C_AO_BGA::SpinRoulette () { double r = RNDfromCI (roulette [0].start, roulette [parentPopSize - 1].end); for (int s = 0; s < parentPopSize; s++) { if (roulette [s].start <= r && r < roulette [s].end) return s; } return 0; } //——————————————————————————————————————————————————————————————————————————————
3. Результаты тестов
Распечатка работы бинарного генетического алгоритма BGA на тестовом стенде:
C_AO_BGA:50:50:1.0:3:0.001:0.7:3
=============================
5 Hilly's; Func runs: 10000; result: 0.9999191151339382
25 Hilly's; Func runs: 10000; result: 0.994841435673127
500 Hilly's; Func runs: 10000; result: 0.5048331764136147
=============================
5 Forest's; Func runs: 10000; result: 1.0
25 Forest's; Func runs: 10000; result: 0.9997457419655973
500 Forest's; Func runs: 10000; result: 0.32054251149158375
=============================
5 Megacity's; Func runs: 10000; result: 0.9066666666666668
25 Megacity's; Func runs: 10000; result: 0.9640000000000001
500 Megacity's; Func runs: 10000; result: 0.23034999999999997
=============================
All score: 6.92090 (76.90%)
Визуализация процесса оптимизации BGA наглядно демонстрирует высокую сходимость алгоритма. Интересно отметить, что в процессе оптимизации некоторая часть популяции остается вдали от глобального экстремума, что указывает на исследование новых неизвестных областей пространства поиска, сохраняя разнообразие решений в популяции. Однако алгоритм сталкивается с определенными трудностями при работе с дискретной функцией "Megacity", которая является проблемной для большинства алгоритмов. Несмотря на некоторый разброс результатов при работе с этой сложной функцией, BGA проявляет себя значительно лучше, чем другие алгоритмы.
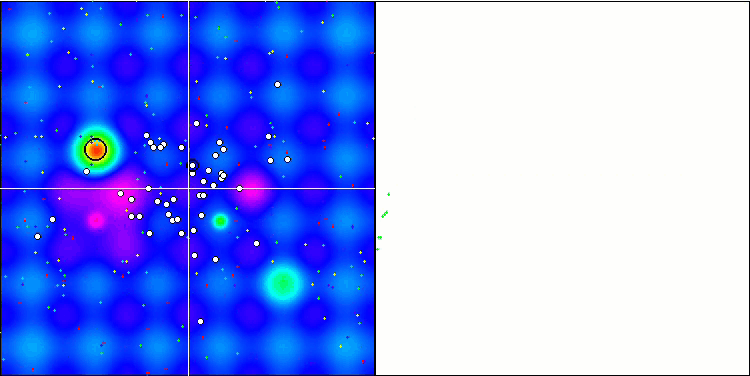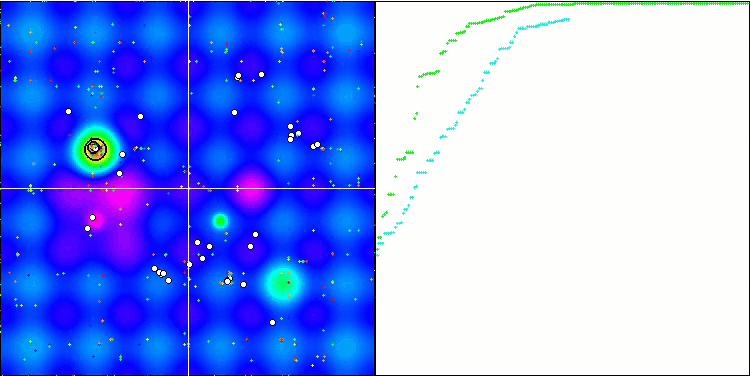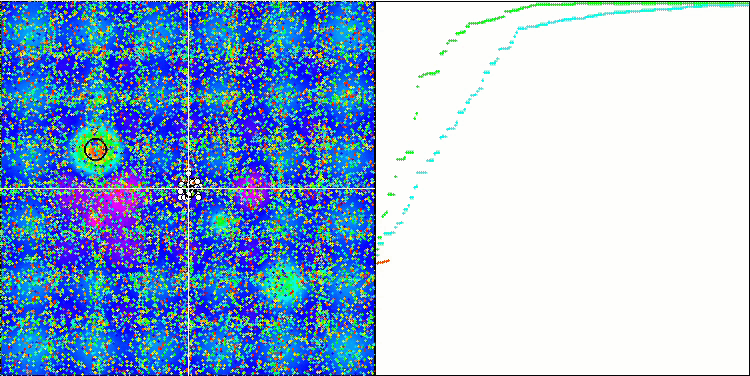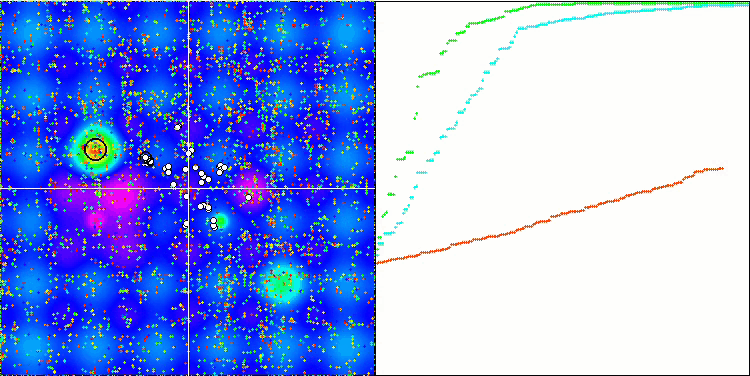
BGA на тестовой функции Hilly.
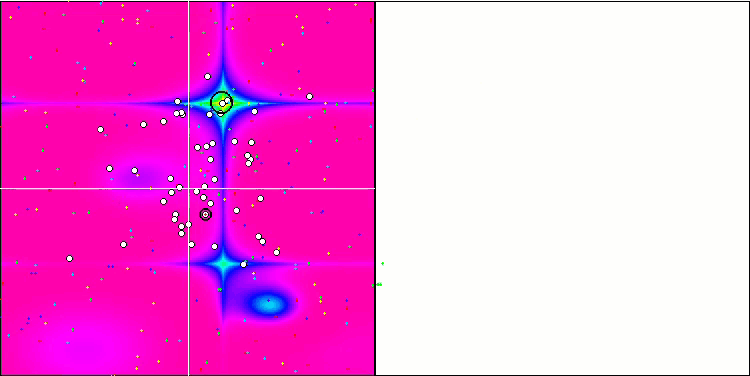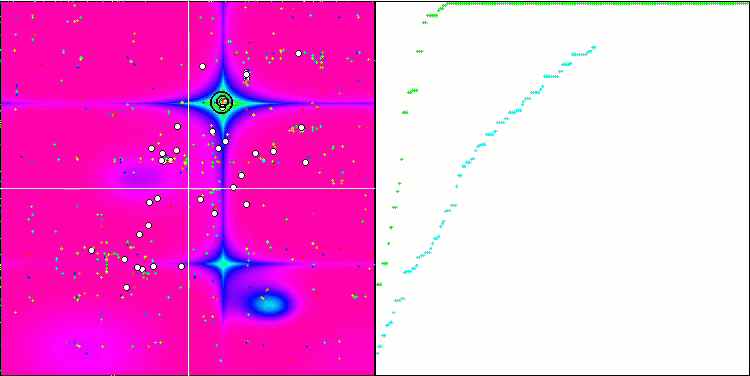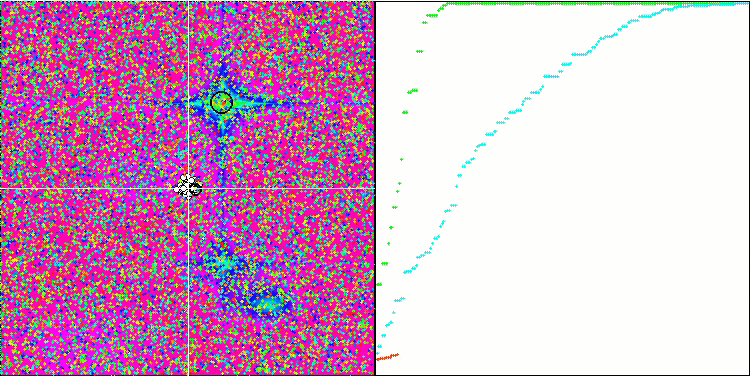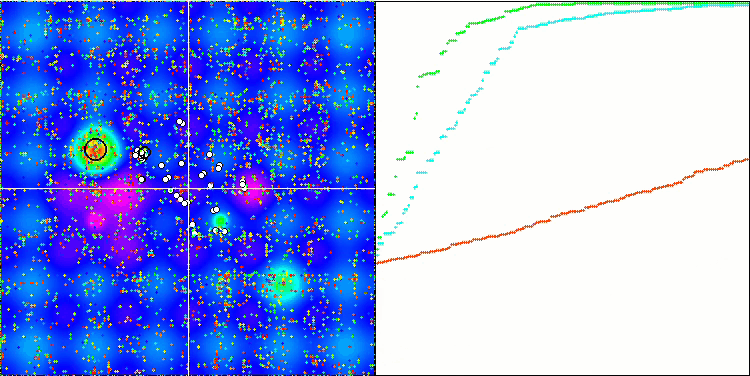
BGA на тестовой функции Forest.
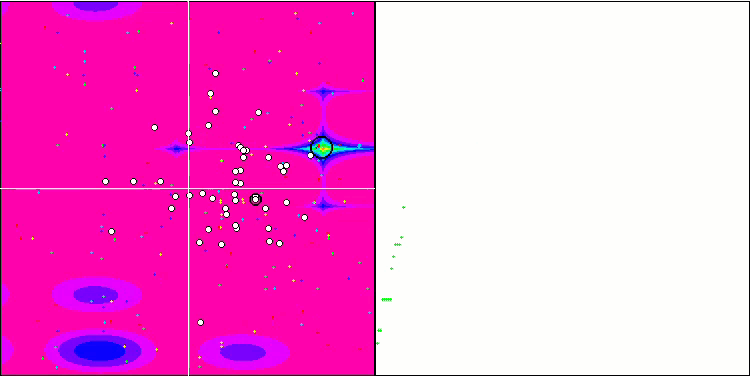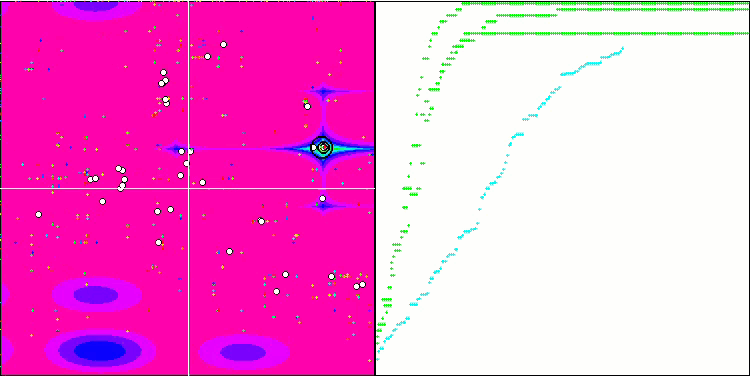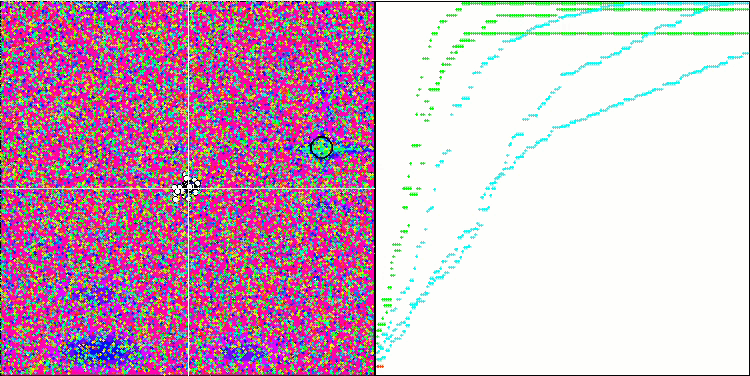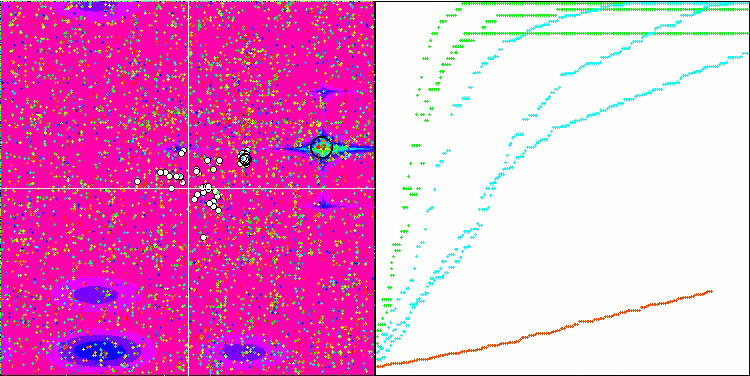
BGA на тестовой функции Megacity.
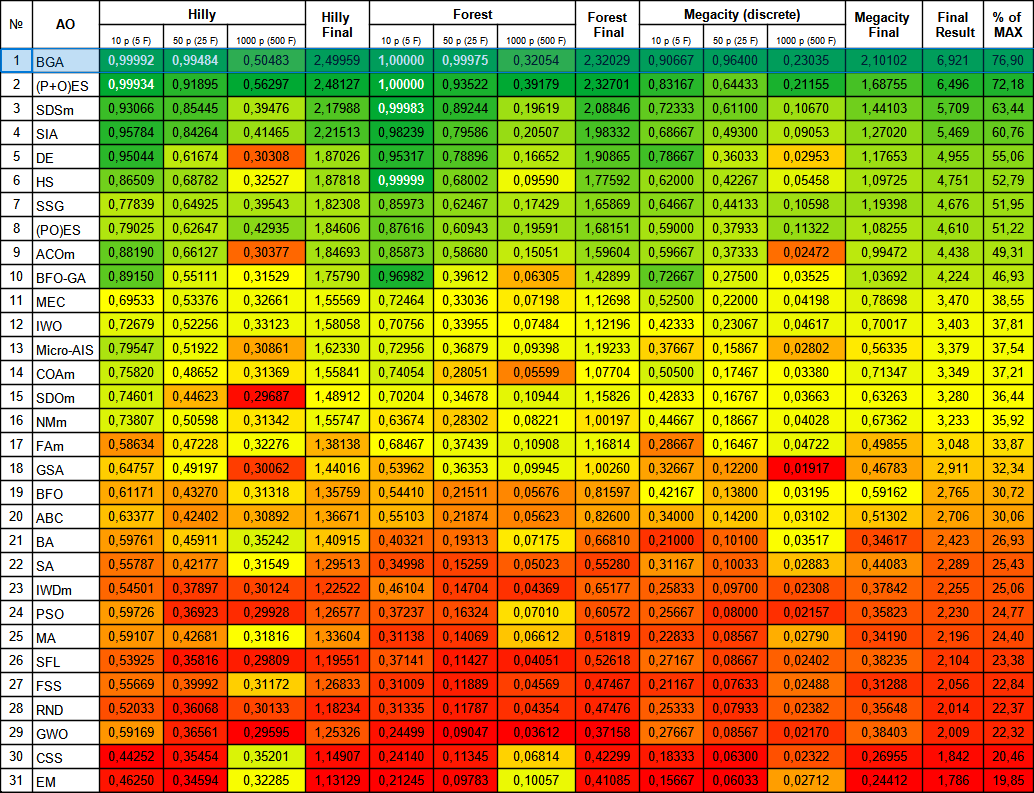
BGA занял первое место в рейтинговой таблице, продемонстрировав лучшие результаты в большинстве тестов для всех трех тестовых функций. Особенно впечатляющие результаты BGA продемонстрировал на дискретной функции Megacity, превзойдя другие алгоритмы.
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Выводы
В этой статье мы рассмотрели классический вариант BGA, частный случай общего класса генетических алгоритмов GA и все выводы относятся именно к нему. Несмотря на давнюю идею двоичного представления решений, подход с использованием двоичного кода остается актуальным до сих пор. Он объединяет независимые пространственные измерения задачи оптимизации в единое целое с помощью кодирования информации в одной хромосоме, что реализовать с помощью обычного вещественного кодирования признаков затруднительно, благодаря чему этот алгоритм выделяется среди других алгоритмов оптимизации.
Хотя математический аппарат и логика стратегии BGA мне полностью понятны, я до сих пор восхищаюсь происходящим с хромосомой. Это подобно тому, как если бы мы сравнили ее с магическим калейдоскопом. Когда мы вращаем калейдоскоп, разнообразные формы и цвета объединяются в уникальные узоры, создавая впечатляющую картину. Точно так же, оператор кроссовера в BGA случайным образом разрезает хромосому на несколько частей, включая внутренние области параметров. Затем эти части соединяются, подобно перемешиванию фрагментов калейдоскопа. Этот процесс позволяет объединить наилучшие характеристики разных решений и создать новую, более оптимальную комбинацию. Как и в случае с калейдоскопом, результаты кроссовера BGA могут быть удивительными и неожиданными, превращая простые хромосомы в истинные "алмазы" оптимальных решений.
Я уверен, что информация двух частей статьи о методах и инструментах, используемых в генетических алгоритмах, поможет вам расширить свои знания и достичь новых высот в работе и исследованиях. В природе, технике и человеческом разуме непрерывно проявляется сила эволюции, и BGA — это один из многих удивительных алгоритмов, которые помогут нам достигать новых высот и достижений.
BGA эффективно решает разнообразные задачи, будь то гладкие поверхности, дискретные проблемы или даже задачи большой размерности, в том числе используя стохастический подход, открывает новые возможности там, где аналитические решения ограничены.
 <
<
Рисунок 2. Цветовая градация алгоритмов по соответствующим тестам. Белым цветом подсвечены результаты больше или равные 0.99.
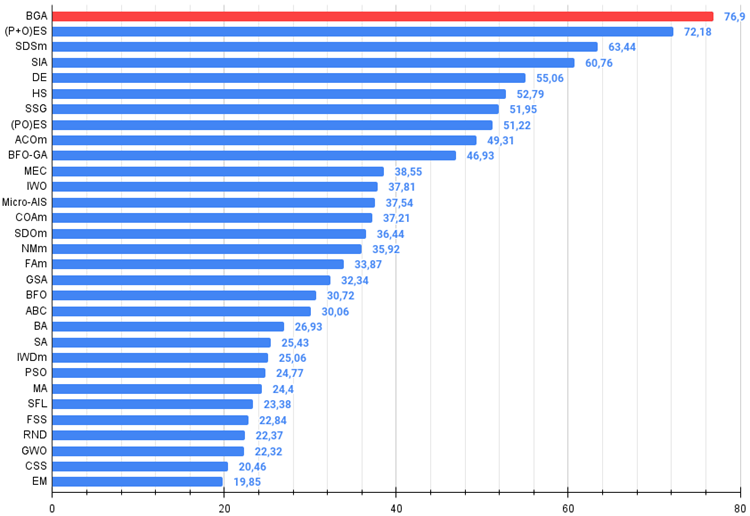
Рисунок 3. Гистограмма результатов тестирования алгоритмов (по шкале от 0 до 100, чем больше, тем лучше,
где 100 - максимально возможный теоретический результат, в архиве скрипт для расчета рейтинговой таблицы).
Плюсы и минусы бинарного генетического алгоритма (BGA), относятся исключительно к представленной реализации:
Плюсы:
- Высокая эффективность при решении разнообразных задач.
- Устойчивость к застреванию.
- Высокие результаты как на гладких, так и на сложных дискретных функциях.
- Высокая сходимость.
Минусы:
- Большое количество внешних параметров.
- Достаточно сложная реализации алгоритма.
- Высокая вычислительная сложность.
К статье прикреплен архив с обновленными актуальными версиями кодов алгоритмов, описанных в предыдущих статьях. Автор статьи не несёт ответственности за абсолютную точность в описании канонических алгоритмов, во многие из них были внесены изменения для улучшения поисковых возможностей. Выводы и суждения, представленные в статьях, основываются на результатах проведённых экспериментов.
Предупреждение: все права на данные материалы принадлежат MetaQuotes Ltd. Полная или частичная перепечатка запрещена.
Данная статья написана пользователем сайта и отражает его личную точку зрения. Компания MetaQuotes Ltd не несет ответственности за достоверность представленной информации, а также за возможные последствия использования описанных решений, стратегий или рекомендаций.
 Нейросети — это просто (Часть 74): Адаптивное прогнозирование траекторий
Нейросети — это просто (Часть 74): Адаптивное прогнозирование траекторий
 Причинно-следственный вывод в задачах классификации временных рядов
Причинно-следственный вывод в задачах классификации временных рядов
 Тип рисования DRAW_ARROW в мультисимвольных мультипериодных индикаторах
Тип рисования DRAW_ARROW в мультисимвольных мультипериодных индикаторах
- Бесплатные приложения для трейдинга
- 8 000+ сигналов для копирования
- Экономические новости для анализа финансовых рынков
Вы принимаете политику сайта и условия использования