
Algoritmo de optimización de reacciones químicas (CRO) (Parte I): Química de procesos en la optimización
Contenido
1. Introducción
La optimización de reacciones químicas (CRO) es un método apasionante e innovador inspirado en la esencia misma de las transformaciones químicas. Imagínese observar una danza de moléculas, donde cada movimiento y colisión juega un papel clave en la solución de problemas complejos. Este método combina inteligentemente los principios de conservación de energía, descomposición y síntesis de moléculas, creando un enfoque flexible y adaptativo para la optimización.
CRO es un método que combina de forma flexible las reacciones químicas con la optimización, utilizando principios generales de interacciones moleculares que están definidos por las dos primeras leyes de la termodinámica. El algoritmo de optimización de reacción química (CRO) fue propuesto y publicado por Lam y Li en 2012.
La primera ley de la termodinámica (la ley de conservación de la energía) establece que la energía no se puede crear ni destruir. Se puede convertir de una forma a otra y pasar de una entidad a otra. En el contexto de CRO, un sistema de reacción química está formado por sustancias y el entorno, y cada partícula tiene energía potencial y cinética.
La segunda ley de la termodinámica establece que la entropía de un sistema tiende a aumentar, donde la entropía es una medida de desorden, el sistema anhela más libertad. La energía potencial es la energía almacenada en una molécula en relación con su configuración molecular. A medida que la energía potencial de las moléculas se libera y se convierte en energía cinética, el sistema se vuelve cada vez más caótico. Pero es en este caos donde CRO encuentra su fuerza, capturando y dirigiendo los flujos de energía hacia la solución óptima.
Por ejemplo, cuando las moléculas con mayor energía cinética (convertida a partir de energía potencial) se mueven más rápido, el sistema se vuelve más desordenado y su entropía aumenta. Así, todos los sistemas reactivos tienden a alcanzar un estado de equilibrio, en el que la energía potencial se reduce al mínimo. En CRO, capturamos este fenómeno convirtiendo la energía potencial en energía cinética y perdiendo gradualmente la energía de las moléculas químicas en el medio ambiente.
Un sistema químico que sufre una reacción química es inestable y, al tener exceso de energía, intenta deshacerse de ella y estabilizarse. El sistema está formado por moléculas, que son las partículas más pequeñas de un compuesto y se clasifican en diferentes tipos según sus propiedades químicas básicas.
Las reacciones químicas forman una danza compleja donde las moléculas chocan, crean y rompen enlaces, cambiando sus estructuras. Cada paso de esta danza es una secuencia de subreacciones que conducen a productos más estables con una energía mínima. Las moléculas almacenan energía en sus enlaces químicos, e incluso pequeños cambios en su estructura pueden conducir a transformaciones sorprendentes que dan como resultado productos más estables. La estructura molecular influye en el comportamiento químico de un compuesto, e incluso cambios menores en la estructura pueden generar diferencias significativas en las propiedades químicas.
Las reacciones químicas se inician por colisiones de moléculas, que pueden ser unimoleculares (con sustancias externas) o bimoleculares (con otras moléculas). Estas colisiones pueden ser eficientes o ineficientes, dependiendo de factores como la energía de activación y los efectos estéricos. De esta manera, las moléculas bailan, entrelazándose y separándose, creando nuevas formas y estructuras.
Es importante señalar que las reacciones químicas a menudo dan lugar a la formación de productos más estables con energías de Gibbs más bajas. Este proceso ocurre a través de pasos secuenciales y/o paralelos que implican la formación de compuestos intermedios y el paso por estados de transición. Comprender estos procesos es importante para encontrar las condiciones óptimas para las reacciones químicas y aclarar los mecanismos de transformación de los compuestos químicos.
Además, la síntesis química puede ocurrir como resultado de colisiones efectivas e ineficaces de reactivos. La eficiencia de las colisiones depende de factores como la energía de activación, los efectos estéricos, etc. La síntesis intermolecular, a diferencia de la intramolecular, conduce a cambios más significativos en las estructuras moleculares.
El algoritmo CRO es similar a un coreógrafo virtuoso que utiliza las leyes de la química como notas musicales para crear soluciones de optimización increíblemente complejas y elegantes. Ahora veamos todo punto por punto: desde los conceptos complejos hasta los más simples.
2. Implementación de operadores químicos
La unidad básica en CRO es la "molécula". En una población, cada uno de ellos tiene ciertas características, como "energías potenciales y cinéticas", "estructuras moleculares", etc. Las reacciones elementales definen interacciones entre moléculas. Podemos definir una clase, en la que los campos de datos representan características de la molécula y los métodos describen reacciones elementales.
Uno de los conceptos principales en CRO es la conservación de energía. Este principio garantiza que la energía total del sistema permanezca constante durante todo el proceso de optimización. La conservación de la energía determina las transiciones entre diferentes estados de las moléculas durante las reacciones, manteniendo un equilibrio de energías que influye en la búsqueda de soluciones óptimas.
Utilizando agentes manipulables (moléculas), reacciones elementales y el concepto de conservación de energía, CRO ofrece un método flexible y adaptativo para resolver problemas de optimización complejos. La capacidad de personalizar operadores, cambiar dinámicamente el tamaño de la población e integrar diferentes atributos hacen de CRO un enfoque prometedor en el campo de las metaheurísticas. Sus características únicas y su flexibilidad permiten a los investigadores explorar nuevas oportunidades en optimización.
El algoritmo CRO utiliza los siguientes operadores:
1. Colisión intermolecular ineficaz. Este operador modela el proceso en el que dos moléculas chocan pero permanecen intactas y sus estructuras cambian ligeramente. Esto permite que el algoritmo realice una búsqueda local en la proximidad de las soluciones actuales.
2. Descomposición. Este operador modela el proceso en el que una molécula choca con una pared y se rompe en dos nuevas moléculas. Esto permite que el algoritmo explore nuevas áreas del espacio de soluciones.
3. Reacción intramolecular. Este operador modela el proceso en el que una molécula choca con una pared y permanece intacta, pero su estructura cambia ligeramente. Esto permite que el algoritmo realice una búsqueda local en la proximidad de la solución actual.
4. Síntesis. Este operador modela el proceso en el que dos moléculas chocan y se combinan para formar una única molécula nueva. Esto permite que el algoritmo combine buenas soluciones para crear soluciones potencialmente mejores.
Cada uno de estos operadores juega un papel importante en el algoritmo CRO, permitiéndole explorar el espacio de búsqueda y encontrar soluciones óptimas. Su colaboración proporciona un equilibrio entre la exploración (búsqueda de nuevas áreas del espacio de búsqueda) y la explotación (mejora de las mejores soluciones actuales).
Consideremos cada operador químico individual en el algoritmo (búsqueda del mínimo global):
Algoritmo nº 1: Colisión ineficaz en la pared
1. Datos de entrada:Mi
molécula2. Generando una nueva posición de la molécula: ω = N(ω)
3. Cálculo de la energía potencial de la nueva posición: PEω = f(ω)
4. Aumentar el contador de colisiones: NumHitω = NumHitω + 1
5. Si la energía potencial de una nueva posición + energía cinética ≥ energía potencial de la posición actual, entonces:
6. Generar un número aleatorio 'a' en el rango[KELossRate, 1]
7. Actualización de la energía cinética: KEω = (PEω - PEω + KEω) × a
8. Actualización del búfer: buffer = buffer + (PEω - PEω + KEω) × (1 - a)
9. Manteniendo la posición y energías actuales
10. Si la energía potencial de una nueva posición < energía potencial mínima, entonces actualice los valores mínimos
11. Condición final
Algoritmo nº 2: Descomposición
1. Datos de entrada:Mi
molécula2. Creación de dos nuevas moléculas Mω1 y Mω2
.3. Obtención de posiciones para nuevas moléculas: ω1 y ω2 a partir de ω
4. Cálculo de la energía potencial para nuevas moléculas: PEω1 = f(ω1) y PEω2 = f(ω2)
5. Si energía potencial de la posición actual + energía cinética ≥ energía potencial total de las nuevas posiciones, entonces:
6. Cálculo de la energía de descomposición: Edec = PEω + KEω - (PEω1 + PEω2)
7. Vaya al paso 13
8. De lo contrario:
9. Generación de números aleatorios δ1, δ2 en el intervalo [0, 1]
. 10. Cálculo de la energía de descomposición: Edec = PEω + KEω + δ1δ2 × buffer - (PEω1 + PEω2)
11. Si Edec ≥ 0, entonces:
12. Actualización de buffer
13. Generación de números aleatorios δ3 en el rango [0, 1]
14. Distribución de la energía cinética entre las nuevas moléculas
15. Mantener valores mínimos para cada nueva molécula
16. Destrucción de la molécula actual
17. De lo contrario:
18. Aumentar el contador de colisiones
19. Destrucción de nuevas moléculas
20. Fin de la condición
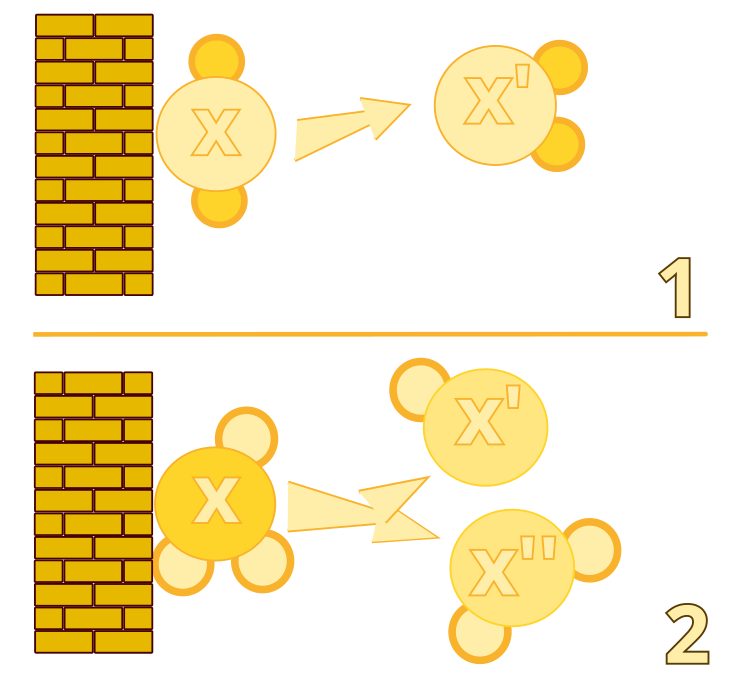
Figura 1. Colisión de paredes ineficiente: algoritmo n.° 1. Descomposición: algoritmo n°2
Algoritmo nº 3: Colisión intermolecular ineficaz
1. Datos de entrada: Mω1 y Mω2
moléculas2. Generación de nuevas posiciones para las moléculas: ω1 = N(ω1) y ω2 = N(ω2)
3. Cálculo de la energía potencial para las nuevas posiciones: PEω1 = f(ω1) y PEω2 = f(ω2)
4. Aumenta los contadores de colisiones:NumHitω1 = NumHitω1 + 1 y NumHitω2 = NumHitω2 + 1
.5. Cálculo de la energía de interacción intermolecular: Einter = (PEω1 + PEω2 + KEω1 + KEω2) - (PEω1 + PEω2)
.6. Si Einter ≥ 0, entonces:
7. Generación de números aleatorios δ4 en el intervalo [0, 1]
. 8. Distribución de la energía cinética entre moléculas: KEω1 = Einter × δ4 y KEω2 = Einter × (1 - δ4)
. 9. Actualizar las posiciones y energías de las moléculas
10. Si la energía potencial de una nueva posición es menor que la energía potencial mínima, entonces actualiza los valores mínimos para cada molécula
11. Fin de la condición
Algoritmo nº 4: Síntesis
1. Datos de entrada: Mω1 y Mω2
moléculas2. Creación de un nuevo Mω
molécula3. Obtención de una posición para una nueva molécula: ω a partir de ω1 y ω2
4. Cálculo de la energía potencial de una nueva molécula: PEω = f(ω)
5. Si la energía potencial y la energía cinética totales de la nueva molécula son mayores o iguales que la energía potencial y la energía cinética totales de las moléculas originales, entonces:
6. Distribución del exceso de energía cinética a una nueva molécula: KEω = (PEω1 + PEω2 + KEω1 + KEω2) - PEω
7. Actualización de los valores mínimos para una nueva molécula
8. Destrucción de las moléculas originales
9. De lo contrario:
10. Aumentar los contadores de colisiones de las moléculas fuente
11. Destrucción de una nueva molécula
12. Fin de la condición
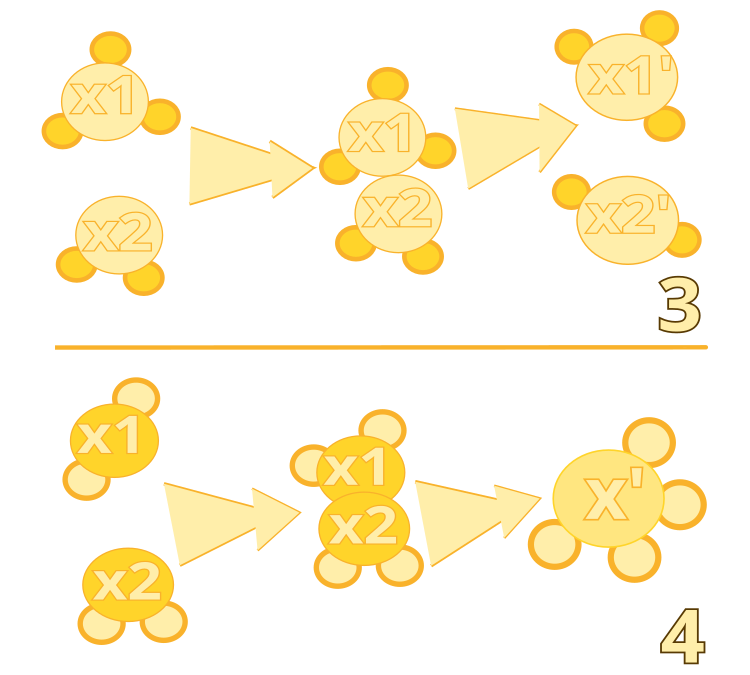
Figura 2. Colisión intermolecular ineficiente: algoritmo nº 3. Síntesis: algoritmo nº 4
La descripción propuesta del algoritmo CRO refleja la visión del autor sobre este enfoque. Sin embargo, no tiene en cuenta algunos puntos importantes que pueden afectar significativamente el rendimiento y las capacidades de búsqueda del algoritmo.
El algoritmo implica la conservación de la energía en un espacio cerrado y la transición de un tipo de energía a otro. Sin embargo, los autores no revelan la correspondencia entre los indicadores numéricos de energía y el valor de la función objetivo (fitness). Es obvio que el papel de la aptitud es definido por los autores como energía potencial, y la energía cinética actúa como un mecanismo para compensar la disminución de la energía potencial (la suma de todas las energías idealmente debería ser una constante).
Por ejemplo, si el criterio de un problema de optimización es el factor de beneficio, entonces los valores de la función objetivo fluctuarán en un rango pequeño en comparación con un problema donde se utiliza el equilibrio como criterio. En este caso, vemos que los valores de la función objetivo variarán dependiendo del criterio de optimización específico, pero los autores utilizan constantes como un parámetro externo del algoritmo que no se puede comparar con las energías calculadas en el algoritmo.
Por tanto, el algoritmo CRO original (sin modificaciones) tiene una lista significativamente limitada de tareas en las que se puede aplicar, por lo que no es universal. En esta serie de artículos consideramos únicamente algoritmos universales aplicables a problemas de optimización en general, y si algún algoritmo no lo permite, normalmente lo modificamos y lo llevamos a una única forma general.
El segundo punto es que el algoritmo original prevé el cálculo de la función de fitness de forma desordenada, en diferentes partes de la lógica, lo que inevitablemente conducirá a problemas cuando se utilice en tareas prácticas y se integre en proyectos. Arreglaremos esto también. Para ello, necesitaremos dividir los operadores químicos en dos partes: los operadores propiamente dichos y los llamados "post-operadores", el primero de los cuales modifica la posición de las moléculas, y el segundo realiza las acciones necesarias con las moléculas después de calcular su aptitud.
El tercer punto es que el algoritmo original prevé un tamaño de población dinámico de moléculas. Aparecen nuevas moléculas y algunas de las antiguas se destruyen. No existen mecanismos para regular el tamaño de la población, y éste puede variar dentro de límites muy amplios. Así, los experimentos han demostrado que con determinados ajustes de parámetros externos la población puede crecer desde las 50 moléculas iniciales a más de mil. También resolvimos este problema llenando secuencialmente la población con moléculas como resultado de la ejecución de operadores, utilizando un contador simple y almacenando el índice de las moléculas principales en la población principal. Esto nos permite mantener constante el tamaño de la población y al mismo tiempo eliminar la necesidad de realizar la operación de eliminación de moléculas: las moléculas hijas, cuando se cumplen las condiciones, simplemente reemplazan a las moléculas madre correspondientes.
Estos cambios permitieron llevar el algoritmo CRO a una forma adecuada para resolver problemas de optimización en general, hacer conveniente la integración del algoritmo en proyectos y, al mismo tiempo, preservar el concepto original general de las reacciones químicas. El algoritmo CRO se describe en detalle. Si lo desea, puede intentar implementar la contabilidad de las energías potencial y cinética en el algoritmo.
Pasemos al código. Describe los tipos de reacciones en la lista para que podamos elegir el postoperador apropiado después de las reacciones y la estructura de las moléculas.
E_ReactionType enumeración:
1. Síntesis es un proceso en el que dos moléculas se combinan para formar una nueva.
2. interMolecularInefColl es una colisión intermolecular ineficaz, en la que las moléculas chocan pero no reaccionan entre sí.
3. Descomposición es un proceso en el que una molécula compleja se descompone en componentes más simples.
4. inefColisión es una colisión ineficiente con la pared, que cambia la estructura de la molécula.
Define la estructura S_CRO_Agent, que representa un modelo de la molécula en el contexto del algoritmo. Veamos qué campos contiene la siguiente estructura:
- structure[] - una matriz que representa la estructura de la molécula.
- NumHit - un contador del número de «hits» o interacciones de moléculas.
- indMolecule_1 y indMolecule_2 - índices de las moléculas que interactúan.
- KE - variable que representa la energía cinética de una molécula.
- f - función de ajuste (ajuste de la molécula).
- rType - E_ReactionType variable de tipo, el tipo de reacción en la que participa una molécula.
"Init" - método que inicializa los campos. Toma el argumento entero coords aplicado para redimensionar el array structure usando la función ArrayResize. NumHit, indMolecule_1, indMolecule_2, f y KE se inicializan utilizando ceros o -DBL_MAX.
Este código representa la estructura básica de datos de las moléculas en el algoritmo CRO e inicializa sus campos cuando se crea una nueva molécula.
enum E_ReactionType { synthesis, interMolecularInefColl, decomposition, inefCollision }; // Molecule structure struct S_CRO_Agent { double structure []; int NumHit; int indMolecule_1; int indMolecule_2; double KE; double f; E_ReactionType rType; // Initialization method void Init (int coords) { ArrayResize (structure, coords); NumHit = 0; indMolecule_1 = 0; indMolecule_2 = 0; f = -DBL_MAX; KE = -DBL_MAX; } };
El método InefCollision de la clase C_AO_CRO es una colisión ineficiente que implica la creación de una nueva molécula por el desplazamiento de una molécula padre. Esto es lo que ocurre en este método:
1. El método acepta el índice de la molécula padre y el enlace al contador de moléculas molCNT. Si molCNT supera o es igual al tamaño de población popSize, el método devuelve false y finaliza su trabajo.
2. El método determina el índice de una nueva molécula index1_, que se creará como resultado de la colisión.
3. La estructura de la molécula madre se copia en la estructura de la nueva.
4. A continuación, se ejecuta la función N para cada coordenada de la nueva molécula. La función genera nuevos valores de coordenadas en las proximidades de los antiguos.
5. Los nuevos valores de coordenadas se ajustan mediante la función SeInDiSp, de forma que permanezcan en el rango especificado de rangeMin a rangeMax.
6. A continuación, se establecen los valores de los campos indMolecule_1, rType y NumHit de la nueva molécula. indMolecule_1 almacena el índice de la molécula padre, rType se establece en inefCollision, mientras que NumHit se restablece.
7. Al final del método, el contador de moléculas molCNT se incrementa en 1 y el método devuelve true.
//—————————————————————————————————————————————————————————————————————————————— // Ineffective collision. Obtaining a new molecule by displacing a parent one. bool C_AO_CRO::InefCollision (int index, int &molCNT) { if (molCNT >= popSize) return false; int index1_ = molCNT; ArrayCopy (Mfilial [index1_].structure, Mparent [index].structure); for (int c = 0; c < coords; c++) { N (Mfilial [index1_].structure [c], c); Mfilial [index1_].structure [c] = u.SeInDiSp (Mfilial [index1_].structure [c], rangeMin [c], rangeMax [c], rangeStep [c]); } Mfilial [index1_].indMolecule_1 = index; // save the parent molecule index Mfilial [index1_].rType = inefCollision; Mfilial [index1_].NumHit = 0; molCNT++; return true; } //——————————————————————————————————————————————————————————————————————————————
El método PostInefCollision de la clase C_AO_CRO sirve para manejar los resultados de la colisión ineficaz de la molécula mol con otras moléculas en la simulación de reacciones químicas. El método hace lo siguiente:
1. Declara la variable ind e inicialízala usando el valor indMolecule_1 del objeto mol.
2. La expresión condicional comprueba si el valor f del objeto mol supera el valor f de la molécula padre con el índice ind.
3. Si la condición es verdadera, entonces la estructura mol se copia en la estructura de la molécula padre con el índice ind.
4. El valor f de la molécula padre se actualiza utilizando el valor f de mol.
5. El contador NumHit de la molécula padre se pone a cero.
6. Si la condición mol.f > Mparent[ind].f es falsa, el contador NumHit de la molécula padre se incrementa en uno.
En general, este método actualiza la estructura y el valor de la función de aptitud de la molécula madre basándose en los resultados de una colisión ineficiente. Si la nueva estructura mol conduce a una mejora de la aptitud, sustituye a la estructura de la molécula padre y se reinicia el contador NumHit. En caso contrario, se incrementa el contador NumHit de la molécula padre.
//—————————————————————————————————————————————————————————————————————————————— // Handling the results of an ineffective collision. void C_AO_CRO::PostInefCollision (S_CRO_Agent &mol) { int ind = mol.indMolecule_1; if (mol.f > Mparent [ind].f) { ArrayCopy (Mparent [ind].structure, mol.structure); Mparent [ind].f = mol.f; Mparent [ind].NumHit = 0; } else { Mparent [ind].NumHit++; } } //——————————————————————————————————————————————————————————————————————————————
El método Decomposition de la clase C_AO_CRO es un proceso de descomposición que implica la creación de dos nuevas moléculas mediante la descomposición de una molécula padre. Esto es lo que ocurre con este método:
1. El método acepta la molécula padre index y un enlace al contador de moléculas molCNT. Si molCNT supera o es igual a popSize - 1, el método devuelve false y finaliza su trabajo.
2. A continuación, el método determina los índices de dos nuevas moléculas - index1_ y index2_, que se crearán como resultado de la descomposición.
3. La estructura de la molécula madre se copia en las estructuras de las nuevas.
4. A continuación, se ejecuta la función N para cada coordenada de las nuevas moléculas. La función genera nuevos valores de coordenadas en las proximidades de los antiguos. Esto se hace por separado para la primera mitad de las coordenadas para index1_ de la primera molécula hija y la segunda mitad de las coordenadas para index2_ de la segunda molécula, respectivamente.
5. Los nuevos valores de coordenadas se ajustan mediante la función SeInDiSp, de forma que permanezcan en el rango especificado de rangeMin a rangeMax.
6. A continuación, se establecen los valores de los campos indMolecule_1, indMolecule_2, rType y NumHit de la nueva molécula. indMolecule_1 y indMolecule_2 guardan los índices de las moléculas padre y hermana, respectivamente, rType se establece en decomposition, mientras que NumHit se establece en cero.
7. Al final del método, el contador de moléculas molCNT se incrementa en 2, y el método devuelve true.
//—————————————————————————————————————————————————————————————————————————————— // Decomposition. Obtaining two new molecules by decomposing a parent one. bool C_AO_CRO::Decomposition (int index, int &molCNT) { if (molCNT >= popSize - 1) return false; // Creating two new molecules M_ω'_1 and M_ω'_2 from M_ω int index1_ = molCNT; int index2_ = molCNT + 1; ArrayCopy (Mfilial [index1_].structure, Mparent [index].structure); ArrayCopy (Mfilial [index2_].structure, Mparent [index].structure); for (int c = 0; c < coords / 2; c++) { N (Mfilial [index1_].structure [c], c); Mfilial [index1_].structure [c] = u.SeInDiSp (Mfilial [index1_].structure [c], rangeMin [c], rangeMax [c], rangeStep [c]); } for (int c = coords / 2; c < coords; c++) { N (Mfilial [index2_].structure [c], c); Mfilial [index2_].structure [c] = u.SeInDiSp (Mfilial [index2_].structure [c], rangeMin [c], rangeMax [c], rangeStep [c]); } Mfilial [index1_].indMolecule_1 = index; // save the parent molecule index Mfilial [index1_].indMolecule_2 = index2_; // save the index of the second daughter molecule Mfilial [index1_].rType = decomposition; Mfilial [index1_].NumHit = 0; Mfilial [index2_].indMolecule_1 = index1_; // save the index of the first daughter molecule Mfilial [index2_].indMolecule_2 = -1; // mark the molecule so we do not handle it twice Mfilial [index2_].rType = decomposition; Mfilial [index2_].NumHit = 0; molCNT += 2; return true; } //——————————————————————————————————————————————————————————————————————————————
El método PostDecomposition de la clase C_AO_CRO maneja los resultados de la descomposición de la molécula. El método hace lo siguiente:
1. El método acepta una referencia a la molécula mol obtenida como resultado de la descomposición. Si indMolecule_2 de la molécula es igual a «-1» (lo que significa que la molécula ya ha sido procesada), el método completa su trabajo.
2. A continuación, se extrae el índice ind de la molécula padre junto con los índices de las dos moléculas «hijas» index1_ y index2_.
3. A continuación, compruebe si el valor de la función de ajuste f de la primera molécula "hija" supera los valores de la función f de la segunda molécula "hija" y de la molécula madre. En ese caso, la primera molécula «hija» sustituye a la molécula madre. Se restablece el valor NumHit de la molécula sustituida y se establece la bandera.
4. Si resulta que la bandera sigue siendo false, se realiza una comprobación similar para la segunda molécula "hija".
5. Si después de todas las comprobaciones, la bandera sigue siendo false, el NumHit de la molécula padre se incrementa en 1.
El método PostDecomposition se encarga de actualizar los estados de la molécula padre tras la descomposición en el algoritmo CRO.
//—————————————————————————————————————————————————————————————————————————————— // Handling decomposition results. void C_AO_CRO::PostDecomposition (S_CRO_Agent &mol) { if (mol.indMolecule_2 == -1) return; int ind = mol.indMolecule_1; int index2_ = mol.indMolecule_2; int index1_ = Mfilial [index2_].indMolecule_1; bool flag = false; if (Mfilial [index1_].f > Mfilial [index2_].f && Mfilial [index1_].f > Mparent [ind].f) { ArrayCopy (Mparent [ind].structure, Mfilial [index1_].structure); Mparent [ind].f = Mfilial [index1_].f; Mparent [ind].NumHit = 0; flag = true; } if (!flag) { if (Mfilial [index2_].f > Mfilial [index1_].f && Mfilial [index2_].f > Mparent [ind].f) { ArrayCopy (Mparent [ind].structure, Mfilial [index2_].structure); Mparent [ind].f = Mfilial [index2_].f; Mparent [ind].NumHit = 0; flag = true; } } if (!flag) { Mparent [ind].NumHit++; } } //——————————————————————————————————————————————————————————————————————————————
El método InterMolInefColl de la clase C_AO_CRO es una colisión ineficiente que implica la creación de dos nuevas moléculas mediante el cambio de dos moléculas padre. Esto es lo que ocurre con este método:
1. El método acepta los índices de las dos moléculas padre - index1 y index2 y un enlace al contador de moléculas molCNT. Si molCNT supera o es igual a popSize - 1, el método devuelve false y finaliza su trabajo.
2. A continuación, el método determina los índices de dos nuevas moléculas hijas index1_ y index2_, que se crearán como resultado de la colisión.
3. Las estructuras de la molécula madre se copian en las estructuras de las nuevas.
4. A continuación, se ejecuta la función N para cada coordenada de las nuevas moléculas. La función genera nuevos valores de coordenadas en las proximidades de los antiguos.
5. A continuación, los nuevos valores de coordenadas se ajustan mediante la función SeInDiSp, de modo que permanezcan en el rango especificado de rangeMin a rangeMax.
6. A continuación, se establecen los valores de los campos indMolecule_1, indMolecule_2, rType y NumHit de la nueva molécula. indMolecule_1 y indMolecule_2 conservan los índices de las moléculas padre, rType se establece en interMolecularInefColl, mientras que NumHit se establece en cero.
7. Al final del método, el contador de moléculas molCNT se incrementa en 2, y el método devuelve true.
//—————————————————————————————————————————————————————————————————————————————— // Intermolecular ineffective collision. Obtaining two new molecules by changing two parent ones bool C_AO_CRO::InterMolInefColl (int index1, int index2, int &molCNT) { if (molCNT >= popSize - 1) return false; int index1_ = molCNT; int index2_ = molCNT + 1; // Obtaining molecules ArrayCopy (Mfilial [index1_].structure, Mparent [index1].structure); ArrayCopy (Mfilial [index2_].structure, Mparent [index2].structure); // Generating new molecules ω'_1 = N(ω1) and ω'_2 = N(ω2) in the vicinity of ω1 and ω2 for (int c = 0; c < coords; c++) { N (Mfilial [index1_].structure [c], c); N (Mfilial [index2_].structure [c], c); } for (int c = 0; c < coords; c++) { Mfilial [index1_].structure [c] = u.SeInDiSp (Mfilial [index1_].structure [c], rangeMin [c], rangeMax [c], rangeStep [c]); Mfilial [index2_].structure [c] = u.SeInDiSp (Mfilial [index2_].structure [c], rangeMin [c], rangeMax [c], rangeStep [c]); } Mfilial [index1_].indMolecule_1 = index1; // save the index of the first parent molecule Mfilial [index1_].indMolecule_2 = index2_; // save the index of the second daughter molecule Mfilial [index1_].rType = interMolecularInefColl; Mfilial [index1_].NumHit = 0; Mfilial [index2_].indMolecule_1 = index2; // save the index of the second parent molecule Mfilial [index2_].indMolecule_2 = -1; // mark the molecule so we do not handle it twice Mfilial [index2_].rType = interMolecularInefColl; Mfilial [index2_].NumHit = 0; molCNT += 2; return true; } //——————————————————————————————————————————————————————————————————————————————
El método PostInterMolInefColl de la clase C_AO_CRO maneja los resultados de las colisiones intermoleculares ineficaces. El método tiene los siguientes objetivos:
1. El método acepta una referencia a la molécula mol obtenida como resultado de la colisión. Si indMolecule_2 de la molécula es igual a «-1» (lo que significa que la molécula ya ha sido procesada), el método completa su trabajo.
2. A continuación, se extraen los índices ind1 y ind2 de las dos moléculas progenitoras.
3. A continuación, comprueba si la f suma de los valores de ajuste de una molécula nueva y su «hermana» supera la f suma de los valores de ajuste de ambas moléculas progenitoras. Si es así, las nuevas moléculas sustituyen a las progenitoras. Se restablecen los valores NumHit de las moléculas sustituidas.
4. En caso contrario, los valores NumHit de las moléculas padre se incrementan en 1.
El método PostInterMolInefColl se encarga de actualizar los estados de las moléculas padre tras la colisión intermolecular ineficiente en el algoritmo CRO.
//—————————————————————————————————————————————————————————————————————————————— // Handling the results of an intermolecular ineffective collision. void C_AO_CRO::PostInterMolInefColl (S_CRO_Agent &mol) { if (mol.indMolecule_2 == -1) return; int ind1 = mol.indMolecule_1; int ind2 = Mfilial [mol.indMolecule_2].indMolecule_1; Mparent [ind1].NumHit++; Mparent [ind2].NumHit++; if (mol.f + Mfilial [mol.indMolecule_2].f > Mparent [ind1].f + Mparent [ind2].f) { ArrayCopy (Mparent [ind1].structure, mol.structure); Mparent [ind1].f = mol.f; ArrayCopy (Mparent [ind2].structure, Mfilial [mol.indMolecule_2].structure); Mparent [ind2].f = Mfilial [mol.indMolecule_2].f; } } //——————————————————————————————————————————————————————————————————————————————
El último operador de reacción química del algoritmo - el método Síntesis de la clase C_AO_CRO - es un proceso de síntesis que implica la creación de una nueva molécula mediante la fusión de dos moléculas progenitoras.
1. El método acepta los índices de las dos moléculas padre - index1 y index2 y un enlace al contador de moléculas molCNT. Si molCNT supera o es igual al tamaño de población popSize, el método devuelve false y finaliza su trabajo.
2. En el bucle, para cada coordenada de la nueva molécula, se hace lo siguiente: si un número aleatorio es menor que 0,5, la coordenada recibe el valor de la coordenada correspondiente de la primera molécula padre Mparent[index1].structure[i]. En caso contrario, recibe el valor de la coordenada correspondiente de la segunda molécula padre Mparent[index2].structure[i].
3. A continuación, se establecen los valores de los campos indMolecule_1, indMolecule_2, rType y NumHit de la nueva molécula. indMolecule_1 y indMolecule_2 conservan los índices de las moléculas padre, rType se establece en synthesis, mientras que NumHit se establece en cero.
4. Al final del método, el contador de moléculas molCNT se incrementa, y el método devuelve true.
//—————————————————————————————————————————————————————————————————————————————— // Synthesis. Obtaining a new molecule by fusing two parent ones bool C_AO_CRO::Synthesis (int index1, int index2, int &molCNT) { if (molCNT >= popSize) return false; // Create a new M_ω' molecule from M_ω1 and M_ω2 for (int i = 0; i < coords; i++) { if (u.RNDprobab () < 0.5) Mfilial [molCNT].structure [i] = Mparent [index1].structure [i]; else Mfilial [molCNT].structure [i] = Mparent [index2].structure [i]; } Mfilial [molCNT].indMolecule_1 = index1; // save the index of the first parent molecule Mfilial [molCNT].indMolecule_2 = index2; // save the index of the second parent molecule Mfilial [molCNT].rType = synthesis; Mfilial [molCNT].NumHit = 0; molCNT++; return true; } //——————————————————————————————————————————————————————————————————————————————
El método PostSynthesis de la clase C_AO_CRO maneja los resultados de la síntesis. Esto es lo que ocurre en él:
1. El método acepta una referencia a la molécula mol obtenida como resultado de la síntesis. A continuación, se extraen los índices ind1 y ind2 de las dos moléculas progenitoras.
2. A continuación, comprueba si el valor de ajuste f de una nueva molécula supera los valores de ajuste f de ambas moléculas progenitoras. Si es así, la nueva molécula sustituye a aquella de las moléculas progenitoras cuyo valor de f sea menor. El valor NumHit de la molécula sustituida se pone a cero.
3. En caso contrario, los valores NumHit de las moléculas padre se incrementan en 1.
El método PostSynthesis se encarga de actualizar los estados de las moléculas padre tras el proceso de síntesis en el algoritmo CRO.
//—————————————————————————————————————————————————————————————————————————————— // Handling synthesis results. void C_AO_CRO::PostSynthesis (S_CRO_Agent &mol) { int ind1 = mol.indMolecule_1; int ind2 = mol.indMolecule_2; if (mol.f > Mparent [ind1].f && mol.f > Mparent [ind2].f) { if (Mparent [ind1].f < Mparent [ind2].f) { ArrayCopy (Mparent [ind1].structure, mol.structure); Mparent [ind1].f = mol.f; Mparent [ind1].NumHit = 0; } else { ArrayCopy (Mparent [ind2].structure, mol.structure); Mparent [ind2].f = mol.f; Mparent [ind2].NumHit = 0; } } else { Mparent [ind1].NumHit++; Mparent [ind2].NumHit++; } } //——————————————————————————————————————————————————————————————————————————————
Por último, vamos a describir el método N de la clase C_AO_CRO utilizado para cambiar la estructura de la molécula en los operadores de reacción química. El método se utiliza para generar un nuevo valor para la coordenada de la molécula dentro de un rango determinado:
1. El método acepta la coordenada de la molécula coord, que necesita ser modificada como referencia, y la posición de la coordenada coordPos en la estructura.
2. A continuación se calcula la distancia dist, que es la diferencia entre los valores máximo y mínimo del rango, multiplicada por el parámetro molecPerturb que indica la dispersión de los valores en las proximidades del valor de la coordenada actual.
3. A continuación, se definen los valores mínimo (min) y máximo (max) de una nueva coordenada. Estos valores son iguales al valor de coordenadas antiguo más o menos la distancia dist, pero no pueden ir más allá del rango especificado de rangeMin[coordPos] a rangeMax[coordPos].
4. Por último, el nuevo valor de la coordenada se genera utilizando la función de distribución gaussiana u.GaussDistribution, que toma el valor antiguo de la coordenada, los valores mínimo y máximo de la desviación típica, igual a 8.
//—————————————————————————————————————————————————————————————————————————————— void C_AO_CRO::N (double &coord, int coordPos) { double dist = (rangeMax [coordPos] - rangeMin [coordPos]) * molecPerturb; double min = coord - dist; if (min < rangeMin [coordPos]) min = rangeMin [coordPos]; double max = coord + dist; if (max > rangeMax [coordPos]) max = rangeMax [coordPos]; coord = u.GaussDistribution (coord, min, max, 8); } //——————————————————————————————————————————————————————————————————————————————
Hemos analizado todos los operadores químicos en función de su funcionalidad y su papel en el proceso de optimización. Este análisis nos ha proporcionado una visión profunda de los mecanismos que definen la dinámica de las moléculas en la modelización de reacciones químicas (CRO).
En el próximo artículo, construiremos el algoritmo y lo probaremos en las funciones de prueba. Configuraremos y combinaremos los operadores estudiados en un algoritmo CRO completo para que esté listo para su uso en tareas prácticas. A continuación, realizaremos una serie de experimentos con una amplia gama de funciones de prueba que nos permitirán evaluar la eficacia y solidez de nuestro planteamiento.
Una vez recibidos los resultados del trabajo, sacaremos conclusiones sobre los puntos fuertes y débiles del algoritmo, así como posibles direcciones para futuras mejoras. Esto nos permitirá no sólo mejorar nuestro método, sino también ofrecer recomendaciones útiles a los investigadores que trabajan en el campo de la optimización y modelización de procesos químicos.
Así pues, el próximo artículo será un paso clave en nuestra investigación, ya que pasaremos de la teoría a la práctica, probando y mejorando nuestro algoritmo para lograr los mejores resultados en la resolución de problemas de optimización complejos.
Traducción del ruso hecha por MetaQuotes Ltd.
Artículo original: https://www.mql5.com/ru/articles/15041
Advertencia: todos los derechos de estos materiales pertenecen a MetaQuotes Ltd. Queda totalmente prohibido el copiado total o parcial.
Este artículo ha sido escrito por un usuario del sitio web y refleja su punto de vista personal. MetaQuotes Ltd. no se responsabiliza de la exactitud de la información ofrecida, ni de las posibles consecuencias del uso de las soluciones, estrategias o recomendaciones descritas.
 Desarrollamos un asesor experto multidivisa (Parte 16): Efecto de diferentes historias de cotizaciones en los resultados de las pruebas
Desarrollamos un asesor experto multidivisa (Parte 16): Efecto de diferentes historias de cotizaciones en los resultados de las pruebas
 Visualización de transacciones en un gráfico (Parte 2): Visualización gráfica de datos
Visualización de transacciones en un gráfico (Parte 2): Visualización gráfica de datos
 La teoría del caos en el trading (Parte 2): Continuamos la inmersión
La teoría del caos en el trading (Parte 2): Continuamos la inmersión
 Desarrollo de un sistema de repetición (Parte 73): Una comunicación inusual (II)
Desarrollo de un sistema de repetición (Parte 73): Una comunicación inusual (II)
- Aplicaciones de trading gratuitas
- 8 000+ señales para copiar
- Noticias económicas para analizar los mercados financieros
Usted acepta la política del sitio web y las condiciones de uso
Aquí es artículo sobre práctica aplicación de en comercio. Leer, estudio y aplicar en práctica. le deseo éxito en comercio.
Artículos
Uso de algoritmos de optimización para configurar parámetros de EA sobre la marcha
Andrey Dik, 2024.06.07 14:30
El artículo discute los aspectos prácticos del uso de algoritmos de optimización para encontrar los mejores parámetros del EA sobre la marcha, así como la virtualización de las operaciones de trading y la lógica del EA. El artículo puede utilizarse como una instrucción para implementar algoritmos de optimización en un EA.Aquí está artículo sobre práctica aplicación de en comercio. Leer, estudio y aplicar en práctica. le deseo éxito en comercio.
Me gusta tu punto de vista, Andrei. Las grandes innovaciones nacen de la investigación. No siempre se reduce a dinero, y aprecio tu contribución a la comunidad.