
Algoritmos de optimización de la población: Algoritmo de forrajeo bacteriano (Bacterial Foraging Optimisation — BFO)
Contenido:
1. Introducción
2. Descripción del algoritmo
3. Resultados de las pruebas
1. Introducción
El algoritmo de forrajeo bacteriano (BFO) es una fascinante técnica de optimización que permite hallar soluciones aproximadas a problemas numéricos extremadamente complejos o imposibles para la maximización o minimización de funciones. El algoritmo es ampliamente reconocido como un algoritmo de optimización global que actualmente supone un interés especial para la optimización y el control distribuidos. El BFO se inspira en el comportamiento social de la Escherichia coli al buscar comida. El BFO ya ha atraído la atención de los investigadores por su eficacia a la hora de resolver problemas de optimización del mundo real derivados de diversas aplicaciones. La biología que subyace en la estrategia de búsqueda de alimentos de E. coli se emula de forma original y se usa como un sencillo algoritmo de optimización.
Bacterias como E.coli o Salmonella son algunos de los organismos más exitosos del planeta. Estas bacterias móviles tienen apéndices semirrígidos llamados flagelos, que usan para propulsarse con un movimiento de rotación. Cuando todos los flagelos giran en sentido contrario a las agujas del reloj, se crea un efecto de hélice y la bacteria se desplaza en una dirección más o menos rectilínea. Al hacerlo, la bacteria realiza un movimiento denominado natación (swims), en el que todos los flagelos giran en la misma dirección.
Los flagelos ayudan a la bacteria E.coli a voltearse o nadar, que son dos de las principales operaciones que efectúa la bacteria mientras busca alimento. Cuando giran el flagelo en el sentido de las agujas del reloj, cada flagelo impulsa la célula. A medida que el flagelo gira en distintas direcciones, la bacteria se da la vuelta, es decir, realiza un movimiento de volteo (tumble). Como resultado, la bacteria se voltea menos en un entorno favorable, mientras que en un lugar nocivo se voltea con mayor frecuencia para encontrar un gradiente de nutrientes. El movimiento de los flagelos en sentido contrario a las agujas del reloj ayuda a las bacterias a nadar a una gran velocidad.
En el algoritmo mencionado, el comportamiento de las bacterias se rige por un mecanismo denominado quimiotaxis bacteriana, que es la reacción motora de estos microorganismos ante un estímulo químico procedente del entorno. Este mecanismo permite a las bacterias moverse hacia los atrayentes (casi siempre nutrientes) y alejarse de los repelentes (sustancias potencialmente nocivas para las bacterias). Los receptores que detectan los atrayentes y repelentes se sitúan en los polos de las bacterias.
A causa de su pequeño tamaño, la bacteria es incapaz de captar la diferencia de concentración de sustancias beneficiosas y perjudiciales entre los polos. Las bacterias determinan los gradientes de dichas sustancias midiendo los cambios en sus concentraciones a medida que se desplazan. La velocidad de este movimiento puede alcanzar varias decenas de longitudes bacterianas por segundo. Por ejemplo, las bacterias E.coli suelen desplazarse a una velocidad que oscila entre 10 y 20 veces su longitud por segundo.
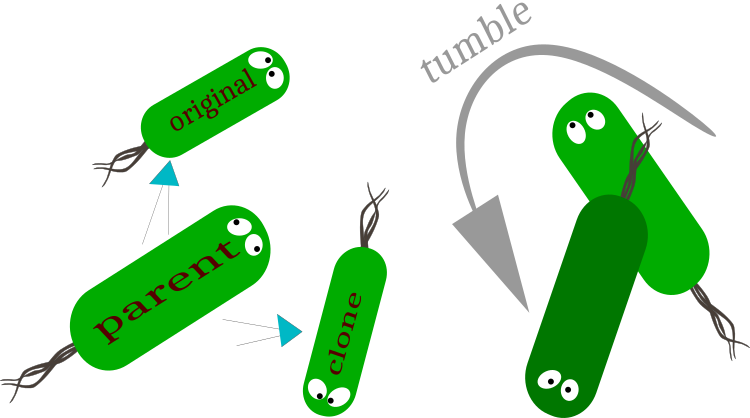
Figura 1. Replicación: división en la bacteria original (conservación del vector de movimiento) y la clonada (cambio del vector de movimiento).
Un volteo supone un cambio en el vector de movimiento de una bacteria.
Si la dirección de movimiento elegida por las bacterias se corresponde con un aumento de la concentración de atrayente (una disminución de la concentración de repelente), el tiempo hasta el siguiente volteo aumentará. Debido al pequeño tamaño de las bacterias, el movimiento browniano afecta mucho a sus movimientos. Como consecuencia, la bacteria solo se desplaza por término medio en dirección hacia las sustancias útiles y se aleja de las nocivas.
El mecanismo de desplazamiento bacteriano anteriormente mencionado no es el único. Por ejemplo, algunas bacterias tienen solo un flagelo. En este caso, las opciones de movimiento de la bacteria ofrecen distintos métodos de rotación y parada. No obstante, en todos los casos, si la bacteria se desplaza en la dirección correcta, la duración de este movimiento aumentará. Así, en general, la quimiotaxis bacteriana puede definirse como una compleja combinación de natación y volteo, que permite a las bacterias permanecer en zonas de alta concentración de nutrientes y evitar concentraciones inadmisibles de sustancias nocivas.
En el contexto de la tarea de optimización del motor de búsqueda, la quimiotaxis bacteriana también puede interpretarse como un mecanismo para optimizar el uso que hacen las bacterias de los recursos alimenticios conocidos, así como para encontrar nuevas zonas potencialmente más valiosas. Una población bacteriana de tamaño suficiente puede formar estructuras espaciales y temporales complejas, el efecto de la formación de estructuras en las poblaciones bacterianas. Este efecto puede deberse tanto a la quimiotaxis como a otras muchas causas.
Para algunas bacterias, la formación de dichas estructuras se debe a las propiedades reguladoras de sus productos metabólicos. Así, resultan posibles efectos similares basados en los fenómenos de magnetotaxis (sensibilidad al campo magnético), bioconvección, geotaxis negativa (movimiento preferencial de los microorganismos en contra de la dirección de la gravedad) y otros fenómenos. Por norma general, las bacterias recorren una mayor distancia en un entorno favorable. Cuando obtienen suficiente alimento, las bacterias crecen en longitud y, dada la temperatura adecuada, se rompen por la mitad, convirtiéndose en una réplica exacta de sí mismas.
Este fenómeno inspiró a Passino a introducir el evento de repetición en BFO. Si se da un cambio repentino en el entorno, o un ataque, el proceso quimiotáctico puede interrumpirse y el grupo de bacterias puede desplazarse a otro lugar. Esto supone un evento de eliminación y dispersión en una población bacteriana real, donde todas las bacterias de una región mueren o el grupo se dispersa en una nueva parte del entorno. Además, los procedimientos considerados de quimiotaxis y reproducción no resultan en general suficientes para encontrar el máximo global de la función objetivo de extremo múltiple, ya que estos procedimientos no permiten a las bacterias salir de los máximos locales de la función que han encontrado. El procedimiento de liquidación y dispersión está diseñado para superar este obstáculo. Según la selección natural (supervivencia del más apto), las bacterias poco adaptables serán aniquiladas y las más adaptables se reproducirán.
2. Descripción del algoritmo
La versión canónica del BFO incluye los siguientes pasos principales
- Inicio de la colonia bacteriana.
- Quimiotaxis.
- Enjambre.
- Reproducción.
- Liquidación y eliminación.
1. Inicialización del parámetro.
Las bacterias pueden formar complejos patrones espacio-temporales estables en algunos nutrientes semisólidos, y pueden sobrevivir en el entorno si inicialmente se colocan juntas en el centro del mismo. Además, en determinadas condiciones, segregarán señales intercelulares atrayentes, de forma que se agrupen y protejan mutuamente.
2. Quimiotaxis.
Las características de las bacterias que se desplazan en busca de alimento pueden determinarse de dos formas: nadando y volteándose juntas, lo que se conoce como quimiotaxis. Se dice que la bacteria "nada" si se mueve en la dirección correcta y "se voltea" si se desplaza hacia un entorno deteriorado.
3. Enjambre.
Para que la bacteria llegue al lugar más rico en alimentos, es deseable que la bacteria óptima, hasta un momento del periodo de búsqueda, intente atraer a otras bacterias para que converjan juntas más rápidamente en el lugar deseado. Para ello, añadiremos una función de penalización a la función de coste original, basada en la distancia relativa de cada bacteria respecto a la bacteria más adaptada a esta duración de búsqueda. Por último, cuando todas las bacterias se fusionen en un punto de decisión, esta función de penalización pasará a ser cero. El efecto del enjambre consiste en que las bacterias se reúnen en grupos y se mueven en patrones concéntricos con una alta densidad de bacterias.
4. Reproducción.
El conjunto inicial de bacterias, tras pasar por varias fases quimiotácticas, llega a la fase de reproducción. Aquí, el mejor conjunto de bacterias se divide en dos grupos: la mitad más sana es sustituida por la otra mitad de las bacterias, que se destruyen debido a su menor capacidad para encontrar alimento. Esto hace que la población bacteriana sea constante durante la evolución.
5. Eliminación y dispersión.
Durante la evolución, puede producirse algún imprevisto que altere drásticamente el proceso evolutivo fluido y provoque la eliminación y/o dispersión de muchas bacterias en un nuevo entorno. Irónicamente, en lugar de interrumpir el crecimiento quimiotáctico normal de un conjunto de bacterias, este acontecimiento desconocido puede situar a un nuevo conjunto de bacterias más cerca de donde se ubica el alimento. Desde una perspectiva amplia, la eliminación y la dispersión forman parte del comportamiento a largo plazo de cualquier población. Aplicadas a la optimización, ayudan a reducir el estancamiento que a menudo se observa en estos algoritmos de búsqueda paralela.
Nuestra implementación del BFO difiere ligeramente de la versión canónica: dedicaremos más atención a las diferencias al analizar los fragmentos específicos del código con la justificación de estos cambios. En general, los cambios en la implementación no pueden considerarse significativos, por lo que no asignaremos el marcador "m" (versión modificada) al nombre del algoritmo. Eso sí, cabe señalar que los resultados han mejorado tras introducir los cambios en el algoritmo.
A continuación, veremos el algoritmo y el código de nuestra implementación.
Pasos del algoritmo:
1. Inicialización de la colonia de bacterias.
2. Medición de la salud de las bacterias (adaptabilidad).
3. ¿Replicación?
3.1. Sí. Realizamos la replicación.
3.2. No. p4.
4. ¿Vieja (límite de vida alcanzado)?
4.1. Sí. Realizamos el volteo (cambio de vector de movimiento).
4.2. No. p.5.
5. ¿Se mueve en la dirección correcta?
5.1. Sí. Continuación del movimiento con el mismo vector.
5.2. No. Realizamos el volteo (cambio de vector de movimiento).
6. Medición de la salud de las bacterias (adaptabilidad).
7. Continuamos con el punto 3 hasta que se cumpla el criterio de parada.
El esquema lógico del algoritmo se muestra en la figura 1.
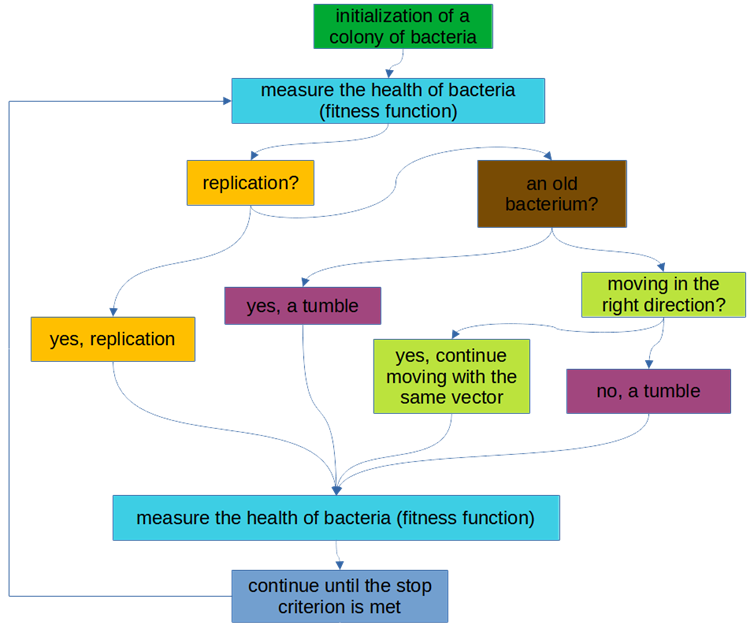
Figura 2. Esquema de bloques con la lógica del algoritmo de BFO.
Vamos a analizar el código.
Para describir una bacteria, lo más adecuado será usar una estructura que contenga arrays de coordenadas y vectores de movimiento. Los valores de salud actuales y anteriores de la bacteria y el contador de vida. En esencia, necesitamos un contador de vida para limitar la cantidad de movimiento secuencial en una dirección, a diferencia de la versión original, donde una vez se alcance el límite de vida la bacteria será destruida y se creará una nueva en una ubicación aleatoria en el espacio de búsqueda. Pero como hemos tocado este tema en artículos anteriores, crear un nuevo agente en una ubicación aleatoria carecerá de sentido práctico y solo degradará las capacidades de búsqueda. En este caso, resulta más apropiado crear un nuevo agente en la ubicación de la mejor solución, o proseguir desde la ubicación actual pero cambiando el vector direccional. La segunda opción ha obtenido mejores resultados.
La versión canónica usa un vector de movimiento constante y, con un gran número de vidas, esto haría que la bacteria se moviera a lo largo de una línea recta a través del espacio de búsqueda; si esta línea no pasara por ningún extremo mejor entonces este proceso de movimiento rectilíneo ocurriría indefinidamente, pero aquí el contador actúa como un volteo forzoso, permitiendo que la bacteria evite el movimiento rectilíneo a lo largo de su vida, y en características sin gradiente, eventualmente todavía conducirá a un lugar donde pueda mejorar su adaptabilidad.
//—————————————————————————————————————————————————————————————————————————————— struct S_Bacteria { double c []; //coordinates double v []; //vector double f; //current health double fLast; //previous health double lifeCNT; //life counter }; //——————————————————————————————————————————————————————————————————————————————
Declaramos la clase del algoritmo BFO como C_AO_BFO. La clase contiene las declaraciones de la colonia de bacterias, los límites del espacio de búsqueda, el valor de la mejor solución y el array de coordenadas de la mejor solución. También declaramos los valores constantes de los parámetros del algoritmo.
//—————————————————————————————————————————————————————————————————————————————— class C_AO_BFO { //---------------------------------------------------------------------------- public: S_Bacteria b []; //bacteria public: double rangeMax []; //maximum search range public: double rangeMin []; //manimum search range public: double rangeStep []; //step search public: double cB []; //best coordinates public: double fB; //FF of the best coordinates public: void Init (const int paramsP, //number of opt. parameters const int populationSizeP, //population size const double lambdaP, //lambda const double reproductionP, //probability of reproduction const int lifeCounterP); //life counter public: void Swimming (); public: void Evaluation (); //---------------------------------------------------------------------------- private: double NewVector (int paramInd); private: S_Bacteria bT []; //bacteria private: double v []; private: int ind []; private: double val []; private: int populationSize; //population size private: int parameters; //number of optimized parameters private: double lambda; //lambda private: double reproduction; //probability of reproduction private: int lifeCounter; //life counter private: bool evaluation; private: void Sorting (); private: double SeInDiSp (double In, double InMin, double InMax, double Step); private: double RNDfromCI (double min, double max); }; //——————————————————————————————————————————————————————————————————————————————
El método abierto Init () está diseñado para inicializar las variables constantes, asignar los tamaños de los arrays, restablecer las banderas y valorar la mejor solución.
//—————————————————————————————————————————————————————————————————————————————— void C_AO_BFO::Init (const int paramsP, //number of opt. parameters const int populationSizeP, //population size const double lambdaP, //lambda const double reproductionP, //probability of reproduction const int lifeCounterP) //life counter { fB = -DBL_MAX; evaluation = false; parameters = paramsP; populationSize = populationSizeP; lambda = lambdaP; reproduction = reproductionP; lifeCounter = lifeCounterP; ArrayResize (rangeMax, parameters); ArrayResize (rangeMin, parameters); ArrayResize (rangeStep, parameters); ArrayResize (v, parameters); ArrayResize (ind, populationSize); ArrayResize (val, populationSize); ArrayResize (b, populationSize); ArrayResize (bT, populationSize); for (int i = 0; i < populationSize; i++) { ArrayResize (b [i].c, parameters); ArrayResize (b [i].v, parameters); b [i].f = -DBL_MAX; b [i].fLast = -DBL_MAX; ArrayResize (bT [i].c, parameters); ArrayResize (bT [i].v, parameters); } ArrayResize (cB, parameters); } //——————————————————————————————————————————————————————————————————————————————
El primer método abierto obligatorio que se llama en cada iteración es Swimming(), o natación bacteriana, que incorpora toda la lógica básica del algoritmo.
void C_AO_BFO::Swimming ()
{}
Vamos a echar un vistazo detallado al método, por partes. En la primera iteración, cuando la bandera evaluation es igual a false, dispersamos las bacterias aleatoriamente por todo el espacio de búsqueda con una distribución uniforme. En este punto la salud actual (aptitud) y la anterior no son conocidas, así que asignaremos a las variables correspondientes el valor - DBL_MAX. Luego comprobamos si hay valores atípicos en las coordenadas generadas aleatoriamente y aplicamos un cambio de paso a los parámetros que se van a optimizar. En esta iteración, deberemos configurar un vector de movimiento individual para cada bacteria utilizando el método privado NewVector() (que se explica más adelante). Todas las bacterias nadan uniformemente hacia delante y en línea recta con el mismo vector individual hasta que se den las condiciones para su cambio.
//---------------------------------------------------------------------------- if (!evaluation) { fB = -DBL_MAX; for (int s = 0; s < populationSize; s++) { for (int k = 0; k < parameters; k++) { b [s].c [k] = RNDfromCI (rangeMin [k], rangeMax [k]); b [s].c [k] = SeInDiSp (b [s].c [k], rangeMin [k], rangeMax [k], rangeStep [k]); v [k] = rangeMax [k] - rangeMin [k]; b [s].v [k] = NewVector (k); b [s].f = -DBL_MAX; b [s].fLast = -DBL_MAX; b [s].lifeCNT = 0; } } evaluation = true; }
En la segunda iteración y en las siguientes, se realizan operaciones de natación, volteo, replicación y eliminación de bacterias cuando se alcanza el límite de vida. Lo primero en la lógica es la operación de reproducción (con una probabilidad especificada en los parámetros); esta supone que la colonia bacteriana se ha clasificado en orden descendente de salud en la iteración anterior.
La reproducción en el algoritmo cumple una función importante: acelerar la convergencia del algoritmo mejorando el "acervo genético" de la colonia de bacterias. La operación consiste en una división imaginaria de la bacteria en dos partes; en este caso, además, solo se permite que se divida la primera mitad (la mejor) de la colonia. La primera mitad de la colonia se divide en dos, y la versión original de los padres se coloca en la segunda mitad de la colonia. La bacteria madre seguirá moviéndose con el mismo vector que su clon. El clon permanecerá en su lugar en la colonia y se le asignará un nuevo vector de movimiento. La bacteria madre y su clon continuarán a partir del punto del espacio donde se produjo la división.
r = RNDfromCI (0.0, 1.0); //========================================================================== if (r < reproduction) { int st = populationSize / 2; for (int s = 0; s < st; s++) { //bacterium original-------------------------------------------------- for (int k = 0; k < parameters; k++) { b [st + s].v [k] = b [s].v [k]; b [st + s].c [k] = b [s].c [k] + b [s].v [k]; b [st + s].c [k] = SeInDiSp (b [st + s].c [k], rangeMin [k], rangeMax [k], rangeStep [k]); b [st + s].fLast = b [s].f; b [st + s].lifeCNT++; } //bacterium clone------------------------------------------------------- for (int k = 0; k < parameters; k++) { b [s].v [k] = NewVector (k); b [s].c [k] = b [s].c [k] + b [s].v [k]; b [s].c [k] = SeInDiSp (b [s].c [k], rangeMin [k], rangeMax [k], rangeStep [k]); b [s].fLast = b [s].f; b [s].lifeCNT = 0; } } }
A continuación, si no se cumple la probabilidad de replicación, realizaremos una comprobación para asegurarnos de que la bacteria ha alcanzado su límite de vida. En la versión canónica del algoritmo, hay que eliminar la bacteria "vieja" y crear en su lugar, en la lista de bacterias, una nueva bacteria en un lugar aleatorio del espacio de búsqueda. En general, las operaciones de reproducción y quimiotaxis no son suficientes para encontrar el máximo global de la función de aptitud de extremo múltiple, ya que
estos procedimientos impiden que las bacterias abandonen los mínimos locales encontrados. El procedimiento de liquidación está precisamente diseñado para superar esta desventaja. No obstante, la experimentación con este y otros algoritmos ha demostrado que, en tal caso, resulta más eficaz limitarse a cambiar el vector de movimiento. En este caso, el contador de vidas se pondrá a cero. El contador es un disparador para cambiar de dirección tras un número determinado de pasos (vidas). El número total de bacterias resultante de la replicación y la eliminación se mantiene constante.
if (b [s].lifeCNT >= lifeCounter) { for (int k = 0; k < parameters; k++) { b [s].v [k] = NewVector (k); b [s].c [k] = b [s].c [k] + b [s].v [k]; b [s].c [k] = SeInDiSp (b [s].c [k], rangeMin [k], rangeMax [k], rangeStep [k]); b [s].fLast = b [s].f; b [s].lifeCNT = 0; } }
Si el límite de vidas no se ha agotado, entonces implementaremos una comprobación para ver si la bacteria se está moviendo hacia un gradiente mejorado de la función de aptitud. Es decir, comprobaremos dos valores de salud, la iteración actual y la iteración anterior. Si la salud ha mejorado, el vector de movimiento se mantendrá, de lo contrario la bacteria tendrá que voltearse (cambiar el vector de movimiento).
En la versión canónica hay una comprobación estricta de "más que" de la salud actual y la anterior, mientras que en mi versión hay una comprobación de "más que o igual a", lo cual permite a las bacterias seguir moviéndose incluso en una sección completamente "horizontal" de la superficie investigada, donde no hay gradiente de función de aptitud; de lo contrario, las bacterias darían tumbos sin fin en un mismo lugar (sin cambio de salud y, por tanto, sin dirección hacia la que nadar). Este paso también debe comprobar si las nuevas coordenadas se encuentran fuera del espacio de búsqueda.
else { if (b [s].f >= b [s].fLast) { for (int k = 0; k < parameters; k++) { b [s].c [k] = b [s].c [k] + b [s].v [k]; b [s].c [k] = SeInDiSp (b [s].c [k], rangeMin [k], rangeMax [k], rangeStep [k]); b [s].fLast = b [s].f; b [s].lifeCNT++; } } else { for (int k = 0; k < parameters; k++) { b [s].v [k] = NewVector (k); b [s].c [k] = b [s].c [k] + b [s].v [k]; b [s].c [k] = SeInDiSp (b [s].c [k], rangeMin [k], rangeMax [k], rangeStep [k]); b [s].fLast = b [s].f; b [s].lifeCNT++; } } }
NewVecror() es un método privado para cambiar el vector de movimiento (volteo). El método se aplica a cada coordenada. La idea es sencilla: multiplicamos la diferencia entre los límites de búsqueda de la coordenada v correspondiente por un número aleatorio r del intervalo [-1,0;1,0] y multiplicamos por el factor de escala lambda (uno de los parámetros del algoritmo). La bacteria usa el vector de movimiento sin cambios hasta que se agota el límite de vida (se crea una nueva bacteria en el mismo lugar, pero con un nuevo vector de movimiento), al replicarse (el clon tiene un nuevo vector), o al deteriorarse su salud (intenta encontrar un nuevo entorno más favorable).
//—————————————————————————————————————————————————————————————————————————————— double C_AO_BFO::NewVector (int paramInd) { double r = RNDfromCI (-1.0, 1.0); return lambda * v [paramInd] * r; } //——————————————————————————————————————————————————————————————————————————————
El segundo método abierto obligatorio en cada iteración, Evaluation(), llama al método de clasificación, y luego comprueba las actualizaciones de la solución global, comparando la salud de la mejor bacteria en la colonia clasificada con la mejor solución encontrada.
//—————————————————————————————————————————————————————————————————————————————— void C_AO_BFO::Evaluation () { Sorting (); if (b [0].f > fB) { fB = b [0].f; ArrayCopy (cB, b [0].c, 0, 0, WHOLE_ARRAY); } } //——————————————————————————————————————————————————————————————————————————————
3. Resultados de las pruebas
Resultados del banco de pruebas para BFO:
2023.01.21 12:52:46.501 Test_AO_BFO (.US500Cash,M1) C_AO_BFO:50;0.01;0.8;100
2023.01.21 12:52:46.501 Test_AO_BFO (.US500Cash,M1) =============================
2023.01.21 12:52:48.451 Test_AO_BFO (.US500Cash,M1) 5 Rastrigin's; Func runs 10000 result: 72.94540549092933
2023.01.21 12:52:48.451 Test_AO_BFO (.US500Cash,M1) Score: 0.90383
2023.01.21 12:52:51.778 Test_AO_BFO (.US500Cash,M1) 25 Rastrigin's; Func runs 10000 result: 54.75744312933767
2023.01.21 12:52:51.778 Test_AO_BFO (.US500Cash,M1) Score: 0.67848
2023.01.21 12:53:21.197 Test_AO_BFO (.US500Cash,M1) 500 Rastrigin's; Func runs 10000 result: 40.668487609790404
2023.01.21 12:53:21.197 Test_AO_BFO (.US500Cash,M1) Score: 0.50391
2023.01.21 12:53:21.197 Test_AO_BFO (.US500Cash,M1) =============================
2023.01.21 12:53:23.211 Test_AO_BFO (.US500Cash,M1) 5 Forest's; Func runs 10000 result: 0.8569098398505888
2023.01.21 12:53:23.211 Test_AO_BFO (.US500Cash,M1) Score: 0.48471
2023.01.21 12:53:26.878 Test_AO_BFO (.US500Cash,M1) 25 Forest's; Func runs 10000 result: 0.37618151461180294
2023.01.21 12:53:26.878 Test_AO_BFO (.US500Cash,M1) Score: 0.21279
2023.01.21 12:54:22.339 Test_AO_BFO (.US500Cash,M1) 500 Forest's; Func runs 10000 result: 0.08748190028975696
2023.01.21 12:54:22.339 Test_AO_BFO (.US500Cash,M1) Score: 0.04948
2023.01.21 12:54:22.339 Test_AO_BFO (.US500Cash,M1) =============================
2023.01.21 12:54:28.849 Test_AO_BFO (.US500Cash,M1) 5 Megacity's; Func runs 10000 result: 4.8
2023.01.21 12:54:28.849 Test_AO_BFO (.US500Cash,M1) Score: 0.40000
2023.01.21 12:54:40.089 Test_AO_BFO (.US500Cash,M1) 25 Megacity's; Func runs 10000 result: 2.216
2023.01.21 12:54:40.089 Test_AO_BFO (.US500Cash,M1) Score: 0.18467
2023.01.21 12:55:24.640 Test_AO_BFO (.US500Cash,M1) 500 Megacity's; Func runs 10000 result: 0.4232
2023.01.21 12:55:24.640 Test_AO_BFO (.US500Cash,M1) Score: 0.03527
Si nos fijamos en los resultados impresos del banco de pruebas, resulta demasiado pronto para sacar conclusiones inequívocas; debemos analizar los resultados en relación con los de los demás participantes en la prueba.
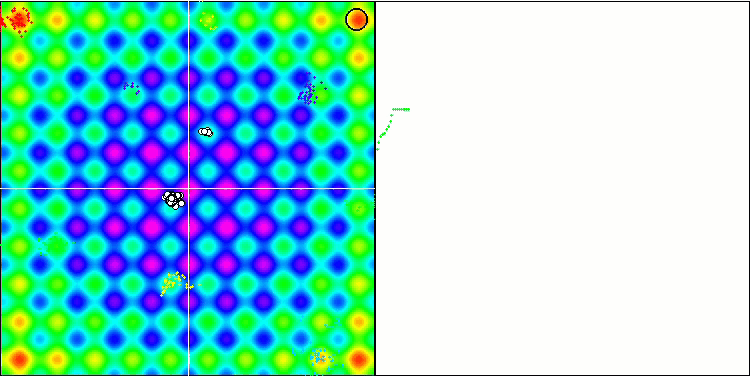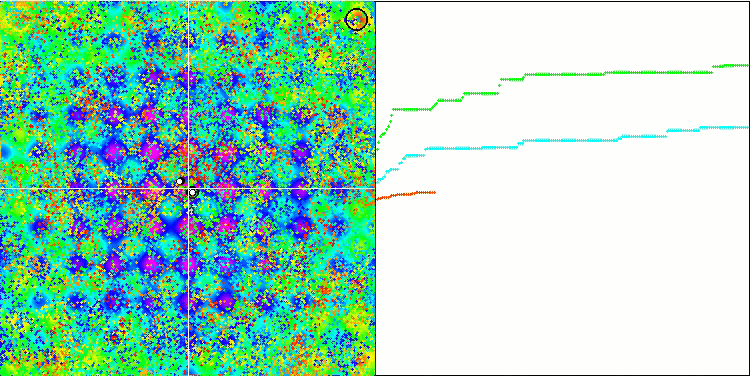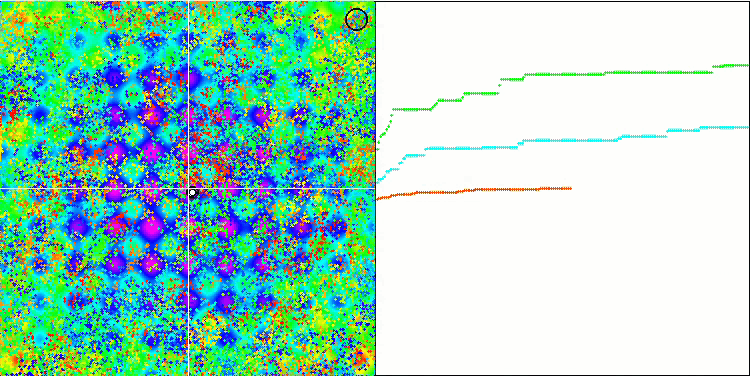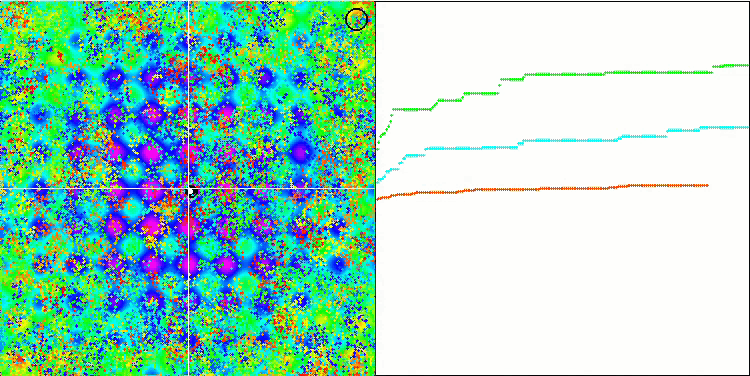
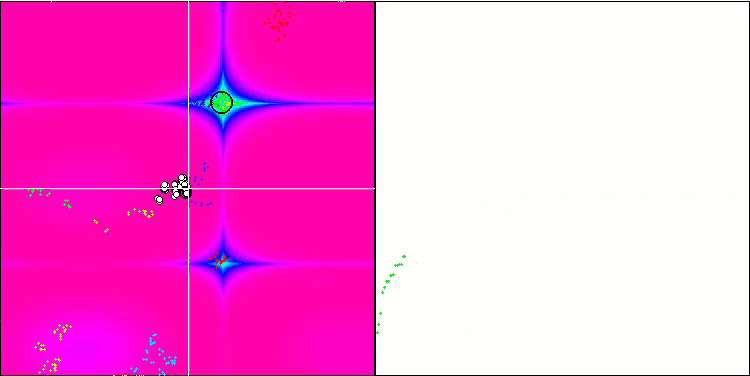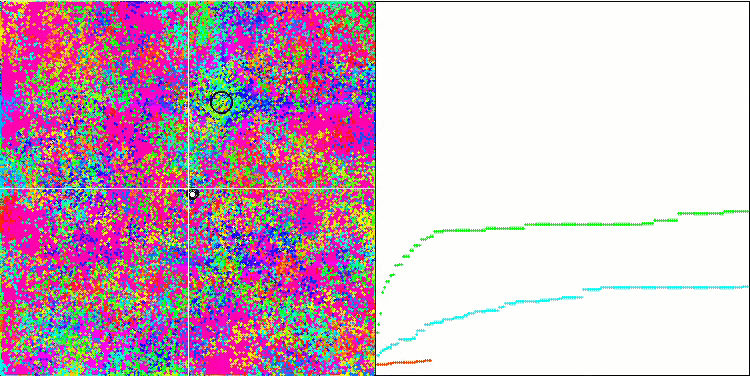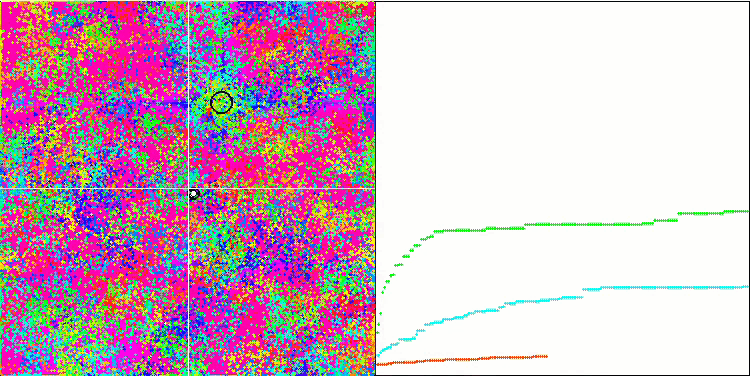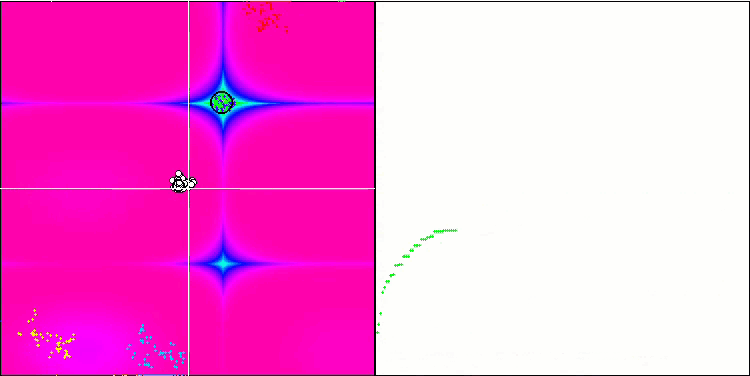
BFO en la función de prueba Rastrigin.
BFO en la función de prueba Forest.
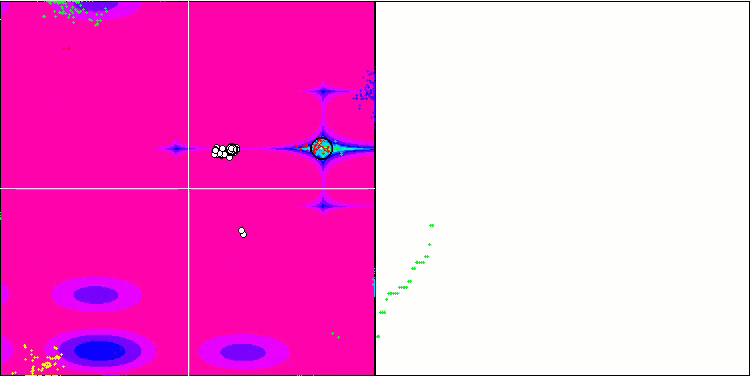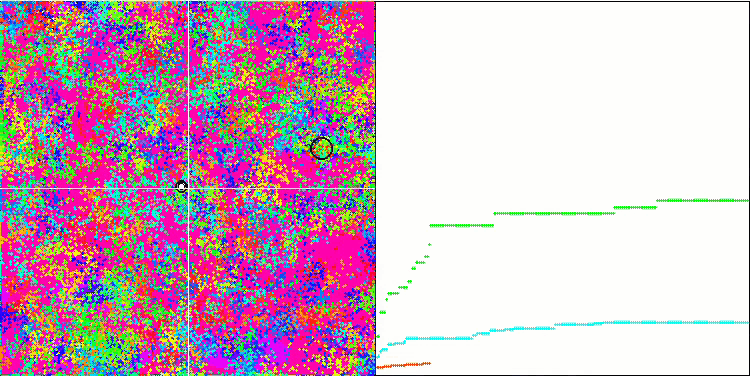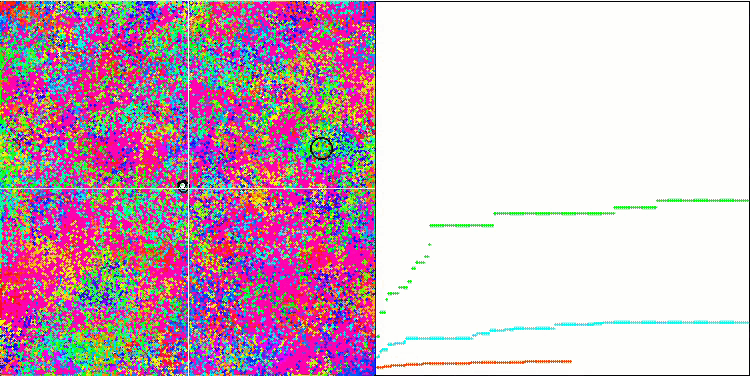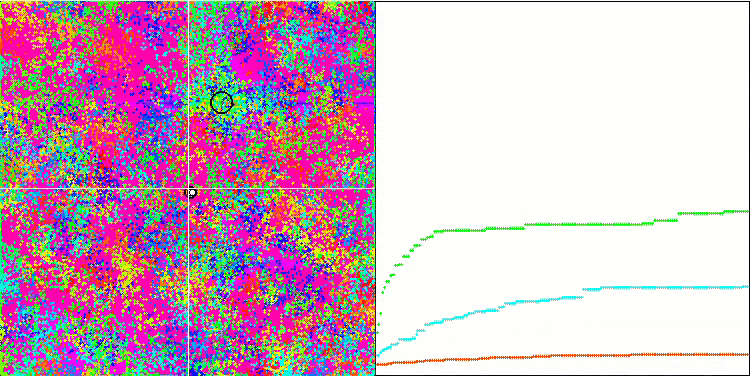
BFO en la función de prueba Megacity.
Prestemos atención a la representación de las pruebas. La animación ha confirmado la decisión de sustituir el signo "más que" por "más que o igual a" en nuestro algoritmo: esto ha permitido a las bacterias seguir moviéndose en las secciones horizontales de las funciones de prueba. Esto se nota especialmente en las funciones Forest y Megacity, las bacterias intentan seguir adelante incluso donde no hay gradiente de función en absoluto. También debemos destacar la capacidad de la colonia bacteriana para dividirse en varias colonias distintas separadas territorialmente de forma visual en extremos locales diferentes, lo cual puede considerarse claramente una propiedad positiva, aunque no existan técnicas lógicas para la formación de subcolonias en el algoritmo. En general, podemos observar un movimiento uniforme de las bacterias a través del espacio de búsqueda sin intentar un salto brusco en ninguna dirección, lo cual se explica por un movimiento progresivo uniforme: la quimiotaxis.
| AO | Description | Rastrigin | Rastrigin final | Forest | Forest final | Megacity (discrete) | Megacity final | Final result | ||||||
| 10 params (5 F) | 50 params (25 F) | 1000 params (500 F) | 10 params (5 F) | 50 params (25 F) | 1000 params (500 F) | 10 params (5 F) | 50 params (25 F) | 1000 params (500 F) | ||||||
| IWO | invasive weed optimization | 1,00000 | 1,00000 | 0,33519 | 2,33519 | 0,79937 | 0,46349 | 0,41071 | 1,67357 | 0,75912 | 0,44903 | 0,94088 | 2,14903 | 100,000 |
| ACOm | ant colony optimization M | 0,36118 | 0,26810 | 0,17991 | 0,80919 | 1,00000 | 1,00000 | 1,00000 | 3,00000 | 1,00000 | 1,00000 | 0,10959 | 2,10959 | 95,996 |
| COAm | cuckoo optimization algorithm M | 0,96423 | 0,69756 | 0,28892 | 1,95071 | 0,64504 | 0,34034 | 0,21362 | 1,19900 | 0,67153 | 0,34273 | 0,45422 | 1,46848 | 74,204 |
| FAm | firefly algorithm M | 0,62430 | 0,50653 | 0,18102 | 1,31185 | 0,55408 | 0,42299 | 0,64360 | 1,62067 | 0,21167 | 0,28416 | 1,00000 | 1,49583 | 71,024 |
| BA | bat algorithm | 0,42290 | 0,95047 | 1,00000 | 2,37337 | 0,17768 | 0,17477 | 0,33595 | 0,68840 | 0,15329 | 0,07158 | 0,46287 | 0,68774 | 59,650 |
| ABC | artificial bee colony | 0,81573 | 0,48767 | 0,22588 | 1,52928 | 0,58850 | 0,21455 | 0,17249 | 0,97554 | 0,47444 | 0,26681 | 0,35941 | 1,10066 | 57,237 |
| BFO | bacterial foraging optimization | 0,70129 | 0,46155 | 0,11627 | 1,27911 | 0,41251 | 0,26623 | 0,26695 | 0,94569 | 0,42336 | 0,34491 | 0,50973 | 1,27800 | 55,516 |
| FSS | fish school search | 0,48850 | 0,37769 | 0,11006 | 0,97625 | 0,07806 | 0,05013 | 0,08423 | 0,21242 | 0,00000 | 0,01084 | 0,18998 | 0,20082 | 20,109 |
| PSO | particle swarm optimisation | 0,21339 | 0,12224 | 0,05966 | 0,39529 | 0,15345 | 0,10486 | 0,28099 | 0,53930 | 0,08028 | 0,02385 | 0,00000 | 0,10413 | 14,232 |
| RND | random | 0,17559 | 0,14524 | 0,07011 | 0,39094 | 0,08623 | 0,04810 | 0,06094 | 0,19527 | 0,00000 | 0,00000 | 0,08904 | 0,08904 | 8,142 |
| GWO | grey wolf optimizer | 0,00000 | 0,00000 | 0,00000 | 0,00000 | 0,00000 | 0,00000 | 0,00000 | 0,00000 | 0,18977 | 0,04119 | 0,01802 | 0,24898 | 1,000 |
Es hora de analizar los resultados de las pruebas. El BFO se sitúa a la mitad de la tabla de clasificación, con una puntuación total de algo más de 55 como parte de la lista actual de algoritmos participantes. No podemos decir que los resultados sean impresionantes, pero tampoco son malos. En particular, hemos encontrado buenos resultados para la función Rastrigin con 10 variables; con 50 y 1000 variables los resultados son notablemente peores. El algoritmo tampoco ha funcionado especialmente bien en la función Forest. Sorprende ver un comportamiento relativamente bueno en la función Megacity discreta, a pesar de que el algoritmo no ofrece ningún mecanismo para trabajar con este tipo de funciones. Asimismo, muestra una buena escalabilidad en comparación con otros algoritmos (prueba de Megacity con 1000 parámetros).
El BFO es un campo científico que ofrece un amplio abanico de posibilidades. Hay una serie de aspectos del forrajeo bacteriano y de la búsqueda de alimentos en animales en general que pueden modelarse para mejorar el rendimiento de la optimización. Para el algoritmo de BFO, la adaptación automática de los parámetros de ajuste puede ser especialmente importante, ya que hay muchos parámetros y el algoritmo puede ofrecer mejores resultados, lo cual es motivo para experimentar adicionalmente.
El BFO presenta una serie de ventajas, como su baja sensibilidad a los valores iniciales de las coordenadas durante la inicialización y la selección de parámetros, su buena fiabilidad, su lógica sencilla, su fácil implementación, su capacidad de paralelizar y su búsqueda global. Así, el algoritmo de BFO se utiliza para resolver una amplia gama de problemas de optimización. Sin embargo, el BFO también presenta una serie de desventajas, como la lentitud de convergencia, la incapacidad para superar los óptimos locales en algunos casos y la longitud fija de los pasos. El BFO es una metaheurística, es decir, supone simplemente una estructura conceptual que puede utilizarse para desarrollar modificaciones de un algoritmo. La versión de BFO que hemos presentado aquí es solo una de las muchas variantes posibles, y debe considerarse como un punto de partida para la experimentación y no como la última palabra sobre el tema.
Histograma de los resultados de la prueba del algoritmo en la figura 3.
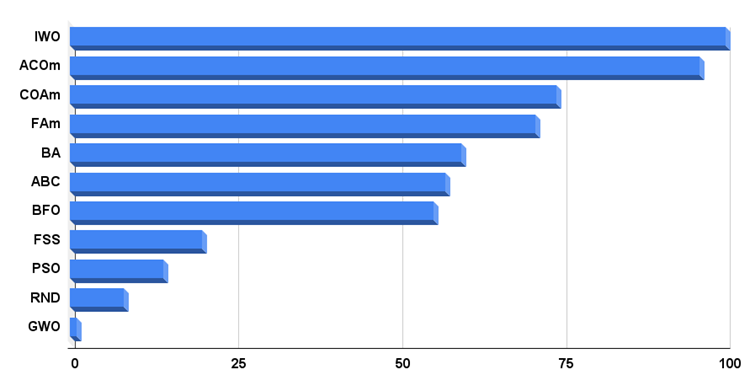
Figura 3. Histograma con los resultados finales de los algoritmos de prueba.
Conclusiones sobre las propiedades del algoritmo de optimización de forrajeo bacteriano (BFO):
Ventajas:
1. Es rápido.
2. Su lógica es simple.
3. Aunque sea lento, sigue convergiendo a lo largo de las iteraciones.
Desventajas:
1. Su convergencia es lenta.
2. No se ofrecen métodos para escapar de los extremos locales.
Traducción del ruso hecha por MetaQuotes Ltd.
Artículo original: https://www.mql5.com/ru/articles/12031
Advertencia: todos los derechos de estos materiales pertenecen a MetaQuotes Ltd. Queda totalmente prohibido el copiado total o parcial.
Este artículo ha sido escrito por un usuario del sitio web y refleja su punto de vista personal. MetaQuotes Ltd. no se responsabiliza de la exactitud de la información ofrecida, ni de las posibles consecuencias del uso de las soluciones, estrategias o recomendaciones descritas.
 Recetas MQL5 - Base de datos de eventos macroeconómicos
Recetas MQL5 - Base de datos de eventos macroeconómicos
- Aplicaciones de trading gratuitas
- 8 000+ señales para copiar
- Noticias económicas para analizar los mercados financieros
Usted acepta la política del sitio web y las condiciones de uso
1- Es el número de parámetros:
2- Es el tamaño de la población:
3. Se estima el número de épocas:
¿Lo he sacado todo bien del código?
1. No. Las funciones de prueba son bidimensionales, es decir, por ejemplo:
significa en ruso 5 funciones de prueba, por lo que se multiplica por 2 - 10 parámetros optimizados25 - 50 parámetros optimizados
500 - 1000 parámetros optimizados
2. Sí.
3. Sí, así es, el número estimado de épocas se hace para que el número total de ejecuciones FF sea el mismo y no dependa de la elección del tamaño de la población en los algoritmos, es decir, para que las pruebas de los algoritmos sean justas con diferentes parámetros de tamaño de población en diferentes algoritmos.
De acuerdo, gracias.
Todos estos algoritmos, de la serie de artículos, son parralelables, no he mirado los otros? Creo que me vendría bien, es útil, sólo que la función a optimizar es más compleja, tiene un número dinámico de parámetros, tanto reales como enteros, y con distintos rangos, hay que resolver el problema a posteriori
Ya veo, gracias.
Todos estos algoritmos, de la serie de artículos, son parralelables, no he mirado los otros? Creo que me vendría bien, es útil, sólo que la función que se optimiza es más compleja, tiene un número dinámico de parámetros, tanto reales como enteros, y con distintos rangos, hay que resolver el problema después.
gran trabajo 👏