
Algoritmo de otimização por reações químicas — Chemical Reaction Optimization, CRO (Parte I): A química dos processos na otimização
Conteúdo:
1. Introdução
A otimização por reações químicas (CRO) é um método fascinante e inovador, inspirado pela própria essência das transformações químicas. Imagine observar a dança das moléculas, onde cada movimento e colisão desempenha um papel essencial na resolução de problemas complexos. Este método combina habilmente os princípios de conservação de energia, decomposição e síntese molecular, criando uma abordagem flexível e adaptativa para a otimização.
CRO é uma técnica que liga livremente as reações químicas à otimização, usando princípios gerais de interação molecular, determinados pelos dois primeiros princípios da termodinâmica. O algoritmo Chemical Reaction Optimization (CRO) foi proposto e publicado por Lam e Li em 2012.
A primeira lei da termodinâmica (lei da conservação da energia) afirma que a energia não pode ser criada ou destruída, mas pode ser transformada de uma forma para outra e transferida de uma entidade para outra. No contexto do CRO, um sistema químico reagente é composto por substâncias e pelo ambiente, e cada partícula possui energia potencial e cinética.
A segunda lei da termodinâmica afirma que a entropia de um sistema tende a aumentar, sendo a entropia uma medida de desordem; o sistema anseia por mais liberdade. A energia potencial é a energia armazenada em uma molécula em relação à sua configuração molecular. Quando a energia potencial das moléculas é liberada e transformada em energia cinética, o sistema se torna cada vez mais caótico. No entanto, é nesse caos que o CRO encontra sua força, capturando e direcionando os fluxos de energia para uma solução ideal.
Por exemplo, quando moléculas com maior energia cinética (convertida a partir de energia potencial) se movem mais rapidamente, o sistema se torna mais desordenado, e sua entropia aumenta. Assim, todos os sistemas reagentes buscam atingir um estado de equilíbrio, onde a energia potencial é minimizada. No CRO, capturamos esse fenômeno, convertendo a energia potencial em energia cinética e, gradualmente, dissipando a energia das moléculas químicas no ambiente.
Um sistema químico, quando submetido a uma reação química, torna-se instável e, ao possuir energia excessiva, tenta se livrar dela para se estabilizar. Ele é composto por moléculas, que são as menores partículas de um composto e são classificadas em diferentes tipos com base em suas principais propriedades químicas.
As reações químicas formam uma dança complexa, na qual moléculas colidem, criam e rompem ligações, alterando suas estruturas. Cada passo dessa dança é uma sequência de subreações que conduzem a produtos mais estáveis, com energia mínima. As moléculas armazenam energia em suas ligações químicas, e até mesmo pequenas alterações em suas estruturas podem resultar em transformações surpreendentes, levando à formação de produtos mais estáveis. A estrutura molecular influencia o comportamento químico de um composto, e pequenas mudanças nessa estrutura podem resultar em diferenças significativas em suas propriedades químicas.
As reações químicas são iniciadas por colisões moleculares, que podem ser: unimoleculares, com substâncias externas; bimoleculares, com outras moléculas. Essas colisões podem ser eficientes ou ineficientes, dependendo de fatores como a energia de ativação e os efeitos estéricos. Dessa forma, as moléculas dançam, entrelaçando-se e separando-se, criando novas formas e estruturas.
É importante observar que as reações químicas frequentemente, mas nem sempre, resultam na formação de produtos mais estáveis com energia de Gibbs mais baixa. Esse processo ocorre por meio de estágios sequenciais e/ou paralelos, envolvendo a formação de compostos intermediários e a passagem por estados de transição. Compreender esses processos é necessário para determinar as condições ideais para as reações químicas e desvendar os mecanismos de transformação dos compostos.
Além disso, a síntese química pode ocorrer como resultado de colisões eficientes ou ineficientes dos reagentes. A eficiência das colisões depende de fatores como energia de ativação, efeitos estéricos, entre outros. A síntese intermolecular, ao contrário da intramolecular, leva a mudanças mais significativas nas estruturas moleculares.
O algoritmo CRO é como um coreógrafo virtuoso, que utiliza as leis da química como notas para criar soluções de otimização incrivelmente complexas e elegantes. Agora, vamos decompor tudo de forma organizada, do complicado ao simples.
2. Implementação de operadores químicos
A unidade fundamental no CRO é a "molécula", onde cada uma em uma população possui características definidas, como "energia potencial e cinética", "estruturas moleculares", entre outras. As reações elementares definem as interações entre as moléculas. Podemos definir uma classe em que os campos de dados representam as características das moléculas e os métodos descrevem as reações elementares.
Um dos conceitos fundamentais no CRO é a conservação de energia. Esse princípio garante que a energia total do sistema permaneça constante durante todo o processo de otimização. A conservação de energia estabelece as transições entre os diferentes estados moleculares durante as reações, mantendo o equilíbrio energético que influencia a busca por soluções ideais.
Ao utilizar agentes manipuláveis (moléculas), reações elementares e o conceito de conservação de energia, o CRO oferece um método flexível e adaptativo para a resolução de problemas complexos de otimização. A possibilidade de ajustar operadores, alterar dinamicamente o tamanho da população e integrar diferentes atributos torna o CRO uma abordagem promissora no campo das meta-heurísticas. Suas características únicas e flexibilidade permitem que os pesquisadores explorem novas oportunidades de otimização.
No algoritmo CRO, são utilizados os seguintes operadores:
1. Colisão Ineficiente Intermolecular (InterMolecularIneffectiveCollision). Este operador simula o processo em que duas moléculas colidem, mas permanecem inteiras, com leves mudanças em suas estruturas. Isso permite que o algoritmo realize uma busca local nas proximidades das soluções atuais.
2. Decomposição (Decomposition). Este operador modela o processo em que uma molécula colide com uma parede e se divide em duas novas moléculas. Isso permite que o algoritmo explore novas regiões do espaço de soluções.
3. Reação Ineficiente Intramolecular (IntraMolecularReaction). Este operador simula o processo em que uma molécula colide com uma parede, permanecendo intacta, mas com uma leve modificação em sua estrutura. Isso permite que o algoritmo faça uma busca local próxima à solução atual.
4. Síntese (Synthesis). Este operador representa o processo em que duas moléculas colidem e se fundem em uma nova molécula. Isso permite que o algoritmo combine boas soluções para criar soluções potencialmente melhores.
Cada um desses operadores desempenha um papel crucial no algoritmo CRO, possibilitando a exploração do espaço de busca e a identificação de soluções ótimas. A colaboração entre esses operadores garante o equilíbrio entre a exploração (busca em novas áreas do espaço de soluções) e a exploração aprimorada das soluções atuais.
Vamos analisar cada operador químico no algoritmo de forma mais detalhada (busca pelo mínimo global):
Algoritmo #1: On-wall Ineffective Collision (Colisão Ineficiente com Parede)
1. Dados de entrada: molécula Mω
2. Geração de uma nova posição para a molécula: ω = N(ω)
3. Cálculo da energia potencial da nova posição: PEω = f(ω)
4. Incremento do contador de colisões: NumHitω = NumHitω + 1
5. Se a energia potencial da nova posição + energia cinética ≥ energia potencial da posição atual, então:
6. Geração de um número aleatório a no intervalo [KELossRate, 1]
7. Atualização da energia cinética: KEω = (PEω − PEω + KEω) × a
8. Atualização do buffer: buffer = buffer + (PEω − PEω + KEω) × (1 − a)
9. Salvamento da posição atual e das energias
10. Se a energia potencial da nova posição < menor energia potencial, então atualização dos valores mínimos
11. Fim da condição
Algoritmo #2: Decomposition (Decomposição)
1. Dados de entrada: molécula Mω
2. Criação de duas novas moléculas Mω1 e Mω2
3. Definição das posições para as novas moléculas: ω1 e ω2 a partir de ω
4. Cálculo da energia potencial das novas moléculas: PEω1 = f(ω1) e PEω2 = f(ω2)
5. Se a energia potencial da posição atual + energia cinética ≥ energia potencial total das novas posições, então:
6. Cálculo da energia para a decomposição: Edec = PEω + KEω − (PEω1 + PEω2)
7. Avanço para o passo 13
8. Caso contrário:
9. Geração de números aleatórios δ1, δ2 no intervalo [0, 1]
10. Cálculo da energia para a decomposição: Edec = PEω + KEω + δ1δ2 × buffer − (PEω1 + PEω2)
11. Se Edec ≥ 0, então:
12. Atualização do buffer
13. Geração de um número aleatório δ3 no intervalo [0, 1]
14. Distribuição da energia cinética entre as novas moléculas
15. Salvamento dos valores mínimos para cada nova molécula
16. Destruição da molécula atual
17. Caso contrário:
18. Incremento do contador de colisões
19. Destruição das novas moléculas
20. Fim da condição
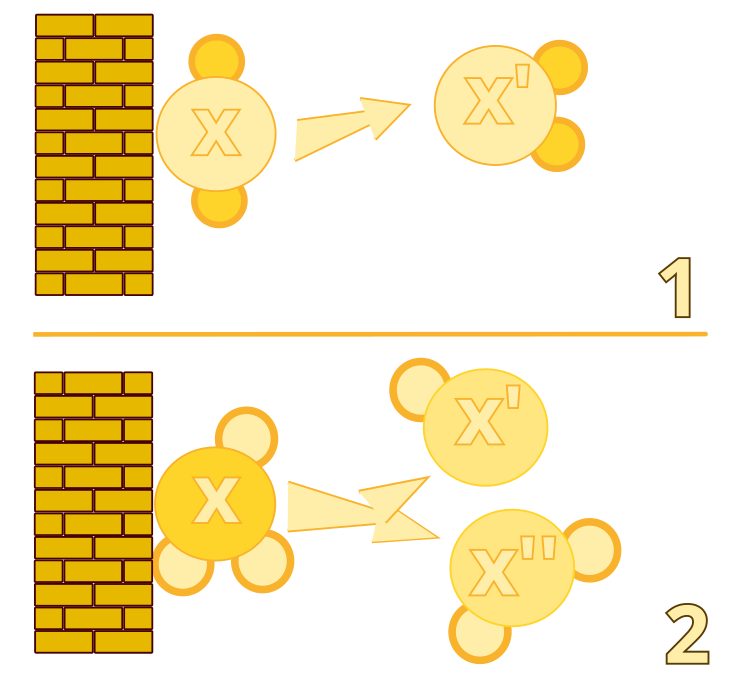
Figura 1. Colisão Ineficiente com Parede: Algoritmo #1. Decomposição: Algoritmo #2.
Algoritmo #3: Intermolecular Ineffective Collision (Colisão Ineficiente Intermolecular)
1. Dados de entrada: moléculas Mω1 e Mω2
2. Geração de novas posições para as moléculas: ω1 = N(ω1) e ω2 = N(ω2)
3. Cálculo da energia potencial das novas posições: PEω1 = f(ω1) e PEω2 = f(ω2)
4. Incremento dos contadores de colisões: NumHitω1 = NumHitω1 + 1 e NumHitω2 = NumHitω2 + 1
5. Cálculo da energia de interação intermolecular: Einter = (PEω1 + PEω2 + KEω1 + KEω2) − (PEω1 + PEω2)
6. Se Einter ≥ 0, então:
7. Geração de um número aleatório δ4 no intervalo [0, 1]
8. Distribuição da energia cinética entre as moléculas: KEω1 = Einter × δ4 e KEω2 = Einter × (1 − δ4)
9. Atualização das posições e energias das moléculas
10. Se a energia potencial da nova posição for menor que a menor energia potencial, então atualização dos valores mínimos para cada molécula
11. Fim da condição
Algoritmo #4: Synthesis (Síntese)
1. Dados de entrada: moléculas Mω1 e Mω2
2. Criação de uma nova molécula Mω
3. Definição da posição da nova molécula: ω a partir de ω1 e ω2
4. Cálculo da energia potencial da nova molécula: PEω = f(ω)
5. Se a soma da energia potencial e cinética da nova molécula for maior ou igual à soma da energia potencial e cinética das moléculas originais, então:
6. Distribuição do excesso de energia cinética para a nova molécula: KEω = (PEω1 + PEω2 + KEω1 + KEω2) − PEω
7. Atualização dos valores mínimos para a nova molécula
8. Destruição das moléculas originais
9. Caso contrário:
10. Incremento dos contadores de colisões para as moléculas originais
11. Destruição da nova molécula
12. Fim da condição
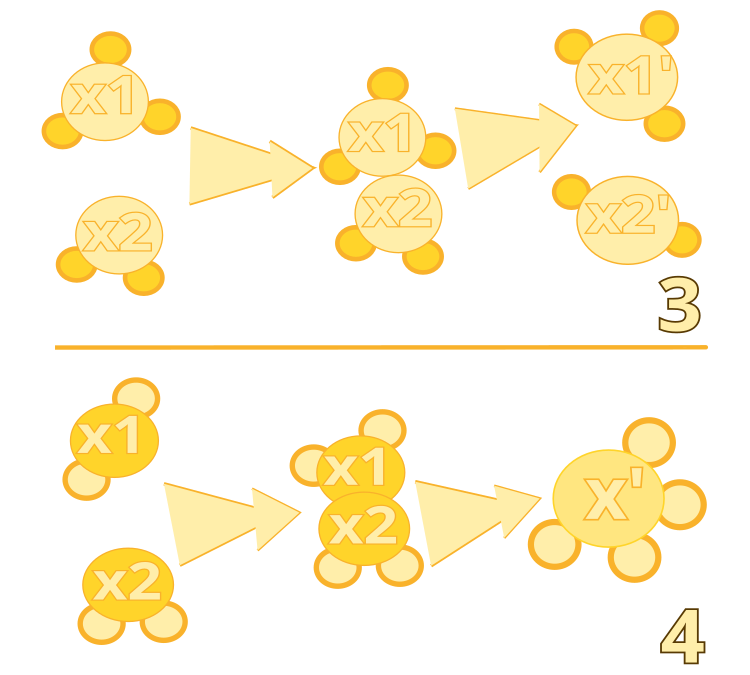
Figura 2. Colisão Ineficiente Intermolecular: Algoritmo #3. Síntese: Algoritmo #4.
A descrição proposta do algoritmo de otimização por reações químicas (CRO) reflete a visão dos autores sobre essa abordagem. No entanto, ela não aborda alguns pontos importantes que podem impactar significativamente o desempenho e a capacidade de busca do algoritmo.
O algoritmo parte do princípio da conservação de energia em um espaço fechado e da transição de uma forma de energia para outra. Contudo, os autores não detalham a correspondência entre os valores numéricos da energia e o significado da função objetivo (fitness function). Fica evidente que o indicador de adequação (fitness) é definido como a energia potencial, enquanto a energia cinética serve como um mecanismo para compensar a redução da energia potencial (a soma de todas as energias deve ser idealmente constante).
Por exemplo, se o critério da tarefa de otimização for o fator de lucro, os valores da função objetivo oscilarão em um intervalo menor do que se o critério utilizado for o saldo. Nesse caso, observamos que os valores da função objetivo variam conforme o critério específico de otimização, mas os autores utilizam como parâmetro externo do algoritmo constantes que não podem ser comparadas às energias calculadas no próprio algoritmo.
Assim, o algoritmo CRO, na versão original dos autores, sem ajustes, possui uma lista significativamente limitada de problemas aos quais pode ser aplicado, ou seja, não é universal. Dentro desta série de artigos, focamos exclusivamente em algoritmos universais, aplicáveis a problemas de otimização de forma geral, e quando um algoritmo não atende a essa exigência, nós geralmente o modificamos e o tornamos mais generalizado.
O segundo ponto é que o algoritmo original calcula a função de aptidão de maneira desordenada, em diferentes partes da lógica, o que inevitavelmente resultará em problemas quando for aplicado em situações práticas e integrado a projetos. Para corrigir isso, é necessário dividir os operadores químicos em duas partes: os próprios operadores, que realizam modificações na posição das moléculas, e os chamados “pós-operadores”, que executam as ações necessárias com as moléculas após o cálculo de sua aptidão.
O terceiro ponto é que o algoritmo dos autores prevê um tamanho dinâmico da população de moléculas, com a geração de novas moléculas e a destruição de algumas antigas. No entanto, não há mecanismos para regular o tamanho da população, que pode variar significativamente. Experimentos mostraram que, com determinadas configurações de parâmetros externos, a população pode crescer de 50 moléculas para mais de mil. Para resolver esse problema, preenchemos a população sequencialmente com moléculas resultantes da execução dos operadores, usando um contador simples e mantendo o índice das moléculas parentais na população original. Isso mantém o tamanho da população constante e elimina a necessidade de remover moléculas — as moléculas filhas, quando as condições são atendidas, simplesmente substituem as moléculas parentais correspondentes.
Essas alterações permitiram adaptar o algoritmo CRO para resolver problemas de otimização de forma geral, facilitando sua integração em projetos sem que o conceito original das reações químicas seja perdido. O algoritmo CRO está detalhadamente descrito, e os entusiastas podem tentar implementá-lo para controlar as energias potencial e cinética, embora eu tenha optado por não adotar esses procedimentos.
A seguir, iniciaremos a escrita do código. Descreveremos os tipos de reações em uma lista, para que possamos escolher o pós-operador apropriado após cada uma e detalharemos a estrutura das moléculas.
Enumeração "E_ReactionType":
1. "synthesis" — refere-se à síntese, o processo em que duas moléculas se combinam para formar uma nova molécula.
2. "interMolecularInefColl" — refere-se à colisão ineficiente intermolecular, o processo em que moléculas colidem, mas não reagem entre si.
3. "decomposition" — refere-se à decomposição, o processo em que uma molécula complexa se divide em componentes mais simples.
4. "inefCollision" — refere-se à colisão ineficiente com a parede, na qual a estrutura da molécula é alterada.
Definindo a estrutura "S_CRO_Agent", que representa o modelo de uma molécula no contexto do algoritmo. Vamos detalhar os campos presentes nesta estrutura:
- structure[] — um array que representa a estrutura da molécula.
- NumHit — um contador que registra o número de "impactos" ou interações da molécula.
- indMolecule_1 e indMolecule_2 — índices das moléculas que interagem.
- KE — uma variável que representa a energia cinética da molécula.
- f — a função de aptidão (fitness) da molécula.
- rType — uma variável do tipo "E_ReactionType", indicando o tipo de reação em que a molécula está envolvida.
"Init" — o método da estrutura que inicializa seus campos. Ele recebe um argumento inteiro "coords", usado para redimensionar o array "structure" com a função "ArrayResize". Os valores de "NumHit", "indMolecule_1", "indMolecule_2", "f", e "KE" são inicializados com zeros ou "-DBL_MAX".
Esse código representa uma estrutura de dados básica para moléculas no algoritmo CRO e inicializa os campos ao criar uma nova molécula.
enum E_ReactionType
{
synthesis,
interMolecularInefColl,
decomposition,
inefCollision
}; // Структура молекулы struct S_CRO_Agent { double structure []; int NumHit; int indMolecule_1; int indMolecule_2; double KE; double f; E_ReactionType rType; // Метод инициализации void Init (int coords) { ArrayResize (structure, coords); NumHit = 0; indMolecule_1 = 0; indMolecule_2 = 0; f = -DBL_MAX; KE = -DBL_MAX; } };
O método "InefCollision" da classe "C_AO_CRO" simula o processo de colisão ineficiente, que envolve a criação de uma nova molécula a partir do deslocamento de uma molécula parental. Aqui está o que acontece nesse método:
1. O método recebe o índice da molécula parental "index" e uma referência ao contador de moléculas "molCNT". Se "molCNT" for maior ou igual ao tamanho da população "popSize", o método retorna "false" e termina.
2. O método define o índice da nova molécula "index1_", que será criada como resultado da colisão.
3. A estrutura da molécula parental é copiada para a estrutura da nova molécula.
4. Em seguida, para cada coordenada da nova molécula, a função "N" é executada, gerando novos valores de coordenadas próximos aos valores antigos.
5. Os novos valores de coordenadas são ajustados com a função "SeInDiSp" para garantir que permaneçam dentro do intervalo especificado de "rangeMin" a "rangeMax".
6. Os campos "indMolecule_1", "rType" e "NumHit" da nova molécula são configurados. "indMolecule_1" armazena o índice da molécula parental, "rType" é definido como "inefCollision", e "NumHit" é redefinido para zero.
7. Por fim, o contador de moléculas "molCNT" é incrementado em 1, e o método retorna "true".
//—————————————————————————————————————————————————————————————————————————————— // Неэффективное столкновение. Получение новой молекулы путём смещения одной родительской. bool C_AO_CRO::InefCollision (int index, int &molCNT) { if (molCNT >= popSize) return false; int index1_ = molCNT; ArrayCopy (Mfilial [index1_].structure, Mparent [index].structure); for (int c = 0; c < coords; c++) { N (Mfilial [index1_].structure [c], c); Mfilial [index1_].structure [c] = u.SeInDiSp (Mfilial [index1_].structure [c], rangeMin [c], rangeMax [c], rangeStep [c]); } Mfilial [index1_].indMolecule_1 = index; //сохраним индекс родительской молекулы Mfilial [index1_].rType = inefCollision; Mfilial [index1_].NumHit = 0; molCNT++; return true; } //——————————————————————————————————————————————————————————————————————————————
O método "PostInefCollision" na classe "C_AO_CRO" é responsável por lidar com os resultados da colisão ineficiente da molécula "mol" com outras moléculas na simulação de reações químicas. O método realiza as seguintes ações:
1. Declara-se a variável "ind" e ela é inicializada com o valor de "indMolecule_1" do objeto "mol".
2. Uma expressão condicional verifica se o valor de "f" do objeto "mol" é maior que o valor de "f" da molécula parental com o índice "ind".
3. Se a condição for verdadeira, a estrutura de "mol" é copiada para a estrutura da molécula parental no índice "ind".
4. O valor "f" da molécula parental é atualizado com o valor de "f" de "mol".
5. O contador "NumHit" da molécula parental é redefinido para zero.
6. Se a condição "mol.f > Mparent[ind].f" for falsa, o contador "NumHit" da molécula parental é incrementado em uma unidade.
Em resumo, esse método atualiza a estrutura e o valor da função de aptidão da molécula parental com base nos resultados da colisão ineficiente. Se a nova estrutura "mol" melhora a aptidão, ela substitui a estrutura da molécula parental, e o contador "NumHit" é reiniciado. Caso contrário, o contador "NumHit" da molécula parental é incrementado.
//—————————————————————————————————————————————————————————————————————————————— // Обработка результатов неэффективного столкновения. void C_AO_CRO::PostInefCollision (S_CRO_Agent &mol) { int ind = mol.indMolecule_1; if (mol.f > Mparent [ind].f) { ArrayCopy (Mparent [ind].structure, mol.structure); Mparent [ind].f = mol.f; Mparent [ind].NumHit = 0; } else { Mparent [ind].NumHit++; } } //——————————————————————————————————————————————————————————————————————————————
O método "Decomposition" da classe "C_AO_CRO" simula o processo de decomposição, que envolve a criação de duas novas moléculas a partir da decomposição de uma molécula parental. Aqui está o que acontece nesse método:
1. O método recebe o índice da molécula parental "index" e uma referência ao contador de moléculas "molCNT". Se "molCNT" for maior ou igual a "popSize - 1", o método retorna "false" e encerra a execução.
2. Em seguida, o método define os índices de duas novas moléculas "index1_" e "index2_", que serão criadas como resultado da decomposição.
3. A estrutura da molécula parental é copiada para as estruturas das novas moléculas.
4. Para cada coordenada das novas moléculas, a função "N" é executada, gerando novos valores de coordenadas próximos aos valores antigos. Isso é feito separadamente: para a primeira metade das coordenadas da molécula "index1_" e para a segunda metade das coordenadas da molécula "index2_", respectivamente.
5. Os novos valores de coordenadas são ajustados com a função "SeInDiSp" para garantir que permaneçam dentro do intervalo especificado de "rangeMin" a "rangeMax".
6. Os campos "indMolecule_1", "indMolecule_2", "rType" e "NumHit" das novas moléculas são configurados. "indMolecule_1" e "indMolecule_2" armazenam os índices das moléculas parentais e irmãs, respectivamente, "rType" é definido como "decomposition", e "NumHit" é reiniciado.
7. No final do método, o contador de moléculas "molCNT" é incrementado em 2, e o método retorna "true".
//—————————————————————————————————————————————————————————————————————————————— // Разложение. Получение новых двух молекул путём разложения одной родительской. bool C_AO_CRO::Decomposition (int index, int &molCNT) { if (molCNT >= popSize - 1) return false; // Создание двух новых молекул M_ω'_1 и M_ω'_2 из M_ω int index1_ = molCNT; int index2_ = molCNT + 1; ArrayCopy (Mfilial [index1_].structure, Mparent [index].structure); ArrayCopy (Mfilial [index2_].structure, Mparent [index].structure); for (int c = 0; c < coords / 2; c++) { N (Mfilial [index1_].structure [c], c); Mfilial [index1_].structure [c] = u.SeInDiSp (Mfilial [index1_].structure [c], rangeMin [c], rangeMax [c], rangeStep [c]); } for (int c = coords / 2; c < coords; c++) { N (Mfilial [index2_].structure [c], c); Mfilial [index2_].structure [c] = u.SeInDiSp (Mfilial [index2_].structure [c], rangeMin [c], rangeMax [c], rangeStep [c]); } Mfilial [index1_].indMolecule_1 = index; //сохраним индекс родительской молекулы Mfilial [index1_].indMolecule_2 = index2_; //сохраним индекс второй дочерней молекулы Mfilial [index1_].rType = decomposition; Mfilial [index1_].NumHit = 0; Mfilial [index2_].indMolecule_1 = index1_; //сохраним индекс первой дочерней молекулы Mfilial [index2_].indMolecule_2 = -1; //пометим молекулу, чтобы не обрабатывать её дважды Mfilial [index2_].rType = decomposition; Mfilial [index2_].NumHit = 0; molCNT += 2; return true; } //——————————————————————————————————————————————————————————————————————————————
O método "PostDecomposition" da classe "C_AO_CRO" processa os resultados da decomposição da molécula. Neste método, o seguinte ocorre:
1. O método recebe uma referência à molécula "mol", que foi gerada a partir da decomposição. Se "indMolecule_2" dessa molécula for igual a "-1" (o que significa que a molécula já foi processada), o método encerra a execução.
2. Em seguida, são extraídos o índice da molécula parental "ind" e os índices das duas moléculas "filhas", "index1_" e "index2_".
3. Depois, verifica-se se o valor da função de aptidão "f" da primeira molécula "filha" supera os valores da função "f" da segunda molécula "filha" e da molécula parental. Se isso for verdadeiro, a primeira molécula "filha" substitui a molécula parental. O valor de "NumHit" da molécula substituída é reiniciado, e um indicador "flag" é definido.
4. Se "flag" ainda for "false", uma verificação semelhante é realizada para a segunda molécula "filha".
5. Se, após todas as verificações, "flag" ainda for "false", o valor de "NumHit" da molécula parental é incrementado em 1.
Esse método "PostDecomposition" é responsável por atualizar os estados da molécula parental após o processo de decomposição no algoritmo CRO.
//—————————————————————————————————————————————————————————————————————————————— // Обработка результатов разложения. void C_AO_CRO::PostDecomposition (S_CRO_Agent &mol) { if (mol.indMolecule_2 == -1) return; int ind = mol.indMolecule_1; int index2_ = mol.indMolecule_2; int index1_ = Mfilial [index2_].indMolecule_1; bool flag = false; if (Mfilial [index1_].f > Mfilial [index2_].f && Mfilial [index1_].f > Mparent [ind].f) { ArrayCopy (Mparent [ind].structure, Mfilial [index1_].structure); Mparent [ind].f = Mfilial [index1_].f; Mparent [ind].NumHit = 0; flag = true; } if (!flag) { if (Mfilial [index2_].f > Mfilial [index1_].f && Mfilial [index2_].f > Mparent [ind].f) { ArrayCopy (Mparent [ind].structure, Mfilial [index2_].structure); Mparent [ind].f = Mfilial [index2_].f; Mparent [ind].NumHit = 0; flag = true; } } if (!flag) { Mparent [ind].NumHit++; } } //——————————————————————————————————————————————————————————————————————————————
O método "InterMolInefColl" da classe "C_AO_CRO" representa o processo de colisão ineficiente intermolecular, que envolve a criação de duas novas moléculas a partir da modificação de duas moléculas parentais. Aqui está o que acontece nesse método:
1. O método recebe os índices de duas moléculas parentais "index1" e "index2" e uma referência ao contador de moléculas "molCNT". Se "molCNT" for maior ou igual a "popSize - 1", o método retorna "false" e termina.
2. Em seguida, o método define os índices das duas novas moléculas filhas "index1_" e "index2_", que serão criadas como resultado da colisão.
3. As estruturas das moléculas parentais são copiadas para as estruturas das novas moléculas.
4. Para cada coordenada das novas moléculas, a função "N" é executada, gerando novos valores de coordenadas próximos aos valores antigos.
5. Os novos valores de coordenadas são então ajustados com a função "SeInDiSp" para garantir que permaneçam dentro do intervalo especificado, de "rangeMin" a "rangeMax".
6. Os campos "indMolecule_1", "indMolecule_2", "rType" e "NumHit" das novas moléculas são definidos. "indMolecule_1" e "indMolecule_2" armazenam os índices das moléculas parentais, "rType" é definido como "interMolecularInefColl", e "NumHit" é reiniciado.
7. No final do método, o contador de moléculas "molCNT" é incrementado em 2, e o método retorna "true".
//—————————————————————————————————————————————————————————————————————————————— // Межмолекулярное неэффективное столкновение. Получение новых двух молекул путём изменения двух родительских bool C_AO_CRO::InterMolInefColl (int index1, int index2, int &molCNT) { if (molCNT >= popSize - 1) return false; int index1_ = molCNT; int index2_ = molCNT + 1; // Получение молекул ArrayCopy (Mfilial [index1_].structure, Mparent [index1].structure); ArrayCopy (Mfilial [index2_].structure, Mparent [index2].structure); // Генерация новых молекул ω'_1 = N(ω1) и ω'_2 = N(ω2) в окрестности ω1 и ω2 for (int c = 0; c < coords; c++) { N (Mfilial [index1_].structure [c], c); N (Mfilial [index2_].structure [c], c); } for (int c = 0; c < coords; c++) { Mfilial [index1_].structure [c] = u.SeInDiSp (Mfilial [index1_].structure [c], rangeMin [c], rangeMax [c], rangeStep [c]); Mfilial [index2_].structure [c] = u.SeInDiSp (Mfilial [index2_].structure [c], rangeMin [c], rangeMax [c], rangeStep [c]); } Mfilial [index1_].indMolecule_1 = index1; //сохраним индекс первой родительской молекулы Mfilial [index1_].indMolecule_2 = index2_; //сохраним индекс второй дочерней молекулы Mfilial [index1_].rType = interMolecularInefColl; Mfilial [index1_].NumHit = 0; Mfilial [index2_].indMolecule_1 = index2; //сохраним индекс второй родительской молекулы Mfilial [index2_].indMolecule_2 = -1; //пометим молекулу, чтобы не обрабатывать её дважды Mfilial [index2_].rType = interMolecularInefColl; Mfilial [index2_].NumHit = 0; molCNT += 2; return true; } //——————————————————————————————————————————————————————————————————————————————
O método "PostInterMolInefColl" da classe "C_AO_CRO" processa os resultados da colisão ineficiente intermolecular. O funcionamento deste método é o seguinte:
1. O método recebe uma referência à molécula "mol", que foi gerada como resultado da colisão. Se "indMolecule_2" dessa molécula for igual a "-1" (o que significa que a molécula já foi processada), o método encerra a execução.
2. Em seguida, são extraídos os índices das duas moléculas parentais "ind1" e "ind2".
3. Depois, verifica-se se a soma dos valores de aptidão "f" da nova molécula e de sua "irmã" supera a soma dos valores de aptidão "f" de ambas as moléculas parentais. Se isso for verdadeiro, as novas moléculas substituem as parentais. Os valores de "NumHit" das moléculas substituídas são reiniciados.
4. Caso contrário, os valores de "NumHit" das moléculas parentais são incrementados em 1.
Esse método "PostInterMolInefColl" é responsável por atualizar os estados das moléculas parentais após o processo de colisão ineficiente intermolecular no algoritmo CRO.
//—————————————————————————————————————————————————————————————————————————————— // Обработка результатов межмолекулярного неэффективного столкновения. void C_AO_CRO::PostInterMolInefColl (S_CRO_Agent &mol) { if (mol.indMolecule_2 == -1) return; int ind1 = mol.indMolecule_1; int ind2 = Mfilial [mol.indMolecule_2].indMolecule_1; Mparent [ind1].NumHit++; Mparent [ind2].NumHit++; if (mol.f + Mfilial [mol.indMolecule_2].f > Mparent [ind1].f + Mparent [ind2].f) { ArrayCopy (Mparent [ind1].structure, mol.structure); Mparent [ind1].f = mol.f; ArrayCopy (Mparent [ind2].structure, Mfilial [mol.indMolecule_2].structure); Mparent [ind2].f = Mfilial [mol.indMolecule_2].f; } } //——————————————————————————————————————————————————————————————————————————————
O último operador de reações químicas no algoritmo é o método "Synthesis" da classe "C_AO_CRO", que representa o processo de síntese, envolvendo a criação de uma nova molécula pela fusão de duas moléculas parentais.
1. O método recebe os índices de duas moléculas parentais "index1" e "index2" e uma referência ao contador de moléculas "molCNT". Se "molCNT" for maior ou igual ao tamanho da população "popSize", o método retorna "false" e termina.
2. Em um loop, para cada coordenada da nova molécula, é realizado o seguinte: se um número aleatório for menor que 0.5, a coordenada recebe o valor correspondente da primeira molécula parental "Mparent[index1].structure[i]"; caso contrário, recebe o valor da coordenada correspondente da segunda molécula parental "Mparent[index2].structure[i]".
3. Em seguida, os campos "indMolecule_1", "indMolecule_2", "rType" e "NumHit" da nova molécula são configurados. "indMolecule_1" e "indMolecule_2" armazenam os índices das moléculas parentais, "rType" é definido como "synthesis", e "NumHit" é reiniciado.
4. No final do método, o contador de moléculas "molCNT" é incrementado, e o método retorna "true".
//—————————————————————————————————————————————————————————————————————————————— // Синтез. Получение новой молекулы путём слияния двух родительских bool C_AO_CRO::Synthesis (int index1, int index2, int &molCNT) { if (molCNT >= popSize) return false; // Создание новой молекулы M_ω' из M_ω1 и M_ω2 for (int i = 0; i < coords; i++) { if (u.RNDprobab () < 0.5) Mfilial [molCNT].structure [i] = Mparent [index1].structure [i]; else Mfilial [molCNT].structure [i] = Mparent [index2].structure [i]; } Mfilial [molCNT].indMolecule_1 = index1; //сохраним индекс первой родительской молекулы Mfilial [molCNT].indMolecule_2 = index2; //сохраним индекс второй родительской молекулы Mfilial [molCNT].rType = synthesis; Mfilial [molCNT].NumHit = 0; molCNT++; return true; } //——————————————————————————————————————————————————————————————————————————————
O método "PostSynthesis" da classe "C_AO_CRO" processa os resultados da síntese. Veja o que acontece neste método:
1. O método recebe uma referência à molécula "mol", que foi gerada a partir da síntese. Dessa molécula, são extraídos os índices das duas moléculas parentais "ind1" e "ind2".
2. Em seguida, verifica-se se o valor de aptidão "f" da nova molécula é superior aos valores de aptidão "f" das duas moléculas parentais. Se for o caso, a nova molécula substitui aquela das moléculas parentais que tem o menor valor de "f". O valor de "NumHit" da molécula substituída é reiniciado.
3. Caso contrário, os valores de "NumHit" de ambas as moléculas parentais são incrementados em 1.
Esse método "PostSynthesis" é responsável por atualizar os estados das moléculas parentais após o processo de síntese no algoritmo Chemical Reaction Optimisation (CRO).
//—————————————————————————————————————————————————————————————————————————————— // Обработка результатов синтеза. void C_AO_CRO::PostSynthesis (S_CRO_Agent &mol) { int ind1 = mol.indMolecule_1; int ind2 = mol.indMolecule_2; if (mol.f > Mparent [ind1].f && mol.f > Mparent [ind2].f) { if (Mparent [ind1].f < Mparent [ind2].f) { ArrayCopy (Mparent [ind1].structure, mol.structure); Mparent [ind1].f = mol.f; Mparent [ind1].NumHit = 0; } else { ArrayCopy (Mparent [ind2].structure, mol.structure); Mparent [ind2].f = mol.f; Mparent [ind2].NumHit = 0; } } else { Mparent [ind1].NumHit++; Mparent [ind2].NumHit++; } } //——————————————————————————————————————————————————————————————————————————————
Por fim, vamos descrever o método "N" da classe "C_AO_CRO", que é utilizado para modificar a estrutura das moléculas nos operadores de reações químicas. O método é empregado para gerar um novo valor para a coordenada da molécula dentro do intervalo especificado:
1. O método recebe a coordenada da molécula "coord" a ser modificada como referência, e a posição dessa coordenada "coordPos" na estrutura.
2. Em seguida, é calculada a distância "dist", que é a diferença entre os valores máximos e mínimos do intervalo, multiplicada pelo parâmetro "molecPerturb", que indica a variação dos valores nas proximidades do valor atual da coordenada.
3. Em seguida, são determinados os valores mínimos "min" e máximos "max" para a nova coordenada. Esses valores correspondem ao valor antigo da coordenada mais ou menos a distância "dist", mas não podem ultrapassar os limites do intervalo especificado, que vai de "rangeMin[coordPos]" a "rangeMax[coordPos]".
4. Por fim, o novo valor da coordenada é gerado usando a função de distribuição Gaussiana "u.GaussDistribution", que recebe o valor antigo da coordenada, os valores mínimo e máximo, e um desvio padrão de 8.
//—————————————————————————————————————————————————————————————————————————————— void C_AO_CRO::N (double &coord, int coordPos) { double dist = (rangeMax [coordPos] - rangeMin [coordPos]) * molecPerturb; double min = coord - dist; if (min < rangeMin [coordPos]) min = rangeMin [coordPos]; double max = coord + dist; if (max > rangeMax [coordPos]) max = rangeMax [coordPos]; coord = u.GaussDistribution (coord, min, max, 8); } //——————————————————————————————————————————————————————————————————————————————
Assim, analisamos todos os operadores químicos, entendendo sua funcionalidade e seu papel no processo de otimização. Essa análise nos proporcionou uma compreensão profunda dos mecanismos que governam a dinâmica das moléculas na simulação de reações químicas (CRO).
No próximo artigo, criaremos o algoritmo e o testaremos em funções de teste. Ajustaremos e integraremos os operadores estudados em um algoritmo CRO completo, pronto para ser aplicado a problemas práticos. Em seguida, conduziremos uma série de experimentos em uma ampla gama de funções de teste, permitindo-nos avaliar a eficácia e a confiabilidade do nosso método.
Com os resultados obtidos, tiraremos conclusões sobre os pontos fortes e fracos do algoritmo, além de sugerir possíveis direções para melhorias futuras. Isso nos ajudará não apenas a aperfeiçoar nosso método, mas também a oferecer recomendações valiosas para pesquisadores que trabalham com otimização e modelagem de processos químicos.
Portanto, a próxima parte será um marco importante em nosso estudo, onde passaremos da teoria para a prática, testando e refinando nosso algoritmo para alcançar os melhores resultados na solução de problemas complexos de otimização.
Traduzido do russo pela MetaQuotes Ltd.
Artigo original: https://www.mql5.com/ru/articles/15041
Aviso: Todos os direitos sobre esses materiais pertencem à MetaQuotes Ltd. É proibida a reimpressão total ou parcial.
Esse artigo foi escrito por um usuário do site e reflete seu ponto de vista pessoal. A MetaQuotes Ltd. não se responsabiliza pela precisão das informações apresentadas nem pelas possíveis consequências decorrentes do uso das soluções, estratégias ou recomendações descritas.
 Algoritmo de otimização por reações químicas — Chemical Reaction Optimisation, CRO (Parte II): Montagem e resultados
Algoritmo de otimização por reações químicas — Chemical Reaction Optimisation, CRO (Parte II): Montagem e resultados
 Desenvolvendo um sistema de Replay (Parte 74): Um novo Chart Trade (I)
Desenvolvendo um sistema de Replay (Parte 74): Um novo Chart Trade (I)
 Redes neurais de maneira fácil (Parte 95): Redução do consumo de memória em modelos Transformer
Redes neurais de maneira fácil (Parte 95): Redução do consumo de memória em modelos Transformer
 Simplificando a negociação com base em notícias (Parte 2): Gerenciando riscos
Simplificando a negociação com base em notícias (Parte 2): Gerenciando riscos
- Aplicativos de negociação gratuitos
- 8 000+ sinais para cópia
- Notícias econômicas para análise dos mercados financeiros
Você concorda com a política do site e com os termos de uso
Aqui está oartigo sobre aplicaçãoprática de nanegociação . Leia, estude e aplique naprática . Desejo avocê sucesso na negociação .
Artigos
Uso de algoritmos de otimização para configurar os parâmetros do EA em tempo real
Andrey Dik, 2024.06.07 14:30
O artigo discute os aspectos práticos do uso de algoritmos de otimização para encontrar os melhores parâmetros do EA em tempo real, bem como a virtualização das operações de negociação e da lógica do EA. O artigo pode ser usado como uma instrução para a implementação de algoritmos de otimização em um EA.Aqui está oartigo sobre aplicaçãoprática de em negociação. Leia, estude e aplique naprática . Desejoavocê sucesso na negociação .
Gosto do seu ponto de vista, Andrei! Grandes inovações nascem da pesquisa. Nem sempre tudo se resume a dinheiro, e eu aprecio sua contribuição para a comunidade.