
Redes neuronales: así de sencillo (Parte 9): Documentamos el trabajo realizado
Contenido
- Introducción
- 1.Principios básicos para documentar los desarrollos
- 2.Seleccionando los instrumentos
- 3.Métodos de documentación en el código
- 4.Trabajo preparatorio en el archivo del código fuente
- 5.Generando la documentación
- Conclusión
- Enlaces
- Programas utilizados en el artículo
Introducción
A lo largo de 8 artículos, hemos añadido nuevos objetos y ampliado la funcionalidad de los existentes, aumentando constantemente nuestra biblioteca. Hemos añadido el archivo de programa OpenCL. Ahora nuestro código es 10 veces mayor que el original, por lo que resulta complicado monitorear las relaciones entre los objetos en el código. Y probablemente, el código le parecerá al lector muy confuso y complicado de entender. En cada artículo, intentamos describir con detalle la cadena lógica de acción, pero la demostración de las cadenas individuales no ofrece una descripción general.
Por eso mismo, hemos decido mostrar cómo crear una documentación técnica para el código que nos permita ver este desde otro punto de vista, posibilitando que generalicemos todos los objetos y métodos de la biblioteca y construyamos la jerarquía de los objetos y la herencia de los métodos. Asimismo, nos ofrecerá una idea general del trabajo realizado.
1. Principios básicos para documentar los desarrollos
¿Por qué necesitamos documentación técnica para los desarrollos de TI? En primer lugar, la documentación de trabajo nos ofrece una idea general del dispositivo, así como de la arquitectura y el funcionamiento del programa. Dicha documentación nos permite diseñar correctamente las áreas de responsabilidad dentro del equipo de desarrollores, monitorear todos los cambios en el código y su influencia en el algoritmo completo y la integridad de la arquitectura de desarrollo. Facilita el proceso de transferencia de conocimientos. Comprender la integridad de la arquitectura del programa nos permite analizar y encontrar formas de desarrollo para los proyectos.
Una documentación técnica adecuadamente preparada debe considerar el nivel de cualificación de su usuario objetivo. La información presentada debe resultar comprensible y no contener explicaciones innecesarias. La documentación debe tener toda la información que el usuario necesita. Al mismo tiempo, debe ser lo más concisa posible, a la vez que simple de leer. El contenido sobrante conlleva que el lector pierda tiempo en su lectura, lo cual resulta molesto. Pero resulta aún más molesto cuando, después de una larga relectura de la enorme documentación, el lecto no encuentra la información que necesita. Por eso, deberemos ceñirnos a la siguiente regla: la documentación deberá tener la funcionalidad adecuada para encontrar información. Es decir, una interfaz amigable y referencias cruzadas que faciliten la búsqueda de la información que necesitamos.
La documentación debe contener la arquitectura completa de la solución y una descripción de las soluciones técnicas implementadas. La integridad y el nivel de detalle de la descripción de las soluciones facilitará el desarrollo de la solución y su soporte adicional. Y resulta vital mantener la documentación actualizada en todo momento. Una representación distorsionada de la información puede redundar en decisiones de gestión contradictorias y, como resultado, en un desequilibrio del desarrollo completo.
La documentación debe describir obligatoriamente las interfaces entre los componentes, valiéndose para ello de un alto nivel de detalle.
2. Seleccionando los instrumentos
En sí, el proceso de documentación de los desarrollos puede simplificarse utilizando programas especializados. Los más comunes, en nuestra opinión, son Doxygen, Sphinx y Latex, pero hay otros. Todos ellos se diseñaron para reducir los costes laborales derivados de la creación de la documentación y ofrecer herramientas propias. Obviamente, cada programa fue creado por desarrolladores para resolver problemas específicos. Por ejemplo, Doxygen se ofrece como un programa capaz de crear documentación para el desarrollo en C ++ y lenguajes de programación similares. A su vez, Sphinx fue creado para documentar Python. Pero esto no significa que sean programas altamente especializados en lenguajes de programación, aunque ambos programas funcionen bien con una amplia gama de lenguajes de programación. En el sitio web de cada uno de los programas anteriores, se ofrece ayuda detallada sobre la utilización de los programas, y cualquiera puede seleccionar el más conveniente para él.
En MQL5, el tema de la documentación de los desarrollos también se planteó anteriormente en el artículo "Documentación generada automáticamente para el código de MQL5". En este artículo, se proponía utilizar Doxygen. En nuestro trabajo, también usamos este programa. La sintaxis de MQL5 es muy similar a la de C ++, por lo que resulta bastante lógico utilizar Doxygen para documentarlo. En nuestra opinión, resulta impresionante que para crear documentación, baste con comentar a fondo el código del programa y que el software especializado se encargue del resto. Además, Doxygen permite insertar hipervínculos y fórmulas matemáticas, lo cual resulta importante para el tema de los artículos. En el artículo, discutiremos las características del uso de la funcionalidad usando ejemplos específicos.
3. Métodos de documentación en el código
Como se hemos mencionado antes, para generar la documentación, deberemos añadir comentarios al código del programa. Usando estos comentarios como base, Doxygen compilará la documentación. Resulta bastante natural que no todos los comentarios del código sean incluidos en la documentación. En algún lugar puede haber notas del desarrollador, en algún otro se comenta un código no usado. Los desarrolladores de Doxygen más previsores han descubierto formas de destacar los comentarios con fines documentales. A su vez, existen varias opciones entre las que los usuarios pueden seleccionar la que más les convenga.
Por analogía con MQL5, los comentarios para la documentación pueden tener una o varias líneas. Y para que no surjan interferencias con el uso posterior del código según el propósito previsto, usaremos opciones estándar para insertar comentarios añadiendo una barra diagonal adicional para los comentarios de una sola línea, o un asterisco para los comentarios de varias líneas. También podemos identificar los bloques para la documentación utilizando un signo de exclamación.
/// A single-line comment for documentation /** A multi-line block for documentation */ //! An alternative single-line comment for documentation /*! An alternative multi-line block for documentation */
Aquí, cabe señalar que un comentario de varias líneas no implica una representación de varias líneas en la documentación. Si necesitamos separar la descripción breve y la detallada del objeto de programa, podemos crear varios bloques de comentarios o utilizar los comandos especiales indicados con el símbolo "\" o "@". Para forzar los finales de línea, podemos utilizar el comando "\n".
Option 1: Separate blocks /// Short description /** Detailed description */ Option 2: Use of special commands /** \brief Brief description \details Detailed description */
En general, asumimos que el objeto de documentación se encuentra en el archivo inmediatamente posterior al bloque de comentarios. Pero en la práctica, es posible que también queramos realizar comentarios sobre el objeto anterior al bloque de comentarios. En este caso, deberemos usar el símbolo "<" para que Doxygen sepa que el objeto de comentario se encuentra delante del bloque. Para crear referencias cruzadas en un comentario, deberemos poner el signo "#" delante del objeto de enlace. A continuación, mostramos al lector un ejemplo de código y un bloque generado por el mismo en la documentación. En este caso, en la plantilla "CConnection" generada, hay un puntero de enlace a la página de la documentación de la clase correspondiente.
#define defConnect 0x7781 ///<Connection \details Identified class #CConnection
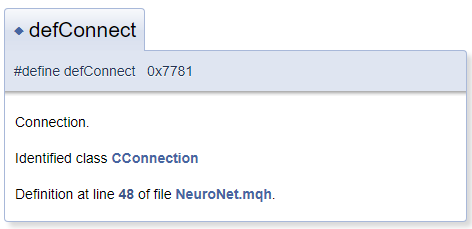
En general, las capacidades de Doxygen son bastante amplias; en la sección documentación de la página del programa se ofrece una lista completa de comandos con sus descripciones correspondientes. Asimismo, Doxygen comprende tanto el marcado HTML como el XML. Todo esto nos permite resolver muchos problemas a la hora de documentar desarrollos.
4. Trabajo preparatorio en el archivo del código fuente
Una vez nos hemos familiarizado con las posibilidades de nuestra herramienta, podemos comenzar a trabajar con la documentación. En primer lugar, vamos a describir nuestros archivos.
/// \file /// \brief NeuroNet.mqh /// Library for creating Neural network for use in MQL5 experts /// \author [DNG](https://www.mql5.com/en/users/dng) /// \copyright Copyright 2019, DNG
y
/// \file /// \brief NeuroNet.cl /// Library consist OpenCL kernels /// \author <A HREF="https://www.mql5.com/en/users/dng"> DNG </A> /// \copyright Copyright 2019, DNG
Debemos tener en cuenta que, en el primer caso, el marcado ofrecido por Doxygen viene después del puntero \author, y en el segundo, se usa el marcado HTML. Lo hemos hecho así para demostrar las diversas opciones de creación de hipervínculos. En ambos casos, el resultado será idéntico: habremos creado un enlace al perfil del autor en este sitio.
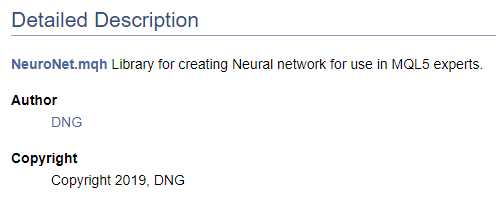
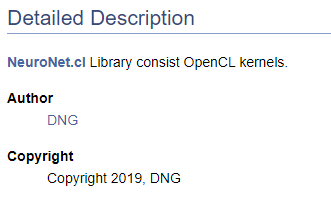
Obviamente, al comenzar a trabajar con la documentación del código, al menos deberemos tener una estructura de alto nivel con el resultado deseado. Precisamente la comprensión de la estructura final nos permitirá agrupar correctamente los objetos de documentación. Vamos a destacar las enumeraciones creadas en un grupo aparte. Para declarar un grupo, usaremos el comando "\defgroup", acotando los límites del grupo con los símbolos "@{" y "@}".
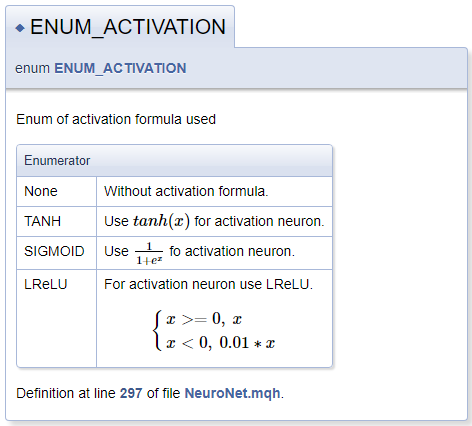
///\defgroup enums ENUM ///@{ //+------------------------------------------------------------------+ /// Enum of activation formula used //+------------------------------------------------------------------+ enum ENUM_ACTIVATION { None=-1, ///< Without activation formula TANH, ///< Use \f$tanh(x)\f$ for activation neuron SIGMOID, ///< Use \f$\frac{1}{1+e^x}\f$ fo activation neuron LReLU ///< For activation neuron use LReLU \f[\left\{ \begin{array} a x>=0, \ x \\x<0, \ 0.01*x \end{array} \right.\f] }; //+------------------------------------------------------------------+ /// Enum of optimization method used //+------------------------------------------------------------------+ enum ENUM_OPTIMIZATION { SGD, ///< Stochastic gradient descent ADAM ///< Adam }; ///@}
Al describir las funciones de activación, hemos mostrado la funcionalidad para declarar fórmulas matemáticas usando MathJax. Las descripciones de dichas fórmulas se deben colocar entre la pareja de comandos "\f$" para mostrar la fórmula en una línea de texto o entre los comandos "\f[" y "\f]" para mostrar la fórmula en una línea aparte. El comando "\frac" nos permite describir una fracción. El comando va seguido del numerador y denominador de la fracción entre corchetes.
Al describir LReLU, necesitábamos un corchete izquierdo unificador, para crearlo, usaremos los comandos "\left\{" y "\right\.". El comando "\right" va seguido de "\.", dado que no necesitamos el corchete correcto en la fórmula. De lo contrario, reemplazaríamos el periodo con un corchete de cierre. Dentro del bloque, declaramos una matriz de líneas utilizando los comandos "\begin{array} a" y "\end{array}"; los elementos de la matriz están separados por el comando "\\". Para forzar la adición de un espacio, usaremos el conjunto de caracteres "\ ".
A continuación, presentamos el bloque de documentación generado.
El siguiente paso será separar los identificadores de clase en la biblioteca en un grupo aparte. Dentro del grupo, destacaremos los subgrupos de matrices, las neuronas con cálculo de operaciones en la CPU y las neuronas con cálculo de operaciones en la GPU. Y, obviamente, como hemos descrito antes, añadiremos un enlace a la clase correspondiente a cada constante.
///\defgroup ObjectTypes Defines Object types identified ///Used to identify classes in a library ///@{ //+------------------------------------------------------------------+ ///\defgroup arr Arrays ///Used to identify array classes ///\{ #define defArrayConnects 0x7782 ///<Array of connections \details Identified class #CArrayCon #define defLayer 0x7787 ///<Layer of neurons \details Identified class #CLayer #define defArrayLayer 0x7788 ///<Array of layers \details Identified class #CArrayLayer #define defNet 0x7790 ///<Neuron Net \details Identified class #CNet ///\} ///\defgroup cpu CPU ///Used to identify classes with CPU calculation ///\{ #define defConnect 0x7781 ///<Connection \details Identified class #CConnection #define defNeuronBase 0x7783 ///<Neuron base type \details Identified class #CNeuronBase #define defNeuron 0x7784 ///<Full connected neuron \details Identified class #CNeuron #define defNeuronConv 0x7785 ///<Convolution neuron \details Identified class #CNeuronConv #define defNeuronProof 0x7786 ///<Proof neuron \details Identified class #CNeuronProof #define defNeuronLSTM 0x7791 ///<LSTM Neuron \details Identified class #CNeuronLSTM ///\} ///\defgroup gpu GPU ///Used to identify classes with GPU calculation ///\{ #define defBufferDouble 0x7882 ///<Data Buffer OpenCL \details Identified class #CBufferDouble #define defNeuronBaseOCL 0x7883 ///<Neuron Base OpenCL \details Identified class #CNeuronBaseOCL #define defNeuronConvOCL 0x7885 ///<Conolution neuron OpenCL \details Identified class #CNeuronConvOCL #define defNeuronProofOCL 0x7886 ///<Proof neuron OpenCL \details Identified class #CNeuronProofOCL #define defNeuronAttentionOCL 0x7887 ///<Attention neuron OpenCL \details Identified class #CNeuronAttentionOCL ///\} ///@}
La división en grupos en la documentación generada tiene el siguiente aspecto.
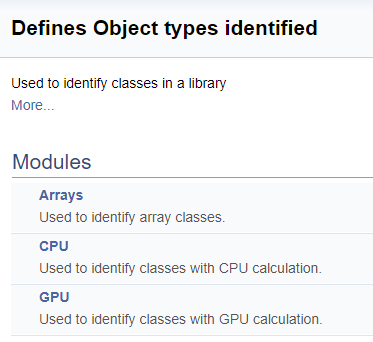
A continuación, elaboraremos un gran grupo de definiciones para trabajar con los kernels OpenCL. En este bloque, asignamos nombres nemotécnicos a los índices de los kernels y los parámetros de estos que se usan al llamar a los kernels desde el programa principal. Utilizando la tecnología descrita antes, dividiremos este grupo según la clase de neuronas desde las que se llama al kernel, y luego según el contenido de las operaciones en el kernel (propagación hacia adelante, propagación inversa del gradiente, actualización de los coeficientes de peso). No vamos a ofrecer el código completo aquí: el lector podrá encontrarlo en el archivo adjunto. La lógica de construcción de los subgrupos es similar a la del ejemplo anterior. La siguiente captura de pantalla muestra la estructura completa de los grupos.
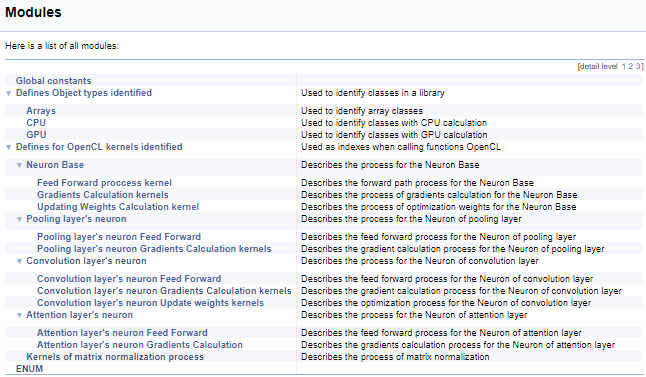
Continuando el trabajo con los kernels, vamos a pasar a los comentarios sobre el programa OpenCL. Para crear una estructura de documentación coherente y obtener una imagen general de nuestra documentación, usaremos otro comando de Doxygen, "\ingroup", que nos permite añadir nuevos objetos de documentación a grupos creados previamente. Con su ayuda, añadiremos kernels a los grupos de índices creados anteriormente para trabajar con los kernels. En la descripción del kernel, añadimos un enlace a la clase que lo llama y al artículo en este sitio web con la descripción del proceso. A continuación, vamos a describir los parámetros del kernel usando el código. El uso de los punteros "[in]" y "[out]" nos indicará la dirección del flujo de información. Y las referencias cruzadas nos dirán el formato de los datos.
///\ingroup neuron_base_ff Feed forward process kernel /// Describes the forward path process for the Neuron Base (#CNeuronBaseOCL). ///\details Detailed description on <A HREF="https://www.mql5.com/ru/articles/8435#para41">the link.</A> //+------------------------------------------------------------------+ __kernel void FeedForward(__global double *matrix_w,///<[in] Weights matrix (m+1)*n, where m - number of neurons in layer and n - number of outputs (neurons in next layer) __global double *matrix_i,///<[in] Inputs tesor __global double *matrix_o,///<[out] Output tensor int inputs,///< Number of inputs int activation///< Activation type (#ENUM_ACTIVATION) )
El código anterior generará el siguiente bloque de documentación.
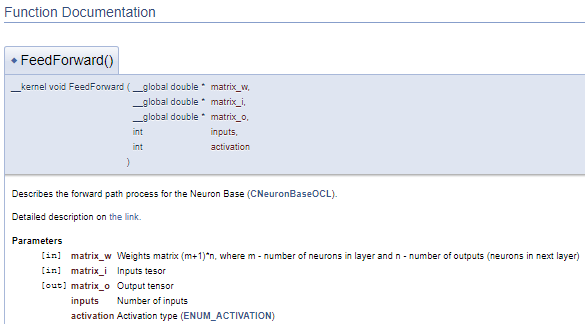
En el ejemplo anterior, la descripción de los parámetros se ofrece justo después de su declaración. Pero este enfoque con frecuencia satura el código. En tales casos, sugerimos usar el comando "\param" para describir los parámetros. Su uso nos permite describir los parámetros en cualquier parte del archivo, pero requiere que indiquemos directamente el nombre del parámetro.
///\ingroup neuron_atten_gr Attention layer's neuron Gradients Calculation kernel /// Describes the gradients calculation process for the Neuron of attention layer (#CNeuronAttentionOCL). ///\details Detailed description on <A HREF="https://www.mql5.com/ru/articles/8765#para44">the link.</A> /// @param[in] querys Matrix of Querys /// @param[out] querys_g Matrix of Querys' Gradients /// @param[in] keys Matrix of Keys /// @param[out] keys_g Matrix of Keys' Gradients /// @param[in] values Matrix of Values /// @param[out] values_g Matrix of Values' Gradients /// @param[in] scores Matrix of Scores /// @param[in] gradient Matrix of Gradients from previous iteration //+------------------------------------------------------------------+ __kernel void AttentionIsideGradients(__global double *querys,__global double *querys_g, __global double *keys,__global double *keys_g, __global double *values,__global double *values_g, __global double *scores, __global double *gradient)
Este enfoque genera un bloque de documentación similar, pero al mismo tiempo nos permite separar el bloque de comentarios del código del programa, lo cual hace que el código sea más legible.
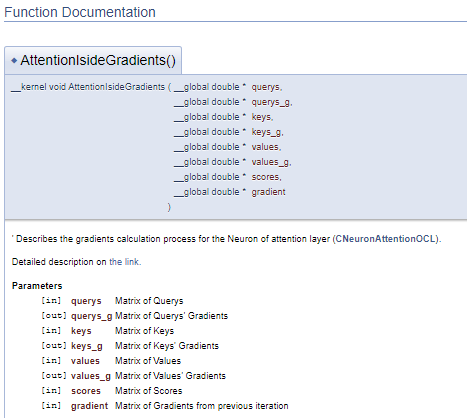
Y, por supuesto, el trabajo principal consistirá en documentar las clases de nuestra biblioteca y sus métodos. Aquí, tenemos que describir todas las clases usadas y sus métodos. Esto requerirá usar todos los comandos anteriores en diferentes variaciones y añadir algunos nuevos. Primero, añadiremos la clase al grupo correspondiente, como mostramos con los kernels (comando \ingroup). El comando "\class" indicará a Doxygen que la siguiente descripción es para una clase. Deberemos indicar el nombre de la clase en los parámetros del comando para vincular la descripción al objeto correcto.
Utilizando los comandos "\brief" y "\details", daremos una descripción breve y ampliada de la clase. En la descripción detallada, crearemos un hipervínculo al artículo correspondiente. Aquí, implementaremos un enlace de anclaje a un apartado específico del artículo, lo cual nos ayudará a encontrar más rápido la información que necesitamos.
Directamente en la línea de declaración de variables, añadiremos su descripción. A continuación, añadiremos los enlaces a los objetos explicativos, si fuera necesario. Cabe señalar que no necesitamos indicar en los comentarios los punteros a las clases de los objetos declarados: Doxygen los añadirá automáticamente.
Vamos a describir de la misma manera los métodos de las clases. No obstante, a diferencia de las variables, deberemos añadir a los comentarios una descripción de los parámetros. Para hacer esto, utilizaremos el comando "\param" ya descrito anteriormente y los punteros "[in]", "[out]", "[in,out]". Describiremos el resultado de la ejecución del método usando el comando "\return".
También podemos vincular a los grupos métodos individuales según la pertenencia. Por ejemplo, combinar según la funcionalidad.
Todo lo anterior se muestra en el siguiente código.
///\ingroup neuron_base ///\class CNeuronBaseOCL ///\brief The base class of neuron for GPU calculation. ///\details Detailed description on <A HREF="https://www.mql5.com/ru/articles/8435#para45">the link.</A> //+------------------------------------------------------------------+ class CNeuronBaseOCL : public CObject { protected: COpenCLMy *OpenCL; ///< Object for working with OpenCL CBufferDouble *Output; ///< Buffer of Output tenzor CBufferDouble *PrevOutput; ///< Buffer of previous iteration Output tenzor CBufferDouble *Weights; ///< Buffer of weights matrix CBufferDouble *DeltaWeights; ///< Buffer of last delta weights matrix (#SGD) CBufferDouble *Gradient; ///< Buffer of gradient tenzor CBufferDouble *FirstMomentum; ///< Buffer of first momentum matrix (#ADAM) CBufferDouble *SecondMomentum; ///< Buffer of second momentum matrix (#ADAM) //--- const double alpha; ///< Multiplier to momentum in #SGD optimization int t; ///< Count of iterations //--- int m_myIndex; ///< Index of neuron in layer ENUM_ACTIVATION activation; ///< Activation type (#ENUM_ACTIVATION) ENUM_OPTIMIZATION optimization; ///< Optimization method (#ENUM_OPTIMIZATION) //--- ///\ingroup neuron_base_ff virtual bool feedForward(CNeuronBaseOCL *NeuronOCL); ///< \brief Feed Forward method of calling kernel ::FeedForward().@param NeuronOCL Pointer to previos layer. ///\ingroup neuron_base_opt virtual bool updateInputWeights(CNeuronBaseOCL *NeuronOCL); ///< Method for updating weights.\details Calling one of kernels ::UpdateWeightsMomentum() or ::UpdateWeightsAdam() in depends of optimization type (#ENUM_OPTIMIZATION).@param NeuronOCL Pointer to previos layer. public: /** Constructor */CNeuronBaseOCL(void); /** Destructor */~CNeuronBaseOCL(void); virtual bool Init(uint numOutputs, uint myIndex, COpenCLMy *open_cl, uint numNeurons, ENUM_OPTIMIZATION optimization_type); ///< Method of initialization class.@param[in] numOutputs Number of connections to next layer.@param[in] myIndex Index of neuron in layer.@param[in] open_cl Pointer to #COpenCLMy object. #param[in] numNeurons Number of neurons in layer @param optimization_type Optimization type (#ENUM_OPTIMIZATION)@return Boolen result of operations. virtual void SetActivationFunction(ENUM_ACTIVATION value) { activation=value; } ///< Set the type of activation function (#ENUM_ACTIVATION) //--- virtual int getOutputIndex(void) { return Output.GetIndex(); } ///< Get index of output buffer @return Index virtual int getPrevOutIndex(void) { return PrevOutput.GetIndex(); } ///< Get index of previous iteration output buffer @return Index virtual int getGradientIndex(void) { return Gradient.GetIndex(); } ///< Get index of gradient buffer @return Index virtual int getWeightsIndex(void) { return Weights.GetIndex(); } ///< Get index of weights matrix buffer @return Index virtual int getDeltaWeightsIndex(void) { return DeltaWeights.GetIndex(); } ///< Get index of delta weights matrix buffer (SGD)@return Index virtual int getFirstMomentumIndex(void) { return FirstMomentum.GetIndex(); } ///< Get index of first momentum matrix buffer (Adam)@return Index virtual int getSecondMomentumIndex(void) { return SecondMomentum.GetIndex();} ///< Get index of Second momentum matrix buffer (Adam)@return Index //--- virtual int getOutputVal(double &values[]) { return Output.GetData(values); } ///< Get values of output buffer @param[out] values Array of data @return number of items virtual int getOutputVal(CArrayDouble *values) { return Output.GetData(values); } ///< Get values of output buffer @param[out] values Array of data @return number of items virtual int getPrevVal(double &values[]) { return PrevOutput.GetData(values); } ///< Get values of previous iteration output buffer @param[out] values Array of data @return number of items virtual int getGradient(double &values[]) { return Gradient.GetData(values); } ///< Get values of gradient buffer @param[out] values Array of data @return number of items virtual int getWeights(double &values[]) { return Weights.GetData(values); } ///< Get values of weights matrix buffer @param[out] values Array of data @return number of items virtual int Neurons(void) { return Output.Total(); } ///< Get number of neurons in layer @return Number of neurons virtual int Activation(void) { return (int)activation; } ///< Get type of activation function @return Type (#ENUM_ACTIVATION) virtual int getConnections(void) { return (CheckPointer(Weights)!=POINTER_INVALID ? Weights.Total()/(Gradient.Total()) : 0); } ///< Get number of connections 1 neuron to next layer @return Number of connections //--- virtual bool FeedForward(CObject *SourceObject); ///< Dispatch method for defining the subroutine for feed forward process. @param SourceObject Pointer to the previous layer. virtual bool calcHiddenGradients(CObject *TargetObject); ///< Dispatch method for defining the subroutine for transferring the gradient to the previous layer. @param TargetObject Pointer to the next layer. virtual bool UpdateInputWeights(CObject *SourceObject); ///< Dispatch method for defining the subroutine for updating weights.@param SourceObject Pointer to previos layer. ///\ingroup neuron_base_gr ///@{ virtual bool calcHiddenGradients(CNeuronBaseOCL *NeuronOCL); ///< Method to transfer gradient to previous layer by calling kernel ::CalcHiddenGradient(). @param NeuronOCL Pointer to next layer. virtual bool calcOutputGradients(CArrayDouble *Target); ///< Method of output gradients calculation by calling kernel ::CalcOutputGradient().@param Target Traget value ///@} //--- virtual bool Save(int const file_handle);///< Save method @param[in] file_handle handle of file @return logical result of operation virtual bool Load(int const file_handle);///< Load method @param[in] file_handle handle of file @return logical result of operation //--- virtual int Type(void) const { return defNeuronBaseOCL; }///< Identificator of class.@return Type of class };
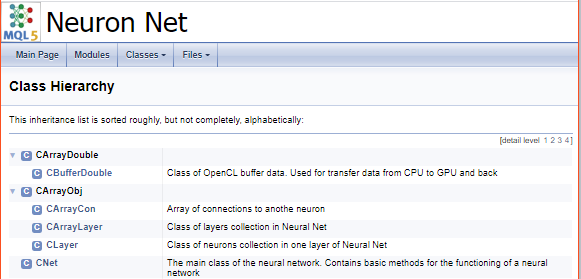
Para terminar de trabajar con el código, crearemos una página de título. Para identificar el bloque de la página de título, usaremos el comando "\mainpage". El comando debe ir seguido del nombre de la página de título. A continuación describiremos nuestro proyecto y crearemos una lista de enlaces. Los elementos de la lista se destacarán con el símbolo "-", y para crear los enlaces a los grupos creados previamente, utilizaremos el comando "\ref". Al generar la documentación de Doxygen, se crearán las páginas de la jerarquía de clases (hierarchy.html) y los archivos usados (files.html). Vamos a añadir a nuestra lista los hipervínculos a las páginas indicadas. A continuación, mostramos el código final de la página de título.
///\mainpage NeuronNet /// Library for creating Neural network for use in MQL5 experts. /// - \ref const /// - \ref enums /// - \ref ObjectTypes /// - \ref group1 /// - [<b>Class Hierarchy</b>](hierarchy.html) /// - [<b>Files</b>](files.html)
Usando como base el código anterior, generaremos la siguiente página.
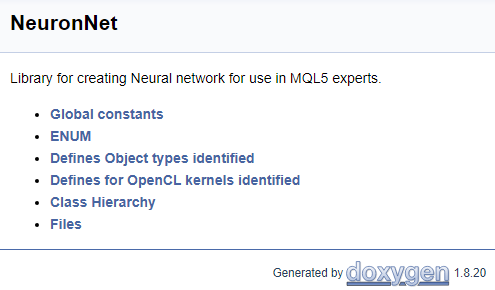
El lector podrá familiarizarse con el código completo de los comentarios en los anexos.
5. Generando la documentación
Una vez hemos terminado de trabajar con el código, podemos pasar a la siguiente etapa. La instalación y configuración de Doxygen se describen con detalle en el artículo [9]. Vamos a detenernos en la configuración de algunos parámetros del programa. Primero, deberemos indicarle a Doxygen con qué archivos debe trabajar; para ello, en el tema Input de la pestaña Expert, añadiremos las máscaras de archivo necesarias al parámetro FILE_PATTERNS. En este caso, hemos añadido "*.mqh" y "*.cl".
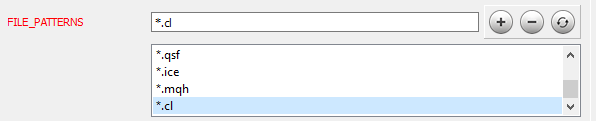
Ahora, deberemos indicarle a Doxygen cómo analizar los archivos añadidos. Para hacerlo, entraremos al tema Project de la misma pestaña Expert y editaremos el parámetro EXTENSION_MAPPING como se muestra en la siguiente figura.
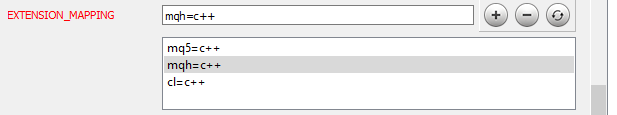
Para que Doxygen pueda generar fórmulas matemáticas, deberemos activar el uso de MathJax. Para hacer esto, en el tema HTML de la pestaña Expert, deberemos activar el parámetro USE_MATHJAX, como se muestra en la siguiente figura.
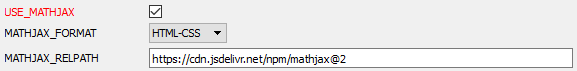
Después de configurar el programa, iremos a la pestaña Wizard e indicaremos el nombre del proyecto, la ruta a los archivos fuente y la ruta para mostrar la documentación generada (estos pasos están bien ilustrados en el artículo [9]). Pasamos a la pestaña Run y ejecutamos el programa de generación de la documentación.
Después de elaborar el programa, obtendremos la documentación lista para usar. A continuación, mostramos algunas capturas de pantalla. El lector podrá encontrar la documentación completa en los anexos.
Conclusión
La documentación de los desarrollos no suele figurar entre las tareas principales del programador. No obstante, a la hora de desarrollar proyectos complejos, la documentación se convierte en un componente fundamental. Su presencia ayuda a monitorear la implementación de las tareas, coordina el trabajo del equipo de desarrollo y simplemente ofrece una visión integral del desarrollo. La documentación es insustituible a la hora de transmitir conocimientos.
El artículo ofrece un mecanismo para documentar los desarrollos en el lenguaje MQL5. Todos los pasos del mecanismo mostrado se describen e ilustran con detalle. Los resultados del trabajo realizado están en el anexo, a la completa disposición del lector para eventuales valoraciones.
Esperamos que la experiencia del autor le resulte de ayuda.
Enlaces
- Redes neuronales: así de sencillo
- Redes neuronales: así de sencillo (Parte 2): Entrenamiento y prueba de la red
- Redes neuronales: así de sencillo (Parte 3): Redes convolucionales
- Redes neuronales: así de sencillo (Parte 4): Redes recurrentes
- Redes neuronales: así de sencillo (Parte 5): Cálculos multihilo en OpenCL
- Redes neuronales: así de sencillo (Parte 6): Experimentos con la tasa de aprendizaje de la red neuronal
- Redes neuronales: así de sencillo (Parte 7): Métodos de optimización adaptativos
- Redes neuronales: así de sencillo (Parte 8): Mecanismos de atención
- Creación automática de documentación para programas en MQL5
- Doxygen
Programas utilizados en el artículo
| # | Nombre | Tipo | Descripción |
|---|---|---|---|
| 1 | NeuroNet.mqh | Biblioteca de clase | Biblioteca de clases para crear la red neuronal |
| 2 | NeuroNet.cl | Biblioteca | Biblioteca de código del programa OpenCL |
| 3 | html.zip | Fichero ZIP | Fichero con la documentación generada por Doxygen |
| 4 | NN.chm | Guía de ayuda de HTML | Archivo convertido de la guía de ayuda HTML. |
| 5 | Doxyfile | Archivo con los parámetros de Doxygen |
Traducción del ruso hecha por MetaQuotes Ltd.
Artículo original: https://www.mql5.com/ru/articles/8819
Advertencia: todos los derechos de estos materiales pertenecen a MetaQuotes Ltd. Queda totalmente prohibido el copiado total o parcial.
Este artículo ha sido escrito por un usuario del sitio web y refleja su punto de vista personal. MetaQuotes Ltd. no se responsabiliza de la exactitud de la información ofrecida, ni de las posibles consecuencias del uso de las soluciones, estrategias o recomendaciones descritas.
 El mercado y la física de sus patrones globales
El mercado y la física de sus patrones globales
 Desarrollando un algoritmo de autoadaptación (Parte II): Aumentando la efectividad
Desarrollando un algoritmo de autoadaptación (Parte II): Aumentando la efectividad
- Aplicaciones de trading gratuitas
- 8 000+ señales para copiar
- Noticias económicas para analizar los mercados financieros
Usted acepta la política del sitio web y las condiciones de uso
La verdad es que no. Comprobamos los valores del objetivo, igual que en las capas ocultas añadimos outpuVal al gradiente para obtener el objetivo y comprobamos su valor. La cuestión es que sigmoide tiene un rango limitado de resultados: función logística de 0 a 1, tanh - de -1 a 1. Si penalizamos la neurona por desviación y aumentamos el factor de ponderación indefinidamente, llegaremos al desbordamiento del peso. Después de todo, si llegamos a un valor de la neurona igual a 1, y la capa posterior de transmitir un error dice que debemos aumentar el valor a 1,5. La neurona obedientemente aumentará los pesos en cada iteración, y la función de activación cortará los valores en el nivel de 1. Por lo tanto, limito los valores del objetivo a los rangos de valores aceptables de la función de activación. Y dejo el ajuste fuera del rango a los pesos de la capa posterior.
Creo que lo he conseguido. Pero me sigo preguntando si este es el enfoque correcto, un ejemplo como este:
si la red se equivoca dando 0 cuando en realidad es 1. A partir de la última capa entonces el gradiente ponderado en la capa anterior viene (lo más probable, según tengo entendido) positivo y puede ser más de 1, digamos 1,6.
Supongamos que hay una neurona en la capa anterior que produjo +0,6, es decir, produjo el valor correcto - su peso debe aumentar en más. Y con esta normalización cortamos el cambio en su peso.
El resultado es norm(1,6)=1. 1-0,6=0,4, y si lo normalizamos como he sugerido, será 1. En este caso, inhibimos la amplificación de la neurona correcta.
¿Qué piensa usted?
Sobre el aumento infinito de pesos, algo así como que he oído que ocurre en caso de "mala función de error", cuando hay muchos mínimos locales y ningún global expreso, o la función no es convexa, algo así, no soy un super experto, sólo creo que se puede y se debe luchar con pesos infinitos y otros métodos.
Pido un experimento para probar ambas variantes. Si se me ocurre como formular la prueba )
Al guardar-lectura de una red desde un archivo, hay un error en la capa de transformadores.
en el método
bool CLayer::CreateElement(int index)
en la línea
int type=FileReadInteger(iFileHandle);
lee 0 y el switch pasa a false por defecto.
(Aparentemente, hay una asincronía de escritura).
Si ya está solucionado, por favor, dadme una pista para agilizarlo, o enviadme el fichero.
Bueno que lo mismo no es arreglar dos veces y simplemente no quieren hacer un montón de cambios en la biblioteca.
Para el trabajo en el probador realizado cambios, y luego se olvida cuando sale un nuevo artículo que usted necesita para transferir a la nueva versión de sus ediciones).
Al guardar y leer una red desde un archivo, se produce un error en la capa de transformadores.
en el método
bool CLayer::CreateElement(int index)
en la cadena
int tipo=FileReadInteger(iFileHandle);
lee 0 y el interruptor pasa a false por defecto.
(Aparentemente hay una asincronía de escritura)
Si ya está arreglado, por favor dame una pista para la velocidad, o envíame el archivo.
Bueno, que uno y el mismo no arreglar dos veces y simplemente no quieren hacer un montón de cambios en la biblioteca.
Para el trabajo en el probador hecho cambios, y luego se olvida cuando sale un nuevo artículo que usted necesita para transferir sus ediciones a la nueva versión).
Buenos días,
Ahora un nuevo artículo está siendo revisado por un moderador. Este error se soluciona allí.
Buenas tardes,
Hay un nuevo artículo que está siendo revisado por un moderador. Este error ha sido corregido allí.
Genial, esperemos.