
最も注目すべき人工協調探索アルゴリズムの修正(ACSm)
内容
1. はじめに
前回の記事では、人工協調探索(ACS)と呼ばれる興味深く将来性のある最適化アルゴリズムについて説明しました。このアルゴリズムは、自然界の生物の相互作用と協力を観察し、それをヒントにしています。生物は共通の目標を達成し、相互利益を得るために団結します。ACSの基本的な考え方は、複雑な多次元問題を最適化するために、「捕食者」と「獲物」の間の相互関係をモデル化することです。
ACSの基本バージョンについて理解した後は、アルゴリズムの修正版を用いてその機能をさらに拡張していきます。これらの拡張バージョンでは、自然の生態系から得た追加のメカニズムを活用し、最適解を見つける効率を向上させます。
ACSの修正版を研究することにより、生物の協力と相互に有益な共存の原理を複雑な最適化問題の解決に適用する方法について深く理解でき、人工知能の分野における新たな視点を得ることができます。これにより、この刺激的な分野のさらなる発展が促進されることが期待されます。
2. アルゴリズムの実装
ACS(修正人工協調検索アルゴリズム)の最初のマイナーな修正には、以下の2つの大きな変更が含まれています。
1. A行列とB行列(母集団)の拡張形式の使用
・この変更では、A行列とB行列に追加の列を設けました。各行にはN個の変数と、その最初のN個の変数に基づいて計算された関数値を格納する追加の列が含まれます。この変更により、関数値の追跡が容易になり、解の精度が向上します。
2. Mバイナリ行列の作成方法の変更
・元のアルゴリズムでは、M行列は「0」値で埋められ、その後いくつかの要素が選択されて「1」に設定されていました。今回の変更により、少なくとも実験で使用したテスト関数において、回の精度がわずかに向上する結果となりました。
これらの最初のマイナーな修正は、最適化問題を解決する際に解の精度とアルゴリズムの効率を向上させることを目的としています。著者が提案する方法では、個体の適応度を格納するための追加の列を設ける代わりに、集団内の個体を記述するエージェント構造において、既に知られている「f」フィールドを使用しています。
次に、ACSアルゴリズムの2番目の重要な変更には、元のバージョンとは異なる次の2つの変更が含まれています。
1. 捕食者行列と被食者行列の埋め方の変更
・拡張されたA行列とB行列の各行が、適応度関数の値に基づいて並び替えられます。その後、並び替えられた行列を比較し、比較結果に基づいて捕食者行列と被食者行列を構築します。この方法により、ランダム選択ではなく、メリット(特徴値)に基づいて候補が選択されます。
2. ランダム比較によるA行列とB行列の更新
・変更により、メインループの各反復でランダム比較によってA行列とB行列が更新されます。つまり、両方の行列が更新される確率が等しくなり、このアプローチにより、両集団の更新が均等に行われることになります。これにより、最適解探索の多様性が向上します。
これらの修正により、候補選択とAおよびB集団の更新方法が改善され、最適解探索の効率と多様性が向上します。このバージョンでは、捕食者集団と被食者集団の形成方法と、行列更新におけるランダム比較の使用方法が異なります。
ACSアルゴリズムの3番目の主要な変更は、捕食者と被食者の行列の形成に対するまったく異なるアプローチを示しています。この変更について詳述します。
1. Pop集団の作成
・拡張されたA行列とB行列を統合し、新たにPop行列を作成します。PopにはA行列とB行列のすべての行が含まれ、適応度関数に基づいて並べ替えられます。その後、Popの最初のpopSize行が捕食者集団、最後のpopSize行が被食者集団として選ばれます。
2. 捕食者と被食者の更新
・捕食者と被食者の集団が形成された後、アルゴリズムは各個体の適応度に基づいて更新をおこないます。選ばれた最良の捕食者が優先され、アルゴリズムの精度と収束速度が向上します。
3.ランダム比較の使用
・2番目の変更と同様に、メインループの各反復の最後に、ランダム比較によりA行列とB行列が更新されます。これにより、両集団の更新機会が均等になり、最適解探索の多様性が促進されます。
この3番目の修正は、捕食者と被食者の集団を適応度に基づいて形成し、より効率的で高度なアプローチを用いて最適解を検索し、集団を更新する方法を提供します。
アルゴリズムの基本バージョン(ACS)への変更を検討した後、この有望な最適化方法の最初の改良版を実装します。目標は、最適解を見つける効率を向上させることです。これから、ACSの基本構造に新しい要素を段階的に導入し、その影響を慎重に分析します。
最初の修正バージョンを実装し、その可能性を探りましょう。アルゴリズムの以降の各バージョンでは、前のバージョンと異なるコードの部分が緑色で示されます。
C_AO_ACSm1クラスコードの説明
1. C_AO_ACSm1は、C_AO基底クラスから派生したクラスです。
2. クラスにはpublicコンストラクタとデストラクタがあります。
3.次のデータメンバーはコンストラクタで初期化されます。
- ao_name、ao_desc、ao_link:アルゴリズムに関する説明行
- popSize:母集団の大きさ
- bioProbab:生物学的相互作用の確率
- params:アルゴリズムパラメータの配列。ここでは、popSizeとbioProbabの2つのパラメータがあります。
4. SetParams():このメソッドは、params配列のパラメータに基づいてpopSizeとbioProbabの値を設定します。
5. Init():このメソッドは、範囲と検索ステップ、およびエポック数を入力として受け取り、初期化の成功を示す論理値を返します。
6. Moving()およびRevision()メソッドには、エージェントを移動および修正するためのアルゴリズムの基本ロジックが含まれています。
7. Privateデータメンバーには、S_AO_Agent型およびS_C型の構造体の配列、およびアルゴリズムで使用されるKey変数およびphase変数が含まれます。
8. ArrayShuffle():このprivateメソッドは配列要素をシャッフルします。
//—————————————————————————————————————————————————————————————————————————————— class C_AO_ACSm1 : public C_AO { public: //-------------------------------------------------------------------- ~C_AO_ACSm1 () { } C_AO_ACSm1 () { ao_name = "ACS"; ao_desc = "Artificial Cooperative Search m1"; ao_link = "https://www.mql5.com/ru/articles/15014"; popSize = 1; //population size bioProbab = 0.9; //biological interaction probability ArrayResize (params, 2); params [0].name = "popSize"; params [0].val = popSize; params [1].name = "bioProbab"; params [1].val = bioProbab; } void SetParams () { popSize = (int)params [0].val; bioProbab = params [1].val; } bool Init (const double &rangeMinP [], //minimum search range const double &rangeMaxP [], //maximum search range const double &rangeStepP [], //step search const int epochsP = 0); //number of epochs void Moving (); void Revision (); //---------------------------------------------------------------------------- double bioProbab; //biological interaction probability private: //------------------------------------------------------------------- S_AO_Agent A []; S_AO_Agent B []; S_AO_Agent Predator []; S_AO_Agent Prey []; S_C M []; int Key; int phase; void ArrayShuffle (double &arr []); }; //——————————————————————————————————————————————————————————————————————————————
C_AO_ACSm1クラスのInitメソッドはオブジェクトの初期化を実行します。
1. Init():メソッド宣言。最小検索範囲rangeMinP、最大検索範囲rangeMaxP、検索ステップrangeStepP、エポック数epochsP(デフォルトでは0)の4つの引数があります。
2. if (!StandardInit (rangeMinP, rangeMaxP, rangeStepP)) return false:基底クラスの標準初期化を実行するStandardInit関数を呼び出します。StandardInitがfalseを返した場合、Initメソッドは直ちに終了し、falseを返します。
3.for (int i = 0; i < popSize; i++)ループ:A、B、Predator、Prey、M配列の各要素がInitメソッドを使用して初期化されます。
4. ArrayResize (A, popSize)などの行:A、B、Predator、Prey、M配列のサイズをpopSizeに変更します。
5. ネストされたループfor (int j = 0; j < coords; j++)では、A配列とB配列の各オブジェクトの各c要素(個々の座標)が、指定された範囲内のランダムな値を使用して初期化されます。
6. phase = 0:phaseを0に設定します。
7. 初期化が成功した場合、メソッドはtrueを返します。
コードは、初期設定と、その後のアルゴリズム操作に必要なデータの準備を担当します。オブジェクトの配列を作成し、その値を初期化し、アルゴリズムの初期状態を設定します。
//—————————————————————————————————————————————————————————————————————————————— bool C_AO_ACSm1::Init (const double &rangeMinP [], //minimum search range const double &rangeMaxP [], //maximum search range const double &rangeStepP [], //step search const int epochsP = 0) //number of epochs { if (!StandardInit (rangeMinP, rangeMaxP, rangeStepP)) return false; //---------------------------------------------------------------------------- ArrayResize (A, popSize); ArrayResize (B, popSize); ArrayResize (Predator, popSize); ArrayResize (Prey, popSize); ArrayResize (M, popSize); for (int i = 0; i < popSize; i++) { A [i].Init (coords); B [i].Init (coords); Predator [i].Init (coords); Prey [i].Init (coords); M [i].Init (coords); } // Initialization for (int i = 0; i < popSize; i++) { for (int j = 0; j < coords; j++) { A [i].c [j] = rangeMin [j] + (rangeMax [j] - rangeMin [j]) * u.RNDprobab (); B [i].c [j] = rangeMin [j] + (rangeMax [j] - rangeMin [j]) * u.RNDprobab (); } } phase = 0; return true; } //——————————————————————————————————————————————————————————————————————————————
C_AO_ACSm1クラスのMovingメソッドは、アルゴリズム内で個体を移動する基本ロジックを実装します。
1. if (phase == 0):個体はA集団からa配列にコピーされます(「а」配列は、適応度関数の計算のために個体を渡すために使用されます)。
- ループ(int i = 0; i < popSize; i++)内の各要素に対して、個々の座標がAからa配列にコピーされます。
- phase変数の値が1増加し、メソッドから戻ります。
2. if (phase == 1):適応度がA集団の個体に割り当てられ、B集団の個体がコピーされます。
- ループfor (int i = 0; i < popSize; i++)内の各要素に対して、a [i].f適応値がA [i].fにコピーされます。
- 次に、ループfor (int i = 0; i < popSize; i++)内の各要素について、個体の適応度をさらに計算するために、個体座標がBからa配列にコピーされます。
- phase値が1増加し、メソッドから戻ります。
3. if (phase == 2):適応度がB集団の個体に返されます。
- for (int i = 0; i < popSize; i++)ループ内の各要素に対して、a [i].fの適合値がB [i].fにコピーされます。
- phase値が1増加し、さらにアルゴリズムロジックが実行されます。
4. //Selection:ここで選択がおこなわれます。
- u.RNDprobab()のランダム値が0.5未満の場合、(int i = 0; i < popSize; i++)ループ内の各要素に対して、A [i]の個体がPredator [i]にコピーされ、Keyが1に設定されます。
- それ以外の場合、for (int i = 0; i < popSize; i++)ループ内の各要素に対して、B [i]の個体がPredator [i]にコピーされ、Keyが2に設定されます。
- Prey配列の選択も同様の方法で実行されます。
5. //Permutation of Prey:ここでは、Prey集団の各個体の座標がランダムに並べ替えられます。
- for (int i = 0; i < popSize; i++)ループ内の各個体に対して、個体座標を混合してArrayShuffle (Prey [i].c)を実行します(各個体の座標は個別に混合されますが、個体間の座標は混合されません)。
6. //Mutationと//Crossover:ここで突然変異と交差が実行されます。
- R値は乱数に応じて計算されます。
- Mバイナリ行列は1で埋められます。
- 生物学的相互作用のbioProbab確率に応じて、M行列の一部の要素を0または1に変更することにより、追加の操作が実行されます。
- (int i = 0; i < popSize; i++)ループ内の各個体に対して突然変異と交差が実行され、a集団配列の値が更新されます。
7. //Boundary control:このブロックは境界制御を実行し、最適化されたパラメータの指定された範囲外にあるa集団個体の座標を、範囲内のランダムな値に置き換えます。
このメソッドは、選択、突然変異、交差、境界制御などのアルゴリズムの基本的な操作を実装し、最適解を見つけるために個体の集団に適用します。
//—————————————————————————————————————————————————————————————————————————————— void C_AO_ACSm1::Moving () { //---------------------------------------------------------------------------- if (phase == 0) { for (int i = 0; i < popSize; i++) ArrayCopy (a [i].c, A [i].c); phase++; return; } //---------------------------------------------------------------------------- if (phase == 1) { for (int i = 0; i < popSize; i++) A [i].f = a [i].f; for (int i = 0; i < popSize; i++) ArrayCopy (a [i].c, B [i].c); phase++; return; } //---------------------------------------------------------------------------- if (phase == 2) { for (int i = 0; i < popSize; i++) B [i].f = a [i].f; phase++; } //---------------------------------------------------------------------------- // Selection if (u.RNDprobab () < 0.5) { for (int i = 0; i < popSize; i++) { Predator [i] = A [i]; } Key = 1; } else { for (int i = 0; i < popSize; i++) { Predator [i] = B [i]; } Key = 2; } if (u.RNDprobab () < 0.5) { for (int i = 0; i < popSize; i++) { Prey [i] = A [i]; } } else { for (int i = 0; i < popSize; i++) { Prey [i] = B [i]; } } // Permutation of Prey for (int i = 0; i < popSize; i++) { ArrayShuffle (Prey [i].c); } double R; if (u.RNDprobab () < 0.5) { R = 4 * u.RNDprobab () * u.RNDfromCI (-1.0, 1.0); } else R = 1 / MathExp (4 * MathRand () / 32767.0); // Fill binary matrix M with 0s for (int i = 0; i < popSize; i++) { for (int j = 0; j < coords; j++) { M [i].c [j] = 0; } } // Additional operations with matrix M for (int i = 0; i < popSize; i++) { for (int j = 0; j < coords; j++) { if (u.RNDprobab () < bioProbab) { M [i].c [j] = 1; } } } for (int i = 0; i < popSize; i++) { for (int j = 0; j < coords; j++) { if (u.RNDprobab () < bioProbab) { M [i].c [j] = 1; } } } for (int i = 0; i < popSize; i++) { int sum = 0; for (int c = 0; c < coords; c++) sum += M [i].c [c]; if (sum == coords) { int j = MathRand () % coords; M [i].c [j] = 0; } } // Mutation for (int i = 0; i < popSize; i++) { for (int j = 0; j < coords; j++) { a [i].c [j] = Predator [i].c [j] + R * (Prey [i].c [j] - Predator [i].c [j]); // Crossover if (M [i].c [j] > 0) { a [i].c [j] = Predator [i].c [j]; } // Boundary control if (a [i].c [j] < rangeMin [j] || a [i].c [j] > rangeMax [j]) { a [i].c [j] = rangeMin [j] + (rangeMax [j] - rangeMin [j]) * u.RNDprobab (); } } } //---------------------------------------------------------------------------- for (int i = 0; i < popSize; i++) { for (int j = 0; j < coords; j++) { a [i].c [j] = u.SeInDiSp (a [i].c [j], rangeMin [j], rangeMax [j], rangeStep [j]); } } } //——————————————————————————————————————————————————————————————————————————————
次に、C_AO_ACSm1クラスのRevisionメソッドコードについて考えてみましょう。このメソッドは次の操作を実行します。
1. phase母集団の準備フェーズがすでに完了しているかどうかを確認します(phase >= 3)。この条件が満たされない場合、メソッドはそれ以上のアクションを実行せずに戻ります。
2. //Selection update:個体の選択を更新します:
- a集団内の各個体について、そのf適応度値がPredator配列内の対応する値を超えているかどうかを確認します。超えている場合、Predatorの値を更新します。
- Key変数の値に応じて、Predator集団の個体がA集団またはB集団にコピーされます。
3.Predator配列内のYbest最適適応度値とそのYbestインデックスを決定します。
- Predator配列を反復処理し、最大適合値とそのインデックスを見つけます。
4. fB大域的最適解を更新します。
- Ybestが現在のfBの最適値を超えると、fBが更新され、適切なaおよびcB配列要素がPredator配列からコピーされます。
このメソッドは、個々の選択を更新し、最適解を決定し、最適化アルゴリズム内で大域的最適解を更新します。
//—————————————————————————————————————————————————————————————————————————————— void C_AO_ACSm1::Revision () { if (phase < 3) return; // Selection update for (int i = 0; i < popSize; i++) { double d = a [i].f; if (d > Predator [i].f) { Predator [i] = a [i]; } } if (Key == 1) { for (int i = 0; i < popSize; i++) { A [i] = Predator [i]; } } else { for (int i = 0; i < popSize; i++) { B [i] = Predator [i]; } } double Ybest = -DBL_MAX; int Ibest = -1; for (int i = 0; i < popSize; i++) { if (Predator [i].f > Ybest) { Ybest = Predator [i].f; Ibest = i; } } if (Ybest > fB) { fB = Ybest; ArrayCopy (a [Ibest].c, Predator [Ibest].c); ArrayCopy (cB, Predator [Ibest].c); } } //——————————————————————————————————————————————————————————————————————————————
C_AO_ACSクラスのArrayShuffleメソッドは、3つの変更すべてに適用され、配列内の要素のランダムなシャッフルを実行します。
1. ArraySize関数を使用して、arr配列のサイズを定義します。
2. 最後の要素から始めて、arr配列を逆順に反復します。
3.各i要素に対して、0からiの範囲のランダムなインデックスjを生成します。
4. tmp一時変数を使用してarr[i]要素とarr[j]要素を交換します。
その結果、arr配列の要素はランダムにシャッフルされます。
//—————————————————————————————————————————————————————————————————————————————— void C_AO_ACSm1::ArrayShuffle (double &arr []) { int n = ArraySize (arr); for (int i = n - 1; i > 0; i--) { int j = MathRand () % (i + 1); double tmp = arr [i]; arr [i] = arr [j]; arr [j] = tmp; } } //——————————————————————————————————————————————————————————————————————————————
ここで、ACSアルゴリズムの2番目の変更のコードを分析します。以下は、変更に応じたC_AO_ACSm2クラスのMovingメソッドコードです。
1. Phase 0
- phaseが0に等しい場合、集団a内の各個体は集団Aから値を取得します。
- phase値が増分され、メソッドから戻ります。
2. Phase 1
- phaseが1の場合、集団a内の各個体は集団A内でfの適応度値を取得します。
- 集団Aの各個体は集団Bから値を取得します。
- phase値が増分され、メソッドから戻ります。
3.Phase 2
- phaseが2の場合、B集団内の各個体はa集団からf適応度値を取得します。
- phase値が増加し、さらにアルゴリズムロジックが続行されます。
4. //Selection:
- A集団とB集団の各個体について、f適応度値が比較されます。
- Aのf値がBを超える場合、Aの個体はPredatorにコピーされ、Bの個体はPreyにコピーされます。それ以外の場合は、逆になります(最も適応力のある個体は「捕食者」に指定され、最も適応力のない個体は「獲物」になります)。
5. //Permutation of Prey
- Prey集団内の各個体の座標は、ArrayShuffle関数を使用して並べ替えられます。
6. //Mutationおよび//Crossover
- R値は乱数に応じて定義されます。
- Mバイナリ行列は0で埋められます。
- M行列に対して追加の操作が実行され、bioProbab確率に応じて一部の要素が1に設定されます。
a集団内の各個体について
- 突然変異の実行:a[i].c[j]要素は、Predator[i].c[j]にPrey[i].c[j]とPredator[i].c[j]の差にRを乗算した値を加算して計算されます。
- 交差の実行:M[i].c[j]が1に等しい場合、a[i].c[j]はPredator[i].c[j]に置き換えられます。
- 境界チェック:a[i].c[j]がrangeMin[j]~rangeMax[j]の範囲外の場合、範囲内のランダムな値に置き換えられます。
7. 座標の確認:
- a集団内の各個体については、SeInDiSp関数を使用して座標がチェックされます。
//—————————————————————————————————————————————————————————————————————————————— void C_AO_ACSm2::Moving () { //---------------------------------------------------------------------------- if (phase == 0) { for (int i = 0; i < popSize; i++) ArrayCopy (a [i].c, A [i].c); phase++; return; } //---------------------------------------------------------------------------- if (phase == 1) { for (int i = 0; i < popSize; i++) A [i].f = a [i].f; for (int i = 0; i < popSize; i++) ArrayCopy (a [i].c, B [i].c); phase++; return; } //---------------------------------------------------------------------------- if (phase == 2) { for (int i = 0; i < popSize; i++) B [i].f = a [i].f; phase++; } //---------------------------------------------------------------------------- // Selection for (int i = 0; i < popSize; i++) { if (A [i].f > B [i].f) { Predator [i] = A [i]; Prey [i] = B [i]; } else { Predator [i] = B [i]; Prey [i] = A [i]; } } // Permutation of Prey for (int i = 0; i < popSize; i++) { ArrayShuffle (Prey [i].c); } double R; if (u.RNDprobab () < 0.5) { R = 4 * u.RNDprobab () * u.RNDfromCI (-1.0, 1.0); } else R = 1 / MathExp (4 * MathRand () / 32767.0); // Fill binary matrix M with 0s for (int i = 0; i < popSize; i++) { for (int j = 0; j < coords; j++) { M [i].c [j] = 0; } } // Additional operations with matrix M for (int i = 0; i < popSize; i++) { for (int j = 0; j < coords; j++) { if (u.RNDprobab () < bioProbab) { M [i].c [j] = 1; } } } for (int i = 0; i < popSize; i++) { for (int j = 0; j < coords; j++) { if (u.RNDprobab () < bioProbab) { M [i].c [j] = 1; } } } for (int i = 0; i < popSize; i++) { int sum = 0; for (int c = 0; c < coords; c++) sum += M [i].c [c]; if (sum == coords) { int j = MathRand () % coords; M [i].c [j] = 0; } } // Mutation for (int i = 0; i < popSize; i++) { for (int j = 0; j < coords; j++) { a [i].c [j] = Predator [i].c [j] + R * (Prey [i].c [j] - Predator [i].c [j]); // Crossover if (M [i].c [j] > 0) { a [i].c [j] = Predator [i].c [j]; } // Boundary control if (a [i].c [j] < rangeMin [j] || a [i].c [j] > rangeMax [j]) { a [i].c [j] = rangeMin [j] + (rangeMax [j] - rangeMin [j]) * u.RNDprobab (); } } } //---------------------------------------------------------------------------- for (int i = 0; i < popSize; i++) { for (int j = 0; j < coords; j++) { a [i].c [j] = u.SeInDiSp (a [i].c [j], rangeMin [j], rangeMax [j], rangeStep [j]); } } } //——————————————————————————————————————————————————————————————————————————————
2番目のアルゴリズム変更のRevisionメソッドはそのまま残っているため、ここでは考慮しません。
3番目の変更に移りましょう。C_AO基底クラスから派生したC_AO_ACSm3の3番目の変更クラスの定義に次の変更が加えられました。
データメンバーのリストが変更されました。
- bioProbab:生物学的相互作用の確率
- A[]、B[]、Predator[]、Prey[]、Pop[]、pTemp[]:さまざまな個体群を格納する配列
- M[]:個人に関連付けられた追加データを格納する配列
- phase:アルゴリズムの現在のフェーズ
//—————————————————————————————————————————————————————————————————————————————— class C_AO_ACSm3 : public C_AO { public: //-------------------------------------------------------------------- ~C_AO_ACSm3 () { } C_AO_ACSm3 () { ao_name = "ACS"; ao_desc = "Artificial Cooperative Search m3"; ao_link = "https://www.mql5.com/ru/articles/15014"; popSize = 10; //population size bioProbab = 0.9; //biological interaction probability ArrayResize (params, 2); params [0].name = "popSize"; params [0].val = popSize; params [1].name = "bioProbab"; params [1].val = bioProbab; } void SetParams () { popSize = (int)params [0].val; bioProbab = params [1].val; } bool Init (const double &rangeMinP [], //minimum search range const double &rangeMaxP [], //maximum search range const double &rangeStepP [], //step search const int epochsP = 0); //number of epochs void Moving (); void Revision (); //---------------------------------------------------------------------------- double bioProbab; //biological interaction probability private: //------------------------------------------------------------------- S_AO_Agent A []; S_AO_Agent B []; S_AO_Agent Predator []; S_AO_Agent Prey []; S_AO_Agent Pop []; S_AO_Agent pTemp []; S_C M []; int phase; void ArrayShuffle (double &arr []); }; //——————————————————————————————————————————————————————————————————————————————
C_AO_ACSm3クラスのInit初期化メソッドに次の変更が加えられました。
1. popSizeまたはpopSize * 2の配列A、B、Predator、Prey、Pop、pTemp、Mにメモリが割り当てられます。
2. A、B、Predator、Prey、Pop、M配列の各要素は、Initメソッドを使用して初期化されます。
アルゴリズムのオリジナルバージョンの著者は、ロジックをフェーズに分割していません。すべての母集団アルゴリズムに採用したスタイルでアルゴリズムを実装するために、フェーズへの分割を追加しました。ACSはA母集団とB母集団のエージェントの適応度の事前計算を使用するため、これが必要でした。これらの操作はメソッド内で順番に実行する必要があります。
//—————————————————————————————————————————————————————————————————————————————— bool C_AO_ACSm3::Init (const double &rangeMinP [], //minimum search range const double &rangeMaxP [], //maximum search range const double &rangeStepP [], //step search const int epochsP = 0) //number of epochs { if (!StandardInit (rangeMinP, rangeMaxP, rangeStepP)) return false; //---------------------------------------------------------------------------- ArrayResize (A, popSize); ArrayResize (B, popSize); ArrayResize (Predator, popSize); ArrayResize (Prey, popSize); ArrayResize (Pop, popSize * 2); ArrayResize (pTemp, popSize * 2); ArrayResize (M, popSize); for (int i = 0; i < popSize; i++) { A [i].Init (coords); B [i].Init (coords); Predator [i].Init (coords); Prey [i].Init (coords); M [i].Init (coords); } for (int i = 0; i < popSize * 2; i++) Pop [i].Init (coords); // Initialization for (int i = 0; i < popSize; i++) { for (int j = 0; j < coords; j++) { A [i].c [j] = rangeMin [j] + (rangeMax [j] - rangeMin [j]) * u.RNDprobab (); B [i].c [j] = rangeMin [j] + (rangeMax [j] - rangeMin [j]) * u.RNDprobab (); } } phase = 0; return true; } //——————————————————————————————————————————————————————————————————————————————
次に、3番目の変更のC_AO_ACSm3クラス内のMovingメソッドのコードを見てみましょう。変更は次のコード領域に影響しました。
- A集団とB集団を1つのPop集団に統合します。
- Pop集団を個体の適応度値で並び替えます。
- 並び替え済みのPop配列からPredatorとPreyの個体群を入力します。個体群の最も優れた半分が「捕食者」になり、残りの半分が「獲物」になります。
- Mバイナリ行列を1で埋めます。
- M行列の追加操作。乱数と生物学的相互作用のbioProbab確率に応じて、一部の要素が0または1に変化します。
//—————————————————————————————————————————————————————————————————————————————— void C_AO_ACSm3::Moving () { //---------------------------------------------------------------------------- if (phase == 0) { for (int i = 0; i < popSize; i++) ArrayCopy (a [i].c, A [i].c); phase++; return; } //---------------------------------------------------------------------------- if (phase == 1) { for (int i = 0; i < popSize; i++) A [i].f = a [i].f; for (int i = 0; i < popSize; i++) ArrayCopy (a [i].c, B [i].c); phase++; return; } //---------------------------------------------------------------------------- if (phase == 2) { for (int i = 0; i < popSize; i++) B [i].f = a [i].f; phase++; } //---------------------------------------------------------------------------- // Merge matrices A and B to create matrix Pop for (int i = 0; i < popSize; i++) { Pop [i] = A [i]; Pop [i + popSize] = B [i]; } // Sort matrix Pop using column N as sort key values u.Sorting (Pop, pTemp, popSize * 2); // Populate Predator and Prey matrices for (int i = 0; i < popSize; i++) { Predator [i] = Pop [i]; Prey [i] = Pop [i + popSize]; } // Permutation of Prey for (int i = 0; i < popSize; i++) { ArrayShuffle (Prey [i].c); } double R; if (u.RNDprobab () < 0.5) { R = 4 * u.RNDprobab () * u.RNDfromCI (-1.0, 1.0); } else R = 1 / MathExp (4 * MathRand () / 32767.0); // Fill binary matrix M with 1s for (int i = 0; i < popSize; i++) { for (int j = 0; j < coords; j++) { M [i].c [j] = 1; } } // Additional operations with matrix M for (int i = 0; i < popSize; i++) { for (int j = 0; j < coords; j++) { if (u.RNDprobab () < bioProbab) { M [i].c [j] = 0; } } } for (int i = 0; i < popSize; i++) { for (int j = 0; j < coords; j++) { if (u.RNDprobab () < bioProbab) { if (u.RNDprobab() < bioProbab) { M[i].c[j] = 1; } else { M[i].c[j] = 0; } } } } for (int i = 0; i < popSize; i++) { int sum = 0; for (int c = 0; c < coords; c++) sum += M [i].c [c]; if (sum == coords) { int j = MathRand () % coords; M [i].c [j] = 0; } } // Mutation for (int i = 0; i < popSize; i++) { for (int j = 0; j < coords; j++) { a [i].c [j] = Predator [i].c [j] + R * (Prey [i].c [j] - Predator [i].c [j]); // Crossover if (M [i].c [j] > 0) { a [i].c [j] = Predator [i].c [j]; } // Boundary control if (a [i].c [j] < rangeMin [j] || a [i].c [j] > rangeMax [j]) { a [i].c [j] = rangeMin [j] + (rangeMax [j] - rangeMin [j]) * u.RNDprobab (); } } } //---------------------------------------------------------------------------- for (int i = 0; i < popSize; i++) { for (int j = 0; j < coords; j++) { a [i].c [j] = u.SeInDiSp (a [i].c [j], rangeMin [j], rangeMax [j], rangeStep [j]); } } } //——————————————————————————————————————————————————————————————————————————————
3.テスト結果
このアルゴリズムの既知のバージョンをすべて注意深く検討し、分析しました。次は、それらの実際の結果とそこから得られる結論に焦点を当てます。それぞれの変更が生産性、精度、効率性にどのような影響を与えたかを詳しく検討したいと思います。これにより、どの変更が最も成功したか、次にどこへ進むべきかをよりよく理解できるようになります。
修正の最初のバージョンでは、全体的なスコアではほぼ同等の結果を示しましたが、1000個の変数を持つHilly関数と、50個および1000個の変数を持つForest関数では結果が大幅に改善されました。
ACS|Artificial Cooperative Search m1|1.0|0.7|
=============================
5 Hilly's; Func runs:10000; result:0.7130880902279995
25 Hilly's; Func runs:10000; result:0.7565145335137569
500 Hilly's; Func runs:10000; result:0.31899537529427235
=============================
5 Forest's; Func runs:10000; result:0.9999999999866176
25 Forest's; Func runs:10000; result:0.9555551824899264
500 Forest's; Func runs:10000; result:0.24186829565864398
=============================
5 Megacity's; Func runs:10000; result:0.6607692307692307
25 Megacity's; Func runs:10000; result:0.5038461538461539
500 Megacity's; Func runs:10000; result:0.13922307692307825
=============================
All score:5.28986 (58.78%)
アルゴリズムの2番目のバージョンでは、50および1000変数のすべてのテスト関数で結果が顕著に改善されましたが、基本バージョンと比較して10変数のMegacityでは結果が大幅に悪化し(結果のばらつきが非常に大きい)、この方向への動きが有望であること(議論の余地はあるものの)を示しています。したがって、さらなる変更に取り組む価値があると言えます。
ACS|Artificial Cooperative Search m2|2.0|0.7|
=============================
5 Hilly's; Func runs:10000; result:0.7682797921658492
25 Hilly's; Func runs:10000; result:0.7664893907210706
500 Hilly's; Func runs:10000; result:0.31831672493319296
=============================
5 Forest's; Func runs:10000; result:0.9999997349437437
25 Forest's; Func runs:10000; result:0.9534110489423269
500 Forest's; Func runs:10000; result:0.24425762117784502
=============================
5 Megacity's; Func runs:10000; result:0.6176923076923077
25 Megacity's; Func runs:10000; result:0.5384615384615385
500 Megacity's; Func runs:10000; result:0.14057692307692446
=============================
All score:5.34749 (59.42%)
残念ながら、最も有望に思えた3番目の修正は期待に応えられず、アルゴリズムの基本バージョンよりも全体的にやや悪い結果を示しました。ただし、10個の変数を持つ滑らかなHilly関数で結果が大幅に改善されたことは注目に値します。このアルゴリズムは、少数の変数を持つ滑らかな関数に対してのみ、より高い収束精度を示します。
ACS|Artificial Cooperative Search m3|10.0|0.9|
=============================
5 Hilly's; Func runs:10000; result:0.8836798635515516
25 Hilly's; Func runs:10000; result:0.715438105666966
500 Hilly's; Func runs:10000; result:0.3011611038405591
=============================
5 Forest's; Func runs:10000; result:0.9893902762645717
25 Forest's; Func runs:10000; result:0.7954795408759185
500 Forest's; Func runs:10000; result:0.21910399769909533
=============================
5 Megacity's; Func runs:10000; result:0.6315384615384615
25 Megacity's; Func runs:10000; result:0.49876923076923074
500 Megacity's; Func runs:10000; result:0.12683076923077044
=============================
All score:5.16139 (57.35%)
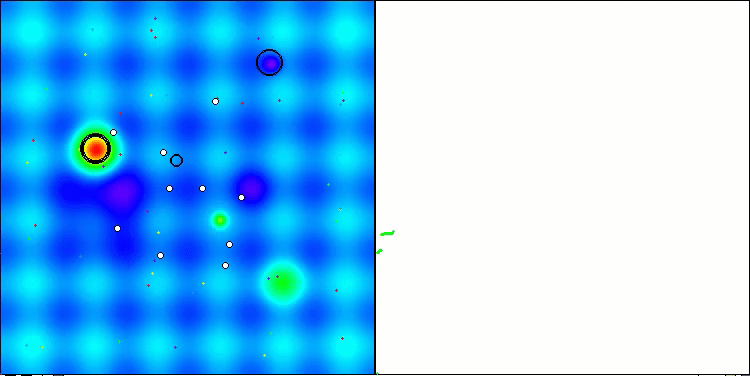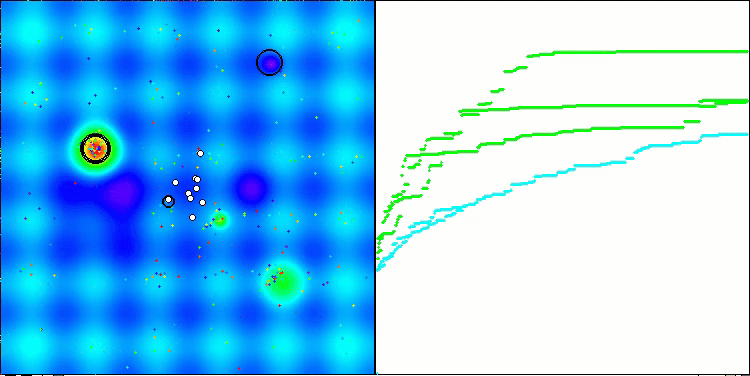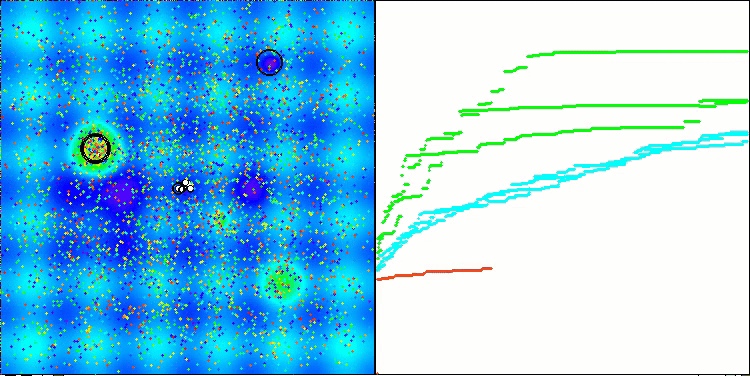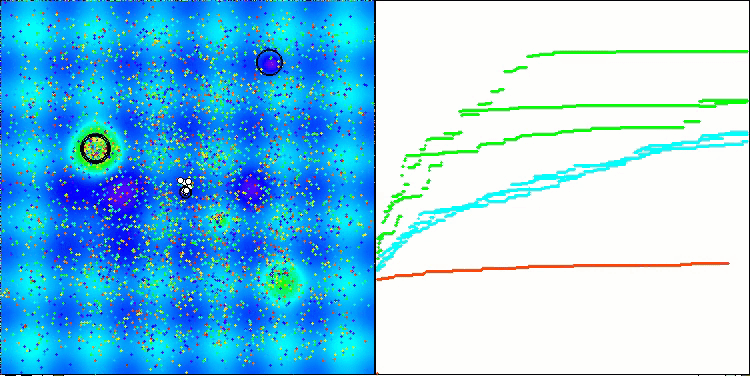
以下は、アルゴリズムの2番目の変更の可視化です。変数の数が少ないため、Hilly関数とMegacity関数の結果にはかなり大きなばらつきがありますが、スパイク状の滑らかなForest関数ではアルゴリズムが優れているように感じられます。
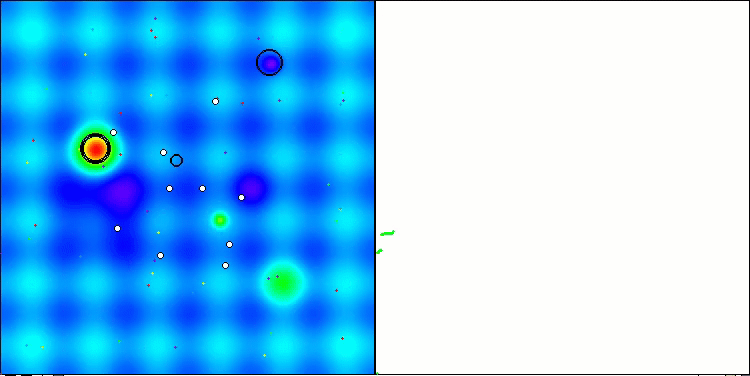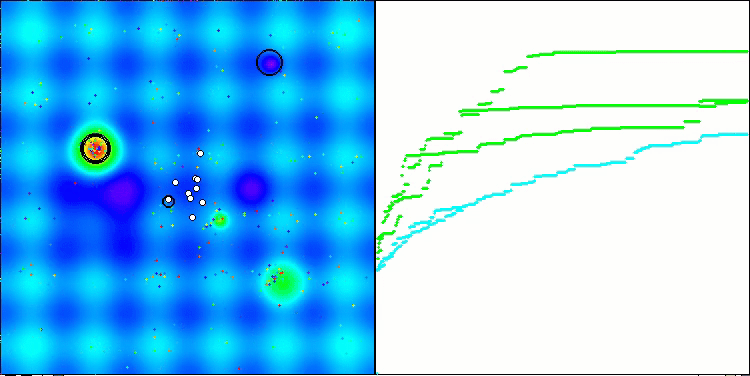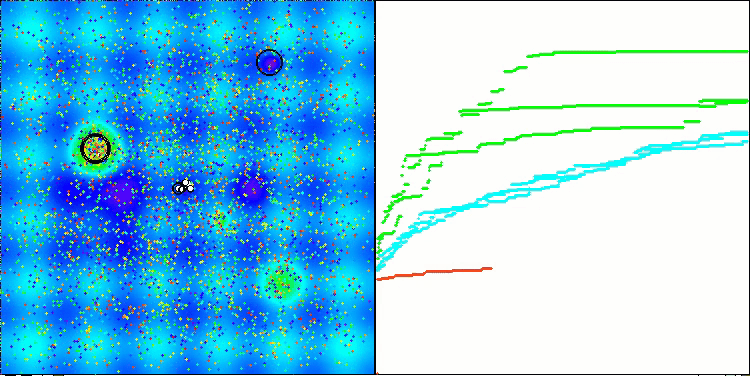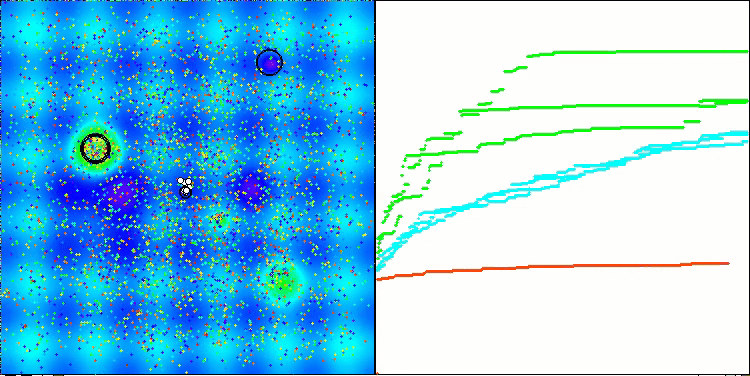
Hillyテスト関数のACSm2
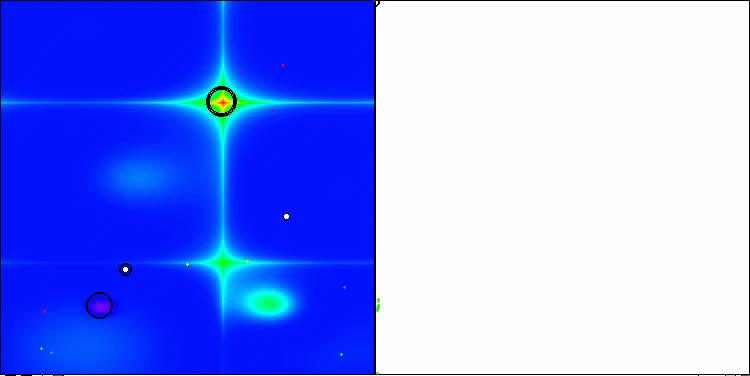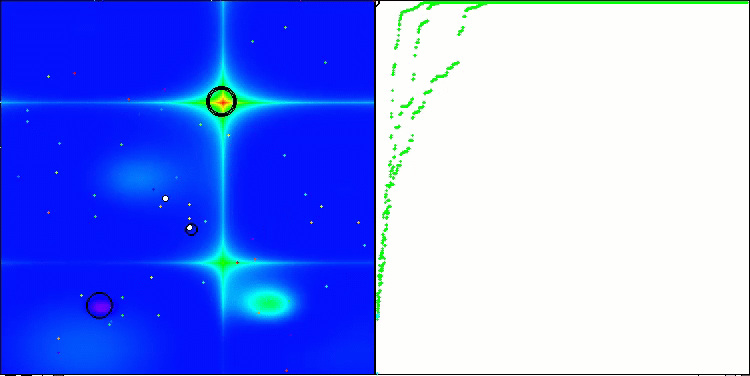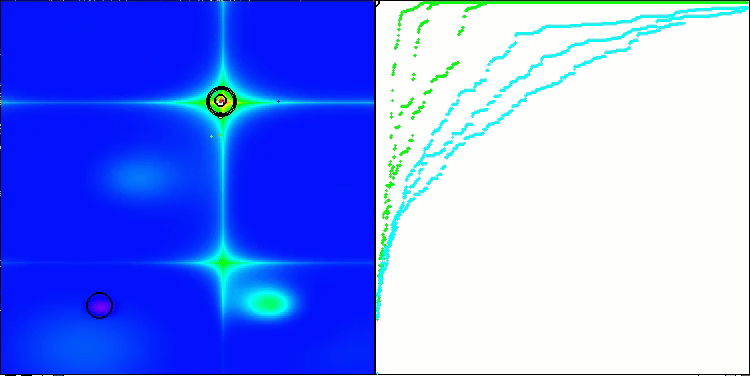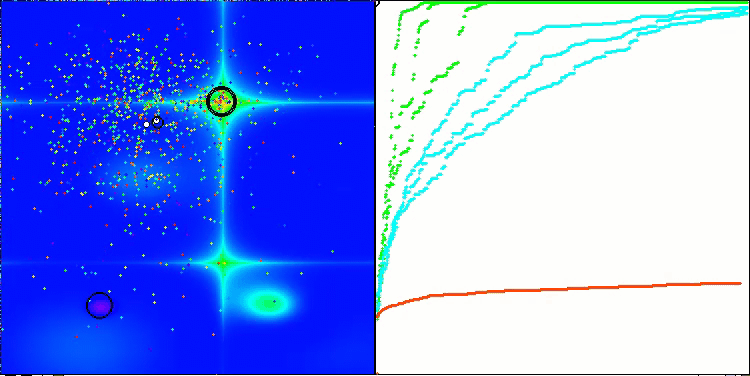
Forestテスト関数のACSm2
Megacityテスト関数のACSm2
まとめ
全体的な結論は次のとおりです。
1. ACSアルゴリズムの最初の修正では、A行列とB行列の拡張形式が追加され、特に1000変数のHilly関数と50および1000変数のForest関数でソリューションの精度が向上しました。
2. 2番目の変更では、Predator行列とPrey行列を埋める方法を変更し、ランダムな比較を使用して行列を更新しました。この結果、50および1000変数を持つすべてのテスト関数で顕著に改善されましたが、10変数のMegacity関数では結果のばらつきが大きくなりました。
3.3番目の修正では、適応度を使用して捕食者と被食者の集団を形成することに重点を置きましたが、期待に応えられませんでした。少数の変数を持つ滑らかな関数の収束精度は向上したものの、全体的な結果はアルゴリズムの基本バージョンよりもやや劣っていました。
このように、各修正にはそれぞれ長所と短所があり、アルゴリズムをさらに開発するには、これらの特徴を考慮して、ACSアルゴリズムのより効率的で汎用的なバージョンを作成する必要があります。
改善の可能性がある領域
1. 検索メカニズムの変更
- 現在の検索メカニズムを分析し、さらに最適化できる可能性を検討します。
- 検索効率を向上させるために、適応型検索ステップなどの追加メカニズムを導入できるかどうかを検討します。
2. ハイブリッドアプローチの検討
- さまざまなアプローチの長所を活用するために、現在のアルゴリズムを他の最適化方法と組み合わせることを検討します。たとえば、進化アルゴリズムや群知能手法の要素を統合して、多様性と収束速度を向上させることができます。
3.集団間の相互作用のメカニズムの分析と改善
- A集団とB集団の相互作用方法を改善し、検索中に両者がより効果的に補完し合う方法を見つけます。集団間の情報共有や協調学習のより複雑な構造を検討する価値があるかもしれません。
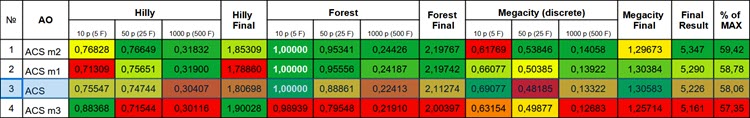
図1:基本バージョンとの比較のための、関連するテストに応じたアルゴリズムの変更のカラーグラデーション(0.99以上の結果は白で強調表示)
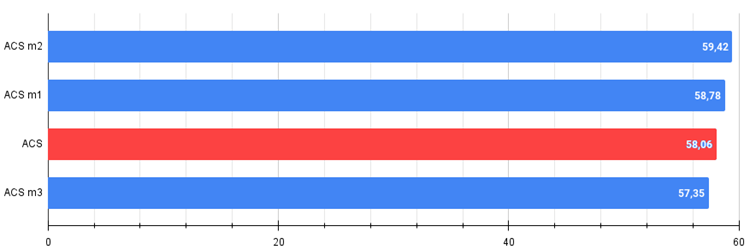
図2:アルゴリズム変更結果のヒストグラム(0~100のスケールで、数値が大きいほど良い)
ここで、100は理論的に可能な最大の結果であり、アーカイブにはレーティング表を計算するスクリプトが含まれている)
修正されたACSアルゴリズムバージョンの一般的な長所と短所
長所
- 外部パラメータの数が少ない(1つだけ)。
- 様々な種類の関数に対して収束性が良い。
短所
- 低次元関数に関する結果が散らばる。
この記事には、最新版のアルゴリズムコードを含むアーカイブが添付されています。記事の著者は、正規アルゴリズムの説明の絶対的な正確さについて責任を負いません。検索機能を向上させるために、それらの多くに変更が加えられています。記事に示された結論と判断は、実験結果に基づいています。
MetaQuotes Ltdによってロシア語から翻訳されました。
元の記事: https://www.mql5.com/ru/articles/15014
警告: これらの資料についてのすべての権利はMetaQuotes Ltd.が保有しています。これらの資料の全部または一部の複製や再プリントは禁じられています。
この記事はサイトのユーザーによって執筆されたものであり、著者の個人的な見解を反映しています。MetaQuotes Ltdは、提示された情報の正確性や、記載されているソリューション、戦略、または推奨事項の使用によって生じたいかなる結果についても責任を負いません。
 MQL5における相関分析の要素:ピアソンのカイ二乗検定による独立性と相関比
MQL5における相関分析の要素:ピアソンのカイ二乗検定による独立性と相関比
 ニューラルネットワークが簡単に(第92回):周波数および時間領域における適応的予測
ニューラルネットワークが簡単に(第92回):周波数および時間領域における適応的予測
 取引履歴を気にせずにチャート上で直接取引を表示する方法
取引履歴を気にせずにチャート上で直接取引を表示する方法
 リプレイシステムの開発(第51回):物事は複雑になる(III)
リプレイシステムの開発(第51回):物事は複雑になる(III)
- 無料取引アプリ
- 8千を超えるシグナルをコピー
- 金融ニュースで金融マーケットを探索