
化学反应优化(CRO)算法(第一部分):在优化中处理化学
目录
1.概述
化学反应优化(CRO)是一种令人兴奋的创新方法,其灵感来自化学转化的本质。想象一下,观看分子的舞蹈,其中每个运动和碰撞都在解决复杂问题中发挥着关键作用。这种方法巧妙地将分子的能量守恒、分解和合成原理编织在一起,创造了一种灵活自适应的优化方法。
CRO是一种将化学反应与优化松散耦合的方法,使用由热力学前两定律定义的分子相互作用的一般原理。化学反应优化(CRO)算法由 Lam 和 Li 于 2012 年提出并发表。
热力学第一定律(能量守恒定律)指出,能量不能被创造或毁灭。它可以从一种形式转换为另一种形式,并从一个实体传递到另一个实体。在 CRO 的背景下,化学反应系统由物质和环境组成,每个粒子都有势能和动能。
热力学第二定律指出,系统的熵趋于增加,其中熵是无序的度量,系统渴望更多的自由。势能是相对于分子构型储存在分子中的能量。随着分子的势能被释放并转化为动能,系统变得越来越混乱。但正是在这种混乱中,CRO 找到了自己的力量,捕捉并引导能量流向最优解。
例如,当具有较高动能(从势能转换而来)的分子移动得更快时,系统会变得更加无序,熵也会增加。因此,所有反应系统都倾向于达到平衡状态,在这种状态下,势能降至最低。在 CRO 中,我们通过将势能转化为动能并逐渐失去环境中化学分子的能量来捕捉这一现象。
一个正在进行化学反应的化学系统是不稳定的,它有多余的能量,试图摆脱它并稳定下来。该系统由分子组成,分子是化合物的最小颗粒,根据其基本化学性质分为不同类型。
化学反应形成了一个复杂的舞蹈,分子碰撞、形成和断裂键,改变了它们的结构。这种舞蹈的每一步都是一系列子动作,以最小的能量产生更稳定的产品。分子将能量储存在其化学键中,即使其结构的微小变化也会导致惊人的转变,从而生成更稳定的产物。分子结构会影响化合物的化学行为,即使结构的微小变化也会导致化学性质的显著差异。
化学反应是由分子碰撞引发的,分子可以是单分子(与外部物质)或双分子(与其他分子)。这些碰撞可能是高效的,也可能是低效的,这取决于活化能和空间效应等因素。就这样,分子舞动、交织和分离,创造出新的形状和结构。
值得注意的是,化学反应通常会导致形成吉布斯能较低的更稳定的产物。该过程通过顺序和/或平行步骤发生,涉及中间化合物的形成和过渡态的通过。了解这些过程对于找到化学反应的最佳条件和阐明化合物转化的机制非常重要。
此外,化学合成可能是反应物有效和无效碰撞的结果。碰撞的效率取决于活化能、空间效应等因素。与分子内合成相比,分子间合成会导致分子结构发生更显著的变化。
CRO算法类似于一位技艺精湛的编舞家,他使用化学定律(如音符)来创建极其复杂和优雅的优化解决方案。现在让我们逐一讨论一切 — 从复杂到简单的概念。
2.化学操作符的实现
CRO 的基本单位是“分子”。在一个种群中,每个生物体都具有一定的特征,比如“势能和动能”、“分子结构”等。基本反应定义了分子之间的相互作用。我们可以定义一个类,其中数据字段表示分子的特征,方法描述基本反应。
CRO 的主要概念之一是节能。该原理确保系统中的总能量在整个优化过程中保持恒定。能量守恒决定了反应过程中分子不同状态之间的转变,保持了影响寻找最佳解决方案的能量平衡。
使用可操纵的试剂(分子)、基本反应和能量守恒的概念,CRO 为解决复杂的优化问题提供了一种灵活和自适应的方法。定制操作符、动态改变种群规模以及整合不同属性的能力使 CRO 成为元启发式领域一种很有前途的方法。其独特的功能和灵活性使研究人员能够探索优化的新机会。
CRO 算法使用以下运算符:
1.分子间无效碰撞。该运算符模拟了两个分子碰撞但保持完整,其结构略有变化的过程。这允许算法在当前解附近执行局部搜索。
2.分解。该运算符模拟了分子与壁碰撞并分裂成两个新分子的过程。这允许算法探索解空间的新区域。
3.分子内反应。该运算符模拟了分子与壁碰撞并保持完整,但其结构略有变化的过程。这允许算法在当前解决方案附近执行局部搜索。
4.合成。该运算符模拟了两个分子碰撞并结合形成一个新分子的过程。这允许算法结合好的解决方案来创建可能更好的解决方案。
这些运算符中的每一个都在 CRO 算法中起着重要作用,使其能够探索搜索空间并找到最优解。它们的合作在探索(搜索搜索空间的新领域)和利用(改进当前最佳解决方案)之间提供了平衡。
让我们探讨算法中的每个单独的化学运算符(搜索全局最小值):
算法#1:单分子碰撞
1.输入数据:Mω
2.产生分子的新位置: ω = N(ω)
3.新位置的势能计算:PEω = f(ω)
4.增加碰撞计数器:NumHitω = NumHitω + 1
5.如果新位置的势能 + 动能 ≥ 当前位置的势能,则:
6.生成 [KELossRate, 1] 范围内的随机数“a”
7.动能更新:KEω = (PEω − PEω + KEω) × a
8.缓冲区更新: buffer = buffer + (PEω − PEω + KEω) × (1 − a)
9.保持当前位置和能量
10.如果新位置的势能 < 最小势能,则更新最小值
11.条件结束
算法#2:分解
1.输入数据:Mω
2.创建两个新分子 Mω1 和 Mω2
3.从 ω 获得新分子的位置:ω1 和 ω2
4.计算新分子的势能:PEω1 = f(ω1) 且 PEω2 = f(ω2)
5.如果当前位置的势能 + 动能 ≥ 新位置的总势能,则:
6.分解能量的计算:Edec = PEω + KEω − (PEω1 + PEω2)
7.继续执行步骤 13
8.否则:
9.在 [0,1] 范围内生成随机数 δ1、δ2
10.分解能量的计算:Edec = PEω + KEω + δ1δ2 × buffer − (PEω1 + PEω2)
11.如果 Edec ≥ 0,则:
12.缓冲区更新
13.在 [0,1] 范围内生成随机数 δ3
14.新分子之间的动能分布
15.保持每个新分子的最小值
16.销毁当前分子
17.否则:
18.增加碰撞计数器
19.销毁新分子
20.条件結束
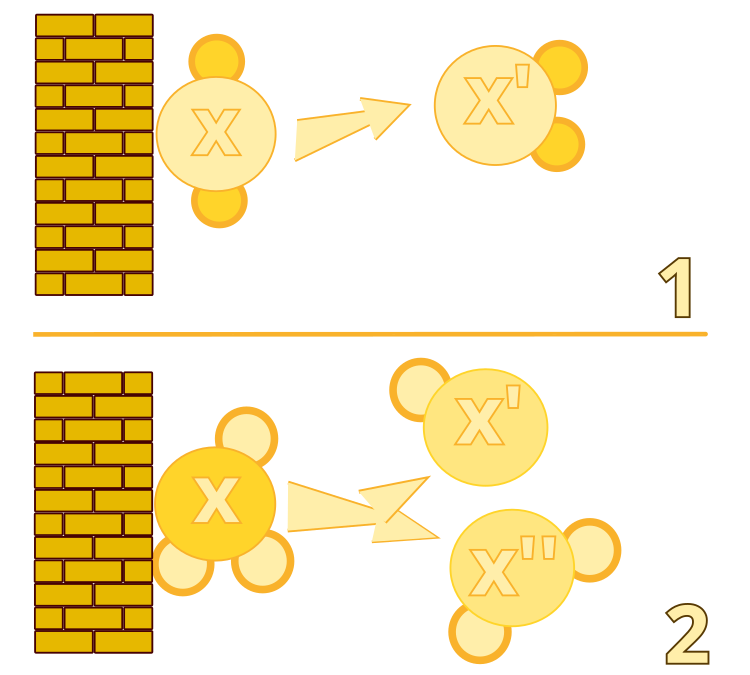
图 1.无效的墙壁碰撞:算法#1。分解:算法 #2
算法 #3:分子间无效碰撞
1.输入数据:Mω1 和 Mω2
2.为分子生成新位置:ω1 = N(ω1) 和 ω2 = N(ω2)
3.新位置势能计算:PEω1 = f(ω1) 且 PEω2 = f(ω2)
4.增加碰撞计数器:NumHitω1 = NumHitω1 + 1 且 NumHitω2 = NumHitω2 + 1
5.分子间相互作用能量的计算:Einter = (PEω1 + PEω2 + KEω1 + KEω2) − (PEω1 + PEω2)
6.如果 Einter ≥ 0,则:
7.在 [0,1] 范围内生成随机数 δ4
8.分子间的动能分布:KEω1 = Einter × δ4 且 KEω2 = Einter × (1 − δ4)
9.更新分子位置和能量
10.如果新位置的势能小于最小势能,则更新每个分子的最小值
11.条件結束
算法#4:合成
1.输入数据:Mω1 和 Mω2
2.创建一个新的 Mω
3.获取新分子的位置:ω 来自 ω1 和 ω2
4.计算新分子的势能:PEω = f(ω)
5.如果新分子的总势能和动能大于或等于原始分子的总动能和势能,则:
6.将过剩动能分配给新分子:KEω = (PEω1 + PEω2 + KEω1 + KEω2) − PEω
7.更新新分子的最小值
8.销毁原始分子
9.否则:
10.增加源分子的碰撞计数器
11.销毁新分子
12.条件結束
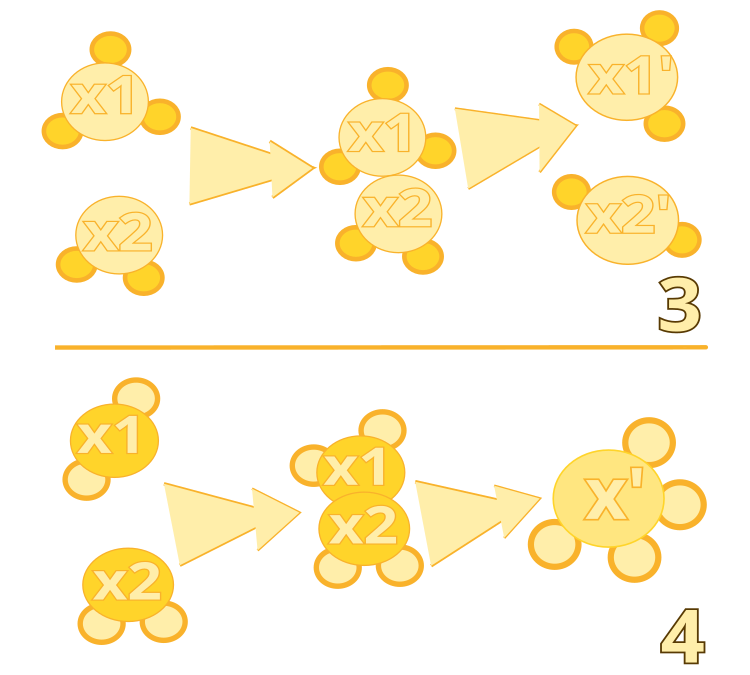
图 2.无效的分子间碰撞:算法#3。合成:算法#4
对 CRO 算法提出的描述反映了作者对这种方法的看法。然而,它没有考虑到一些可能会显著影响算法性能和搜索能力的重要点。
该算法涉及封闭空间中的能量守恒以及从一种能量到另一种能量的转换。然而,作者并没有揭示数值能量指标与目标(适应度)函数值之间的对应关系。很明显,作者将适应度的作用定义为势能,动能作为补偿势能减少的机制(理想情况下,所有能量的总和应该是常数)。
例如,如果优化问题的标准是利润因子,那么与使用余额作为标准的问题相比,目标函数的值将在很小的范围内波动。在这种情况下,我们看到目标函数的值将根据特定的优化标准而变化,但作者使用常数作为算法的外部参数,不能与算法中计算的能量进行比较。
因此,原始的 CRO 算法(未经修改)可以应用的任务列表非常有限,因此它不是通用的。在本系列文章中,我们只考虑适用于一般优化问题的通用算法,如果任何算法不允许这样做,我们通常会对其进行修改并将其转化为单一的通用形式。
第二点是,原始算法在逻辑的不同部分以无序的方式提供适应度函数的计算,这在实际任务中使用和集成到项目中时不可避免地会导致问题。我们也会修复此问题。为此,我们需要将化学运算符分为两部分:运算符本身和所谓的“后运算符”,第一部分修改分子的位置,第二部分在计算分子的适应度后对分子执行必要的操作。
第三点是,原始算法提供了分子的动态种群大小。新分子出现,一些旧分子被破坏。没有调节群体规模的机制,群体规模可以在非常大的范围内变化。因此,实验表明,在一定的外部参数设置下,种群可以从最初的 50 个分子增长到 1000 多个。我们还通过执行运算符、使用简单的计数器并将父分子的索引存储在父总体中,顺序地用分子填充总体来解决这个问题。这使我们能够保持种群大小恒定,同时消除了执行去除分子操作的需要 — 当条件满足时,子分子只需替换相应的母分子即可。
这些变化使CRO算法成为一种适合解决一般优化问题的形式,便于将算法集成到项目中,同时保留了化学反应的一般原始概念。我们已经详细描述了CRO算法。如果您愿意,您可以尝试在算法中实现势能和动能的核算。
让我们继续讨论代码。描述列表中的反应类型,以便我们在反应和分子结构之后选择合适的后运算符。
E_ReactionType 枚举:
1. synthesis 是两个分子结合形成一个新分子的过程。
2. interMolecularInefColl 是分子间无效碰撞,其中分子发生碰撞但不发生反应。
3. decomposition 是将复杂分子分解成更简单成分的过程。
4. inefCollision 是与壁面的低效碰撞,会改变分子的结构。
它定义了 S_CRO_Agent 结构,该结构表示算法环境中的分子模型。让我们来看看下面的结构包含哪些字段:
- structure[] - 表示分子结构的数组。
- NumHit -“命中”次数或分子相互作用的计数器。
- indMolecule_1 和 indMolecule_2 - 相互作用分子的索引。
- KE - 表示分子动能的变量。
- f - 适应度函数(分子适应度)。
- rType - E_ReactionType 类型变量,分子参与的反应类型。
“Init”- 初始化字段的结构方法。它采用 coords 整数参数,使用 ArrayResize 函数来调整 structure 数组的大小。NumHit、indMolecule_1、indMolecule_2、f 和 KE 使用零或 -DBL_MAX 初始化。
该代码代表 CRO 算法中分子的基本数据结构,并在创建新分子时初始化其字段。
enum E_ReactionType { synthesis, interMolecularInefColl, decomposition, inefCollision }; // Molecule structure struct S_CRO_Agent { double structure []; int NumHit; int indMolecule_1; int indMolecule_2; double KE; double f; E_ReactionType rType; // Initialization method void Init (int coords) { ArrayResize (structure, coords); NumHit = 0; indMolecule_1 = 0; indMolecule_2 = 0; f = -DBL_MAX; KE = -DBL_MAX; } };
C_AO_CRO 类的 InefCollision 方法是一种无效碰撞,它涉及通过一个父分子的位移来创建一个新分子。此方法中发生的情况如下:
1.该方法接受父分子的 index 和与 molCNT 分子计数器的链接。如果 molCNT 超过或等于 popSize 种群规模,该方法则返回 false 并完成其工作。
2.该方法确定了碰撞后产生的新分子 index1_ 的索引。
3.母分子的结构被复制到新分子的结构中。
4.接下来,对新分子的每个坐标执行 N 函数,该函数在旧坐标值附近生成新的坐标值。
5.使用 SeInDiSp 函数调整新的坐标值,以便使它们保持在从 rangeMin 到 rangeMax 的指定范围内。
6.然后设置新分子的 indMolecule_1、rType 和 NumHit 字段值。indMolecule_1 存储父分子的索引,rType 设置为 inefCollision,而 NumHit 被重置。
7.在方法结束时,molCNT 分子计数器增加 1,并且方法返回 true。
//—————————————————————————————————————————————————————————————————————————————— // Ineffective collision. Obtaining a new molecule by displacing a parent one. bool C_AO_CRO::InefCollision (int index, int &molCNT) { if (molCNT >= popSize) return false; int index1_ = molCNT; ArrayCopy (Mfilial [index1_].structure, Mparent [index].structure); for (int c = 0; c < coords; c++) { N (Mfilial [index1_].structure [c], c); Mfilial [index1_].structure [c] = u.SeInDiSp (Mfilial [index1_].structure [c], rangeMin [c], rangeMax [c], rangeStep [c]); } Mfilial [index1_].indMolecule_1 = index; // save the parent molecule index Mfilial [index1_].rType = inefCollision; Mfilial [index1_].NumHit = 0; molCNT++; return true; } //——————————————————————————————————————————————————————————————————————————————
C_AO_CRO 类中的 PostInefCollision 方法用于处理化学反应模拟中 mol 分子与其他分子发生无效碰撞的结果。该方法执行以下操作:
1.声明 ind 变量并使用来自 mol 对象中的 indMolecule_1 值对其进行初始化。
2.条件表达式检查 mol 对象的 f 值是否超过具有 ind 索引的父分子的 f 值。
3.如果条件为 true,则将 mol 结构复制到具有 ind 索引的父分子结构中。
4.使用 mol中的 f 值更新父分子的 f 值。
5.父分子的 NumHit 计数器被重置为零。
6.如果 mol.f > Mparent [ind].f 条件为 false,则父分子的 NumHit 计数器增加 1。
一般来说,该方法根据无效碰撞的结果来更新父分子的结构和适应度函数值。如果新的 mol 结构导致适应度提高,它将取代父分子的结构并且 NumHit 计数器被重置。否则,父分子的 NumHit 计数器会增加。
//—————————————————————————————————————————————————————————————————————————————— // Handling the results of an ineffective collision. void C_AO_CRO::PostInefCollision (S_CRO_Agent &mol) { int ind = mol.indMolecule_1; if (mol.f > Mparent [ind].f) { ArrayCopy (Mparent [ind].structure, mol.structure); Mparent [ind].f = mol.f; Mparent [ind].NumHit = 0; } else { Mparent [ind].NumHit++; } } //——————————————————————————————————————————————————————————————————————————————
C_AO_CRO 类的 Decomposition 方法是一个分解过程,通过分解一个父分子来创建两个新分子。此方法中发生的情况如下:
1.该方法接受 index 父分子和到 molCNT 分子计数器的链接。如果 molCNT 超过或等于 popSize - 1,该方法将返回 false 并完成其工作。
2.然后,该方法确定两个新分子的索引 - index1_ 和 index2_,它们将是分解的结果。
3.父分子的结构被复制到新分子的结构中。
4.接下来,对新分子的每个坐标执行 N 函数。该函数在旧坐标值附近生成新的坐标值。这分别针对第一个子分子的 index1_ 坐标的前半部分和第二个分子的 index2_ 坐标的后半部分分别进行。
5.使用 SeInDiSp 函数调整新的坐标值,以便使它们保持在从 rangeMin 到 rangeMax 的指定范围内。
6.然后设置新分子的 indMolecule_1、indMolecule_2、rType 和 NumHit 字段值。indMolecule_1 和 indMolecule_2 分别保存父分子和姊妹分子的索引,rType 设置为 分解,而 NumHit 设置为零。
7.在方法结束时,molCNT 分子计数器增加 2,并且方法返回 true。
//—————————————————————————————————————————————————————————————————————————————— // Decomposition. Obtaining two new molecules by decomposing a parent one. bool C_AO_CRO::Decomposition (int index, int &molCNT) { if (molCNT >= popSize - 1) return false; // Creating two new molecules M_ω'_1 and M_ω'_2 from M_ω int index1_ = molCNT; int index2_ = molCNT + 1; ArrayCopy (Mfilial [index1_].structure, Mparent [index].structure); ArrayCopy (Mfilial [index2_].structure, Mparent [index].structure); for (int c = 0; c < coords / 2; c++) { N (Mfilial [index1_].structure [c], c); Mfilial [index1_].structure [c] = u.SeInDiSp (Mfilial [index1_].structure [c], rangeMin [c], rangeMax [c], rangeStep [c]); } for (int c = coords / 2; c < coords; c++) { N (Mfilial [index2_].structure [c], c); Mfilial [index2_].structure [c] = u.SeInDiSp (Mfilial [index2_].structure [c], rangeMin [c], rangeMax [c], rangeStep [c]); } Mfilial [index1_].indMolecule_1 = index; // save the parent molecule index Mfilial [index1_].indMolecule_2 = index2_; // save the index of the second daughter molecule Mfilial [index1_].rType = decomposition; Mfilial [index1_].NumHit = 0; Mfilial [index2_].indMolecule_1 = index1_; // save the index of the first daughter molecule Mfilial [index2_].indMolecule_2 = -1; // mark the molecule so we do not handle it twice Mfilial [index2_].rType = decomposition; Mfilial [index2_].NumHit = 0; molCNT += 2; return true; } //——————————————————————————————————————————————————————————————————————————————
C_AO_CRO 类的 PostDecomposition 方法处理分子分解的结果。该方法执行以下操作:
1.该方法接受对分解结果获得的 mol 分子的引用。如果分子的 indMolecule_2 等于“-1”(意味着该分子已被处理),则该方法完成其工作。
2.然后将父分子的 ind 索引与两个“子”分子 index1_ 和 index2_ 的索引一起提取。
3.接下来检查第一个“子”分子的 f 适应度函数值是否超过第二个“子”分子和父分子的 f 函数值。如果确实如此,那么第一个“子”分子就会取代父分子。被替换的分子的 NumHit 值被重置并且 flag 被设置。
4.如果 flag 仍然为 false,则对第二个“子”分子进行类似的检查。
5.如果经过所有检查后, flag 仍然为 false,则父分子的 NumHit 增加 1。
PostDecomposition 方法负责在 CRO 算法中分解后更新母分子的状态。
//—————————————————————————————————————————————————————————————————————————————— // Handling decomposition results. void C_AO_CRO::PostDecomposition (S_CRO_Agent &mol) { if (mol.indMolecule_2 == -1) return; int ind = mol.indMolecule_1; int index2_ = mol.indMolecule_2; int index1_ = Mfilial [index2_].indMolecule_1; bool flag = false; if (Mfilial [index1_].f > Mfilial [index2_].f && Mfilial [index1_].f > Mparent [ind].f) { ArrayCopy (Mparent [ind].structure, Mfilial [index1_].structure); Mparent [ind].f = Mfilial [index1_].f; Mparent [ind].NumHit = 0; flag = true; } if (!flag) { if (Mfilial [index2_].f > Mfilial [index1_].f && Mfilial [index2_].f > Mparent [ind].f) { ArrayCopy (Mparent [ind].structure, Mfilial [index2_].structure); Mparent [ind].f = Mfilial [index2_].f; Mparent [ind].NumHit = 0; flag = true; } } if (!flag) { Mparent [ind].NumHit++; } } //——————————————————————————————————————————————————————————————————————————————
C_AO_CRO 类的 InterMolInefColl 方法是一种无效碰撞,它涉及通过改变两个父分子来创建两个新分子。此方法中发生的情况如下:
1.该方法接受两个父分子的索引 -index1 和 index2 以及到 molCNT 分子计数器的链接。如果 molCNT 超过或等于 popSize - 1,该方法将返回 false 并完成其工作。
2.然后,该方法确定碰撞后产生的两个新子分子 index1_ 和 index2_ 的索引。
3.父分子的结构被复制到新分子的结构中。
4.接下来,对新分子的每个坐标执行 N 函数。该函数在旧坐标值附近生成新的坐标值。
5.然后使用 SeInDiSp 函数调整新的坐标值,以便它们保持在从 rangeMin 到 rangeMax 的指定范围内。
6.然后设置新分子的 indMolecule_1、indMolecule_2、rType 和 NumHit 字段值 。indMolecule_1 和 indMolecule_2 保留母分子的索引,rType 设置为 interMolecularInefColl,而 NumHit 设置为零。
7.在方法结束时,molCNT 分子计数器增加 2,并且方法返回 true。
//—————————————————————————————————————————————————————————————————————————————— // Intermolecular ineffective collision. Obtaining two new molecules by changing two parent ones bool C_AO_CRO::InterMolInefColl (int index1, int index2, int &molCNT) { if (molCNT >= popSize - 1) return false; int index1_ = molCNT; int index2_ = molCNT + 1; // Obtaining molecules ArrayCopy (Mfilial [index1_].structure, Mparent [index1].structure); ArrayCopy (Mfilial [index2_].structure, Mparent [index2].structure); // Generating new molecules ω'_1 = N(ω1) and ω'_2 = N(ω2) in the vicinity of ω1 and ω2 for (int c = 0; c < coords; c++) { N (Mfilial [index1_].structure [c], c); N (Mfilial [index2_].structure [c], c); } for (int c = 0; c < coords; c++) { Mfilial [index1_].structure [c] = u.SeInDiSp (Mfilial [index1_].structure [c], rangeMin [c], rangeMax [c], rangeStep [c]); Mfilial [index2_].structure [c] = u.SeInDiSp (Mfilial [index2_].structure [c], rangeMin [c], rangeMax [c], rangeStep [c]); } Mfilial [index1_].indMolecule_1 = index1; // save the index of the first parent molecule Mfilial [index1_].indMolecule_2 = index2_; // save the index of the second daughter molecule Mfilial [index1_].rType = interMolecularInefColl; Mfilial [index1_].NumHit = 0; Mfilial [index2_].indMolecule_1 = index2; // save the index of the second parent molecule Mfilial [index2_].indMolecule_2 = -1; // mark the molecule so we do not handle it twice Mfilial [index2_].rType = interMolecularInefColl; Mfilial [index2_].NumHit = 0; molCNT += 2; return true; } //——————————————————————————————————————————————————————————————————————————————
C_AO_CRO 类的 PostInterMolInefColl 方法处理分子间无效碰撞的结果。该方法具有以下目的:
1.该方法接受对碰撞获得的 mol 分子的引用。如果分子的 indMolecule_2 等于“-1”(意味着该分子已被处理),则该方法完成其工作。
2.然后提取两个父分子的 ind1 和 ind2 索引。
3.接下来,检查新分子及其“姐妹”的适应度值的 f 总和是否超过两个父分子的适应度值的 f 总和。如果事实确实如此,新分子就会取代父分子。被替换的分子的 NumHit 值被重置。
4.否则,父分子的 NumHit 值增加 1。
PostInterMolInefColl 方法负责在CRO算法中分子间低效碰撞后更新父分子的状态。
//—————————————————————————————————————————————————————————————————————————————— // Handling the results of an intermolecular ineffective collision. void C_AO_CRO::PostInterMolInefColl (S_CRO_Agent &mol) { if (mol.indMolecule_2 == -1) return; int ind1 = mol.indMolecule_1; int ind2 = Mfilial [mol.indMolecule_2].indMolecule_1; Mparent [ind1].NumHit++; Mparent [ind2].NumHit++; if (mol.f + Mfilial [mol.indMolecule_2].f > Mparent [ind1].f + Mparent [ind2].f) { ArrayCopy (Mparent [ind1].structure, mol.structure); Mparent [ind1].f = mol.f; ArrayCopy (Mparent [ind2].structure, Mfilial [mol.indMolecule_2].structure); Mparent [ind2].f = Mfilial [mol.indMolecule_2].f; } } //——————————————————————————————————————————————————————————————————————————————
算法中的最后一个化学反应运算符 -C_AO_CRO 类的 合成 方法 - 是一个通过融合两个父分子来创建新分子的合成过程。
1.该方法接受两个父分子的索引 -index1 和 index2 以及到 molCNT 分子计数器的链接。如果 molCNT 超过或等于 popSize 种群规模,该方法则返回 false 并完成其工作。
2.在循环中,对于新分子的每个坐标,执行以下操作:如果随机数小于 0.5,则该坐标接收第一个父分子 Mparent[index1].structure[i] 的相应坐标的值。否则,它将接收第二个父分子 Mparent[index2].structure[i] 的相应坐标的值。
3.然后设置新分子的 indMolecule_1、indMolecule_2、rType 和 NumHit 字段值。indMolecule_1 和 indMolecule_2 保留母分子的索引,rType 设置为 synthesis,而 NumHit 设置为零。
4.在方法结束时,molCNT 分子计数器增加,并且方法返回 true。
//—————————————————————————————————————————————————————————————————————————————— // Synthesis. Obtaining a new molecule by fusing two parent ones bool C_AO_CRO::Synthesis (int index1, int index2, int &molCNT) { if (molCNT >= popSize) return false; // Create a new M_ω' molecule from M_ω1 and M_ω2 for (int i = 0; i < coords; i++) { if (u.RNDprobab () < 0.5) Mfilial [molCNT].structure [i] = Mparent [index1].structure [i]; else Mfilial [molCNT].structure [i] = Mparent [index2].structure [i]; } Mfilial [molCNT].indMolecule_1 = index1; // save the index of the first parent molecule Mfilial [molCNT].indMolecule_2 = index2; // save the index of the second parent molecule Mfilial [molCNT].rType = synthesis; Mfilial [molCNT].NumHit = 0; molCNT++; return true; } //——————————————————————————————————————————————————————————————————————————————
C_AO_CRO 类的 PostSynthesis 方法处理合成结果。以下是其中发生的事情:
1.该方法接受对合成结果所获得的 mol 分子的引用。然后从中提取两个父分子的 ind1 和 ind2 索引。
2.接下来,检查新分子的 f 适应度值是否超过两个父分子的 f 适应度值。如果是这种情况,那么新分子将取代 f 值较小的父分子。被替换的分子的 NumHit 值设置为零。
3.否则,父分子的 NumHit 值增加 1。
PostSynthesis 方法负责在 CRO 算法中合成过程后更新父分子的状态。
//—————————————————————————————————————————————————————————————————————————————— // Handling synthesis results. void C_AO_CRO::PostSynthesis (S_CRO_Agent &mol) { int ind1 = mol.indMolecule_1; int ind2 = mol.indMolecule_2; if (mol.f > Mparent [ind1].f && mol.f > Mparent [ind2].f) { if (Mparent [ind1].f < Mparent [ind2].f) { ArrayCopy (Mparent [ind1].structure, mol.structure); Mparent [ind1].f = mol.f; Mparent [ind1].NumHit = 0; } else { ArrayCopy (Mparent [ind2].structure, mol.structure); Mparent [ind2].f = mol.f; Mparent [ind2].NumHit = 0; } } else { Mparent [ind1].NumHit++; Mparent [ind2].NumHit++; } } //——————————————————————————————————————————————————————————————————————————————
最后,我们来描述一下 C_AO_CRO 类中用于改变化学反应运算符中的分子结构的 N 方法。该方法用于为给定范围内的分子坐标生成新值:
1.该方法接受需要修改为参考的 coord 分子坐标,以及结构中的 coordPos 坐标位置。
2.接下来计算 dist 距离,它是范围的最大值和最小值之间的差值,乘以 molecPerturb 参数,该参数表示当前坐标值附近的值的扩展。
3.然后定义新坐标的最小值(min)和最大值(max)。这些值等于旧坐标值加上或减去 dist 距离,但它们不能超出从 rangeMin[coordPos] 到 rangeMax[coordPos] 的指定范围。
4.最后,使用 u.GaussDistribution 高斯分布函数生成新的坐标值,该函数取坐标的旧值、标准差的最小值和最大值,等于 8。
//—————————————————————————————————————————————————————————————————————————————— void C_AO_CRO::N (double &coord, int coordPos) { double dist = (rangeMax [coordPos] - rangeMin [coordPos]) * molecPerturb; double min = coord - dist; if (min < rangeMin [coordPos]) min = rangeMin [coordPos]; double max = coord + dist; if (max > rangeMax [coordPos]) max = rangeMax [coordPos]; coord = u.GaussDistribution (coord, min, max, 8); } //——————————————————————————————————————————————————————————————————————————————
我们对所有化学运算符的功能及其在优化过程中的作用进行了分析。该分析为我们提供了对化学反应模型(CRO)中定义分子动力学的机制的深入了解。
在下一篇文章中,我们将构建该算法并在测试函数上对其进行测试。我们将配置所研究的运算符并将其组合成一个完整的 CRO 算法,以便在实际任务中使用。然后,我们将对各种测试函数进行一系列实验,以评估我们方法的效率和鲁棒性。
在收到工作结果后,我们将得出关于算法优缺点的结论,以及进一步改进的可能方向。这将使我们不仅可以改进我们的方法,还可以为化学过程优化和建模领域的研究人员提供有用的建议。
因此,下一篇文章将是我们研究的关键一步,因为我们将从理论转向实践,测试和改进我们的算法,以在解决复杂优化问题时达到最佳效果。
本文由MetaQuotes Ltd译自俄文
原文地址: https://www.mql5.com/ru/articles/15041
注意: MetaQuotes Ltd.将保留所有关于这些材料的权利。全部或部分复制或者转载这些材料将被禁止。
本文由网站的一位用户撰写,反映了他们的个人观点。MetaQuotes Ltd 不对所提供信息的准确性负责,也不对因使用所述解决方案、策略或建议而产生的任何后果负责。
 您应当知道的 MQL5 向导技术(第 17 部分):多币种交易
您应当知道的 MQL5 向导技术(第 17 部分):多币种交易
 人工电场算法(AEFA)
人工电场算法(AEFA)
 MQL5 向导技巧须知(第27部分):移动平均线与攻击角度
MQL5 向导技巧须知(第27部分):移动平均线与攻击角度

这里是 关于 在 交易中的实际应用的文章 。 阅读、 学习 和 在 实践中应用 。 我祝愿您 在 交易中取得成功。
文章
使用优化算法即时配置 EA 参数
Andrey Dik, 2024.06.07 14:30
这篇文章讨论了使用优化算法即时找到最佳 EA 参数的实际问题,以及交易操作和 EA 逻辑的虚拟化。这篇文章可作为在 EA 中实施优化算法的指导。以下是 关于 在 交易中的实际应用的文章 。 阅读、 学习 和 在 实践中应用 。 我祝愿您 在 交易中取得成功。
我喜欢你的观点,安德烈!伟大的创新源于研究。这并不总是钱的问题,我很欣赏你对社区的贡献。