
Algorithmus einer chemischen Reaktionsoptimierung (CRO) (Teil I): Prozesschemie in der Optimierung
Inhalt
1. Einführung
Chemische Reaktionsoptimierung (Chemical reaction optimization, CRO) ist eine aufregende und innovative Methode, die sich am Wesen chemischer Umwandlungen orientiert. Stellen Sie sich vor, Sie beobachten einen Tanz der Moleküle, bei dem jede Bewegung und jeder Zusammenstoß eine Schlüsselrolle bei der Lösung komplexer Probleme spielt. Bei dieser Methode werden die Grundsätze der Energieerhaltung, der Zerlegung und der Synthese von Molekülen geschickt miteinander verknüpft, sodass ein flexibler und anpassungsfähiger Ansatz zur Optimierung entsteht.
CRO ist eine Methode, die chemische Reaktionen locker mit der Optimierung koppelt und allgemeine Prinzipien molekularer Wechselwirkungen verwendet, die durch die ersten beiden Gesetze der Thermodynamik definiert sind. Der Algorithmus Chemical Reaction Optimization (CRO) wurde von Lam und Li im Jahr 2012 vorgeschlagen und veröffentlicht.
Der erste Hauptsatz der Thermodynamik (der Energieerhaltungssatz) besagt, dass Energie weder erzeugt noch zerstört werden kann. Es kann von einer Form in eine andere umgewandelt und von einer Einheit an eine andere weitergegeben werden. Im Rahmen von CRO besteht ein chemisches Reaktionssystem aus Stoffen und der Umgebung, und jedes Teilchen hat potenzielle und kinetische Energie.
Der zweite Hauptsatz der Thermodynamik besagt, dass die Entropie eines Systems tendenziell zunimmt, wobei die Entropie ein Maß für die Unordnung ist und das System nach mehr Freiheit strebt. Die potenzielle Energie ist die in einem Molekül gespeicherte Energie im Verhältnis zu seiner molekularen Konfiguration. Da die potenzielle Energie der Moleküle freigesetzt und in kinetische Energie umgewandelt wird, wird das System zunehmend chaotisch. Aber gerade in diesem Chaos findet CRO seine Stärke, indem es die Energieströme einfängt und auf die optimale Lösung lenkt.
Wenn sich beispielsweise Moleküle mit höherer kinetischer Energie (umgewandelt aus potenzieller Energie) schneller bewegen, wird das System ungeordneter und seine Entropie steigt. Daher streben alle reagierenden Systeme danach, einen Gleichgewichtszustand zu erreichen, in dem die potenzielle Energie auf ein Minimum reduziert ist. Bei der CRO erfassen wir dieses Phänomen, indem wir potenzielle Energie in kinetische Energie umwandeln und die Energie der chemischen Moleküle in der Umgebung allmählich verlieren.
Ein chemisches System, das eine chemische Reaktion durchläuft, ist instabil und versucht, überschüssige Energie loszuwerden und sich zu stabilisieren. Das System besteht aus Molekülen, den kleinsten Teilchen einer Verbindung, die je nach ihren grundlegenden chemischen Eigenschaften in verschiedene Typen eingeteilt werden.
Chemische Reaktionen sind ein komplexer Tanz, bei dem Moleküle zusammenstoßen, Bindungen eingehen und aufbrechen und dabei ihre Struktur verändern. Jeder Schritt dieses Tanzes ist eine Abfolge von Unterreaktionen, die zu stabileren Produkten mit minimaler Energie führen. Moleküle speichern Energie in ihren chemischen Bindungen, und selbst kleine Veränderungen in ihrer Struktur können zu erstaunlichen Umwandlungen führen, die stabilere Produkte hervorbringen. Die Molekülstruktur beeinflusst das chemische Verhalten einer Verbindung, und selbst geringfügige Änderungen der Struktur können zu erheblichen Unterschieden in den chemischen Eigenschaften führen.
Chemische Reaktionen werden durch Zusammenstöße von Molekülen ausgelöst, die unimolekular (mit externen Substanzen) oder bimolekular (mit anderen Molekülen) sein können. Diese Kollisionen können entweder effizient oder ineffizient sein, abhängig von Faktoren wie der Aktivierungsenergie und sterischen Effekten. Auf diese Weise tanzen die Moleküle, verflechten und trennen sich und schaffen neue Formen und Strukturen.
Es ist wichtig zu wissen, dass bei chemischen Reaktionen häufig stabilere Produkte mit niedrigeren Gibbs-Energien gebildet werden. Dieser Prozess erfolgt in aufeinander folgenden und/oder parallelen Schritten, bei denen Zwischenverbindungen gebildet werden und Übergangszustände durchlaufen werden. Das Verständnis dieser Prozesse ist wichtig, um optimale Bedingungen für chemische Reaktionen zu finden und die Mechanismen der Umwandlung chemischer Verbindungen zu klären.
Außerdem kann eine chemische Synthese sowohl durch effektive als auch durch ineffektive Zusammenstöße von Reaktionspartner erfolgen. Die Effizienz von Kollisionen hängt von Faktoren wie der Aktivierungsenergie, sterischen Effekten usw. ab. Die intermolekulare Synthese führt im Gegensatz zur intramolekularen Synthese zu stärkeren Veränderungen der Molekülstrukturen.
Der CRO-Algorithmus gleicht einem virtuosen Choreographen, der die Gesetze der Chemie wie Musiknoten einsetzt, um unglaublich komplexe und elegante Optimierungslösungen zu schaffen. Gehen wir nun Punkt für Punkt vor - von komplexen zu einfachen Konzepten.
2. Implementierung von chemischen Vorgängen bzw. Operatoren
Die Grundeinheit der CRO ist das „Molekül“. In einer „Population“ hat jeder von ihnen bestimmte Eigenschaften, wie „potenzielle und kinetische Energien“, „molekulare Strukturen“, usw. Elementare Reaktionen definieren die Wechselwirkungen zwischen Molekülen. Wir können eine Klasse definieren, in der die Datenfelder Merkmale des Moleküls darstellen und die Methoden elementare Reaktionen beschreiben.
Eines der wichtigsten Konzepte im CRO-Bereich ist die Energieeinsparung. Dieses Prinzip gewährleistet, dass die Gesamtenergie im System während des gesamten Optimierungsprozesses konstant bleibt. Die Energieerhaltung bestimmt die Übergänge zwischen den verschiedenen Zuständen der Moleküle während der Reaktionen und sorgt für ein Gleichgewicht der Energien, das die Suche nach optimalen Lösungen beeinflusst.
Unter Verwendung manipulierbarer Agenten (Moleküle), elementarer Reaktionen und dem Konzept der Energieerhaltung bietet CRO eine flexible und anpassungsfähige Methode zur Lösung komplexer Optimierungsprobleme. Die Möglichkeit, diese Vorgänge bzw. Operatoren anzupassen, die Populationsgröße dynamisch zu verändern und verschiedene Attribute zu integrieren, machen CRO zu einem vielversprechenden Ansatz im Bereich der Metaheuristik. Seine einzigartigen Funktionen und seine Flexibilität ermöglichen es den Forschern, neue Möglichkeiten der Optimierung zu erkunden.
Der CRO-Algorithmus verwendet die folgenden Operatoren:
1. Intermolekulare, unwirksame Kollision. Dieser Operator modelliert den Prozess, bei dem zwei Moleküle zusammenstoßen, aber intakt bleiben, und ihre Strukturen leicht verändert werden. Dadurch kann der Algorithmus eine lokale Suche in der Nähe der aktuellen Lösungen durchführen.
2. Zerlegung. Dieser Operator modelliert den Prozess, bei dem ein Molekül mit einer Wand zusammenstößt und in zwei neue Moleküle zerfällt. Dadurch kann der Algorithmus neue Bereiche des Lösungsraums erkunden.
3. Intra-molekulare Reaktion. Dieser Operator modelliert den Prozess, bei dem ein Molekül mit einer Wand zusammenstößt und intakt bleibt, aber seine Struktur leicht verändert. Dadurch kann der Algorithmus eine lokale Suche in der Nähe der aktuellen Lösung durchführen.
4. Synthese. Dieser Operator modelliert den Prozess, bei dem zwei Moleküle zusammenstoßen und sich zu einem einzigen neuen Molekül verbinden. Auf diese Weise kann der Algorithmus gute Lösungen kombinieren, um potenziell bessere Lösungen zu schaffen.
Jeder dieser Operatoren spielt eine wichtige Rolle im CRO-Algorithmus, da er es ihm ermöglicht, den Suchraum zu erkunden und optimale Lösungen zu finden. Ihre Zusammenarbeit sorgt für ein Gleichgewicht zwischen Exploration (Suche nach neuen Bereichen des Suchraums) und Exploitation (Verbesserung der derzeit besten Lösungen).
Betrachten wir nun jeden einzelnen chemischen Vorgang im Algorithmus (Suche nach dem globalen Minimum):
Algorithmus #1: On-wall Ineffective Collision (Unwirksamer Aufprall auf die Wand)
1. Eingangsdaten: Mω
molecule2. Erzeugen einer neuen Position des Moleküls:ω = N(ω)
3. Berechne die potenziellen Energie der neuen Position: PEω = f(ω)
4. Erhöhe den Kollisionszähler: NumHitω = NumHitω + 1
5. Wenn die potentielle Energie einer neuen Position + kinetische Energie ≥ potentielle Energie der aktuellen Position ist, dann:
6. Erzeuge eine Zufallszahl „a“ im Bereich [KELossRate, 1]
7. Aktualisiere die kinetischen Energie: KEω = (PEω − PEω + KEω) × a
8. Aktualisiere den Puffer: buffer = buffer + (PEω − PEω + KEω) × (1 − a)
9. Behalte die derzeitigen Energien und die Position
10. Wenn die potenzielle Energie einer neuen Position < minimale potenzielle Energie ist, werden die Mindestwerte aktualisiert.
11. Bedingung für das Ende
Algorithmus #2: Zerlegung
1. Eingangsdaten: Mω
molecule2. Schaffen von zwei neuen Molekülen Mω1 and Mω2
3. Ermittle die Positionen für neue Moleküle: ω1 and ω2 aus ω
4. Berechne die potenziellen Energie der neuen Moleküle: PEω1 = f(ω1) and PEω2 = f(ω2)
5. Wenn die potentielle Energie der aktuellen Position + die kinetische Energie ≥ die gesamte potentielle Energie der neuen Positionen ist, dann:
6. Berechne die Energie für die Zerlegung: Edec = PEω + KEω − (PEω1 + PEω2)
7. Weiter zu Schritt 13
8. sonst:
9. Erzeuge die Zufallszahlen δ1, δ2 im Bereich [0, 1]
10. Berechne die Energie für die Zerlegung: Edec = PEω + KEω + δ1δ2 × buffer − (PEω1 + PEω2)
11. Wenn Edec ≥ 0 ist, dann:
12. Aktualisiere den Puffer
13. Erzeuge die Zufallszahl δ3 im Bereich [0, 1]
14. Verteile die kinetischen Energie zwischen neuen Molekülen
15. Behalte die Mindestwerte für jedes neue Molekül
16. Zerstöre das aktuelle Molekül
17. sonst:
18. Erhöhe den Kollisionszähler
19. Zerstöre die neuen Moleküle
20. Ende der Bedingung
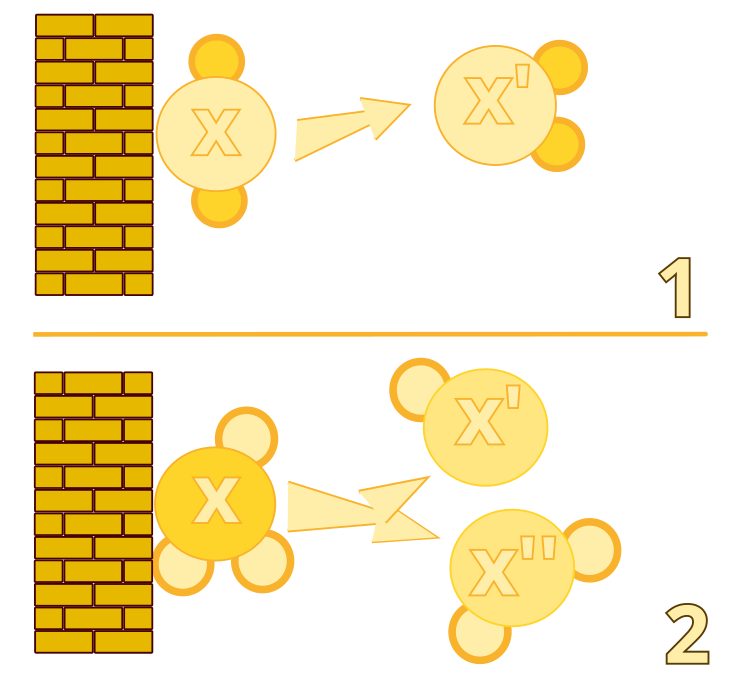
Abbildung 1. Ineffiziente Wandkollision: Algorithmus #1. Zerlegung: Algorithmus Nr. 2
Algorithmus #3: Ineffektive, intermolekulare Kollision
1. Eingangsdaten: Mω1 und Mω2
Moleküle2. Generieren neuer Positionen für die Moleküle: ω1 = N(ω1) und ω2 = N(ω2)
3. Berechne die potenzielle Energie für neue Positionen: PEω1 = f(ω1) and PEω2 = f(ω2)
4. Erhöhe den Kollisionszähler: NumHitω1 = NumHitω1 + 1 and NumHitω2 = NumHitω2 + 1
5. Berechne die Energie der intermolekularen Wechselwirkung: Einter = (PEω1 + PEω2 + KEω1 + KEω2) − (PEω1 + PEω2)
6. Wenn Einter ≥ 0 ist, dann:
7. Erzeuge die Zufallszahl δ4 im Bereich [0, 1]
8. Verteile die kinetische Energie zwischen den Molekülen: KEω1 = Einter × δ4 and KEω2 = Einter × (1 − δ4)
9. Aktualisiere die Molekülpositionen und Energien
10. Wenn die potenzielle Energie einer neuen Position geringer ist als die minimale potenzielle Energie, dann aktualisiere die Mindestwerte für jedes Molekül
11. Ende der Bedingung
Algorithmus #4: Synthese
1. Eingangsdaten: Mω1 und Mω2
Moleküle2. Erstellen eines neuen Mω
Molekül3. Ermittele eine Position für ein neues Molekül: ω aus ω1 und ω2
4. Berechne die potenzielle Energie für ein neues Molekül: PEω = f(ω)
5. Wenn die gesamte potenzielle Energie und kinetische Energie des neuen Moleküls größer oder gleich der gesamten potenziellen Energie und kinetischen Energie der ursprünglichen Moleküle ist, dann:
6. Verteile die überschüssige, kinetische Energie auf ein neues Molekül: KEω = (PEω1 + PEω2 + KEω1 + KEω2) − PEω
7. Aktualisiere die Mindestwerte für neue Moleküle
8. Zerstöre die ursprünglichen Moleküle
9. sonst:
10. Kollisionszähler für Quellmoleküle erhöhen
11. Zerstöre ein neues Moleküls
12. Ende der Bedingung
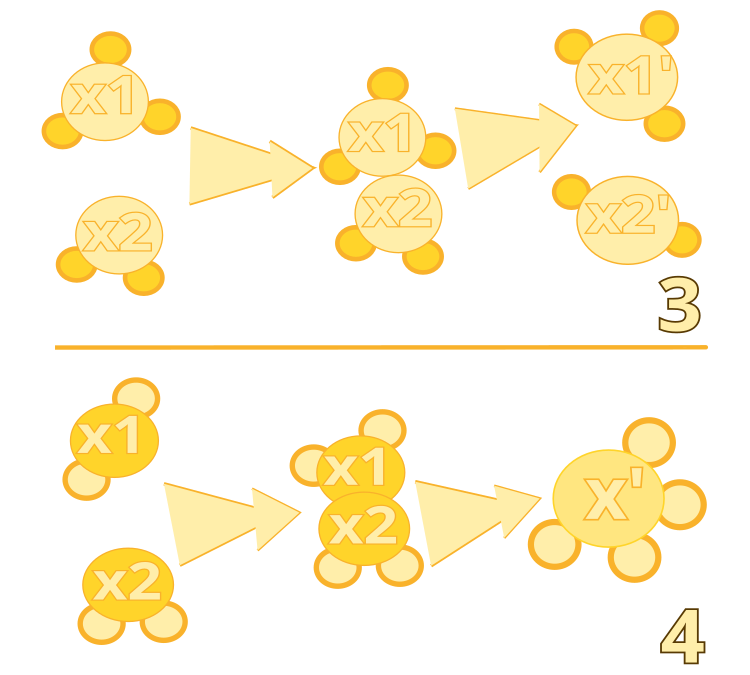
Bild 2. Ineffiziente, intermolekulare Kollision: Algorithmus #3. Synthese: Algorithmus #4
Die vorgeschlagene Beschreibung des CRO-Algorithmus spiegelt die Vision des Autors von diesem Ansatz wider. Dabei bleiben jedoch einige wichtige Punkte unberücksichtigt, die die Leistung und die Suchmöglichkeiten des Algorithmus erheblich beeinträchtigen können.
Der Algorithmus beinhaltet die Erhaltung der Energie in einem geschlossenen Raum und den Übergang von einer Energieart zu einer anderen. Allerdings geben die Autoren nicht an, wie die numerischen Energieindikatoren mit dem Wert der Zielfunktion (Fitness) korrespondieren. Es ist offensichtlich, dass die Autoren die Rolle der Fitness als potenzielle Energie definieren und die kinetische Energie als Mechanismus zum Ausgleich der Abnahme der potenziellen Energie dient (die Summe aller Energien sollte idealerweise eine Konstante sein).
Wenn zum Beispiel das Kriterium eines Optimierungsproblems der Gewinnfaktor ist, werden die Werte der Zielfunktion in einem kleinen Bereich schwanken, verglichen mit einem Problem, bei dem die Bilanz als Kriterium verwendet wird. In diesem Fall sehen wir, dass die Werte der Zielfunktion je nach dem spezifischen Optimierungskriterium variieren, aber die Autoren verwenden Konstanten als externe Parameter des Algorithmus, die nicht mit den im Algorithmus berechneten Energien verglichen werden können.
Der ursprüngliche CRO-Algorithmus (ohne Änderungen) hat also eine deutlich eingeschränkte Liste von Aufgaben, auf die er angewendet werden kann, ist also nicht universell. In dieser Artikelserie betrachten wir nur universelle Algorithmen, die auf allgemeine Optimierungsprobleme anwendbar sind, und wenn ein Algorithmus dies nicht zulässt, modifizieren wir ihn normalerweise und bringen ihn in eine einzige allgemeine Form.
Der zweite Punkt ist, dass der ursprüngliche Algorithmus eine ungeordnete Berechnung der Fitnessfunktion in verschiedenen Teilen der Logik vorsieht, was bei der Anwendung in der Praxis und bei der Integration in Projekte unweigerlich zu Problemen führen wird. Auch dies werden wir beheben. Dazu müssen wir die chemischen Operatoren in zwei Teile aufspalten: die Operatoren selbst und die so genannten „Post-Operatoren“, von denen der erste die Position der Moleküle verändert und der zweite die notwendigen Aktionen mit den Molekülen durchführt, nachdem ihre Fitness berechnet wurde.
Der dritte Punkt ist, dass der ursprüngliche Algorithmus eine dynamische Populationsgröße von Molekülen vorsieht. Neue Moleküle entstehen und einige der alten werden zerstört. Es gibt keine Mechanismen zur Regulierung der Populationsgröße, und sie kann in sehr weiten Grenzen schwanken. So haben Experimente gezeigt, dass die Population bei bestimmten Einstellungen der externen Parameter von anfänglich 50 Molekülen auf mehr als tausend anwachsen kann. Wir haben dieses Problem auch dadurch gelöst, dass wir der Population nacheinander Molekülen hinzufügen, die aus der Ausführung von Operatoren resultieren, und dabei einen einfachen Zähler verwenden und den Index der Elternmoleküle in der Elternpopulation speichern. Auf diese Weise kann die Größe der Population konstant gehalten werden, und gleichzeitig entfällt die Notwendigkeit, Moleküle zu entfernen - die Tochtermoleküle ersetzen, wenn die Bedingungen erfüllt sind, einfach die entsprechenden Elternmoleküle.
Diese Änderungen ermöglichten es, den CRO-Algorithmus in eine Form zu bringen, die für die Lösung von Optimierungsproblemen im Allgemeinen geeignet ist, die Integration des Algorithmus in Projekte zu erleichtern und gleichzeitig das allgemeine ursprüngliche Konzept der chemischen Reaktionen zu bewahren. Der CRO-Algorithmus wird im Detail beschrieben. Wenn Sie möchten, können Sie versuchen, die Berücksichtigung von potenzieller und kinetischer Energie in den Algorithmus zu integrieren.
Kommen wir nun zum Code. Beschreiben wir die Reaktionstypen in der Liste, damit wir nach den Reaktionen und der Struktur der Moleküle den geeigneten Postoperator auswählen können.
Die Enumeration E_ReactionType:
1. synthesis ist ein Prozess, bei dem sich zwei Moleküle zu einem neuen Molekül verbinden.
2. interMolecularInefColl ist eine unwirksame, intermolekulare Kollision, bei der Moleküle zusammenstoßen, aber nicht miteinander reagieren.
3. decomposition ist ein Prozess, bei dem ein komplexes Molekül in einfachere Bestandteile zerlegt wird.
4. inefCollision ist ein ineffizienter Zusammenstoß mit der Wand, der die Struktur des Moleküls verändert.
Definition der Struktur S_CRO_Agent, die ein Modell des Moleküls im Kontext des Algorithmus darstellt. Schauen wir uns an, welche Felder die folgende Struktur enthält:
- structure[] - ein Array, das die Molekülstruktur darstellt.
- NumHit - ein Zähler für die Anzahl der „hits“ (Zusammenstöße) oder Molekülwechselwirkungen.
- indMolecule_1 und indMolecule_2 - Indizes der interagierenden Moleküle.
- KE - eine Variable, die die kinetische Energie eines Moleküls darstellt.
- f - Fitnessfunktion (Fitness des Moleküls).
- rType - Variable vom Typ E_ReactionType, die die Art der Reaktion angibt, an der ein Molekül beteiligt ist.
„Init“ - Strukturmethode, die die Felder initialisiert. Sie nimmt das Ganzzahlargument coords, um die Größe des Strukturarrays mit der Funktion ArrayResize zu ändern. NumHit, indMolecule_1, indMolecule_2, f und KE werden mit Nullen oder -DBL_MAX initialisiert.
Dieser Code stellt die grundlegende Datenstruktur für Moleküle im CRO-Algorithmus dar und initialisiert ihre Felder, wenn ein neues Molekül erstellt wird.
enum E_ReactionType { synthesis, interMolecularInefColl, decomposition, inefCollision }; // Molecule structure struct S_CRO_Agent { double structure []; int NumHit; int indMolecule_1; int indMolecule_2; double KE; double f; E_ReactionType rType; // Initialization method void Init (int coords) { ArrayResize (structure, coords); NumHit = 0; indMolecule_1 = 0; indMolecule_2 = 0; f = -DBL_MAX; KE = -DBL_MAX; } };
Die Methode InefCollision der Klasse C_AO_CRO ist eine ineffiziente Kollision, bei der ein neues Molekül durch die Verdrängung eines Elternmoleküls entsteht. Bei dieser Methode geschieht Folgendes:
1. Die Methode akzeptiert den Index des übergeordneten Moleküls und den Link zum molCNT-Molekülzähler. Wenn molCNT größer oder gleich der Populationsgröße popSize ist, gibt die Methode false zurück und beendet ihre Arbeit.
2. Die Methode bestimmt den Index eines neuen Moleküls index1_, das als Ergebnis der Kollision entsteht.
3. Die Struktur des Elternmoleküls wird in die Struktur des neuen Moleküls kopiert.
4. Anschließend wird die Funktion N für jede Koordinate des neuen Moleküls ausgeführt. Die Funktion erzeugt neue Koordinatenwerte in der Nähe der alten Werte.
5. Neue Koordinatenwerte werden mit der Funktion SeInDiSp so angepasst, dass sie in dem angegebenen Bereich von rangeMin bis rangeMax bleiben.
6. Dann werden die Feldwerte indMolecule_1, rType und NumHit des neuen Moleküls gesetzt. indMolecule_1 speichert den Index des übergeordneten Moleküls, rType wird auf inefCollision gesetzt, während NumHit zurückgesetzt wird.
7. Am Ende der Methode wird der Molekülzähler molCNT um 1 erhöht und die Methode gibt true zurück.
//—————————————————————————————————————————————————————————————————————————————— // Ineffective collision. Obtaining a new molecule by displacing a parent one. bool C_AO_CRO::InefCollision (int index, int &molCNT) { if (molCNT >= popSize) return false; int index1_ = molCNT; ArrayCopy (Mfilial [index1_].structure, Mparent [index].structure); for (int c = 0; c < coords; c++) { N (Mfilial [index1_].structure [c], c); Mfilial [index1_].structure [c] = u.SeInDiSp (Mfilial [index1_].structure [c], rangeMin [c], rangeMax [c], rangeStep [c]); } Mfilial [index1_].indMolecule_1 = index; // save the parent molecule index Mfilial [index1_].rType = inefCollision; Mfilial [index1_].NumHit = 0; molCNT++; return true; } //——————————————————————————————————————————————————————————————————————————————
Die Methode PostInefCollision in der Klasse C_AO_CRO ist für die Behandlung der Ergebnisse der unwirksamen Kollision des Moleküls mol mit anderen Molekülen in der chemischen Reaktionssimulation gedacht. Die Methode geht folgendermaßen vor:
1. Die Variable ind wird deklariert und mit dem Wert indMolecule_1 aus dem Objekt mol initialisiert.
2. Der bedingte Ausdruck prüft, ob der f-Wert des Objekts mol den f-Wert des übergeordneten Moleküls mit dem Index ind übersteigt.
3. Wenn die Bedingung erfüllt ist, wird die Struktur mol in die Struktur des übergeordneten Moleküls mit dem Index ind kopiert.
4. Der Wert f des übergeordneten Moleküls wird mit dem Wert f von mol aktualisiert.
5. Der Zähler NumHit des übergeordneten Moleküls wird auf Null zurückgesetzt.
6. Wenn die Bedingung mol.f > Mparent [ind].f falsch ist, wird der Zähler NumHit des Elternmoleküls um eins erhöht.
Im Allgemeinen aktualisiert diese Methode die Struktur und den Wert der Fitnessfunktion des Elternmoleküls auf der Grundlage der Ergebnisse einer ineffizienten Kollision. Wenn die neue Molstruktur zu einer Verbesserung der Fitness führt, ersetzt sie die Struktur des Elternmoleküls und der Zähler NumHit wird zurückgesetzt. Andernfalls wird der Zähler NumHit des übergeordneten Moleküls erhöht.
//—————————————————————————————————————————————————————————————————————————————— // Handling the results of an ineffective collision. void C_AO_CRO::PostInefCollision (S_CRO_Agent &mol) { int ind = mol.indMolecule_1; if (mol.f > Mparent [ind].f) { ArrayCopy (Mparent [ind].structure, mol.structure); Mparent [ind].f = mol.f; Mparent [ind].NumHit = 0; } else { Mparent [ind].NumHit++; } } //——————————————————————————————————————————————————————————————————————————————
Die Methode Decomposition der Klasse C_AO_CRO ist ein Zerlegungsprozess, bei dem zwei neue Moleküle durch das Zerlegen eines Elternmoleküls entstehen. Bei dieser Methode geschieht Folgendes:
1. Die Methode akzeptiert den Index des übergeordneten Moleküls und einen Link zum Molekülzähler molCNT. Wenn molCNT größer oder gleich popSize - 1 ist, gibt die Methode false zurück und beendet ihre Arbeit.
2. Die Methode bestimmt dann die Indizes von zwei neuen Molekülen - index1_ und index2_, die als Ergebnis der Zerlegung entstehen.
3. Die Struktur des übergeordneten Moleküls wird in die Strukturen der neuen Moleküle kopiert.
4. Anschließend wird die Funktion N für jede Koordinate der neuen Moleküle ausgeführt. Die Funktion erzeugt neue Koordinatenwerte in der Nähe der alten Werte. Dies geschieht getrennt für die erste Hälfte der Koordinaten für index1_ des ersten Tochtermoleküls und für die zweite Hälfte der Koordinaten für index2_ des zweiten Moleküls.
5. Neue Koordinatenwerte werden mit der Funktion SeInDiSp so angepasst, dass sie in dem angegebenen Bereich von rangeMin bis rangeMax bleiben.
6. Dann werden die Feldwerte indMolecule_1, indMolecule_2, rType und NumHit des neuen Moleküls gesetzt. indMolecule_1 und indMolecule_2 speichern die Indizes der Eltern- bzw. Schwestermoleküle, rType wird auf Zerlegung gesetzt, während NumHit auf Null gesetzt wird.
7. Am Ende der Methode wird der Molekülzähler molCNT um 2 erhöht, und die Methode gibt true zurück.
//—————————————————————————————————————————————————————————————————————————————— // Decomposition. Obtaining two new molecules by decomposing a parent one. bool C_AO_CRO::Decomposition (int index, int &molCNT) { if (molCNT >= popSize - 1) return false; // Creating two new molecules M_ω'_1 and M_ω'_2 from M_ω int index1_ = molCNT; int index2_ = molCNT + 1; ArrayCopy (Mfilial [index1_].structure, Mparent [index].structure); ArrayCopy (Mfilial [index2_].structure, Mparent [index].structure); for (int c = 0; c < coords / 2; c++) { N (Mfilial [index1_].structure [c], c); Mfilial [index1_].structure [c] = u.SeInDiSp (Mfilial [index1_].structure [c], rangeMin [c], rangeMax [c], rangeStep [c]); } for (int c = coords / 2; c < coords; c++) { N (Mfilial [index2_].structure [c], c); Mfilial [index2_].structure [c] = u.SeInDiSp (Mfilial [index2_].structure [c], rangeMin [c], rangeMax [c], rangeStep [c]); } Mfilial [index1_].indMolecule_1 = index; // save the parent molecule index Mfilial [index1_].indMolecule_2 = index2_; // save the index of the second daughter molecule Mfilial [index1_].rType = decomposition; Mfilial [index1_].NumHit = 0; Mfilial [index2_].indMolecule_1 = index1_; // save the index of the first daughter molecule Mfilial [index2_].indMolecule_2 = -1; // mark the molecule so we do not handle it twice Mfilial [index2_].rType = decomposition; Mfilial [index2_].NumHit = 0; molCNT += 2; return true; } //——————————————————————————————————————————————————————————————————————————————
Die Methode PostDecomposition der Klasse C_AO_CRO verarbeitet die Ergebnisse der Molekülzerlegung. Die Methode geht folgendermaßen vor:
1. Die Methode akzeptiert einen Verweis auf das Molmolekül, das als Ergebnis der Zersetzung erhalten wird. Wenn indMolecule_2 des Moleküls gleich „-1“ ist (was bedeutet, dass das Molekül bereits verarbeitet wurde), beendet die Methode ihre Arbeit.
2. Der Index ind des Elternmoleküls wird dann zusammen mit den Indizes der beiden „Tochter-Molekülen“ index1_ und index2_ extrahiert.
3. Anschließend wird geprüft, ob der f-Fitness-Funktionswert des ersten „Tochter“-Moleküls die f-Funktionswerte des zweiten „Tochter“- und des Elternmoleküls übersteigt. Wenn dies der Fall ist, ersetzt das erste „Tochter“-Molekül das Muttermolekül. Der NumHit-Wert des ersetzten Moleküls wird zurückgesetzt und das Flag wird gesetzt.
4. Ist das Flag immer noch falsch, so wird eine ähnliche Prüfung für das zweite „Tochter“-Molekül durchgeführt.
5. Wenn nach allen Prüfungen das Flag immer noch falsch ist, wird die Anzahl der Treffer des übergeordneten Moleküls um 1 erhöht.
Die Methode PostDecomposition ist für die Aktualisierung der Zustände des Elternmoleküls nach der Zerlegung im CRO-Algorithmus zuständig.
//—————————————————————————————————————————————————————————————————————————————— // Handling decomposition results. void C_AO_CRO::PostDecomposition (S_CRO_Agent &mol) { if (mol.indMolecule_2 == -1) return; int ind = mol.indMolecule_1; int index2_ = mol.indMolecule_2; int index1_ = Mfilial [index2_].indMolecule_1; bool flag = false; if (Mfilial [index1_].f > Mfilial [index2_].f && Mfilial [index1_].f > Mparent [ind].f) { ArrayCopy (Mparent [ind].structure, Mfilial [index1_].structure); Mparent [ind].f = Mfilial [index1_].f; Mparent [ind].NumHit = 0; flag = true; } if (!flag) { if (Mfilial [index2_].f > Mfilial [index1_].f && Mfilial [index2_].f > Mparent [ind].f) { ArrayCopy (Mparent [ind].structure, Mfilial [index2_].structure); Mparent [ind].f = Mfilial [index2_].f; Mparent [ind].NumHit = 0; flag = true; } } if (!flag) { Mparent [ind].NumHit++; } } //——————————————————————————————————————————————————————————————————————————————
Die Methode InterMolInefColl der Klasse C_AO_CRO ist eine ineffiziente Kollision, bei der zwei neue Moleküle durch die Veränderung von zwei Elternmolekülen entstehen. Bei dieser Methode geschieht Folgendes:
1. Die Methode akzeptiert die Indizes der beiden Elternmoleküle - index1 und index2 - und einen Link zum Molekülzähler molCNT. Wenn molCNT größer oder gleich popSize - 1 ist, gibt die Methode false zurück und beendet ihre Arbeit.
2. Die Methode bestimmt dann die Indizes der beiden neuen Tochtermoleküle index1_ und index2_, die als Ergebnis der Kollision entstehen.
3. Die Strukturen des Muttermoleküls werden in die Strukturen der neuen Moleküle kopiert.
4. Anschließend wird die Funktion N für jede Koordinate der neuen Moleküle ausgeführt. Die Funktion erzeugt neue Koordinatenwerte in der Nähe der alten Werte.
5. Anschließend werden die neuen Koordinatenwerte mit der Funktion SeInDiSp so angepasst, dass sie in dem angegebenen Bereich von rangeMin bis rangeMax bleiben.
6. Dann werden die Feldwerte indMolecule_1, indMolecule_2, rType und NumHit des neuen Moleküls gesetzt. indMolecule_1 und indMolecule_2 behalten die Indizes der Elternmoleküle bei, rType wird auf interMolecularInefColl gesetzt, während NumHit auf Null gesetzt wird.
7. Am Ende der Methode wird der Molekülzähler molCNT um 2 erhöht, und die Methode gibt true zurück.
//—————————————————————————————————————————————————————————————————————————————— // Intermolecular ineffective collision. Obtaining two new molecules by changing two parent ones bool C_AO_CRO::InterMolInefColl (int index1, int index2, int &molCNT) { if (molCNT >= popSize - 1) return false; int index1_ = molCNT; int index2_ = molCNT + 1; // Obtaining molecules ArrayCopy (Mfilial [index1_].structure, Mparent [index1].structure); ArrayCopy (Mfilial [index2_].structure, Mparent [index2].structure); // Generating new molecules ω'_1 = N(ω1) and ω'_2 = N(ω2) in the vicinity of ω1 and ω2 for (int c = 0; c < coords; c++) { N (Mfilial [index1_].structure [c], c); N (Mfilial [index2_].structure [c], c); } for (int c = 0; c < coords; c++) { Mfilial [index1_].structure [c] = u.SeInDiSp (Mfilial [index1_].structure [c], rangeMin [c], rangeMax [c], rangeStep [c]); Mfilial [index2_].structure [c] = u.SeInDiSp (Mfilial [index2_].structure [c], rangeMin [c], rangeMax [c], rangeStep [c]); } Mfilial [index1_].indMolecule_1 = index1; // save the index of the first parent molecule Mfilial [index1_].indMolecule_2 = index2_; // save the index of the second daughter molecule Mfilial [index1_].rType = interMolecularInefColl; Mfilial [index1_].NumHit = 0; Mfilial [index2_].indMolecule_1 = index2; // save the index of the second parent molecule Mfilial [index2_].indMolecule_2 = -1; // mark the molecule so we do not handle it twice Mfilial [index2_].rType = interMolecularInefColl; Mfilial [index2_].NumHit = 0; molCNT += 2; return true; } //——————————————————————————————————————————————————————————————————————————————
Die Methode PostInterMolInefColl der Klasse C_AO_CRO verarbeitet die Ergebnisse der intermolekularen unwirksamen Kollisionen. Mit der Methode werden folgende Ziele verfolgt:
1. Die Methode akzeptiert einen Verweis auf das Molekül mol, das als Ergebnis einer Kollision erhalten wurde. Wenn indMolecule_2 des Moleküls gleich „-1“ ist (was bedeutet, dass das Molekül bereits verarbeitet wurde), beendet die Methode ihre Arbeit.
2. Anschließend werden die Indizes ind1 und ind2 der beiden Ausgangsmoleküle extrahiert.
3. Als Nächstes wird geprüft, ob die f-Summe der Fitnesswerte eines neuen Moleküls und seiner „Schwester“ die f-Summe der Fitnesswerte der beiden Elternmoleküle übersteigt. Wenn dies der Fall ist, ersetzen die neuen Moleküle die alten. Die Werte NumHit der ersetzten Moleküle werden zurückgesetzt.
4. Andernfalls werden die Werte NumHit der Elternmoleküle um 1 erhöht.
Die Methode PostInterMolInefColl ist für die Aktualisierung der Zustände der Elternmoleküle nach der intermolekularen ineffizienten Kollision im CRO-Algorithmus zuständig.
//—————————————————————————————————————————————————————————————————————————————— // Handling the results of an intermolecular ineffective collision. void C_AO_CRO::PostInterMolInefColl (S_CRO_Agent &mol) { if (mol.indMolecule_2 == -1) return; int ind1 = mol.indMolecule_1; int ind2 = Mfilial [mol.indMolecule_2].indMolecule_1; Mparent [ind1].NumHit++; Mparent [ind2].NumHit++; if (mol.f + Mfilial [mol.indMolecule_2].f > Mparent [ind1].f + Mparent [ind2].f) { ArrayCopy (Mparent [ind1].structure, mol.structure); Mparent [ind1].f = mol.f; ArrayCopy (Mparent [ind2].structure, Mfilial [mol.indMolecule_2].structure); Mparent [ind2].f = Mfilial [mol.indMolecule_2].f; } } //——————————————————————————————————————————————————————————————————————————————
Der letzte chemische Reaktionsoperator im Algorithmus - die Synthesemethode der Klasse C_AO_CRO - ist ein Syntheseprozess, bei dem ein neues Molekül durch Verschmelzung zweier Elternmoleküle entsteht.
1. Die Methode akzeptiert die Indizes der beiden Elternmoleküle - index1 und index2 - und einen Link zum Molekülzähler molCNT. Wenn molCNT größer oder gleich der Populationsgröße popSize ist, gibt die Methode false zurück und beendet ihre Arbeit.
2. In der Schleife wird für jede Koordinate des neuen Moleküls Folgendes getan: Wenn eine Zufallszahl kleiner als 0,5 ist, erhält die Koordinate den Wert der entsprechenden Koordinate des ersten Elternmoleküls Mparent[index1].structure[i]. Andernfalls erhält es den Wert der entsprechenden Koordinate des zweiten Elternmoleküls Mparent[index2].structure[i].
3. Dann werden die Feldwerte indMolecule_1, indMolecule_2, rType und NumHit des neuen Moleküls gesetzt. indMolecule_1 und indMolecule_2 behalten die Indizes der Elternmoleküle bei, rType wird auf Synthese gesetzt, während NumHit auf Null gesetzt wird.
4. Am Ende der Methode wird der Molekülzähler molCNT erhöht, und die Methode gibt true zurück.
//—————————————————————————————————————————————————————————————————————————————— // Synthesis. Obtaining a new molecule by fusing two parent ones bool C_AO_CRO::Synthesis (int index1, int index2, int &molCNT) { if (molCNT >= popSize) return false; // Create a new M_ω' molecule from M_ω1 and M_ω2 for (int i = 0; i < coords; i++) { if (u.RNDprobab () < 0.5) Mfilial [molCNT].structure [i] = Mparent [index1].structure [i]; else Mfilial [molCNT].structure [i] = Mparent [index2].structure [i]; } Mfilial [molCNT].indMolecule_1 = index1; // save the index of the first parent molecule Mfilial [molCNT].indMolecule_2 = index2; // save the index of the second parent molecule Mfilial [molCNT].rType = synthesis; Mfilial [molCNT].NumHit = 0; molCNT++; return true; } //——————————————————————————————————————————————————————————————————————————————
Die Methode PostSynthesis der Klasse C_AO_CRO verarbeitet die Syntheseergebnisse. Hier ist, was darin geschieht:
1. Die Methode akzeptiert einen Verweis auf das Molmolekül, das als Ergebnis der Synthese erhalten wurde. Daraus werden dann die Indizes ind1 und ind2 der beiden Elternmoleküle extrahiert.
2. Als Nächstes wird geprüft, ob der Fitness-Wert f eines neuen Moleküls die Fitness-Werte f der beiden Elternmoleküle übersteigt. Wenn dies der Fall ist, ersetzt das neue Molekül dasjenige der Elternmoleküle, dessen f-Wert kleiner ist. Der Wert NumHit des ersetzten Moleküls wird auf Null gesetzt.
3. Andernfalls werden die Werte NumHit der Elternmoleküle um 1 erhöht.
Die Methode PostSynthesis ist für die Aktualisierung der Zustände der Elternmoleküle nach dem Syntheseprozess im CRO-Algorithmus zuständig.
//—————————————————————————————————————————————————————————————————————————————— // Handling synthesis results. void C_AO_CRO::PostSynthesis (S_CRO_Agent &mol) { int ind1 = mol.indMolecule_1; int ind2 = mol.indMolecule_2; if (mol.f > Mparent [ind1].f && mol.f > Mparent [ind2].f) { if (Mparent [ind1].f < Mparent [ind2].f) { ArrayCopy (Mparent [ind1].structure, mol.structure); Mparent [ind1].f = mol.f; Mparent [ind1].NumHit = 0; } else { ArrayCopy (Mparent [ind2].structure, mol.structure); Mparent [ind2].f = mol.f; Mparent [ind2].NumHit = 0; } } else { Mparent [ind1].NumHit++; Mparent [ind2].NumHit++; } } //——————————————————————————————————————————————————————————————————————————————
Abschließend beschreiben wir die Methode N der Klasse C_AO_CRO, die zur Änderung der Molekülstruktur in den chemischen Reaktionsoperatoren verwendet wird. Die Methode wird verwendet, um einen neuen Wert für die Molekülkoordinate innerhalb eines bestimmten Bereichs zu erzeugen:
1. Die Methode akzeptiert die Molekülkoordinate coord, die als Referenz geändert werden muss, und die Koordinatenposition coordPos in der Struktur.
2. Als Nächstes wird der Abstand dist berechnet, d. h. die Differenz zwischen dem Höchst- und dem Mindestwert des Bereichs, multipliziert mit dem Parameter molecPerturb, der die Streuung der Werte in der Nähe des aktuellen Koordinatenwerts angibt.
3. Anschließend werden das Minimum (min) und das Maximum (max) einer neuen Koordinate festgelegt. Diese Werte entsprechen dem alten Koordinatenwert zuzüglich oder abzüglich des Abstandes, können aber nicht über den angegebenen Bereich von rangeMin[coordPos] bis rangeMax[coordPos] hinausgehen.
4. Schließlich wird der neue Koordinatenwert mit Hilfe der Gaußschen Verteilungsfunktion u.GaussDistribution erzeugt, die den alten Wert der Koordinate, den Minimal- und den Maximalwert der Standardabweichung, die gleich 8 sind, übernimmt.
//—————————————————————————————————————————————————————————————————————————————— void C_AO_CRO::N (double &coord, int coordPos) { double dist = (rangeMax [coordPos] - rangeMin [coordPos]) * molecPerturb; double min = coord - dist; if (min < rangeMin [coordPos]) min = rangeMin [coordPos]; double max = coord + dist; if (max > rangeMax [coordPos]) max = rangeMax [coordPos]; coord = u.GaussDistribution (coord, min, max, 8); } //——————————————————————————————————————————————————————————————————————————————
Wir haben alle chemischen Operatoren im Hinblick auf ihre Funktionalität und ihre Rolle im Optimierungsprozess analysiert. Diese Analyse hat uns einen tiefen Einblick in die Mechanismen verschafft, die die Dynamik von Molekülen in der chemischen Reaktionsmodellierung (CRO) bestimmen.
Im nächsten Artikel werden wir den Algorithmus erstellen und ihn anhand der Testfunktionen testen. Wir werden die untersuchten Operatoren zu einem vollwertigen CRO-Algorithmus konfigurieren und kombinieren, sodass er für den Einsatz bei praktischen Aufgaben bereit ist. Anschließend werden wir eine Reihe von Experimenten mit einer Vielzahl von Testfunktionen durchführen, die es uns ermöglichen, die Effizienz und Robustheit unseres Ansatzes zu bewerten.
Nach Erhalt der Arbeitsergebnisse werden wir Schlussfolgerungen über die Stärken und Schwächen des Algorithmus sowie über mögliche Richtungen für weitere Verbesserungen ziehen. Dadurch können wir nicht nur unsere Methode verbessern, sondern auch nützliche Empfehlungen für Forscher geben, die auf dem Gebiet der Optimierung und Modellierung chemischer Prozesse arbeiten.
Der nächste Artikel wird daher ein wichtiger Schritt in unserer Forschung sein, da wir von der Theorie zur Praxis übergehen und unseren Algorithmus testen und verbessern werden, um die besten Ergebnisse bei der Lösung komplexer Optimierungsprobleme zu erzielen.
Übersetzt aus dem Russischen von MetaQuotes Ltd.
Originalartikel: https://www.mql5.com/ru/articles/15041
Warnung: Alle Rechte sind von MetaQuotes Ltd. vorbehalten. Kopieren oder Vervielfältigen untersagt.
Dieser Artikel wurde von einem Nutzer der Website verfasst und gibt dessen persönliche Meinung wieder. MetaQuotes Ltd übernimmt keine Verantwortung für die Richtigkeit der dargestellten Informationen oder für Folgen, die sich aus der Anwendung der beschriebenen Lösungen, Strategien oder Empfehlungen ergeben.
 Neuronale Netze leicht gemacht (Teil 93): Adaptive Vorhersage im Frequenz- und Zeitbereich (letzter Teil)
Neuronale Netze leicht gemacht (Teil 93): Adaptive Vorhersage im Frequenz- und Zeitbereich (letzter Teil)
 Visualisierung der Handelsgeschäfte auf dem Chart (Teil 2): Grafische Anzeige der Daten
Visualisierung der Handelsgeschäfte auf dem Chart (Teil 2): Grafische Anzeige der Daten
 Vom Neuling zum Experten: Umfassende Fehlersuche in MQL5
Vom Neuling zum Experten: Umfassende Fehlersuche in MQL5
 Entwicklung eines Replay Systems (Teil 53): Die Dinge werden kompliziert (V)
Entwicklung eines Replay Systems (Teil 53): Die Dinge werden kompliziert (V)
- Freie Handelsapplikationen
- Über 8.000 Signale zum Kopieren
- Wirtschaftsnachrichten für die Lage an den Finanzmärkte
Sie stimmen der Website-Richtlinie und den Nutzungsbedingungen zu.
Hier ist Artikel über praktische Anwendung von in Handel. Lesen, studieren und anwenden in Praxis. Ich wünsche Sie Erfolg in Handel.
Artikel
Verwendung von Optimierungsalgorithmen zur Konfiguration von EA-Parametern im laufenden Betrieb
Andrey Dik, 2024.06.07 14:30
Der Artikel behandelt die praktischen Aspekte der Verwendung von Optimierungsalgorithmen, um die besten EA-Parameter on the fly zu finden, sowie die Virtualisierung von Handelsoperationen und EA-Logik. Der Artikel kann als Anleitung für die Implementierung von Optimierungsalgorithmen in einen EA verwendet werden.Hier ist Artikel über praktische Anwendung von in Handel. Lesen, studieren und anwenden in Praxis. Ichwünsche Sie Erfolg in Handel.
Mir gefällt Ihr Standpunkt, Andrei! Große Innovationen entstehen aus der Forschung. Es geht nicht immer nur um Geld, und ich schätze Ihren Beitrag für die Gemeinschaft.