
ディープニューラルネットワーク(その4)ニューラルネットワークモデルの作成、訓練、テスト
内容
はじめに
研究と適用の主な方向性
ディープニューラルネットワークの研究と適用には現在2つの主流があり、それぞれでは隠れ層でのニューロンの重みの初期化の手法が異なります。
手法1:ニューラルネットワークは、隠れ層の数が増えた場合(3を超える場合)は特に、隠れ層ニューロンの初期化の方法に非常に敏感です。G.Hynton教授はこの問題の解決を試みた最初の人物でした。彼の手法の背後にあるアイデアは、RBM(restricted Boltzmann machine、制限付きボルツマンマシン)またはAE(autoencoder、自己符号化器)で構成された自己協調的ニューラルネットワークの教師なし訓練中に得られた重みを用いて、隠れ層のニューロンの重みを動かすことでした。これらの積み重ねRBM( stacked RBM、SRBM)及び積み重ねAE(stacked AE、SAE)は、大量のラベルなしデータで訓練されます。そのような訓練の目的は、隠された構造(表現、イメージ)とデータの関係を強調することです。事前訓練中に得られたMLPの重みによるニューロンの初期化によって、MLPは最適解に非常に近い解空間に置かれます。これにより、MLPの次の微調整(訓練)中にラベル付きデータとエポックの数を減らすことができます。これらは、特に多くのデータを処理する場合に、多くの応用分野で非常に重要な利点です。
手法2:Yoshua Bengio氏が率いる別のグループの科学者たちは、隠れニューロンの初期化の特定な方法、活性化の特定の機能、安定化及び訓練の方法を作成しました。この方向の成功は、深層畳み込みニューラルネットワーク(deep convolutional neural network、DCNN)と再帰型ニューラルネットワーク(recurrent neural network、RNN)の広範な発展と結びついています。このようなニューラルネットワークは、画像認識、分析、及びテキストの分類や話し言葉の言語間翻訳において高い効率を示しました。これらのニューラルネットワークのために開発されたアイデアや手法もMLPに使用され始めました。
両方の手法が現在、積極的に使用されています。事前訓練を伴うニューラルネットワークは、より少ない計算リソースと訓練サンプルでほとんど同じ結果を得られることにご注目ください。これは重要な利点です。私は個人的に事前訓練を伴うディープニューラルネットワークを好み、未来が教師なし学習に属すると信じています。
DNNの開発と使用を可能にするRパッケージ
Rには、DNNを作成して使用するための多数の異なるレベルの複雑さと機能セットを持つパッケージがあります。
事前訓練を伴ってDNNを作成、訓練、テストすることができるパッケージは以下のとおりです。
- deepnet は、多くの設定やパラメータを持たないシンプルなパッケージです。事前訓練とSRBMを備え、SAEニューラルネットワークの両方を作成できます。 前回の記事では、このパッケージを使ってエキスパートを実際に実装することを検討しました。事前訓練にRBMを使用すると、結果の安定性が低下します。このパッケージは、このテーマと初めて遭遇しそのようなネットワークの特質について学ぶのに適しています。正しい手法を使えば、エキスパートでも使用できます。RcppDLは、このパッケージをС++で少し短くしたものです。
- darch v.0.12は、多くのパラメータと設定を持つ、複雑で柔軟なパッケージです。推奨設定がデフォルトに設定されています。このパッケージを使用すると、あらゆる複雑さと構成を持つニューラルネットワークを作成して設定できます。事前訓練にSRBMを使用します。このパッケージは上級ユーザー向けです。その機能については後で詳しく説明します。
事前訓練を伴わずにDNNを作成、訓練、テストすることができるパッケージは以下のとおりです。
- H2Oは多量(1M行以上、1K列以上)のデータを処理するためのパッケージで、発展した正則化システムを持つディープニューラルネットワークが使われています。その能力は私たちの分野にとっては過度ですが、これは不使用の理由になるべきではありません。
- mxnetではMLPだけでなく複雑な再帰ネットワークや複雑なLSTMニューラルネットワークを作成することができます。このパッケージには、RやPythonを含む複数の言語のためのAPIがあります。パッケージの視点は上記のものとは異なります。これは、開発者がパッケージを主にPython用に書いたからです。Rのmxnetパッケージは軽量化され、Pythonパッケージよりも機能が少なくなっていますが、これによってこのパッケージが劣るわけではありません。
深層かつ再帰的なネットワークのテーマは、Python環境で十分に発達しています。このRにはないタイプのニューラルネットワークを構築するためには多くの興味深いパッケージがあります。下記はPythonで書かれたプログラムやモジュールを実行できるようにするRパッケージです。
- PythonInRとreticulateは、PythonコードをRで実行できる2つのパッケージです。使用するには、お使いのコンピューターにPython 2/3が必要です。
- kerasrは、人気高いkeras深層学習ライブラリのRインタフェースです。
- tensorflowは、R環境で完全なTensorFow APIへのアクセスを提供するパッケージです。
Microsoftは最近cntk v.2.1(Computatinal Network Toolkit) GitHubライブラリを発表しました。これは、同等のKerasのバックエンドとして使用できます。ここでの問題をテストすることをお勧めします。
Yandexも負けていません。その独自のCatBoostライブラリはオープンソースで入手できます。このライブラリは、異なるタイプのデータを持つモデルの学習に使用できます。これには、雲の種類や商品の種類など、数字として表現するのが困難なデータも含まれます。ソースコード、ドキュメント、ベンチマークと必要なツールはApache 2.0ライセンスでGitHub に公開されています。これらはニューラルネットワークではなくブースティングツリーであるという事実にもかかわらず、特にRのAPIが含まれているので、アルゴリズムをテストすることをお勧めします。
1. パッケージ機能の簡単な説明
darchパッケージバージョン0.12.0は幅広い機能を提供し、モデルを作成して訓練するだけでなく、必要性や好みに合わせて調整することができます。前回の記事で考察されたパッケージのバージョン (0.10.0) からは大きく変更され、新しい活性化機能、初期化機能、安定化機能が追加されました。もっとも注目すべき点は、すべてがdarch()という同時にコンストラクタでもある1つの関数にまとめられたことです。グラフィックカードもサポートされています。この関数は訓練後にDArchクラスオブジェクトを返します。オブジェクトの構造を図1に示します。predict()関数とdarchTest()関数は、新しいデータの予測や分類メトリックを返します。

図1 DArchオブジェクトの構造
すべてのパラメータにはデフォルト値がありますが、これらの値は通常最適ではありません。これらのパラメータはすべて、一般、RBM、NNの3つのグループに分けることができます。このうちいくつかは後ほど詳しく考察します。
| 関数 | 型 |
|---|---|
| 初期化関数 |
|
| 活性化関数 |
|
| 訓練関数 |
|
| 訓練レベル |
|
| 安定化関数 |
|
| モメンタム |
|
| 停止条件 |
|
ディープニューラルネットワークは、自動結合ネットワーク(SRBM)に接続されたn RBM(n =層-1)と、複数の層を持つ実際のニューラルネットワークMLPで構成されています。RBMの層単位の訓練は、ラベルなしデータに対する教師なし訓練です。ニューラルネットワークの微調整には監視が必要であり、ラベル付きデータに対して実行されます。
これらの段階の訓練をパラメータで分割することで、異なる量のデータ(異なる構造ではありません)を使用して、1つの事前訓練に基づいていくつかの微調整モデルを得る機会が得られます。事前訓練のデータと微調整のデータが同じ場合、訓練は2段階に分かれずに一度に実行できます。事前訓練は省略できます(rbm.numEpochs = 0; darch.numEpochs = 10)、その場合、多層ニューラルネットワークのみを使用するか、RBMのみを使用することができます (rbm.numEpochs = 10; darch.numEpochs = 0)。すべての内部パラメータには引き続きアクセスできます。
訓練されたニューラルネットワークは、さらに必要な回数だけ新しいデータで訓練することができます。これは限られた数のモデルでのみ可能です。複合制限ボルツマン装置(DNRBM)で初期化したディープニューラルネットワークの構造図を図2に示します。
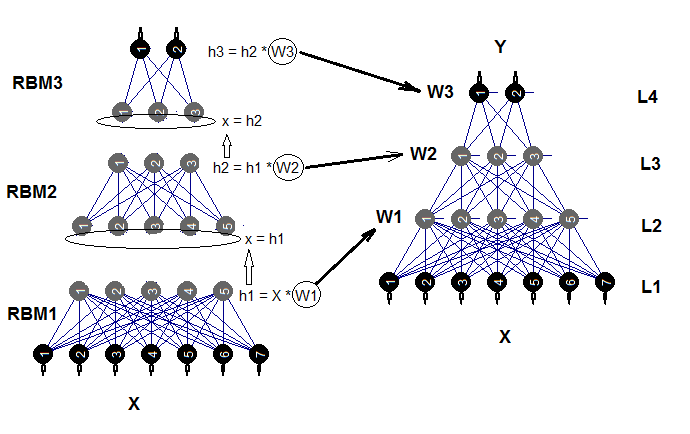
図2 DNSRBMの構造図
1.1. ニューロン初期化関数
パッケージには2つの主要なニューロン初期化関数があります。
- generateWeightsUniform()はrunif(n, min, max)関数を使い下記のように実装されます。
> generateWeightsUniform function (numUnits1, numUnits2, weights.min = getParameter(".weights.min", -0.1, ...), weights.max = getParameter(".weights.max", 0.1, ...), ...) { matrix(runif(numUnits1 * numUnits2, weights.min, weights.max), nrow = numUnits1, ncol = numUnits2) } <environment: namespace:darch>
numUnits1は前の層のニューロン数でnumUnits2は現在の層のニューロン数です。
- generateWeightsNormal() はrnorm(n, mean, sd)関数を使いパッケージで下記のように実装されています。
> generateWeightsNormal function (numUnits1, numUnits2, weights.mean = getParameter(".weights.mean", 0, ...), weights.sd = getParameter(".weights.sd", 0.01, ...), ...) { matrix(rnorm(numUnits1 * numUnits2, weights.mean, weights.sd), nrow = numUnits1, ncol = numUnits2) } <environment: namespace:darch>
他の4つの関数はこれらの2つの関数を使用していますが、特定の関数でmin、max、mean及びsdを定義しています。端末に括弧を付けずに関数名を入力すると、それらを調べることができます。
1.2. ニューロン活性化関数
このパッケージは標準的なものと並んで、新しい活性化関数を幅広く提案します。下記はそれらのいくつかです。
x <- seq(-5, 5, 0.1) par(mfrow = c(2,3)) plot(x, y = 1/(1 + exp(-x)), t = "l", main = "sigmoid") abline(v = 0, col = 2) plot(x, y = tanh(x), t = "l", main = "tanh") abline(v = 0, h = 0, col = 2) plot(x, y = log(1 + exp(x)), t = "l", main = "softplus"); abline(v = 0, col = 2) plot(x, y = ifelse(x > 0, x ,exp(x) - 1), t = "l", main = "ELU") abline(h = 0, v = 0, col = 2) plot(x, y = ifelse(x > 0, x , 0), t = "l", main = "ReLU") abline(h = 0, v = 0, col = 2) par(mfrow = c(1,1))
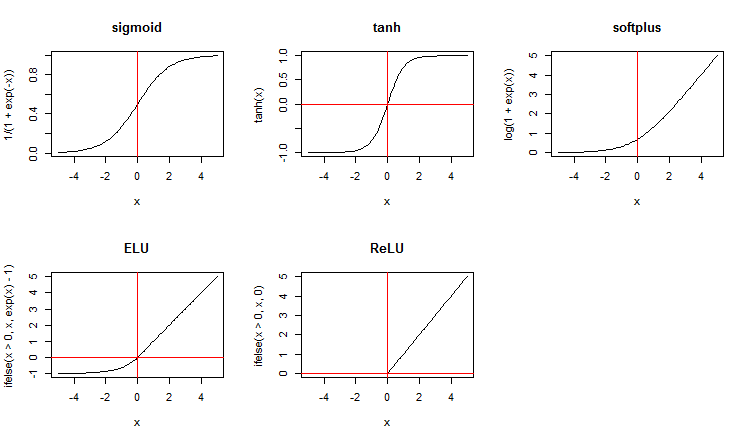
図3 ニューロン活性化関数
maxout活性化関数を別に考えてみましょう。この関数は複雑なネットワークに由来します。ニューラルネットワークの隠れ層は、poolSizeのサイズでモジュールに分割されます。隠れ層のニューロンの数は、プールのサイズで割り切れる必要があります。訓練のためには、最大活性化を有するニューロンがプールから選択され、入力に送られます。プール中のニューロンの活性化関数は別々に設定されます。簡単な言葉で言えば、これはろ過工程における能力が限定された二重層(回旋+最大プール)です。さまざまな出版物によればドロップアウトと一緒に良い結果が得られます。図4は8つのニューロンと2つのサイズのプールを持つ隠れ層を模式的に示しています。
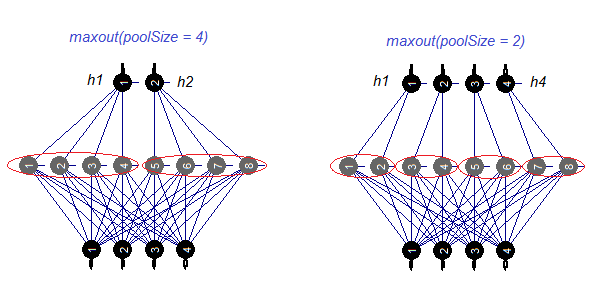
図4 maxout活性化関数
1.3. 訓練法
残念ながら、パッケージ内には、逆伝播と逆伝播中の重み更新を伴わない基本バージョンと伴う改良バージョンのbackpropagationとrpropの2つの訓練方法しかありません。また、bp.learnRateScale.multiplierの助けを借りて訓練レベルを変更する可能性もあります。
1.4. 制御と安定化の方法
- dropoutは学習中の隠れ層のニューロンの一部のドロップアウト(重みをゼロにする)です。ニューロンはランダムな順序でゼロ化します。ドロップアウトするニューロンの相対的な数は、darch.dropoutパラメータで定義します。各隠れ層のドロップアウトのレベルは異なる場合があります。ドロップアウトマスクは、各バッチまたは各エポックごとに生成することができます。
- dropconnectは、現在の層のニューロンの一部と前の層のニューロンとの間の接続を切断します。接続はランダムな順序で切断されます。切断される接続の相対的な数は、同じくdarch.dropoutパラメータ(通常0.5以下)で定義されます。いくつかの出版物によると、dropconnectはdropoutより良い結果を示します。
- ditherは入力データのディザリングによる再訓練を防ぐ方法です。
- weightDecayでは、更新前に各ニューロンの重みに(1 - weightDecay) を乗算します。
- normalizeWeightsは、上からの可能な制限でニューロン重みの入力ベクトルを正規化する方法です(L2ノルム)。
最初の3つのメソッドは別々にしか使用されません。
1.5. RBM訓練メソッドとパラメータ
SRBMを訓練するには2つの方法があります。 RBMはrbm.numEpochsの間に一度に1つずつ訓練されるか、または各RBMは一度に1エポックずつ訓練されます。rbm.consecutive: パラメータによってこれらのメソッドの1つが選ばれます。デフォルトのTRUEが最初のメソッドで、FALSEが2番目のメソッドです。図5は、2つのバリアントの訓練スキームを示します。rbm.lastLayerは、事前訓練を停止すべきSRBMの層を指定するために使用できます。0の場合はすべての層を訓練し、-1の場合は上位層を訓練しないままにします。上層は別に訓練しなければならず、時間がかかるので、これには意味があります。他のパラメータは追加の説明を必要としません。
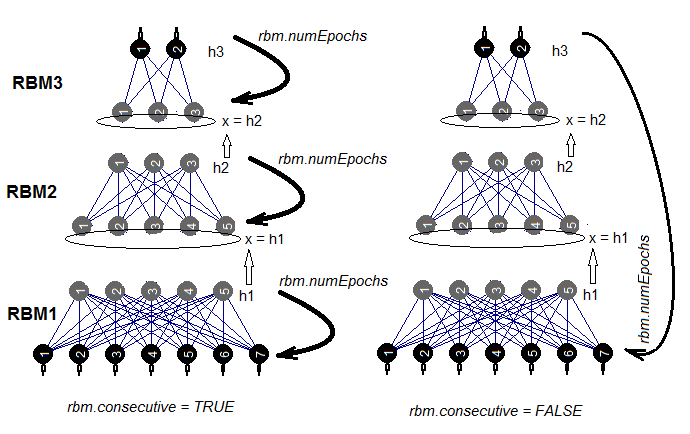
図5 SRBMの2つの訓練方法
1.6. DNN訓練メソッドとパラメータ
DNNは、事前訓練と訓練の2つの方法で訓練することができます。これらの方法で使用されるパラメータはまったく異なります。例えば、事前訓練を伴う訓練では、初期化と正規化の具体的な方法を使用することには意味がありません。実際、これらの方法を使用すると結果が悪化する可能性があります。その背後にある理由は、事前訓練の後、隠れ層のニューロンの重みが最適値に近い領域に配置され、微調整だけが必要になるということです。事前訓練なしで同じ結果を達成するには、使用可能なすべての初期化と正則化の方法を使用する必要があります。通常、この方法でニューラルネットワークを訓練するには、より長い時間がかかります。
そこで、ここでは事前訓練を伴う訓練に焦点を当てます。通常、それには2つの段階があります。
- SRBMを大量のラベルなしデータセットで訓練します。事前訓練パラメータは別々に設定されます。その結果、SRBMの重みによってニューラルネットワークが動きます。次いで、ニューラルネットワークの上位層は、独自の訓練パラメータを用いてラベル付きデータで訓練されます。このようにして、上位層が訓練され、下位層の重み付けによって開始されたニューラルネットワークが得られるので、将来的な使用のために独立したオブジェクトとして保存します。
- 第2段階では、いくつかのラベル付きサンプル、下位の訓練、及び少数の訓練エポックをニューラルネットワークの全層で使用します。これはネットワークの微調整です。ニューラルネットワークはこれで訓練されています。
事前訓練、微調整、さらなる訓練と段階を分ける可能性は、1つのDNNだけでなくDNNコミッティを訓練するための訓練アルゴリズムを作成する際に驚異的な柔軟性をもたらします。図6は、DNNとDNNコミッティ訓練のいくつかのバリアントを表しています。
- バリアントа:微調整中にnエポックごとにDNNを保存します。このようにして、私たちは訓練の異なるDNNを持つことになります。これらのニューラルネットワークの各々は、後に別個にまたはコミッティの一部として使用することができます。このシナリオの欠点は、すべてのDNNが同じデータで訓練されており、同じ訓練パラメータを持っていることです。
- バリアントb:さまざまなデータセット(スライディングウィンドウ、拡張ウィンドウなど)とさまざまなパラメータを使用して、並行して開始されたDNNを微調整します。その結果、バリアントаよりも相関の低い予想が得られます。
- バリアントcは異なるデータセットと異なるパラメータを使用して、起動されたDNNを順番に微調整します。中間モデルを保存します。これが、先に「さらなる訓練」として言及したものです。これは、新しいデータが十分にあるたびに実行できます。
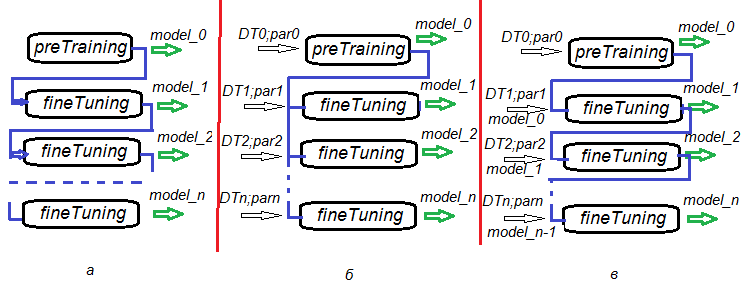
図6DNN訓練のバリアント
2. 使用されるパラメータに応じたDNN作業品質のテスト
2.1. 実験
2.1.1. 入力データ(準備)
ここでは記事の前の部分(1、2、3)のデータと関数を使用します。そこでは、予備データ作成のさまざまなバリあんとについて詳細に検討しました。ここで実行しようとしている予備準備の段階について簡単に触れます。OHLCVは、以前と同じく、初期データです。入力データはデジタルフィルタで、出力データはジグザグです。関数とCotir.RDataの作業空間の画像を使用することができます。
実行するデータを準備する段階は、別々の関数でまとめられます。
- PrepareData() — 初期データセットを作成し、それをNAから消去します。
- SplitData() — 初期データセットを事前訓練、訓練、検証、テストのサブセットに分割します。
- CappingData() — すべての部分集合の外れ値を特定して補完します。
記事のスペースを節約するために、これらの関数をここで一覧するつもりはありませんが、これらは以前の記事で詳細に考察されているので、GitHubからダウンロードすることができます。後で結果を調べる予定です。予備処理でのデータ変換のすべての方法について議論するつもりはありません。それらの多くはよく知られており広く使われています。ここでは離散化(教師付きと教師なし)というあまり知られていない方法を使用します。2番目の記事では、2つの教師付き離散化パッケージ(discretizationと smbinning)を検討 しました。これらには、異なる離散化アルゴリズムが含まれます。
ここでは、連続変数をビンに分割するさまざまな方法と、これらの離散変数をモデルで使用する方法を検討します。
ビニングとは何か
ビニングとはスコアリングモデリングで使用される用語で、機械学習においては離散化として知られています。これは、連続変数を有限数の間隔(ビン)に変換する過程で、バイナリ目標変数との関係とその分布を理解するのに役立ちます。この過程で作成されたビンは、モデルで使用するための予測特性の特徴になります。
ビニングを使う理由
ビニングには、いくつかの欠点もありますが、大きな利点があります。
- モデルに欠損データ(NA)や他の特定の計算(ゼロで割るなど)を含めることが可能
- 外れ値がモデルに及ぼす影響を制御または緩和
- 予測変数の異なるスケールによる問題を解決し、重み付け係数を最終モデルに匹敵する
教師なしの離散化
教師なしの離散化は、他の情報を考慮せずに連続関数をビンに分割します。分割には2つの選択肢があります。長さの同じビンと頻度の同じビンです。
| 選択肢 |
目標 |
例 |
欠点 |
|---|---|---|---|
| 長さの同じビン |
変数の分布を理解する |
異なるルール(sturges、riceets)を使用して計算することができる、同じ長さのバンカーを用いた古典的ヒストグラム |
バンカー内のレコード数が小さすぎて正しい計算ができない |
| 同じ頻度のビン |
不良率などの指標を使用してバイナリ目標変数との関係を分析する |
四分位数または百分位数 | 選択されたカットオフポイントは、目標変数を確認する際にビンの差を最大にすることはできない |
教師付き離散化
教師付き離散化は、連続変数を目標変数に投影されるビンに分割します。ここで重要なアイデアは、グループ間の相違を最大にするようなカットオフポイントを見つけることです。
分析者は、ChiMergeやRecursive Partitioningなどのアルゴリズムを使用して、数秒で最適なポイントを素早く見つけ、証拠の重み(WoE)や情報の価値(IV)などの指標を使用して目標変数との関係を評価することができます。
WoEは、教師あり学習のアルゴリズムの前処理の段階で予測変数を変換するための手段として使用できます。予測変数の離散化の間には、それらを新しい名義変数またはそれらのWoEの値で置き換えることができます。2番目のバリアントは、名義変数(因子)をダミー変数に変換することを可能にするので興味深いものです。これにより、分類の品質が大幅に向上します。
WOEとIVは、データ分析において2つの異なる役割を果たします。
- WOEは、予測変数と2進目標変数の関係を記述し、
- IVはこれらの関係の強さを測定します。
図や式を使用してWOEとIVの正体を見てみましょう。本稿の2番目の部分にある、10の平等な領域に分割されたv.fatl変数のグラフを思い出してください。
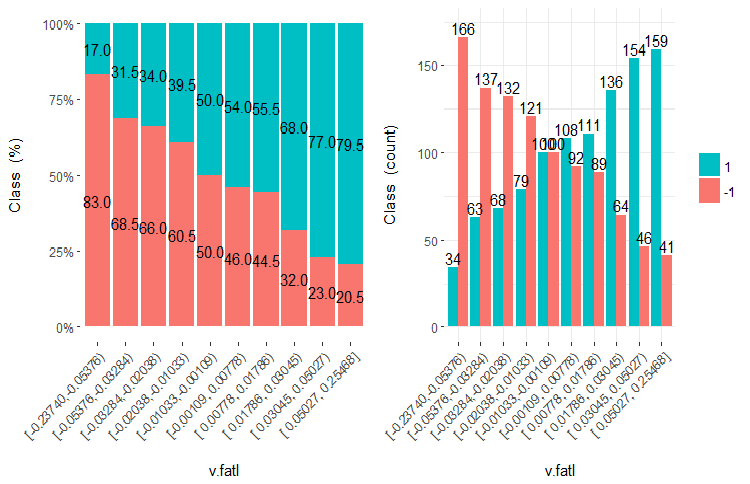
図7 10の平等な領域に分割されたv.fatl変数
データの予測能力 (WOE)
ご覧のように、各ビンには、クラス "1"とクラス "-1"に入るサンプルがあります。WoEiビンの予測能力は式
WoEi = ln(Gi/Bi)*100
で計算されます。ここで
Gi — 変数の各ビン内の「良い」(ここでは "良い" = "1")サンプルの相対頻度
Bi — 変数の各ビン内の「悪い」(ここでは "悪い" = "-1")サンプルの相対頻度
WoEi = 1の場合、このビンの「良い」サンプルと「悪い」サンプルの数がおおよそ同じで、このビンの予測能力は0です。「良い」サンプルが「悪い」サンプルよりも多い場合、WOE> 0で、この逆も同様です。
情報価値 (IV)
これは、変数の有意性を特定し、「良い」及び「悪い」サンプルの分布の差を測定する最も一般的な尺度です。情報価値は次の公式で計算できます。
IV = ∑ (Gi – Bi) ln (Gi/Bi)
変数の情報値は、変数のすべてのビンの合計と等しくなります。この係数の値は次のように解釈できます。
- 0.02以下 - 統計的に有意でない変数
- 0,02 – 0,1 — 統計的に弱い変数
- 0,1 – 0,3 — 統計的に有意な変数
- 0,3 and above — 統計的に強い変数
次に、さまざまなアルゴリズムと最適化基準を使用してビンをマージ/分割し、これらのビンの差を可能な限り大きくします。たとえば、smbinningパッケージは、最適なカットオフポイントを計算するために数値と情報値を分類するために再帰的パーティショニングを使用します。discretizationパッケージは、この問題をChiMergeとMDLで解決します。カットオフポイントは訓練セットで取得され、検証とテストセットの分割に使用されることを覚えておくべきです。
数値変数を何らかの方法で離散化するパッケージには discretization、smbinning、Information、InformationValue 、woebinningといくつかあります。テストデータセットを慎重に作成し、この情報を使用して検証セットとテストセットを分割することが必要です。また、結果を視覚的に管理したいと思います。これらの要件を満たすために、私はwoebinningパッケージを選択しました。
このパッケージは自動的に数値と要素を分割してバイナリ目標変数にバインドします。ここでは、2つの手法が用意されています。
- 細かい分類と粗い分類を実装して、顆粒化されたクラスとレベルを逐次結合する
- ツリーような手法で、バイナリ分割を介して反復初期ビンをセグメント化する
両方の手順は、WOEの類似の値に基づいて分割されたビンをマージし、IV基準に基づいて停止します。パッケージは、スタンドアロン変数またはデータフレーム全体で使用できます。これにより、ビニングのためのさまざまなソリューションを検討し、新しいデータを拡大するための柔軟なツールが提供されます。
計算をしましょう。作業環境にロードされた端末(またはGitHubのCotir.RData作業環境のイメージ)からのクオーツがすでにあります。下記は計算と結果のシーケンスです。
- PrepareData() — 初期データセットdt [7906,14]を作成してNAから削除します。このセットには、一時ラベルData、入力変数(12) 、目標変数Class(2つのレベル「-1」及び「+1」を含む)が含まれます。
- SplitData() — 初期データセットdt []を2000/1000/500/500の割合でpretrain、train、val、testのサブセットに分割し、それらをデータフレームDT [4,4000,14]にまとめます。
- CappingData() — すべての部分集合の外れ値を特定して帰結し、DTcap [4、4000、14]の集合を得ます。離散化は外れ値に対して寛容であるという事実にもかかわらず、それらは補完されます。この段階なしでの実験が可能です。覚えていらっしゃるかもしれませんが、外れ値のパラメータ (pre.outl)は事前訓練サブセットで定義されています。これらのパラメータを使用して訓練/テスト/検証セットを処理します。
- NormData() — caretパッケージのspatialSingメソッドを使ってセットを正規化します。外れ値の補完と同様に、正規化のパラメータ(preproc)は事前訓練サブセットで定義されます。サンプル訓練/テスト/検証は、これらのパラメータを使用して処理されます。結果としてDTcap.n[4, 4000, 14] が得られます。
- DiscretizeData() — WOEとIVの観点から、離散化のパラメータ(preCut)、変数とそのビンの品質を定義します。
evalq({
dt <- PrepareData(Data, Open, High, Low, Close, Volume)
DT <- SplitData(dt, 2000, 1000, 500,500)
pre.outl <- PreOutlier(DT$pretrain)
DTcap <- CappingData(DT, impute = T, fill = T, dither = F,
pre.outl = pre.outl)
preproc <- PreNorm(DTcap, meth = meth)
DTcap.n <- NormData(DTcap, preproc = preproc)
preCut <- PreDiscret(DTcap.n)
}, env)
すべての変数の離散化データを表にして見てみましょう。
evalq(tabulate.binning <- woe.binning.table(preCut), env) > env$tabulate.binning $`WOE Table for v.fatl` Final.Bin Total.Count Total.Distr. 0.Count 1.Count 0.Distr. 1.Distr. 1.Rate WOE IV 1 <= -0.3904381926 154 7.7% 130 24 13.2% 2.4% 15.6% 171.3 0.185 2 <= -0.03713814085 769 38.5% 498 271 50.4% 26.8% 35.2% 63.2 0.149 3 <= 0.1130198981 308 15.4% 141 167 14.3% 16.5% 54.2% -14.5 0.003 4 <= Inf 769 38.5% 219 550 22.2% 54.3% 71.5% -89.7 0.289 6 Total 2000 100.0% 988 1012 100.0% 100.0% 50.6% NA 0.626 $`WOE Table for ftlm` Final.Bin Total.Count Total.Distr. 0.Count 1.Count 0.Distr. 1.Distr. 1.Rate WOE IV 1 <= -0.2344708291 462 23.1% 333 129 33.7% 12.7% 27.9% 97.2 0.204 2 <= -0.01368798447 461 23.1% 268 193 27.1% 19.1% 41.9% 35.2 0.028 3 <= 0.1789073635 461 23.1% 210 251 21.3% 24.8% 54.4% -15.4 0.005 4 <= Inf 616 30.8% 177 439 17.9% 43.4% 71.3% -88.4 0.225 6 Total 2000 100.0% 988 1012 100.0% 100.0% 50.6% NA 0.463 $`WOE Table for rbci` Final.Bin Total.Count Total.Distr. 0.Count 1.Count 0.Distr. 1.Distr. 1.Rate WOE IV 1 <= -0.1718377948 616 30.8% 421 195 42.6% 19.3% 31.7% 79.4 0.185 2 <= -0.09060410462 153 7.6% 86 67 8.7% 6.6% 43.8% 27.4 0.006 3 <= 0.3208178176 923 46.2% 391 532 39.6% 52.6% 57.6% -28.4 0.037 4 <= Inf 308 15.4% 90 218 9.1% 21.5% 70.8% -86.1 0.107 6 Total 2000 100.0% 988 1012 100.0% 100.0% 50.6% NA 0.335 $`WOE Table for v.rbci` Final.Bin Total.Count Total.Distr. 0.Count 1.Count 0.Distr. 1.Distr. 1.Rate WOE IV 1 <= -0.1837437563 616 30.8% 406 210 41.1% 20.8% 34.1% 68.3 0.139 2 <= 0.03581374495 461 23.1% 253 208 25.6% 20.6% 45.1% 22.0 0.011 3 <= 0.2503922644 461 23.1% 194 267 19.6% 26.4% 57.9% -29.5 0.020 4 <= Inf 462 23.1% 135 327 13.7% 32.3% 70.8% -86.1 0.161 6 Total 2000 100.0% 988 1012 100.0% 100.0% 50.6% NA 0.331 $`WOE Table for v.satl` Final.Bin Total.Count Total.Distr. 0.Count 1.Count 0.Distr. 1.Distr. 1.Rate WOE IV 1 <= -0.01840058612 923 46.2% 585 338 59.2% 33.4% 36.6% 57.3 0.148 2 <= 0.3247097195 769 38.5% 316 453 32.0% 44.8% 58.9% -33.6 0.043 3 <= 0.4003869443 154 7.7% 32 122 3.2% 12.1% 79.2% -131.4 0.116 4 <= Inf 154 7.7% 55 99 5.6% 9.8% 64.3% -56.4 0.024 6 Total 2000 100.0% 988 1012 100.0% 100.0% 50.6% NA 0.330 $`WOE Table for v.stlm` Final.Bin Total.Count Total.Distr. 0.Count 1.Count 0.Distr. 1.Distr. 1.Rate WOE IV 1 <= -0.4030051922 154 7.7% 118 36 11.9% 3.6% 23.4% 121.1 0.102 2 <= -0.1867821117 462 23.1% 282 180 28.5% 17.8% 39.0% 47.3 0.051 3 <= 0.1141896118 615 30.8% 301 314 30.5% 31.0% 51.1% -1.8 0.000 4 <= Inf 769 38.5% 287 482 29.0% 47.6% 62.7% -49.4 0.092 6 Total 2000 100.0% 988 1012 100.0% 100.0% 50.6% NA 0.244 $`WOE Table for pcci` Final.Bin Total.Count Total.Distr. 0.Count 1.Count 0.Distr. 1.Distr. 1.Rate WOE IV 1 <= -0.1738420887 616 30.8% 397 219 40.2% 21.6% 35.6% 61.9 0.115 2 <= -0.03163945242 307 15.3% 165 142 16.7% 14.0% 46.3% 17.4 0.005 3 <= 0.2553612644 615 30.8% 270 345 27.3% 34.1% 56.1% -22.1 0.015 4 <= Inf 462 23.1% 156 306 15.8% 30.2% 66.2% -65.0 0.094 6 Total 2000 100.0% 988 1012 100.0% 100.0% 50.6% NA 0.228 $`WOE Table for v.ftlm` Final.Bin Total.Count Total.Distr. 0.Count 1.Count 0.Distr. 1.Distr. 1.Rate WOE IV 1 <= -0.03697698898 923 46.2% 555 368 56.2% 36.4% 39.9% 43.5 0.086 2 <= 0.2437475615 615 30.8% 279 336 28.2% 33.2% 54.6% -16.2 0.008 3 <= Inf 462 23.1% 154 308 15.6% 30.4% 66.7% -66.9 0.099 5 Total 2000 100.0% 988 1012 100.0% 100.0% 50.6% NA 0.194 $`WOE Table for v.rftl` Final.Bin Total.Count Total.Distr. 0.Count 1.Count 0.Distr. 1.Distr. 1.Rate WOE IV 1 <= -0.1578370554 616 30.8% 372 244 37.7% 24.1% 39.6% 44.6 0.060 2 <= 0.1880959621 768 38.4% 384 384 38.9% 37.9% 50.0% 2.4 0.000 3 <= 0.3289762494 308 15.4% 129 179 13.1% 17.7% 58.1% -30.4 0.014 4 <= Inf 308 15.4% 103 205 10.4% 20.3% 66.6% -66.4 0.065 6 Total 2000 100.0% 988 1012 100.0% 100.0% 50.6% NA 0.140 $`WOE Table for stlm` Final.Bin Total.Count Total.Distr. 0.Count 1.Count 0.Distr. 1.Distr. 1.Rate WOE IV 1 <= -0.4586732186 154 7.7% 60 94 6.1% 9.3% 61.0% -42.5 0.014 2 <= -0.1688696056 462 23.1% 266 196 26.9% 19.4% 42.4% 32.9 0.025 3 <= 0.2631157075 922 46.1% 440 482 44.5% 47.6% 52.3% -6.7 0.002 4 <= 0.3592235072 154 7.7% 97 57 9.8% 5.6% 37.0% 55.6 0.023 5 <= 0.4846279843 154 7.7% 81 73 8.2% 7.2% 47.4% 12.8 0.001 6 <= Inf 154 7.7% 44 110 4.5% 10.9% 71.4% -89.2 0.057 8 Total 2000 100.0% 988 1012 100.0% 100.0% 50.6% NA 0.122 $`WOE Table for v.rstl` Final.Bin Total.Count Total.Distr. 0.Count 1.Count 0.Distr. 1.Distr. 1.Rate WOE IV 1 <= -0.4541701981 154 7.7% 94 60 9.5% 5.9% 39.0% 47.3 0.017 2 <= -0.3526306487 154 7.7% 62 92 6.3% 9.1% 59.7% -37.1 0.010 3 <= -0.2496412214 154 7.7% 53 101 5.4% 10.0% 65.6% -62.1 0.029 4 <= -0.08554320418 307 15.3% 142 165 14.4% 16.3% 53.7% -12.6 0.002 5 <= 0.360854678 923 46.2% 491 432 49.7% 42.7% 46.8% 15.2 0.011 6 <= Inf 308 15.4% 146 162 14.8% 16.0% 52.6% -8.0 0.001 8 Total 2000 100.0% 988 1012 100.0% 100.0% 50.6% NA 0.070 $`WOE Table for v.pcci` Final.Bin Total.Count Total.Distr. 0.Count 1.Count 0.Distr. 1.Distr. 1.Rate WOE IV 1 <= -0.4410911486 154 7.7% 92 62 9.3% 6.1% 40.3% 41.9 0.013 2 <= -0.03637567714 769 38.5% 400 369 40.5% 36.5% 48.0% 10.5 0.004 3 <= 0.1801156117 461 23.1% 206 255 20.9% 25.2% 55.3% -18.9 0.008 4 <= 0.2480148615 154 7.7% 84 70 8.5% 6.9% 45.5% 20.6 0.003 5 <= 0.3348752487 154 7.7% 67 87 6.8% 8.6% 56.5% -23.7 0.004 6 <= 0.4397404288 154 7.7% 76 78 7.7% 7.7% 50.6% -0.2 0.000 7 <= Inf 154 7.7% 63 91 6.4% 9.0% 59.1% -34.4 0.009 9 Total 2000 100.0% 988 1012 100.0% 100.0% 50.6% NA 0.042
表の各変数には次の値があります。
- Final.Bin — ビン境界
- Total.Count — ビン内のサンプルの総数
- Total.Distr — ビン内のサンプルの相対数
- 0.Count — クラス"0"に属するサンプルの数
- 1.Count — クラス"1"に属するサンプルの数
- 0.Distr — — クラス"0"に属するサンプルの相対数
- 1.Distr — クラス"1"に属するサンプルの相対数
- 1.Rate — クラス"0"のサンプル数に対するクラス"1"のサンプルの割合
- WOE — ビンの予測能力
- IV — ビンの統計的重要性
図式表現は、より具体的なものになります。この表に基づいて、IVの昇順にすべての変数のWOEグラフをプロットします。
> evalq(woe.binning.plot(preCut), env)
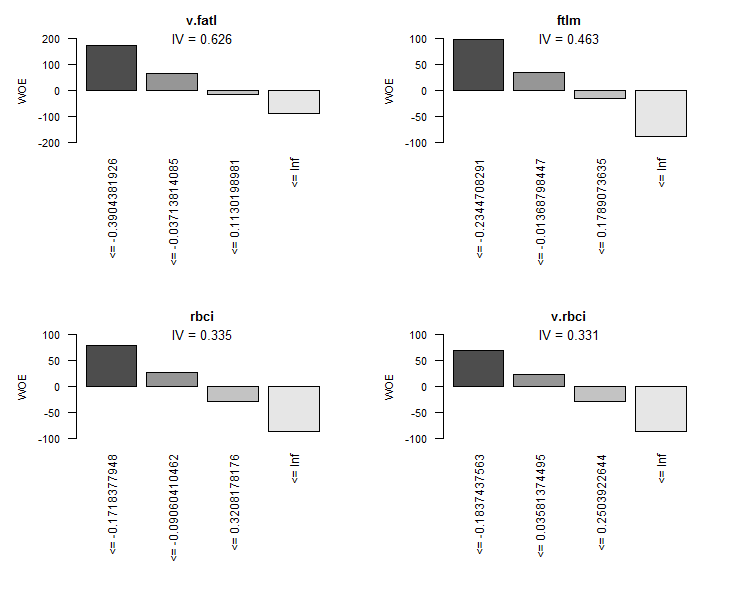
図8 4つの最良変数のWOE
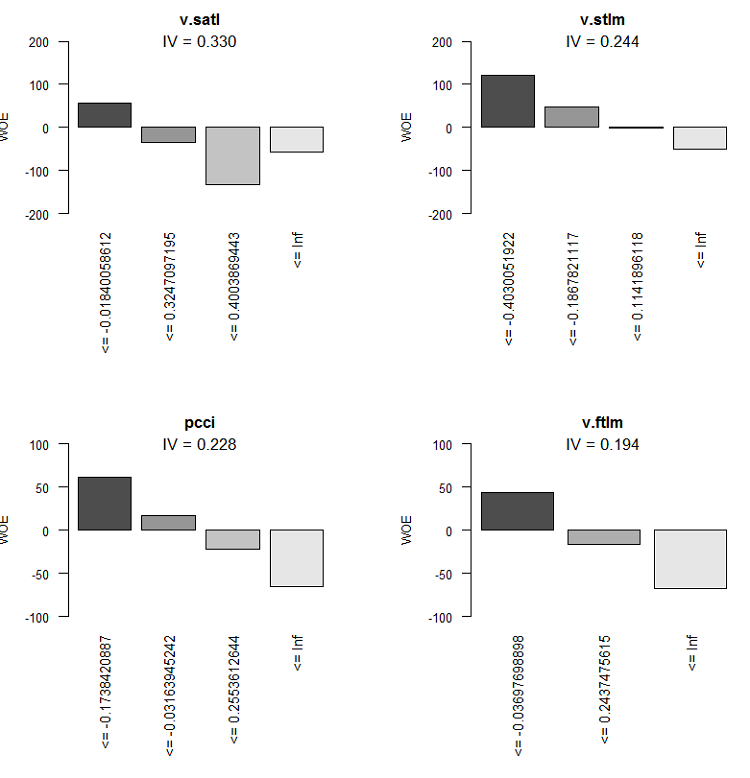
図9 変数5-8のWOE
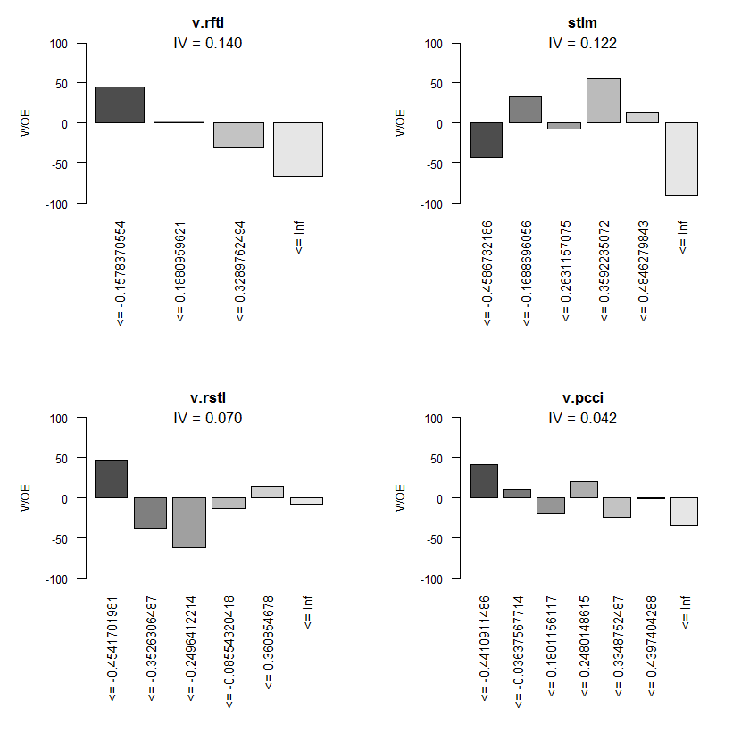
図10 変数9-12のWOE
IVによる変数の総距離のグラフ
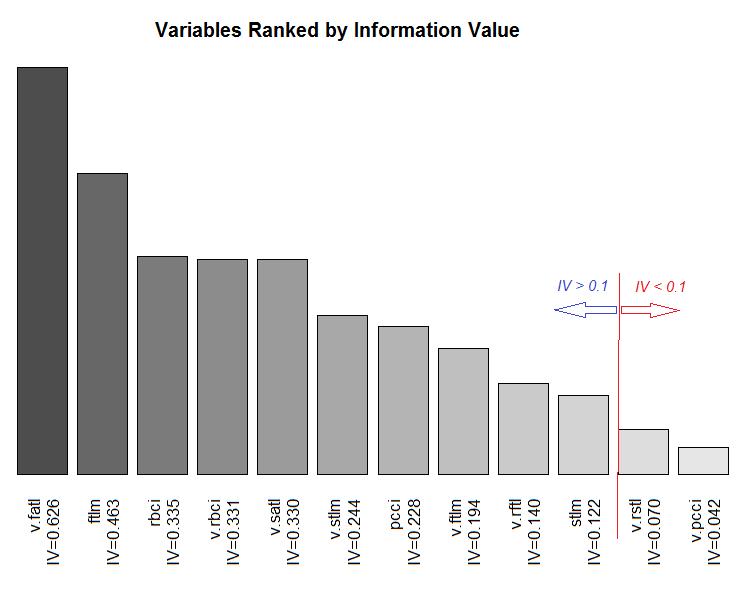
図11 IVによる変数の範囲指定
変数v.rstlとv.pcciはIV < 0.1で重要でないので使用しません。グラフからは、10個の重要変数のうちでv.satlとstlmだけが目標変数と非線形の関係にあることが分かります。他の変数は線形関係にあります。
さらなる実験のために、下記の3つのセットを作成する必要があります。
- DTbinは、連続数値予測変数が分割されるビンの数に等しいレベル数で要素に変換されるデータセットです。
- DTdumは、DTbinデータセットの因子予測変数がダミーバイナリ変数に変換されるデータセットです。
- DTwoeは、因子予測変数がそのレベルをこれらのレベルのWOE値で置き換えることによって数値変数に変換されるデータセットです。
最初のDTbinセットは、基本モデルの訓練と測定値を得るために必要です。2番目のセットと3番目のセットは、DNNの訓練と、これら2つの変換方法の効率の比較に使用されます。
この問題は woebinningパッケージのwoe.binning.deploy()関数によって比較的容易に解決できます。関数には次のデータを渡す必要があります。
- 予測変数と目標変数を持つデータフレーム(目標変数は0または1の値を持つことが必要)
- 前段階で得られた離散化のパラメータ(preCut)
- 分類されなければならない変数の名前。すべての変数を分類する必要がある場合は、データフレームの名前を指定。
- 分類されない変数の最小IVを指定
- 取得したい追加の変数(カテゴリ化されたものを除く)の指定。"woe"と"dum"の2種類。
この関数は、初期変数、カテゴリ化変数、及び追加変数(指定されている場合)を含むデータフレームを返します。新しく作成される変数の名前は、初期変数の名前に対応する接頭辞または接尾辞を追加することによって作成されます。このようにして、すべての追加変数の接頭辞は"dum"または "woe"になり、分類された変数には接尾辞"binned"が付きます。woe.binning.deploy()を使用して初期データセットを変換するDiscretizeData()を書きましょう。
DiscretizeData <- function(X, preCut, var){ require(foreach) require(woeBinning) DTd <- list() foreach(i = 1:length(X)) %do% { X[[i]] %>% select(-Data) %>% targ.int() %>% woe.binning.deploy(preCut, min.iv.total = 0.1, add.woe.or.dum.var = var) -> res return(res) } -> DTd list(pretrain = DTd[[1]] , train = DTd[[2]] , val = DTd[[3]] , test = DTd[[4]] ) -> DTd return(DTd) }
関数の入力パラメータは事前訓練/訓練/検証/テストスロットを持つ初期データ (list X) 、離散化パラメータ preCutと追加変数の型 (string var)です。
関数は各スロットから"Data"変数を削除して目標変数(数値的目標変数"Cl"では "Class"因数)を変更します。それに基づいて、woe.binning.deploy()を関数の入力に送ります。さらに、この関数の入力パラメータで、変数を出力セットに含めるための最小IV = 0.1を指定します。出力には、同じスロット事前訓練/訓練/テスト/検証のリストが表示されます。各スロットには、分類された変数と、必要に応じて、追加の変数が初期変数に追加されます。すべての必要なセットを計算し、それらにDTcap.nセットの生データを追加しましょう。
evalq({
require(dplyr)
require(foreach)
DTbin = DiscretizeData(DTcap.n, preCut = preCut, var = "")
DTwoe = DiscretizeData(DTcap.n, preCut = preCut, var = "woe")
DTdum = DiscretizeData(DTcap.n, preCut = preCut, var = "dum")
X.woe <- list()
X.bin <- list()
X.dum <- list()
foreach(i = 1:length(DTcap.n)) %do% {
DTbin[[i]] %>% select(contains("binned")) -> X.bin[[i]]
DTdum[[i]] %>% select(starts_with("dum")) -> X.dum[[i]]
DTwoe[[i]] %>% select(starts_with("woe")) %>%
divide_by(100) -> X.woe[[i]]
return(list(bin = X.bin[[i]], woe = X.woe[[i]],
dum = X.dum[[i]], raw = DTcap.n[[i]]))
} -> DTcut
list(pretrain = DTcut[[1]],
train = DTcut[[2]],
val = DTcut[[3]],
test = DTcut[[4]] ) -> DTcut
rm(DTwoe, DTdum, X.woe, X.bin, X.dum)
},
env)
WOEはパーセンテージ値なので、WOEを100で除算し、追加の正規化なしにニューラルネットワークの入力に送ることができる変数の値を得ることができます。得られたスロットの構造を見てみましょう。例えばDTcut$valです。
> env$DTcut$val %>% str() List of 4 $ bin:'data.frame': 501 obs. of 10 variables: ..$ v.fatl.binned: Factor w/ 5 levels "(-Inf,-0.3904381926]",..: 1 1 3 2 4 3 4 4 4 4 ... ..$ ftlm.binned : Factor w/ 5 levels "(-Inf,-0.2344708291]",..: 2 1 1 1 2 2 3 4 4 4 ... ..$ rbci.binned : Factor w/ 5 levels "(-Inf,-0.1718377948]",..: 2 1 2 1 2 3 3 3 4 4 ... ..$ v.rbci.binned: Factor w/ 5 levels "(-Inf,-0.1837437563]",..: 1 1 3 2 4 3 4 4 4 4 ... ..$ v.satl.binned: Factor w/ 5 levels "(-Inf,-0.01840058612]",..: 1 1 1 1 1 1 1 1 1 2 ... ..$ v.stlm.binned: Factor w/ 5 levels "(-Inf,-0.4030051922]",..: 2 2 3 2 3 2 3 3 4 4 ... ..$ pcci.binned : Factor w/ 5 levels "(-Inf,-0.1738420887]",..: 1 1 4 2 4 2 4 2 2 3 ... ..$ v.ftlm.binned: Factor w/ 4 levels "(-Inf,-0.03697698898]",..: 1 1 3 2 3 2 3 3 2 2 ... ..$ v.rftl.binned: Factor w/ 5 levels "(-Inf,-0.1578370554]",..: 2 1 1 1 1 1 1 2 2 2 ... ..$ stlm.binned : Factor w/ 7 levels "(-Inf,-0.4586732186]",..: 2 2 2 2 1 1 1 1 1 2 ... $ woe:'data.frame': 501 obs. of 10 variables: ..$ woe.v.fatl.binned: num [1:501] 1.713 1.713 -0.145 0.632 -0.897 ... ..$ woe.ftlm.binned : num [1:501] 0.352 0.972 0.972 0.972 0.352 ... ..$ woe.rbci.binned : num [1:501] 0.274 0.794 0.274 0.794 0.274 ... ..$ woe.v.rbci.binned: num [1:501] 0.683 0.683 -0.295 0.22 -0.861 ... ..$ woe.v.satl.binned: num [1:501] 0.573 0.573 0.573 0.573 0.573 ... ..$ woe.v.stlm.binned: num [1:501] 0.473 0.473 -0.0183 0.473 -0.0183 ... ..$ woe.pcci.binned : num [1:501] 0.619 0.619 -0.65 0.174 -0.65 ... ..$ woe.v.ftlm.binned: num [1:501] 0.435 0.435 -0.669 -0.162 -0.669 ... ..$ woe.v.rftl.binned: num [1:501] 0.024 0.446 0.446 0.446 0.446 ... ..$ woe.stlm.binned : num [1:501] 0.329 0.329 0.329 0.329 -0.425 ... $ dum:'data.frame': 501 obs. of 41 variables: ..$ dum.v.fatl.-Inf.-0.3904381926.binned : num [1:501] 0 0 0 0 0 0 0 0 0 0 ... ..$ dum.v.fatl.-0.03713814085.0.1130198981.binned : num [1:501] 0 0 0 0 0 0 0 0 0 0 ... ..$ dum.v.fatl.-0.3904381926.-0.03713814085.binned: num [1:501] 0 0 0 0 0 0 0 0 0 0 ... ..$ dum.v.fatl.0.1130198981.Inf.binned : num [1:501] 0 0 0 0 0 0 0 0 0 0 ... ..$ dum.ftlm.-0.2344708291.-0.01368798447.binned : num [1:501] 0 0 0 0 0 0 0 0 0 0 ... ..$ dum.ftlm.-Inf.-0.2344708291.binned : num [1:501] 0 0 0 0 0 0 0 0 0 0 ... ..$ dum.ftlm.-0.01368798447.0.1789073635.binned : num [1:501] 0 0 0 0 0 0 0 0 0 0 ... ..$ dum.ftlm.0.1789073635.Inf.binned : num [1:501] 0 0 0 0 0 0 0 0 0 0 ... ....................................................................................... ..$ dum.stlm.-Inf.-0.4586732186.binned : num [1:501] 0 0 0 0 0 0 0 0 0 0 ... ..$ dum.stlm.-0.1688696056.0.2631157075.binned : num [1:501] 0 0 0 0 0 0 0 0 0 0 ... ..$ dum.stlm.0.2631157075.0.3592235072.binned : num [1:501] 0 0 0 0 0 0 0 0 0 0 ... ..$ dum.stlm.0.3592235072.0.4846279843.binned : num [1:501] 0 0 0 0 0 0 0 0 0 0 ... ..$ dum.stlm.0.4846279843.Inf.binned : num [1:501] 0 0 0 0 0 0 0 0 0 0 ... $ raw:'data.frame': 501 obs. of 14 variables: ..$ Data : POSIXct[1:501], format: "2017-02-23 15:30:00" "2017-02-23 15:45:00" ... ..$ ftlm : num [1:501] -0.223 -0.374 -0.262 -0.31 -0.201 ... ..$ stlm : num [1:501] -0.189 -0.257 -0.271 -0.389 -0.473 ... ..$ rbci : num [1:501] -0.0945 -0.1925 -0.1348 -0.1801 -0.1192 ... ..$ pcci : num [1:501] -0.5714 -0.2602 0.4459 -0.0478 0.2596 ... ..$ v.fatl: num [1:501] -0.426 -0.3977 0.0936 -0.1512 0.1178 ... ..$ v.satl: num [1:501] -0.35 -0.392 -0.177 -0.356 -0.316 ... ..$ v.rftl: num [1:501] -0.0547 -0.2065 -0.3253 -0.4185 -0.4589 ... ..$ v.rstl: num [1:501] 0.0153 -0.0273 -0.0636 -0.1281 -0.15 ... ..$ v.ftlm: num [1:501] -0.321 -0.217 0.253 0.101 0.345 ... ..$ v.stlm: num [1:501] -0.288 -0.3 -0.109 -0.219 -0.176 ... ..$ v.rbci: num [1:501] -0.2923 -0.2403 0.1909 0.0116 0.2868 ... ..$ v.pcci: num [1:501] -0.0298 0.3738 0.6153 -0.5643 0.2742 ... ..$ Class : Factor w/ 2 levels "-1","1": 1 1 1 1 2 2 2 2 2 1 ...
ご覧のようにbinスロットには、レベル数の異なる10個の因子変数が含まれており、接尾辞 "binned"を持っています。. woeスロットには、WOEの要因レベルが変更された10個の変数が含まれています(接頭辞は"woe" です)。dumスロットには、1対1のコード変数(接頭辞は "dum")によって得られた値 (0, 1) とともに41個の数値変数dummy が含まれています。raw スロットにはData(タイムスタンプ)、Class — 目標因子変数と12個の数値予測変数である14の変数が含まれています。
さらなる実験に必要なすべてのデータがあります。以下にリストされているオブジェクトは、すでにenv環境にあるはずです。これらのオブジェクトを含む作業領域 PartIV.RDataファイルに保存しましょう。
> ls(env) [1] "Close" "Data" "dt" "DT" "DTbin" "DTcap" "DTcap.n" "DTcut" "High" [10] "i" "Low" "Open" "pre.outl" "preCut" "preproc" "Volume"
2.1.2. 比較の基本モデル
ここでは基本モデルとしてOneRパッケージに実装されているOneRモデルを使います。このモデルは、シンプルで信頼性が高く、解釈しやすいものです。アルゴリズムに関する情報は、パッケージの説明に記載されています。このモデルはビンデータのみで動作しています。このパッケージには、さまざまな方法で数値変数を区別できる補助関数が含まれています。予測変数を要素に変換しているので、必要ではありません。
ここでは以下の計算を詳しく説明します。DTcutから対応するスロットを抽出し、それらにゴール変数Classを追加することによって訓練/検証/テストセットを作成します。モデルを訓練セットで訓練します。
> evalq({
+ require(OneR)
+ require(dplyr)
+ require(magrittr)
+ train <- cbind(DTcut$train$bin, Class = DTcut$train$raw$Class) %>% as.data.frame()
+ val <- cbind(DTcut$val$bin, Class = DTcut$val$raw$Class) %>% as.data.frame()
+ test <- cbind(DTcut$test$bin, Class = DTcut$test$raw$Class) %>% as.data.frame()
+ model <- OneR(data = train, formula = NULL, ties.method = "chisq", #c("first","chisq"
+ verbose = TRUE) #FALSE, TRUE
+ }, env)
Loading required package: OneR
Attribute Accuracy
1 * v.satl.binned 63.14%
2 v.fatl.binned 62.64%
3 ftlm.binned 62.54%
4 pcci.binned 61.44%
5 v.rftl.binned 59.74%
6 v.rbci.binned 58.94%
7 rbci.binned 58.64%
8 stlm.binned 58.04%
9 v.stlm.binned 57.54%
10 v.ftlm.binned 56.14%
---
Chosen attribute due to accuracy
and ties method (if applicable): '*'
Warning message:
In OneR(data = train, formula = NULL, ties.method = "chisq", verbose = TRUE) :
data contains unused factor levels
このモデルはルールを作成するための基礎として63.14%の基本精度を持つ v.satl.binned変数を選びました。このモデルに関する一般的な情報を見てみましょう。
> summary(env$model) Call: OneR(data = train, formula = NULL, ties.method = "chisq", verbose = FALSE) Rules: If v.satl.binned = (-Inf,-0.01840058612] then Class = -1 If v.satl.binned = (-0.01840058612,0.3247097195] then Class = 1 If v.satl.binned = (0.3247097195,0.4003869443] then Class = 1 If v.satl.binned = (0.4003869443, Inf] then Class = 1 Accuracy: 632 of 1001 instances classified correctly (63.14%) Contingency table: v.satl.binned Class (-Inf,-0.01840058612] (-0.01840058612,0.3247097195] (0.3247097195,0.4003869443] (0.4003869443, Inf] Sum -1 * 325 161 28 37 551 1 143 * 229 * 35 * 43 450 Sum 468 390 63 80 1001 --- Maximum in each column: '*' Pearson's Chi-squared test: X-squared = 74.429, df = 3, p-value = 4.803e-16
訓練結果の図式表現は下記です。
plot(env$model)

図12 モデル内のクラスによるv.satl.binned変数のカテゴリの分布
訓練中の予測の精度はあまり高くありません。このモデルが検証セットにどのような正確さを示すかを見てみましょう。
> evalq(res.val <- eval_model(predict(model, val %>% as.data.frame()), val$Class), + env) Confusion matrix (absolute): Actual Prediction -1 1 Sum -1 106 87 193 1 100 208 308 Sum 206 295 501 Confusion matrix (relative): Actual Prediction -1 1 Sum -1 0.21 0.17 0.39 1 0.20 0.42 0.61 Sum 0.41 0.59 1.00 Accuracy: 0.6267 (314/501) Error rate: 0.3733 (187/501) Error rate reduction (vs. base rate): 0.0922 (p-value = 0.04597)
テストセットについては下記です。
> evalq(res.test <- eval_model(predict(model, test %>% as.data.frame()), test$Class), + env) Confusion matrix (absolute): Actual Prediction -1 1 Sum -1 130 102 232 1 76 193 269 Sum 206 295 501 Confusion matrix (relative): Actual Prediction -1 1 Sum -1 0.26 0.20 0.46 1 0.15 0.39 0.54 Sum 0.41 0.59 1.00 Accuracy: 0.6447 (323/501) Error rate: 0.3553 (178/501) Error rate reduction (vs. base rate): 0.1359 (p-value = 0.005976)
結果にはがっかりします。エラーレートの低減は、ベース(0.5)レベルに対して精度がどのように向上したかを示します。p(0.05未満)の低い値は、このモデルが基本レベルより良い予測を生成できることを示します。テストセットの精度は0.6447(323/501)です。これは検証セットの精度よりも高くなっています。テストセットは、検証セットよりも訓練セットから離れています。この結果は、将来のモデルの予測結果を比較するための参照点になります。
2.1.3. DNNの構造
訓練とテストには3つのデータセットを使います。
- DTcut$$raw — 12入力変数(外れ値は補完され正規化される)
- DTcut$$dum — 41バイナリ変数
- DTcut$$woe — 10数値変数
すべてのデータセットで、Class変数= factorを2つのレベルで使用します。下記はニューラルネットワークの構造です。
- DNNraw - layers = c(12, 16, 8(2), 2), 活性化関数 c(tanh, maxout(lin), softmax)
- DNNwoe - layers = c(10, 16, 8(2), 2), 活性化関数 c(tanh, maxout(lin), softmax)
- DNNdum - layers = c(41, 50, 8(2), 2), 活性化関数 c(ReLU, maxout(ReLU), softmax)
次の図は、DNNwoeニューラルネットワークの構造を示しています。ニューラルネットワークには、1つの入力層、2つの隠れ層、1つの出力層があります。2つの他のニューラルネットワーク(DNNdum、DNNraw)も同様の構造を持っています。それらで異なるのは、層及び活性化機能におけるニューロンの数だけです。
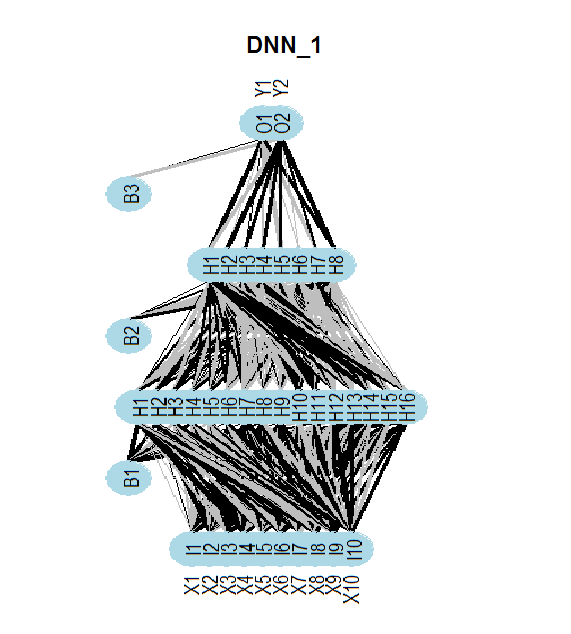
図13 DNNwoeニューラルネットワークの構造
2.1.4. 訓練のバリアント
事前訓練を伴う訓練
訓練は2つの段階で行われます。
- /pretrainセットによるSRBMの事前訓練、それに続くニューラルネットワークの上位層のみの訓練、訓練セットとパラメータによる検証 - par_0
- 訓練/検証セットとパラメータpar_1によるネットワーク全体の微調整
望まれる場合は調整の中間モデルを保存することができます。最良の訓練結果を示すモデルが保存されます。これらの2つの段階のパラメータには、下記が含まれます。
- par_0 — ニューラルネットワークの一般的なパラメータ、RBMの訓練パラメータ、DNNの上位層の訓練パラメータ
- par_1 — DNNのすべての層の訓練パラメータ
DArchのすべてのパラメータにはデフォルト値があります。訓練のある段階で異なるパラメータが必要な場合は、リストで設定し、デフォルトのパラメータを上書きします。訓練の第一段階の後、パラメータと訓練結果(訓練エラー、テストエラーなど)、訓練されたSRBMの重みで開始されたニューラルネットワークを使用してDArch構造を取得します。 第2段階の訓練を完了するためには、この段階の訓練のために、第1段階で得られたDArch構造をパラメータのリストに含める必要があります。もちろん、訓練と検証のセットが必要になります。
訓練の第1段階(SRBMの事前訓練とニューラルネットワークの上位層の訓練)に必要なパラメータを検討し、実行しましょう。
##=====CODE I etap=========================== evalq({ require(darch) require(dplyr) require(magrittr) Ln <- c(0, 16, 8, 0)# // 入力ニューロン及び出力ニューロンの数はデータセットから自動的に識別される nEp_0 <- 25 #------------------ par_0 <- list( layers = Ln, # // このパラメータをリストから外してみる(簡略化のため) seed = 54321,# // f初期化中に同じデータを取得したい場合 logLevel = 5, # // 必要な情報出力レベル # params RBM======================== rbm.consecutive = F, # 各RBMは一度に1エポック訓練される rbm.numEpochs = nEp_0, rbm.batchSize = 50, rbm.allData = TRUE, rbm.lastLayer = -1, # // SRBMの上位層は訓練しない rbm.learnRate = 0.3, rbm.unitFunction = "tanhUnitRbm", # params NN ======================== darch.batchSize = 50, darch.numEpochs = nEp_0,# // このパラメータをリストから外してみる(簡略化のため) darch.trainLayers = c(F,F,T), #обучать // 上位層のみ darch.unitFunction = c("tanhUnit","maxoutUnit", "softmaxUnit"), bp.learnRate = 0.5, bp.learnRateScale = 1, darch.weightDecay = 0.0002, darch.dither = F, darch.dropout = c(0.1,0.2,0.1), darch.fineTuneFunction = backpropagation, #rpropagation normalizeWeights = T, normalizeWeightsBound = 1, darch.weightUpdateFunction = c("weightDecayWeightUpdate", "maxoutWeightUpdate", "weightDecayWeightUpdate"), darch.dropout.oneMaskPerEpoch = T, darch.maxout.poolSize = 2, darch.maxout.unitFunction = "linearUnit") #--------------------------- DNN_default <- darch(darch = NULL, paramsList = par_0, x = DTcut$pretrain$woe %>% as.data.frame(), y = DTcut$pretrain$raw$Class %>% as.data.frame(), xValid = DTcut$train$woe %>% as.data.frame(), yValid = DTcut$train$raw$Class %>% as.data.frame() ) }, env)下記は訓練の第1段階の完了時の結果です。
........................... INFO [2017-09-11 14:12:19] Classification error on Train set (best model): 31.95% (639/2000) INFO [2017-09-11 14:12:19] Train set (best model) Cross Entropy error: 1.233 INFO [2017-09-11 14:12:19] Classification error on Validation set (best model): 35.86% (359/1001) INFO [2017-09-11 14:12:19] Validation set (best model) Cross Entropy error: 1.306 INFO [2017-09-11 14:12:19] Best model was found after epoch 3 INFO [2017-09-11 14:12:19] Final 0.632 validation Cross Entropy error: 1.279 INFO [2017-09-11 14:12:19] Final 0.632 validation classification error: 34.42% INFO [2017-09-11 14:12:19] Fine-tuning finished after 5.975 secs
下記はニューラルネットワークの第2段階の訓練です。
##=====CODE II etap=========================== evalq({ require(darch) require(dplyr) require(magrittr) nEp_1 <- 100 bp.learnRate <- 1 par_1 <- list( layers = Ln, seed = 54321, logLevel = 5, rbm.numEpochs = 0,# SRBMは訓練されるべきではない darch.batchSize = 50, darch.numEpochs = nEp_1, darch.trainLayers = c(T,T,T), #TRUE, darch.unitFunction = c("tanhUnit","maxoutUnit", "softmaxUnit"), bp.learnRate = bp.learnRate, bp.learnRateScale = 1, darch.weightDecay = 0.0002, darch.dither = F, darch.dropout = c(0.1,0.2,0.1), darch.fineTuneFunction = backpropagation, #rpropagation backpropagation normalizeWeights = T, normalizeWeightsBound = 1, darch.weightUpdateFunction = c("weightDecayWeightUpdate", "maxoutWeightUpdate", "weightDecayWeightUpdate"), darch.dropout.oneMaskPerEpoch = T, darch.maxout.poolSize = 2, darch.maxout.unitFunction = exponentialLinearUnit, darch.elu.alpha = 2) #------------------------------ DNN_1 <- darch( darch = DNN_default, paramsList = par_1, x = DTcut$train$woe %>% as.data.frame(), y = DTcut$train$raw$Class %>% as.data.frame(), xValid = DTcut$val$woe %>% as.data.frame(), yValid = DTcut$val$raw$Class %>% as.data.frame() ) }, env)
下記は訓練の第2段階の結果です。
........................... INFO [2017-09-11 15:48:37] Finished epoch 100 of 100 after 0.279 secs (3666 patterns/sec) INFO [2017-09-11 15:48:37] Classification error on Train set (best model): 31.97% (320/1001) INFO [2017-09-11 15:48:37] Train set (best model) Cross Entropy error: 1.225 INFO [2017-09-11 15:48:37] Classification error on Validation set (best model): 31.14% (156/501) INFO [2017-09-11 15:48:37] Validation set (best model) Cross Entropy error: 1.190 INFO [2017-09-11 15:48:37] Best model was found after epoch 96 INFO [2017-09-11 15:48:37] Final 0.632 validation Cross Entropy error: 1.203 INFO [2017-09-11 15:48:37] Final 0.632 validation classification error: 31.44% INFO [2017-09-11 15:48:37] Fine-tuning finished after 37.22 secs
下記は訓練の第2段階における予測誤差の変化のグラフです。
plot(env$DNN_1, y = "raw")
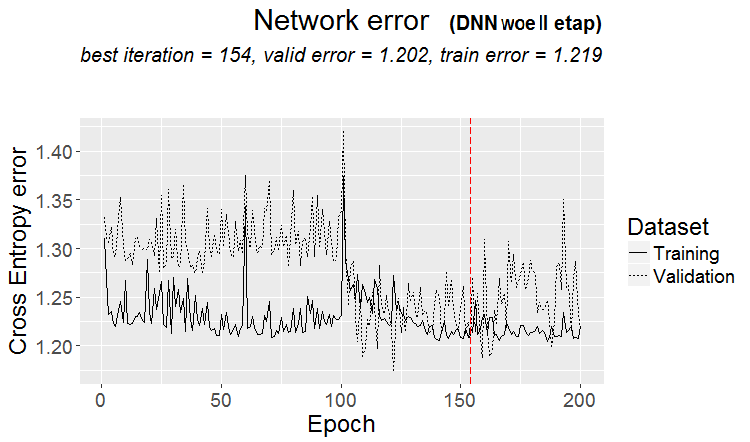
図14 第2段階の訓練中の分類エラーの変更
テストセット上の最終モデルの分類誤差を見てみましょう。
#-----------
evalq({
xValid = DTcut$test$woe %>% as.data.frame()
yValid = DTcut$test$raw$Class %>% as.vector()
Ypredict <- predict(DNN_1, newdata = xValid, type = "class")
numIncorrect <- sum(Ypredict != yValid)
cat(paste0("Incorrect classifications on all examples: ", numIncorrect, " (",
round(numIncorrect/nrow(xValid)*100, 2), "%)\n"))
caret::confusionMatrix(yValid, Ypredict)
}, env)
Incorrect classifications on all examples: 166 (33.13%)
Confusion Matrix and Statistics
Reference
Prediction -1 1
-1 129 77
1 89 206
Accuracy : 0.6687
95% CI : (0.6255, 0.7098)
No Information Rate : 0.5649
P-Value [Acc > NIR] : 1.307e-06
Kappa : 0.3217
Mcnemar's Test P-Value : 0.3932
Sensitivity : 0.5917
Specificity : 0.7279
Pos Pred Value : 0.6262
Neg Pred Value : 0.6983
Prevalence : 0.4351
Detection Rate : 0.2575
Detection Prevalence : 0.4112
Balanced Accuracy : 0.6598
'Positive' Class : -1
#----------------------------------------
このデータセット(woe)はこれらの最適ではないパラメータを用いますが、その精度は基本モデルよりもはるかに高いです。DNNのハイパーパラメータを最適化することによって精度を高める可能性があります。計算が繰り返されると、データは本稿のものと完全に同じではなくなる可能性があります。
スクリプトをよりコンパクトな形式にして、他のデータセットとのさらなる計算を可能にしましょう。woeセットのための関数を書いてみます。
#------------------- DNN.train.woe <- function(param, X){ require(darch) require(magrittr) darch( darch = NULL, paramsList = param[[1]], x = X[[1]]$woe %>% as.data.frame(), y = X[[1]]$raw$Class %>% as.data.frame(), xValid = X[[2]]$woe %>% as.data.frame(), yValid = X[[2]]$raw$Class %>% as.data.frame() ) %>% darch( ., paramsList = param[[2]], x = X[[2]]$woe %>% as.data.frame(), y = X[[2]]$raw$Class %>% as.data.frame(), xValid = X[[3]]$woe %>% as.data.frame(), yValid = X[[3]]$raw$Class %>% as.data.frame() ) -> Darch return(Darch) }
コンパクトな形式でDTcut$$woeデータセットの計算を繰り返します。
evalq({
require(darch)
require(magrittr)
Ln <- c(0, 16, 8, 0)
nEp_0 <- 25
nEp_1 <- 25
rbm.learnRate = c(0.5,0.3,0.1)
bp.learnRate <- c(0.5,0.3,0.1)
list(par_0, par_1) %>% DNN.train.woe(DTcut) -> Dnn.woe
xValid = DTcut$test$woe %>% as.data.frame()
yValid = DTcut$test$raw$Class %>% as.vector()
Ypredict <- predict(Dnn.woe, newdata = xValid, type = "class")
numIncorrect <- sum(Ypredict != yValid)
cat(paste0("Incorrect classifications on all examples: ", numIncorrect, " (",
round(numIncorrect/nrow(xValid)*100, 2), "%)\n"))
caret::confusionMatrix(yValid, Ypredict) -> cM.woe
}, env)
DTcut$$rawデータセットの計算をします。
#-------------------------
DNN.train.raw <- function(param, X){
require(darch)
require(magrittr)
darch( darch = NULL, paramsList = param[[1]],
x = X[[1]]$raw %>% tbl_df %>% select(-c(Data, Class)),
y = X[[1]]$raw$Class %>% as.data.frame(),
xValid = X[[2]]$raw %>% tbl_df %>% select(-c(Data, Class)),
yValid = X[[2]]$raw$Class %>% as.data.frame()
) %>%
darch( ., paramsList = param[[2]],
x = X[[2]]$raw %>% tbl_df %>% select(-c(Data, Class)),
y = X[[2]]$raw$Class %>% as.data.frame(),
xValid = X[[3]]$raw %>% tbl_df %>% select(-c(Data, Class)),
yValid = X[[3]]$raw$Class %>% as.data.frame()
) -> Darch
return(Darch)
}
#-------------------------------
evalq({
require(darch)
require(magrittr)
Ln <- c(0, 16, 8, 0)
nEp_0 <- 25
nEp_1 <- 25
rbm.learnRate = c(0.5,0.3,0.1)
bp.learnRate <- c(0.5,0.3,0.1)
list(par_0, par_1) %>% DNN.train.raw(DTcut) -> Dnn.raw
xValid = DTcut$test$raw %>% tbl_df %>% select(-c(Data, Class))
yValid = DTcut$test$raw$Class %>% as.vector()
Ypredict <- predict(Dnn.raw, newdata = xValid, type = "class")
numIncorrect <- sum(Ypredict != yValid)
cat(paste0("Incorrect classifications on all examples: ", numIncorrect, " (",
round(numIncorrect/nrow(xValid)*100, 2), "%)\n"))
caret::confusionMatrix(yValid, Ypredict) -> cM.raw
}, env)
#----------------------------
以下は、このセットの結果と分類エラーの変化のグラフです。
> env$cM.raw
Confusion Matrix and Statistics
Reference
Prediction -1 1
-1 133 73
1 86 209
Accuracy : 0.6826
95% CI : (0.6399, 0.7232)
No Information Rate : 0.5629
P-Value [Acc > NIR] : 2.667e-08
Kappa : 0.3508
Mcnemar's Test P-Value : 0.3413
Sensitivity : 0.6073
Specificity : 0.7411
Pos Pred Value : 0.6456
Neg Pred Value : 0.7085
Prevalence : 0.4371
Detection Rate : 0.2655
Detection Prevalence : 0.4112
Balanced Accuracy : 0.6742
'Positive' Class : -1
#--------------------------------------
plot(env$Dnn.raw, y = "raw")
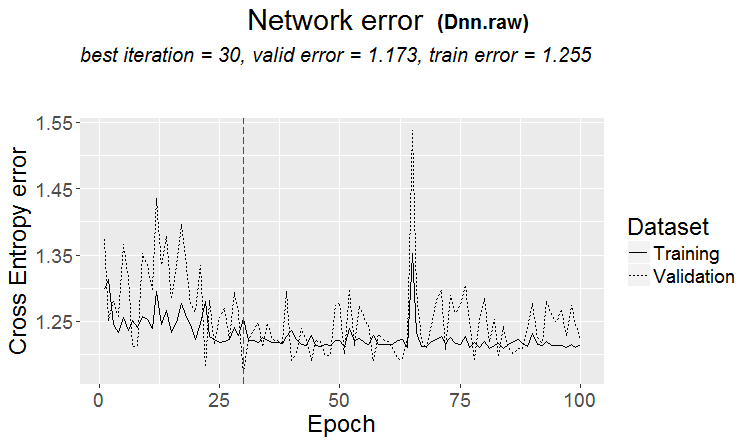
図15 第2段階における分類誤差の変化
DTcut$$dumデータではニューラルネットワークを訓練することができませんでした。ご自分でお試しください。たとえば、DTcut$$binデータを入力し、予測パラメータがダミーに変換されるように訓練パラメータを調整します。
事前訓練なしの訓練
事前訓練/訓練/検証セットと同じデータ(woe、raw)でニューラルネットワークを事前訓練なしで訓練しましょう。結果を見てみます。
#-------WOE---------------- evalq({ require(darch) require(magrittr) Ln <- c(0, 16, 8, 0) nEp_1 <- 100 bp.learnRate <- c(0.5,0.7,0.1) #--param---------------- par_1 <- list( layers = Ln, seed = 54321, logLevel = 5, rbm.numEpochs = 0,# SRBMは訓練されるべきではない darch.batchSize = 50, darch.numEpochs = nEp_1, darch.trainLayers = c(T,T,T), #TRUE, darch.unitFunction = c("tanhUnit","maxoutUnit", "softmaxUnit"), bp.learnRate = bp.learnRate, bp.learnRateScale = 1, darch.weightDecay = 0.0002, darch.dither = F, darch.dropout = c(0.0,0.2,0.1), darch.fineTuneFunction = backpropagation, #rpropagation backpropagation normalizeWeights = T, normalizeWeightsBound = 1, darch.weightUpdateFunction = c("weightDecayWeightUpdate", "maxoutWeightUpdate", "weightDecayWeightUpdate"), darch.dropout.oneMaskPerEpoch = T, darch.maxout.poolSize = 2, darch.maxout.unitFunction = exponentialLinearUnit, darch.elu.alpha = 2) #--訓練--------------------------- darch( darch = NULL, paramsList = par_1, x = DTcut[[1]]$woe %>% as.data.frame(), y = DTcut[[1]]$raw$Class %>% as.data.frame(), xValid = DTcut[[2]]$woe %>% as.data.frame(), yValid = DTcut[[2]]$raw$Class %>% as.data.frame() ) -> Dnn.woe.I #---テスト-------------------------- xValid = DTcut$val$woe %>% as.data.frame() yValid = DTcut$val$raw$Class %>% as.vector() Ypredict <- predict(Dnn.woe.I, newdata = xValid, type = "class") numIncorrect <- sum(Ypredict != yValid) cat(paste0("Incorrect classifications on all examples: ", numIncorrect, " (", round(numIncorrect/nrow(xValid)*100, 2), "%)\n")) caret::confusionMatrix(yValid, Ypredict) -> cM.woe.I }, env) #---------Ris16------------------------------------ plot(env$Dnn.woe.I, type = "class") env$cM.woe.I
下記はメトリクスです。
....................................................... INFO [2017-09-14 10:38:01] Classification error on Train set (best model): 28.7% (574/2000) INFO [2017-09-14 10:38:01] Train set (best model) Cross Entropy error: 1.140 INFO [2017-09-14 10:38:02] Classification error on Validation set (best model): 35.86% (359/1001) INFO [2017-09-14 10:38:02] Validation set (best model) Cross Entropy error: 1.299 INFO [2017-09-14 10:38:02] Best model was found after epoch 67 INFO [2017-09-14 10:38:02] Final 0.632 validation Cross Entropy error: 1.241 INFO [2017-09-14 10:38:02] Final 0.632 validation classification error: 33.23% INFO [2017-09-14 10:38:02] Fine-tuning finished after 37.13 secs Incorrect classifications on all examples: 150 (29.94%) > env$cM.woe.I Confusion Matrix and Statistics Reference Prediction -1 1 -1 144 62 1 88 207 Accuracy : 0.7006 95% CI : (0.6584, 0.7404) No Information Rate : 0.5369 P-Value [Acc > NIR] : 5.393e-14 Kappa : 0.3932 Mcnemar's Test P-Value : 0.04123 Sensitivity : 0.6207 Specificity : 0.7695 Pos Pred Value : 0.6990 Neg Pred Value : 0.7017 Prevalence : 0.4631 Detection Rate : 0.2874 Detection Prevalence : 0.4112 Balanced Accuracy : 0.6951 'Positive' Class : -1
下記は訓練中の分類誤差の変化を表すグラフです。
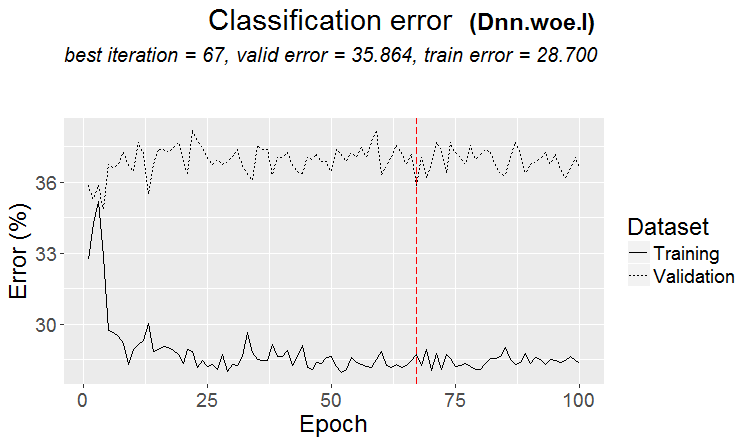
図16 事前訓練をしない場合の$woeセットの分類誤差の変化
/rawセットでも同じです。
evalq({
require(darch)
require(magrittr)
Ln <- c(0, 16, 8, 0)
nEp_1 <- 100
bp.learnRate <- c(0.5,0.7,0.1)
#--param-----------------------------
par_1 <- list(
layers = Ln,
seed = 54321,
logLevel = 5,
rbm.numEpochs = 0,# SRBMは訓練されるべきではない
darch.batchSize = 50,
darch.numEpochs = nEp_1,
darch.trainLayers = c(T,T,T), #TRUE,
darch.unitFunction = c("tanhUnit","maxoutUnit", "softmaxUnit"),
bp.learnRate = bp.learnRate,
bp.learnRateScale = 1,
darch.weightDecay = 0.0002,
darch.dither = F,
darch.dropout = c(0.1,0.2,0.1),
darch.fineTuneFunction = backpropagation, #rpropagation backpropagation
normalizeWeights = T,
normalizeWeightsBound = 1,
darch.weightUpdateFunction = c("weightDecayWeightUpdate",
"maxoutWeightUpdate",
"weightDecayWeightUpdate"),
darch.dropout.oneMaskPerEpoch = T,
darch.maxout.poolSize = 2,
darch.maxout.unitFunction = exponentialLinearUnit,
darch.elu.alpha = 2)
#---訓練------------------------------
darch( darch = NULL, paramsList = par_1,
x = DTcut[[1]]$raw %>% tbl_df %>% select(-c(Data, Class)) ,
y = DTcut[[1]]$raw$Class %>% as.vector(),
xValid = DTcut[[2]]$raw %>% tbl_df %>% select(-c(Data, Class)) ,
yValid = DTcut[[2]]$raw$Class %>% as.vector()
) -> Dnn.raw.I
#---テスト--------------------------------
xValid = DTcut[[3]]$raw %>% tbl_df %>% select(-c(Data, Class))
yValid = DTcut[[3]]$raw$Class %>% as.vector()
Ypredict <- predict(Dnn.raw.I, newdata = xValid, type = "class")
numIncorrect <- sum(Ypredict != yValid)
cat(paste0("Incorrect classifications on all examples: ", numIncorrect, " (",
round(numIncorrect/nrow(xValid)*100, 2), "%)\n"))
caret::confusionMatrix(yValid, Ypredict) -> cM.raw.I
}, env)
#---------Ris17----------------------------------
env$cM.raw.I
plot(env$Dnn.raw.I, type = "class")
下記はメトリクスです。
INFO [2017-09-14 11:06:13] Classification error on Train set (best model): 30.75% (615/2000) INFO [2017-09-14 11:06:13] Train set (best model) Cross Entropy error: 1.189 INFO [2017-09-14 11:06:13] Classification error on Validation set (best model): 33.67% (337/1001) INFO [2017-09-14 11:06:13] Validation set (best model) Cross Entropy error: 1.236 INFO [2017-09-14 11:06:13] Best model was found after epoch 45 INFO [2017-09-14 11:06:13] Final 0.632 validation Cross Entropy error: 1.219 INFO [2017-09-14 11:06:13] Final 0.632 validation classification error: 32.59% INFO [2017-09-14 11:06:13] Fine-tuning finished after 35.47 secs Incorrect classifications on all examples: 161 (32.14%) > #---------Ris17---------------------------------- > env$cM.raw.I Confusion Matrix and Statistics Reference Prediction -1 1 -1 140 66 1 95 200 Accuracy : 0.6786 95% CI : (0.6358, 0.7194) No Information Rate : 0.5309 P-Value [Acc > NIR] : 1.283e-11 Kappa : 0.3501 Mcnemar's Test P-Value : 0.02733 Sensitivity : 0.5957 Specificity : 0.7519 Pos Pred Value : 0.6796 Neg Pred Value : 0.6780 Prevalence : 0.4691 Detection Rate : 0.2794 Detection Prevalence : 0.4112 Balanced Accuracy : 0.6738 'Positive' Class : -1
下記は分類誤差の変化を表すグラフです。
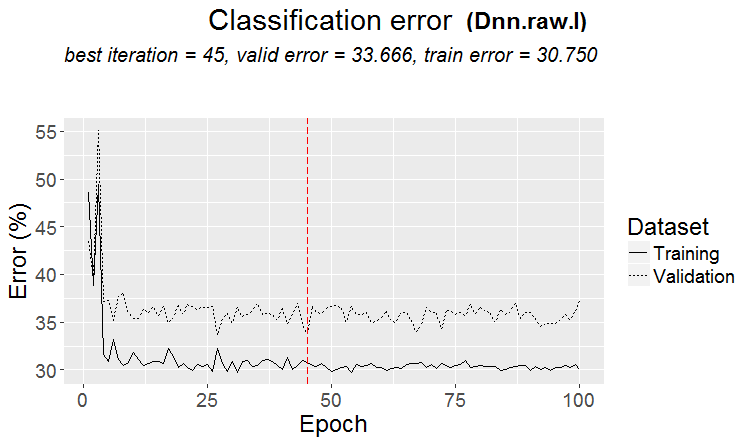
図17 事前訓練をしない場合の$rawセットの分類誤差の変化
2.2. 結果分析
実験の結果を表にまとめてみましょう。
| 訓練の種類 | /woeセット | /rawセット |
|---|---|---|
| 事前訓練を伴う訓練 | 0.6687 (0.6255 - 0.7098) | 0.6826(0.6399 - 0.7232) |
| 事前訓練を伴わない訓練 | 0.7006(0.6589 - 0.7404) | 0.6786(0.6359 - 0.7194) |
事前訓練を伴う場合の分類誤差は両セットでほぼ同じで、30+/-4%の範囲です。誤差がより少ないにもかかわらず、分類誤差の変化のグラフから、事前訓練を伴わない場合には訓練中に再訓練が行われたことが明らかです(検証セットとテストセットの誤差が訓練セットよりもかなり多い)。したがって、これからの実験では事前訓練を伴う訓練を使用します。
結果は、基本モデルと比べてそれほど優れてはいません。いくつかのハイパーパラメータを最適化することによって特性を改善する可能性がありますが、これは次の記事で行います。
終わりに
darchパッケージは、基本的な訓練方法は2つのみであるというような制限を持ちながらも、構造とパラメータが異なるニューラルネットワークの作成を可能にします。このパッケージは、ニューラルネットワークを深く勉強するための良いツールです。
DNNの脆弱性は、主に既定のパラメータまたはそれに近いパラメータの使用によって説明されます。woeセットはrawセットと比べて何の利点も示しませんでした。したがって、次の記事では、
- 先に作成したDNN.woeのハイパーパラメータの一部を最適化します。
- TensorFlowライブラリを使用してDNNを作成し、テストして結果をDNN (darch)と比較します。
- 異なる種類のニューラルネットワークのアンサンブルを作成し(バギング、スタッキング)、これがどのようにして予測の質を改善するかを見ます。
適用
GitHub/PartIVは下記を含みます。
- FunPrepareData.R — データの準備に使われる関数
- RunPrepareData.R — データの準備に使われるスクリプト
- Experiment.R — 実験するためのスクリプト
- Part_IV.RData — データの準備段階から得られた全てのオブジェクトを含む作業領域のイメージ
- SessionInfo.txt — 使用されているソフトウェアに関する情報
- Darch_default.txt — デフォルト値を持つDArch構造体のパラメータのリスト
MetaQuotes Ltdによってロシア語から翻訳されました。
元の記事: https://www.mql5.com/ru/articles/3473
警告: これらの資料についてのすべての権利はMetaQuotes Ltd.が保有しています。これらの資料の全部または一部の複製や再プリントは禁じられています。
この記事はサイトのユーザーによって執筆されたものであり、著者の個人的な見解を反映しています。MetaQuotes Ltdは、提示された情報の正確性や、記載されているソリューション、戦略、または推奨事項の使用によって生じたいかなる結果についても責任を負いません。
 ユニバーサルEA: CUnIndicator と予約オーダーの使用 (その 9)
ユニバーサルEA: CUnIndicator と予約オーダーの使用 (その 9)
 トレードオブジェクト: メタトレーダーのグラフィカルオブジェクトに基づいたトレードの自動化
トレードオブジェクト: メタトレーダーのグラフィカルオブジェクトに基づいたトレードの自動化
- 無料取引アプリ
- 8千を超えるシグナルをコピー
- 金融ニュースで金融マーケットを探索

OneR.comは、OneR.comが提供するOneR.comの最新バージョンです。DNNはOneR!
これは、DNNがデフォルトのパラメータを 持つ場合にのみ当てはまります。最適化されたハイパーパラメータでは、DNNははるかに良い結果を示す。パートVを参照。
幸運を祈る
これは、DNNがデフォルトのパラメーターを使用した場合にのみ当てはまる。最適化されたハイパーパラメータを用いると、DNNははるかに良い結果を示す。パートVを参照。
幸運を祈る。
トレードの結果は?
みんなマッシュに戻って いる。それはまた別の話題だ。