
Algoritmos genéticos - é fácil!
Introdução
Algoritmo genético (GA) se refere ao algoritmo heurístico (EA), o qual dá uma solução aceitável para o problema na maioria dos casos praticamente significativos, mas a correção das decisões não foi provada matematicamente e é usado com mais frequência para problemas, a solução analítica a qual é muito difícil ou até mesmo impossível.
Um exemplo clássico de um problema desta classe (classe NP) é "um problema de um vendedor viajante" (é um dos mais famosos problemas de otimização combinatória). O desafio principal é encontrar a rota mais vantajosa, que passa pelas cidades determinadas ao menos uma vez e, então, retornar para a cidade inicial). Mas nada impede de usá-los para tarefas, o que resulta em formalização.
O EA é amplamente utilizado para resolver problema de alta complexidade computacional, ao invés de passar por todas as opções, o que toma uma quantidade significante de tempo. Eles são usados nos campos de inteligência artificial, como um reconhecimento de padrão, em programas antivírus, engenharia, jogos de computador e outras áreas.
Deve-se mencionar que a MetaQuotes Software Corp. utiliza GA em seus produtos de software do MetaTrader 4/5. Todos sabemos da estratégia verificadora e sobre quanto tempo e esforço podem ser poupados utilizando um otimizador de estratégia integrado, no qual, assim como com o enumerador direto, é possível otimizar com o uso de GA. Além disso, o verificador do MetaTrader 5 nos possibilita utilizar o critério de otimização do usuário. Talvez o leitor esteja interessado em ler os artigos sobre o GA e as vantagens, fornecidas pelo EA, em contrapartida à enumeração direta.
1. Um pouco de história
Pouco mais de um ano atrás, eu precisava de um algoritmo de otimização para treinar redes neurais. Após me familiarizar rapidamente com os vários algoritmos, eu escolhi o GA. Como resultado da minha busca por implementações já prontas - constatei que aquelas abertas para acesso público, ou possuem limitações funcionais, como o número de parâmetros que podem ser otimizados ou são demasiadamente "restritas nos ajustes".
Eu precisava de um instrumento universalmente flexível, não apenas para treinar todos os tipos de redes neurais, mas também para resolver de maneira geral quaisquer problemas de otimização. Após um extenso estudo de "criações genéticas", eu ainda não era capaz de entender como eles funcionavam. A causa disto era tanto um estilo de código elaborado, ou minha falta de experiência em programação, ou possivelmente os dois.
As principais dificuldades que surgiram a partir da codificação de genes a um código binário e, em seguida, trabalhar com eles nesta forma. De qualquer forma, estava decidido a escrever um algoritmo genético a partir do nada, focado em escalabilidade e fácil modificação no futuro.
Eu não queria lidar com transformação binária e decidi trabalhar com genes diretamente, ou seja, representar o cromossomo com um conjunto de genes na forma de números reais. É assim que o código para o meu algoritmo genético, com uma representação de cromossomos por números reais, apareceu. Mais tarde, eu soube que eu não descobri nada de novo e que algoritmos genéticos análogos (chamados GA verdadeiramente codificados) já existiam por mais de 15 anos, desde então, saíram as primeiras publicações sobre eles.
Eu deixo a minha visão sobre a abordagem da implementação e princípios de funcionamento do GA para o leitor julgar, já que é baseada em experiência pessoal de seu uso em problemas práticos.
2. Descrição do GA
O GA contém os princípios, emprestados a partir da própria natureza. Estes são os princípios da hereditariedade e variabilidade. A hereditariedade é a capacidade dos organismos de transferir seus traços e características evolucionárias para os seus descendentes. Graças a esta capacidade, todos os seres vivos deixam para trás características de suas espécies em seus descendentes.
A variabilidade de genes que vivem em organismos assegura a diversidade genética da população e é aleatória, já que a natureza não possui uma maneira de saber com antecedência quais atributos serão mais preferíveis no futuro (alteração do clima, diminuição/aumento dos alimentos, o surgimento de espécies concorrentes, etc.). Esta variabilidade permite o aparecimento de criaturas com novos traços, que podem sobreviver e deixar descendentes nas novas condições de habitat alteradas.
Na biologia, a variabilidade, que originou-se devido ao surgimento de mutações, é chamada mutacional, a variabilidade devido à combinação cruzada adicional de genes por acasalamento, é chamada combinatória. Ambos os tipos de variações são implementadas no GA. Além disso, há uma implementação de mutagênese, que imita o mecanismo natural de mutações (alterações na sequência de nucleotídeo do DNA) - o natural (espontâneo) e artificial (induzido).
A unidade mais simples de transferência de informações nos critérios do algoritmo é o gene - unidade estrutural e funcional de hereditariedade, que controla o desenvolvimento de um traço ou propriedade em particular. Chamaremos uma variável da função de o gene. O gene é representado por um número real. O conjunto de genes - variáveis da função estudada é o atributo característico do - cromossomo.
Vamos concordar em representar o cromossomo na forma de uma colina. Então, o cromossomo para a função f (x) = x ^ 2, se pareceria com isto:
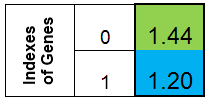
Figura 1. Cromossomo para a função f (x) = x ^ 2
na qual o índice 0-th - valor da função f (x), chamada de adaptação dos indivíduos (chamaremos esta função de função de aptidão - FF e o valor da função - VFF). Convém armazenar o cromossomo em uma série unidimensional. Esta é a série de cromossomos duplos [].
Todos os espécimens da mesma era evolucionária são combinados em uma população. Além disso, a população é dividida arbitrariamente em duas colônias - as colônias pais e as colônias descendentes. Como resultado do cruzamento das espécies pais, que são selecionadas a partir de toda a população e outros operadores do GA, há uma colônia de nova descendência, que é igual à metade do tamanho da população, que substitui a colônia da descendência na população.
A população total de indivíduos durante uma busca pelo mínimo da função f (x) = x ^ 2 pode se parecer com isto:
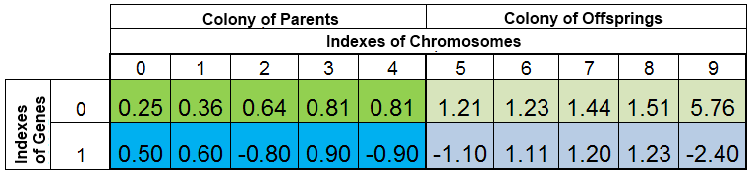
Figura 2. População total de indivíduos
A população é classificada por VFF. Aqui, o índice 0-th do cromossomo é ocupado pelo espécime com o menor VFF. Os novos descendentes substituem completamente apenas os indivíduos na colônia dos descendentes, enquanto a colônia pai permanece intacta. Contudo, a colônia pai pode nem sempre estar completa, já que os espécimes duplicados são destruídos, então, os novos descendentes preenchem as vagas na colônia pai e o restante é posicionado na colônia dos descendentes.
Em outras palavras, o tamanho da população raramente é constante e varia de uma era para outra, quase igual como na natureza. Por exemplo, a população antes da reprodução e depois da reprodução pode ficar assim:
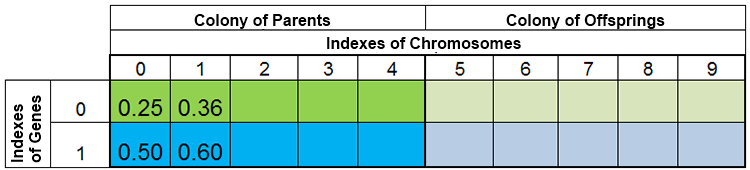
Figura 3. A população antes da reprodução
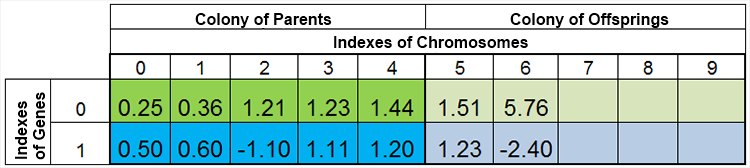
Figura 4. A população após a reprodução
O mecanismo de "meio" preenchimento da população por descendentes descrito, assim como a destruição de duplicados e o banimento da reprodução cruzada de indivíduos com eles mesmos possuem um único objetivo - evitar o "efeito gargalo" (um termo vindo da biologia, que significa uma redução do conjunto de genes como um resultado de uma redução grave devida a um número de razões distintas, que pode levar a uma extinção completa de uma espécie, o GA está enfrentando o fim do aparecimento de cromossomos únicos e "ficando preso" em um dos extremos locais).
3. Descrição da função UGAlib
Algoritmo GA:- Criando uma proto-população. Os genes são gerados aleatoriamente dentro de um dado intervalo.
- Determinando a aptidão de cada indivíduo, ou, em outras palavras, o cálculo do VFF.
- Preparando a população para reprodução, depois de remover cromossomos duplicados.
- Isolamento e preservação do cromossomo de referência (com o melhor VFF).
- Operadores de UGA (seleção, acasalamento, mutação). Para cada acasalamento e mutação, novos pais são selecionados a cada vez. Preparando a população para a próxima era.
- Comparação de genes das melhores descendências com os genes do cromossomo de referência. Se o cromossomo da melhor descendência é melhor do que o cromossomo de referência, então substitua o cromossomo de referência.
Continue a partir do parágrafo 5 até não haver mais cromossomos, melhores do que os de referência que apareçam, para um número de áreas específico.
3.1. Variáveis globais
Anunciadas as seguintes variáveis no nível global:
//----------------------Global variables----------------------------- double Chromosome[]; //A set of optimized arguments of the function - genes //(for example: the weight of the neuron network, etc.)- of the chromosome int ChromosomeCount =0; //Maximum possible amount of chromosomes in a colony int TotalOfChromosomesInHistory=0;//Total number of chromosomes in the history int ChrCountInHistory =0; //Number of unique chromosomes in the base chromosome int GeneCount =0; //Number of genes in the chromosome double RangeMinimum =0.0;//Minimum search range double RangeMaximum =0.0;//Maximum search range double Precision =0.0;//Search step int OptimizeMethod =0; //1-minimum, any other - maximum int FFNormalizeDigits =0; //Number of symbols after the comma in the fitness value int GeneNormalizeDigits =0; //Number of symbols after the comma in the genes value double Population [][1000]; //Population double Colony [][500]; //Offspring colony int PopulChromosCount =0; //The current number of chromosomes in a population int Epoch =0; //Number of epochs without progress int AmountStartsFF=0; //Number of launches of the fitness function //————————————————————————————————————————————————————————————————————————
3.2. UGA. A função principal do GA
Na verdade, a principal função do GA, é chamada a partir do corpo do programa para executar as etapas listadas acima, portanto, não iremos a muitos detalhes sobre isto.
Mediante a conclusão do algoritmo, está registrada a seguinte informação no log:
- Quantos períodos haviam no total (gerações).
- Quantas falhas no total.
- O número de cromossomos únicos.
- O número total de lançamentos de FF.
- O número total de cromossomos no histórico.
- Proporção percentual de duplicados em relação ao número total de cromossomos no histórico.
- Melhor resultado.
"O número de cromossomos únicos" e o "Número total de lançamentos de FF" - têm os mesmos tamanhos, mas são calculados de maneira diferente. Utilize para controle.
//———————————————————————————————————————————————————————————————————————— //Basic function UGA void UGA ( double ReplicationPortion, // Proportion of replication. double NMutationPortion, // Proportion of natural mutations. double ArtificialMutation, // Proportion of artificial mutations. double GenoMergingPortion, // Proportion of borrowed genes. double CrossingOverPortion, // Proportion of cross -over. //--- double ReplicationOffset, // Coefficient of displacement of interval borders double NMutationProbability // Probability of mutation of each gene in% ) { //generator reset takes place only once MathSrand((int)TimeLocal()); //-----------------------Variables------------------------------------- int chromos=0, gene =0;//indexes of chromosomes and genes int resetCounterFF =0;//counter of resets of "Epochs without progress" int currentEpoch =1;//number of the current epoch int SumOfCurrentEpoch=0;//sum of "Epochs without progress" int MinOfCurrentEpoch=Epoch;//minimum of "epochs without progress" int MaxOfCurrentEpoch=0;//maximum of "Epochs without progress" int epochGlob =0;//total number of epochs // Colony [number of traits(genes)][number of individuals in a colony] ArrayResize(Population,GeneCount+1); ArrayInitialize(Population,0.0); // Colony of offspring [number of traits(genes)][number of individuals in a colony] ArrayResize(Colony,GeneCount+1); ArrayInitialize(Colony,0.0); // Chromosome bank // [number of traits (genes)][number of chromosomes in the bank] double historyHromosomes[][100000]; ArrayResize(historyHromosomes,GeneCount+1); ArrayInitialize(historyHromosomes,0.0); //---------------------------------------------------------------------- //--------------Verification of the correctness of incoming parameters---------------- //...the number of chromosomes mus be less than 2 if(ChromosomeCount<=1) ChromosomeCount=2; if(ChromosomeCount>500) ChromosomeCount=500; //---------------------------------------------------------------------- //====================================================================== // 1) Create a proto- population —————1) ProtopopulationBuilding(); //====================================================================== // 2) Determine the fitness of each individual —————2) //For the 1st colony for(chromos=0;chromos<chromosomecount;chromos++) for(gene=1;gene<=GeneCount;gene++) Colony[gene][chromos]=Population[gene][chromos]; GetFitness(historyHromosomes); for(chromos=0;chromos<chromosomecount;chromos++) population[0][chromos]=Colony[0][chromos]; //For the 2nd colony for(chromos=ChromosomeCount;chromos<chromosomecount*2;chromos++) for(gene=1;gene<=GeneCount;gene++) Colony[gene][chromos-ChromosomeCount]=Population[gene][chromos]; GetFitness(historyHromosomes); for(chromos=ChromosomeCount;chromos<chromosomecount*2;chromos++) Population[0][chromos]=Colony[0][chromos-ChromosomeCount]; //====================================================================== // 3) Prepare the population for reproduction ————3) RemovalDuplicates(); //====================================================================== // 4) Extract the reference chromosome —————4) for(gene=0;gene<=GeneCount;gene++) Chromosome[gene]=Population[gene][0]; //====================================================================== ServiceFunction(); //The main cycle The main cycle of the genetic algorithm from 5 to 6 while(currentEpoch<=Epoch) { //==================================================================== // 5) Operators of UGA —————5) CycleOfOperators ( historyHromosomes, //--- ReplicationPortion, //Proportion of replication. NMutationPortion, //Proportion of natural mutation. ArtificialMutation, //Proportion of artificial mutation. GenoMergingPortion, //Proportion of borrowed genes. CrossingOverPortion,//Proportion of cross- over. //--- ReplicationOffset, //Coefficient of displacement of interval borders NMutationProbability//Probability of mutation of each gene in % ); //==================================================================== // 6) Compare the genes of the best offspring with the genes of the reference chromosome. // If the chromosome of the best offspring is better that the reference chromosome, // replace the reference. —————6) //If the optimization mode is - minimization if(OptimizeMethod==1) { //If the best chromosome of the population is better than the reference chromosome if(Population[0][0]<chromosome[0]) { //Replace the reference chromosome for(gene=0;gene<=GeneCount;gene++) Chromosome[gene]=Population[gene][0]; ServiceFunction(); //Rest the counter of "epochs without progress" if(currentEpoch<minofcurrentepoch) minofcurrentepoch="currentEpoch;" if(currentEpoch>MaxOfCurrentEpoch) MaxOfCurrentEpoch=currentEpoch; SumOfCurrentEpoch+=currentEpoch; currentEpoch=1; resetCounterFF++; } else currentEpoch++; } //If the optimization mode is - minimization else { //If the best chromosome of the population is better than the reference chromosome if(Population[0][0]>Chromosome[0]) { //Replace the reference chromosome for(gene=0;gene<=GeneCount;gene++) Chromosome[gene]=Population[gene][0]; ServiceFunction(); //Reset the counter of "epochs without progress" if(currentEpoch<minofcurrentepoch) minofcurrentepoch="currentEpoch;" if(currentEpoch>MaxOfCurrentEpoch) MaxOfCurrentEpoch=currentEpoch; SumOfCurrentEpoch+=currentEpoch; currentEpoch=1; resetCounterFF++; } else currentEpoch++; } //==================================================================== //Another epoch went by.... epochGlob++; } Print("Epochs went by=",epochGlob," Number of resets=",resetCounterFF); Print("MinOfCurrentEpoch",MinOfCurrentEpoch, " AverageOfCurrentEpoch",NormalizeDouble((double)SumOfCurrentEpoch/(double)resetCounterFF,2), " MaxOfCurrentEpoch",MaxOfCurrentEpoch); Print(ChrCountInHistory," - Unique chromosome"); Print(AmountStartsFF," - Total number of launches of FF"); Print(TotalOfChromosomesInHistory," - Total number of chromosomes in the history"); Print(NormalizeDouble(100.0-((double)ChrCountInHistory*100.0/(double)TotalOfChromosomesInHistory),2),"% of duplicates"); Print(Chromosome[0]," - best result"); } //————————————————————————————————————————————————————————————————————————</chromosome[</chromosomecount*</chromosomecount*
3.3. Criando uma proto-população
Já que na maioria dos problemas de otimização, não há como saber com antecedência onde a função argumentos está localizada na linha do número, a melhor opção otimizada é a geração aleatória dentro de um dado intervalo.
//———————————————————————————————————————————————————————————————————————— //Creating a proto- population void ProtopopulationBuilding() { PopulChromosCount=ChromosomeCount*2; //Fill up the population with chromosomes with random //...genes in the range between RangeMinimum...RangeMaximum for(int chromos=0;chromos<populchromoscount;chromos++) {="" //beginning with the 1st indexes (the 0 index is reserved for VFF) for(int gene=1;gene<=GeneCount;gene++) Population[gene][chromos]= NormalizeDouble(SelectInDiscreteSpace(RNDfromCI(RangeMinimum,RangeMaximum),RangeMinimum,RangeMaximum,Precision,3),GeneNormalizeDigits); TotalOfChromosomesInHistory++; } } //————————————————————————————————————————————————————————————————————————
3.4. GetFitness. Obtendo aptidão
Executa a função otimizada para cada cromossomo em ordem.
//------------------------------------------------ ------------------------ // Getting the fitness for each individual. void GetFitness ( double &historyHromosomes[][100000] ) { for(int chromos=0;chromos<chromosomecount;chromos++) checkhistorychromosomes(chromos,historyhromosomes);="" }="" //————————————————————————————————————————————————————————————————————————
3.5. CheckHistoryChromosomes. Verificação do cromossomo através da base do cromossomo
O cromossomo de cada indivíduo é verificado através da base - se o FF tiver sido calculado para ele e, se foi, então o VFF pronto é retirado da base, se não, o FF é acionado para isto. Desta forma, o recurso repetido - cálculos intensivos de FFF são excluídos.
//———————————————————————————————————————————————————————————————————————— //Verification of chromosome through the chromosome base. void CheckHistoryChromosomes ( int chromos, double &historyHromosomes[][100000] ) { //-----------------------Variables------------------------------------- int Ch1=0; //Index of the chromosome from the base int Ge =0; //Index of the gene int cnt=0; //Counter of unique genes. If at least one gene is different //- the chromosome is acknowledged unique //---------------------------------------------------------------------- //If at least one chromosome is stored in the base if(ChrCountInHistory>0) { //Enumerate the chromosomes in the base to find an identical one for(Ch1=0;Ch1<chrcountinhistory &&="" cnt<genecount;ch1++)="" {="" cnt="0; //Compare the genes, while the gene index is less than the number of genes and while there are identical genes for(Ge=1;Ge<=GeneCount;Ge++) { if(Colony[Ge][chromos]!=historyHromosomes[Ge][Ch1]) break; cnt++; } } //If there are enough identical genes then we can use a ready- made solution from the base if(cnt==GeneCount) Colony[0][chromos]=historyHromosomes[0][Ch1-1]; //If there is no such chromosome, then we calculate the FF for it... else { FitnessFunction(chromos); //.. and if there is space in the base, then save it if(ChrCountInHistory<100000) { for(Ge=0;Ge<=GeneCount;Ge++) historyHromosomes[Ge][ChrCountInHistory]=Colony[Ge][chromos]; ChrCountInHistory++; } } } //If the base is empty, calculate the FF for it and save it in the base else { FitnessFunction(chromos); for(Ge=0;Ge<=GeneCount;Ge++) historyHromosomes[Ge][ChrCountInHistory]=Colony[Ge][chromos]; ChrCountInHistory++; } } //————————————————————————————————————————————————————————————————————————
3.6. CycleOfOperators. Ciclo de operadores em UGA
Neste ponto, o destino de literalmente um período inteiro de vida artificial está sendo decidido - uma nova geração nasce e morre. Isto acontece da seguinte maneira: dois pais são selecionados para acasalamento, ou um para praticar ato de mutações sobre si. Para cada operador de GA, um parâmetro apropriado é determinado. Como resultado, obtemos uma descendência. Isto é repetido por várias vezes, até que a colônia de descendentes esteja completamente preenchida. Então, a colônia de descendentes é liberada dentro do habitat, de modo que cada indivíduo possa demonstrar-se tão bem quanto consiga, e nós calculamos o VFF.
Após ser testado por "fogo, água e tubos de cobre", a colônia de descendentes está assentada na população. O próximo passo na evolução artificial será o homicídio sagrado de clones, para evitar o esgotamento de "sangue" em gerações futuras e a classificação subsequente da população renovada pelo grau de aptidão.
//———————————————————————————————————————————————————————————————————————— //Cycle of operators of UGA void CycleOfOperators ( double &historyHromosomes[][100000], //--- double ReplicationPortion, //Proportion of replications. double NMutationPortion, //Proportion of natural mutations. double ArtificialMutation, //Proportion of artificial mutations. double GenoMergingPortion, //Portion of borrowed genes. double CrossingOverPortion,//Proportion of cross-over. //--- double ReplicationOffset, //Coefficient of displacement of interval borders double NMutationProbability//Probability of mutation of each gene in % ) { //-----------------------Variables------------------------------------- double child[]; ArrayResize (child,GeneCount+1); ArrayInitialize(child,0.0); int gene=0,chromos=0, border=0; int i=0,u=0; double p=0.0,start=0.0; double fit[][2]; ArrayResize (fit,6); ArrayInitialize(fit,0.0); //Counter of planting spots in a new population. int T=0; //---------------------------------------------------------------------- //Set a portion of operators of UGA double portion[6]; portion[0]=ReplicationPortion; //Proportion of replications. portion[1]=NMutationPortion; //Proportion of natural mutations. portion[2]=ArtificialMutation; //Proportion of artificial mutations. portion[3]=GenoMergingPortion; //Proportion of borrowed genes. portion[4]=CrossingOverPortion;//Proportion of cross- overs. portion[5]=0.0; //------------------------Cycle of operators of UGA--------- //Fill up the new colony with offspring while(T<chromosomecount) {="" //============================ for(i=0;i<6;i++) { fit[i][0]=start; fit[i][1]=start+MathAbs(portion[i]-portion[5]); start=fit[i][1]; } p=RNDfromCI(fit[0][0],fit[4][1]); for(u=0;u<5;u++) { if((fit[u][0]<=p && p<fit[u][1]) || p==fit[u][1]) break; } //============================ switch(u) { //--------------------- case 0: //------------------------Replication-------------------------------- //If there is space in the new colony, create a new individual if(T<chromosomecount) {="" replication(child,replicationoffset);="" //Settle the new individual into the new colony for(gene=1;gene<=GeneCount;gene++) Colony[gene][T]=child[gene]; //One place is occupied, fast- forward the counter T++; TotalOfChromosomesInHistory++; } //--------------------------------------------------------------- break; //--------------------- case 1: //---------------------Natural mutation------------------------- //If there is space in the new colony, create a new individual if(T<chromosomecount) {="" naturalmutation(child,nmutationprobability);="" //Settle the new individual into the new colony for(gene=1;gene<=GeneCount;gene++) Colony[gene][T]=child[gene]; //One place is occupied, fast- forward the counter T++; TotalOfChromosomesInHistory++; } //--------------------------------------------------------------- break; //--------------------- case 2: //----------------------Artificial mutation----------------------- //If there is space in the new colony, create a new individual if(T<chromosomecount) {="" artificialmutation(child,replicationoffset);="" //Settle the new individual into the new colony for(gene=1;gene<=GeneCount;gene++) Colony[gene][T]=child[gene]; //One place is occupied, fast-forward the counter T++; TotalOfChromosomesInHistory++; } //--------------------------------------------------------------- break; //--------------------- case 3: //-------------The creation of an individual with borrowed genes----------- //If there is space in the new colony, create a new individual if(T<chromosomecount) {="" genomerging(child);="" //Settle the new individual into the new colony for(gene=1;gene<=GeneCount;gene++) Colony[gene][T]=child[gene]; //One space is occupied, fast forward the counter T++; TotalOfChromosomesInHistory++; } //--------------------------------------------------------------- break; //--------------------- default: //---------------------------Crossing-Over--------------------------- //If there is place in the new colony, create a new individual if(T<chromosomecount) {="" crossingover(child);="" //Settle the new individual into the new colony for(gene=1;gene<=GeneCount;gene++) Colony[gene][T]=child[gene]; //One place is occupied, fast forward the counter T++; TotalOfChromosomesInHistory++; } //--------------------------------------------------------------- break; //--------------------- } }//End of the cycle operators of UGA-- //Determine the fitness of each individual in the colony of offspring GetFitness(historyHromosomes); //Settle the offspring into the main population if(PopulChromosCount>=ChromosomeCount) { border=ChromosomeCount; PopulChromosCount=ChromosomeCount*2; } else { border=PopulChromosCount; PopulChromosCount+=ChromosomeCount; } for(chromos=0;chromos<chromosomecount;chromos++) for(gene=0;gene<=GeneCount;gene++) Population[gene][chromos+border]=Colony[gene][chromos]; //Prepare the population for the next reproduction RemovalDuplicates(); }//the end of the function //————————————————————————————————————————————————————————————————————————</fit[u][
3.7. Réplica
O operador está mais próximo ao fenômeno natural, que na biologia é chamado - réplica de DNA, embora, na essência, não seja a mesma coisa. Mas já que não encontrei nenhum equivalente, mais próximo a isto na natureza, decidi manter este título.
A réplica é o operador genético mais importante, que gera novos genes, enquanto transmite os traços dos cromossomos pais. O operador principal, garantindo a convergência do algoritmo. O GA pode funcionar com ele apenas sem a utilização de outros operadores, mas neste caso o número de lançamentos de FF seria muito maior.
Considere o princípio do operador de réplica. Utilizamos dois cromossomos pais. O novo gene da descendência é um número aleatório a partir do intervalo
[C1-((C2-C1)*ReplicationOffset),C2+((C2-C1)*ReplicationOffset)]
no qual os genes pais C1 e C2 ReplicationOffset - Coeficiente de deslocamento das margens internas [C1, C2].
Por exemplo, a partir do indivíduo paternal (azul) e o indivíduo maternal (rosa) uma criança (verde) pode ser criada:
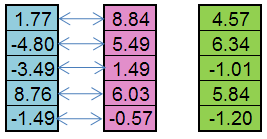
Figura 5. O princípio do trabalho de Réplica do operador
Graficamente, a probabilidade do gene da descendência pode ser resumida:
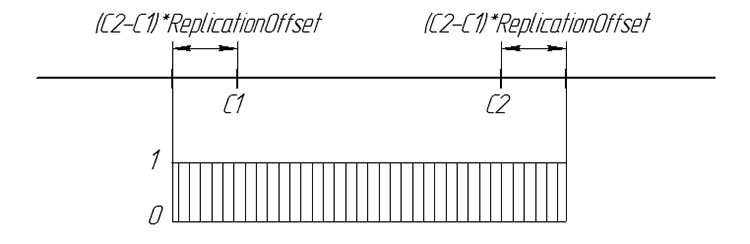
Figura 6. A probabilidade do aparecimento do gene da descendência em uma linha de número
Os outros genes da descendência são gerados da mesma maneira.
//------------------------------------------------ ------------------------ // Replication void Replication ( double &child[], double ReplicationOffset ) { //-----------------------Variables------------------------------------- double C1=0.0,C2=0.0,temp=0.0,Maximum=0.0,Minimum=0.0; int address_mama=0,address_papa=0; //---------------------------------------------------------------------- SelectTwoParents(address_mama,address_papa); //-------------------Cycle of gene enumeration-------------------------------- for(int i=1;i<=GeneCount;i++) { //----figure out where the father and mother came from -------- C1 = Population[i][address_mama]; C2 = Population[i][address_papa]; //------------------------------------------ //Mandatory verification to make sure that the search had not gone over the specified range if(C1 < RangeMinimum) C1 = RangeMinimum; if(C1 > RangeMaximum) C1 = RangeMaximum; if(C2 < RangeMinimum) C2 = RangeMinimum; if(C2 > RangeMaximum) C2 = RangeMaximum; //------------------------------------------------------------------ //....determine the largest and smallest of them, //if we С1>C2, swi if(C1>C2) { temp=C1; C1=C2; C2=temp; } //-------------------------------------------- //Specify the borders of the created gene Minimum = C1-((C2-C1)*ReplicationOffset); Maximum = C2+((C2-C1)*ReplicationOffset); //-------------------------------------------- //Mandatory verification to make sure that the search has not gone over the specified range if(Minimum < RangeMinimum) Minimum = RangeMinimum; if(Maximum > RangeMaximum) Maximum = RangeMaximum; //--------------------------------------------------------------- temp=RNDfromCI(Minimum,Maximum); child[i]= NormalizeDouble(SelectInDiscreteSpace(temp,RangeMinimum,RangeMaximum,Precision,3),GeneNormalizeDigits); } } //————————————————————————————————————————————————————————————————————————3.8. NaturalMutation. Mutação natural
As mutações podem ocorrer de forma constante ao longo do curso dos processos, ocorrendo em células vivas e servem como material para seleção natural. Elas surgem espontaneamente ao longo de toda a vida do organismo em suas condições de habitat normal, com uma frequência de uma vez por 10 gerações de células.
Nós - os pesquisadores curiosos, não necessariamente precisamos aderir à ordem natural e esperar tanto tempo para a próxima mutação do gene. O parâmetro NMutationProbability que é expresso como um percentual e determina a probabilidade de mutação para cada gene no cromossomo nos ajudará a fazer isto.
No operador NaturalMutation, a mutação consiste da geração de um gene aleatório no intervalo [RangeMinimum, RangeMaximum] . NMutationProbability = 100% significaria uma mutação de 100% de todos os genes no cromossomo e NMutationProbability = 0% - ausência completa de mutações. A última opção não está apta a ser usada em problemas práticos.
//------------------------------------------------ ------------------------ // The natural mutation. void NaturalMutation ( double &child[], double NMutationProbability ) { //-----------------------Variables------------------------------------- int address=0; double prob=0.0; //---------------------------------------------------------------------- if(NMutationProbability<0.0) prob=0.0; if(NMutationProbability>100.0) prob=100.0; //-----------------Parent selection------------------------ SelectOneParent(address); //--------------------------------------- for(int i=1;i<=GeneCount;i++) if(RNDfromCI(0.0,100.0)<prob) child[i]="NormalizeDouble( SelectInDiscreteSpace(RNDfromCI(RangeMinimum,RangeMaximum),RangeMinimum,RangeMaximum,Precision,3),GeneNormalizeDigits ); } //————————————————————————————————————————————————————————————————————————
3.9. ArtificialMutation. Mutação artificial
A tarefa principal do operador - é a geração de sangue "novo". Nós usamos dois pais e os genes dos descendentes são selecionados a partir, não alocados por genes pais, dos espaços na linha do número. Protege o GA de ficar preso em um extremo do local. Em uma proporção maior, comparado a outros operadores, acelera a convergência, ou se não - desacelera, aumentando o número de lançamentos de FF.
Assim como na Réplica, usamos dois cromossomos pais. Mas a tarefa do operador do ArtificialMutation não é transportar os traços parentais para a prole, mas sim, fazer a criança diferente deles. Portanto, sendo um oposto completo, utilizando o mesmo coeficiente de intervalo de deslocamento de margem, mas os genes são gerados fora do intervalo, o que seria tomado como Réplica. O novo gene da descendência é um número aleatório a partir dos intervalos [RangeMinimum, C1-(C2-C1) * ReplicationOffset] e [C2 + (C2-C1) * ReplicationOffset, RangeMaximum].
Graficamente, a probabilidade de um gene na descendência ReplicationOffset = 0,25 pode ser representado como:
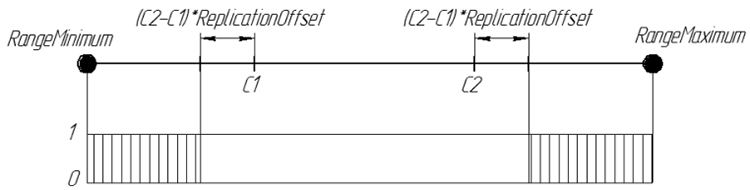
Figura 7. A probabilidade de um gene no ReplicationOffset do descendente = 0,25 no intervalo de linha real [RangeMinimum; RangeMaximum]
//———————————————————————————————————————————————————————————————————————— //Artificial mutation. void ArtificialMutation ( double &child[], double ReplicationOffset ) { //-----------------------Variables------------------------------------- double C1=0.0,C2=0.0,temp=0.0,Maximum=0.0,Minimum=0.0,p=0.0; int address_mama=0,address_papa=0; //---------------------------------------------------------------------- //-----------------Selecting parents------------------------ SelectTwoParents(address_mama,address_papa); //-------------------------------------------------------- //-------------------Cycle of genes enumeration------------------------------ for(int i=1;i<=GeneCount;i++) { //----determine where the mother and father are from -------- C1 = Population[i][address_mama]; C2 = Population[i][address_papa]; //------------------------------------------ //Mandatory verification to make sure that the search doesn't go beyond the specified range if(C1 < RangeMinimum) C1 = RangeMinimum; if(C1 > RangeMaximum) C1 = RangeMaximum; if(C2 < RangeMinimum) C2 = RangeMinimum; if(C2 > RangeMaximum) C2 = RangeMaximum; //------------------------------------------------------------------ //....determine the largest and smallest of them, //if С1>C2, we change their places if(C1>C2) { temp=C1; C1=C2; C2=temp; } //-------------------------------------------- //Specify the borders of creating the new gene Minimum=C1-((C2-C1)*ReplicationOffset); Maximum=C2+((C2-C1)*ReplicationOffset); //-------------------------------------------- //Mandatory verification to make sure that the search doesn't go beyond the specified range if(Minimum < RangeMinimum) Minimum = RangeMinimum; if(Maximum > RangeMaximum) Maximum = RangeMaximum; //--------------------------------------------------------------- p=MathRand(); if(p<16383.5) { temp=RNDfromCI(RangeMinimum,Minimum); child[i]= NormalizeDouble(SelectInDiscreteSpace(temp,RangeMinimum,RangeMaximum,Precision,3),GeneNormalizeDigits); } else { temp=RNDfromCI(Maximum,RangeMaximum); child[i]= NormalizeDouble(SelectInDiscreteSpace(temp,RangeMinimum,RangeMaximum,Precision,3),GeneNormalizeDigits); } } } //————————————————————————————————————————————————————————————————————————
3.10 GenoMerging. Pegando genes emprestado
O determinado operador de GA não possui um equivalente natural. É, de fato, difícil imaginar como este maravilhoso mecanismo funcionaria em organismos vivos. No entanto, ele possui uma notável propriedade de transferir genes a partir de um número de pais (o número de pais é igual ao número de genes) para a prole. O operador não gera novos genes e é um mecanismo de busca combinatório.
Funciona assim: para o primeiro gene da prole, um pai é selecionado e o primeiro gene é tomado dele, então, para o segundo gene, um segundo pai é selecionado e o gene é tomado dele, etc. Isto é aconselhável que se aplique caso o número de genes seja mais alto do que um. Do contrário, ele deve ser desabilitado, já que o operador gerará duplicatas dos cromossomos.
//———————————————————————————————————————————————————————————————————————— //Borrowing genes. void GenoMerging ( double &child[] ) { //-----------------------Variables------------------------------------- int address=0; //---------------------------------------------------------------------- for(int i=1;i<=GeneCount;i++) { //-----------------Selecting parents------------------------ SelectOneParent(address); //-------------------------------------------------------- child[i]=Population[i][address]; } } //————————————————————————————————————————————————————————————————————————
3.11. CrossingOver. Crossing-over
O crossing-over (também conhecido como cruzamento) - é um fenômeno de troca de seções de cromossomos. Assim como no GenoMerging, este é um mecanismo de busca combinatória.
Dois cromossomos parentais são selecionados. Ambos são "cortados" em um lugar aleatório. O cromossomo da prole consistirá de partes dos cromossomos parentais.
É o mais fácil para ilustrar este mecanismo em uma imagem:
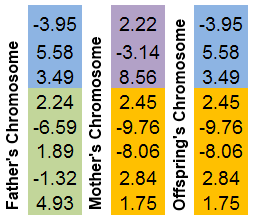
Figura 8. O mecanismo de troca de partes do cromossomo
Isto é aconselhável que se aplique se o número de genes for mais alto do que um. Do contrário, ele deve ser desabilitado, já que o operador gerará duplicatas dos cromossomos.
//———————————————————————————————————————————————————————————————————————— //Crossing-over. void CrossingOver ( double &child[] ) { //-----------------------Variables------------------------------------- int address_mama=0,address_papa=0; //---------------------------------------------------------------------- //-----------------Selecting parents------------------------ SelectTwoParents(address_mama,address_papa); //-------------------------------------------------------- //Determine the breakage point int address_of_gene=(int)MathFloor((GeneCount-1)*(MathRand()/32767.5)); for(int i=1;i<=GeneCount;i++) { //----copy the mother's genes-------- if(i<=address_of_gene+1) child[i]=Population[i][address_mama]; //----copy the father's genes-------- else child[i]=Population[i][address_papa]; } } //————————————————————————————————————————————————————————————————————————
3.12. SelectTwoParents. A seleção de dois pais
Para evitar o esgotamento do conjunto de gene, há um banimento ao cruzamento consigo mesmo. São feitas dez tentativas para encontrar pais diferentes e, se falharmos em encontrar um par, permitimos o cruzamento consigo mesmo. Basicamente, obtemos uma cópia do mesmo espécime.
Por um lado, a probabilidade de clonar indivíduos diminui, por outro - evita-se a circularidade da busca, já que uma situação pode surgir, na qual seria praticamente impossível fazer isto (escolher pais diferentes) em um número razoável de passos.
Utilizado nos operadores de Réplica, ArtificialMutation e CrossingOver.
//———————————————————————————————————————————————————————————————————————— //Selection of two parents. void SelectTwoParents ( int &address_mama, int &address_papa ) { //-----------------------Variables------------------------------------- int cnt=1; address_mama=0;//address of the mother individual in a population address_papa=0;//address of the father individual in a population //---------------------------------------------------------------------- //----------------------------Selection of parents-------------------------- //Ten attempts to chose different parents. while(cnt<=10) { //For the mother individual address_mama=NaturalSelection(); //For the father individual address_papa=NaturalSelection(); if(address_mama!=address_papa) break; } //--------------------------------------------------------------------- } //————————————————————————————————————————————————————————————————————————
3.13. SelectOneParent. A seleção de um pai
Aqui tudo é simples - um pai é selecionado a partir da população.
Utilizado em operadores de NaturalMutation e GenoMerging.
//———————————————————————————————————————————————————————————————————————— //Selection of one parent. void SelectOneParent ( int &address//address of the parent individual in the population ) { //-----------------------Variables------------------------------------- address=0; //---------------------------------------------------------------------- //----------------------------Selecting a parent-------------------------- address=NaturalSelection(); //--------------------------------------------------------------------- } //————————————————————————————————————————————————————————————————————————
3.14. NaturalSelection. Seleção natural
Seleção natural - o processo que leva à sobrevivência e reprodução preferencial de indivíduos, melhor adaptadores a estas condições ambientais, processando traços hereditários úteis.
O operador é semelhante ao operador tradicional de "Roleta" (Seleção por roleta - Seleção de indivíduos com n "lançamentos" da roleta. A roleta contém um setor para cada membro da população. O tamanho do setor i-th é proporcional ao valor correspondente da aptidão), mas possui diferenças significativas. Ele leva em consideração a posição do indivíduo, relativo aos mais e aos menos apropriados. Além disso, mesmo que um indivíduo que possui os piores genes, tem a chance de deixar uma descendência. Isto é justo, não é? Embora isto não seja sobre justiça, mas na verdade sobre o fato que, na natureza, todos os indivíduos têm a oportunidade de deixar prole.
Por exemplo, tome-se 10 indivíduos, que tenham o seguinte VFF no problema de maximização: 256, 128, 64, 32, 16, 8, 4, 2, 0, -1 - no qual os maiores valores correspondem à melhor aptidão. Este exemplo é tomado de modo que possamos ver que a "distância" entre indivíduos vizinhos é 2 vezes maior do que entre os dois indivíduos anteriores. No entanto, no gráfico de pizza, a probabilidade de cada indivíduo deixar uma prole é conforme segue:
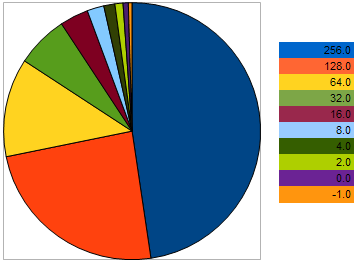
Figura 9. O quadro de probabilidade de selecionar os indivíduos pais
ele demonstra que com a aproximação de indivíduos para o pior, as suas chances se tornam piores. Reciprocamente - quanto mais próximo o indivíduo fica do melhor espécime, melhores chances de reprodução ele tem.

Figura 10. O quadro de probabilidade de selecionar os indivíduos pais
//———————————————————————————————————————————————————————————————————————— //Natural selection. int NaturalSelection() { //-----------------------Variables------------------------------------- int i=0,u=0; double p=0.0,start=0.0; double fit[][2]; ArrayResize (fit,PopulChromosCount); ArrayInitialize(fit,0.0); double delta=(Population[0][0]-Population[0][PopulChromosCount-1])*0.01-Population[0][PopulChromosCount-1]; //---------------------------------------------------------------------- for(i=0;i<populchromoscount;i++) {="" fit[i][0]=start; fit[i][1]=start+MathAbs(Population[0][i]+delta); start=fit[i][1]; } p=RNDfromCI(fit[0][0],fit[PopulChromosCount-1][1]); for(u=0;u<populchromoscount;u++) if((fit[u][0]<=p && p<fit[u][1]) || p==fit[u][1]) break; return(u); } //————————————————————————————————————————————————————————————————————————</fit[u][
3.15. RemovalDuplicates. Remoção de duplicatas
A função remove cromossomos duplicados na população e os cromossomos únicos restantes (únicos para a população do período atual) são classificados em ordem por VFF, determinado pelo tipo de otimização, ou seja, diminuindo ou aumentando.
//———————————————————————————————————————————————————————————————————————— //Removing duplicates sorted by VFF void RemovalDuplicates() { //-----------------------Variables------------------------------------- int chromosomeUnique[1000];//Array stores the unique trait //of each chromosome: 0-duplicate, 1-unique ArrayInitialize(chromosomeUnique,1); //Assume that there are no duplicates double PopulationTemp[][1000]; ArrayResize (PopulationTemp,GeneCount+1); ArrayInitialize(PopulationTemp,0.0); int Ge =0; //Index of the gene int Ch =0; //Index of the chromosome int Ch2=0; //Index of the second chromosome int cnt=0; //Counter //---------------------------------------------------------------------- //----------------------Remove duplicates---------------------------1 //Chose the first from the pair for comparison... for(Ch=0;Ch<populchromoscount;ch++) {="" //If it's not a duplicate... if(chromosomeUnique[Ch]!=0) { //Chose the second from the pair... for(Ch2=0;Ch2<populchromoscount;ch2++) {="" if(Ch!=Ch2 && chromosomeUnique[Ch2]!=0) { //Zeroize the counter of identical genes cnt=0; //Compare the genes. while there are identical genes present for(Ge=1;Ge<=GeneCount;Ge++) { if(Population[Ge][Ch]!=Population[Ge][Ch2]) break; else cnt++; } //If there are the same amount of identical genes as total genes //..the chromosome is considered a duplicate if(cnt==GeneCount) chromosomeUnique[Ch2]=0; } } } } //The counter calculates the number of unique chromosomes cnt=0; //Copy the unique chromosomes into a temporary array for(Ch=0;Ch<populchromoscount;ch++) {="" //If the chromosome is unique, copy it, if not, go to the next if(chromosomeUnique[Ch]==1) { for(Ge=0;Ge<=GeneCount;Ge++) PopulationTemp[Ge][cnt]=Population[Ge][Ch]; cnt++; } } //Assigning the variable "All chromosomes" the value of counter of unique chromosomes PopulChromosCount=cnt; //Return unique chromosomes back to the array for temporary storage //..of combined populations for(Ch=0;Ch<populchromoscount;ch++) for(Ge=0;Ge<=GeneCount;Ge++) Population[Ge][Ch]=PopulationTemp[Ge][Ch]; //=================================================================1 //----------------Ranking the population---------------------------2 PopulationRanking(); //=================================================================2 } //————————————————————————————————————————————————————————————————————————
3.16. PopulationRanking. Classificação da população
A classificação é feita pelo VFF. O método é semelhante ao 'bubbly. O algoritmo consiste de passagens repetidas através da série classificada. Para cada passe os elementos são sucessivamente comparados por pares e, se a ordem de um par estiver errada, ocorre uma troca de lugares. Passagens através de série são repetidas até que uma das passagens mostre que as trocas não são mais necessárias, o que significa que a série foi classificada.
Ao passar por um algoritmo, um elemento que está fora do lugar, "aparece" na posição desejada, assim como uma bolha na água, desta forma, o nome do algoritmo, mas há uma diferença - apenas os índices da série são classificados, não os conteúdos da série. Este método é mais rápido e levemente diferente em velocidade do que simplesmente copiar uma série para outra. Quanto maior o tamanho da série classificada, menor é a diferença.
//———————————————————————————————————————————————————————————————————————— //Population ranking. void PopulationRanking() { //-----------------------Variables------------------------------------- int cnt=1, i = 0, u = 0; double PopulationTemp[][1000]; //Temporary population ArrayResize (PopulationTemp,GeneCount+1); ArrayInitialize(PopulationTemp,0.0); int Indexes[]; //Indexes of chromosomes ArrayResize (Indexes,PopulChromosCount); ArrayInitialize(Indexes,0); int t0=0; double ValueOnIndexes[]; //VFF of corresponding //..chromosome indexes ArrayResize (ValueOnIndexes,PopulChromosCount); ArrayInitialize(ValueOnIndexes,0.0); double t1=0.0; //---------------------------------------------------------------------- //Fill in the indexes in the temporary array temp2 and //...copy the first line from the sorted array for(i=0;i<populchromoscount;i++) {="" indexes[i]="i;" valueonindexes[i]="Population[0][i]; } if(OptimizeMethod==1) { while(cnt>0) { cnt=0; for(i=0;i<populchromoscount-1;i++) { if(ValueOnIndexes[i]>ValueOnIndexes[i+1]) { //----------------------- t0 = Indexes[i+1]; t1 = ValueOnIndexes[i+1]; Indexes [i+1] = Indexes[i]; ValueOnIndexes [i+1] = ValueOnIndexes[i]; Indexes [i] = t0; ValueOnIndexes [i] = t1; //----------------------- cnt++; } } } } else { while(cnt>0) { cnt=0; for(i=0;i<populchromoscount-1;i++) { if(ValueOnIndexes[i]<valueonindexes[i+1]) { //----------------------- t0 = Indexes[i+1]; t1 = ValueOnIndexes[i+1]; Indexes [i+1] = Indexes[i]; ValueOnIndexes [i+1] = ValueOnIndexes[i]; Indexes [i] = t0; ValueOnIndexes [i] = t1; //----------------------- cnt++; } } } } //Create a sorted-out array based on the obtained indexes for(i=0;i<genecount+1;i++) for(u=0;u<populchromoscount;u++) populationtemp[i][u]="Population[i][Indexes[u]];" //Copy the sorted-out array back for(i=0;i<genecount+1;i++) for(u=0;u<populchromoscount;u++) population[i][u]="PopulationTemp[i][u];" }="" //————————————————————————————————————————————————————————————————————————</genecount+</genecount+</valueonindexes[i+</populchromoscount-</populchromoscount-
3.17. RNDfromCustomInterval. A geração de números aleatórios a partir de um dado intervalo
Apenas um atributo conveniente. É conveniente no UGA.
//———————————————————————————————————————————————————————————————————————— //Generator of random numbers from the selected interval. double RNDfromCI(double RangeMinimum,double RangeMaximum) { return(RangeMinimum+((RangeMaximum-RangeMinimum)*MathRand()/32767.5));} //————————————————————————————————————————————————————————————————————————
3.18. SelectInDiscreteSpace. A seleção em espaço discreto
É utilizado para reduzir o espaço de pesquisa. Com o parâmetro etapa = 0,0 a busca é realizada em um espaço contínuo (limitado às limitações de linguagem, em MQL até o 15º símbolo significativo incluso). A utilização do algoritmo do GA com uma precisão maior, você precisa escrever uma biblioteca adicional para trabalhar com número mais longos.
O trabalho da função em RoundMode = 1 pode ser ilustrado pela seguinte figura:
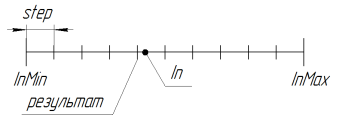
Figura 11. o trabalho da função SelectInDiscreteSpace em RoundMode = 1
//———————————————————————————————————————————————————————————————————————— //Selection in discrete space. //Modes: //1-closest below //2-closest above //any closest double SelectInDiscreteSpace ( double In, double InMin, double InMax, double step, int RoundMode ) { if(step==0.0) return(In); // secure the correctness of borders if( InMax < InMin ) { double temp = InMax; InMax = InMin; InMin = temp; } // during a breach - return the breached border if( In < InMin ) return( InMin ); if( In > InMax ) return( InMax ); if( InMax == InMin || step <= 0.0 ) return( InMin ); // bring to the specified scale step = (InMax - InMin) / MathCeil ( (InMax - InMin) / step ); switch( RoundMode ) { case 1: return( InMin + step * MathFloor ( ( In - InMin ) / step ) ); case 2: return( InMin + step * MathCeil ( ( In - InMin ) / step ) ); default: return( InMin + step * MathRound ( ( In - InMin ) / step ) ); } } //————————————————————————————————————————————————————————————————————————
3.19. FitnessFunction. Função aptidão
Não faz parte do GA. A função recebe o índice do cromossomo na população, para a qual o VFF será calculado. O VFF é escrito no índice zero do cromossomo transmitido. O código desta função é único para cada tarefa.
3,20. ServiceFunction. Função serviço
Não faz parte do GA. O código desta função é único para cada tarefa. Pode ser usado para implementar controles sobre períodos. Por exemplo, a fim de exibir o melhor VFF para o período corrente.
4. Exemplos de trabalho de UGA
Todos os problemas de otimização são resolvidos pelos meios de EA e são divididos em dois tipos:
- O genótipo consiste em um fenótipo. Os valores dos genes do cromossomo são apontados diretamente pelos argumentos de uma função de otimização. Exemplo 1.
- O genótipo não corresponde ao fenótipo. A interpretação do significado dos genes do cromossomo é exigida para calcular a função otimizada. Exemplo 2.
4.1. Exemplo 1
Considere o problema com uma resposta conhecida, a fim de certificar-se que o algoritmo funciona e, então, siga adiante para resolver o problema, a solução do qual é de interesse para muitos negociadores.
Problema: Encontrar a função mínima e máxima "Pele":
no segmento [-5, 5].
Resposta: fmin (3.07021,3.315935) = -4.3182, fmax (-3.315699; -3.072485) = 14.0606.
Figura 12. O gráfico da "Pele" no segmento [-5, 5]
Para resolver o problema, escrevemos o seguinte script:
#property script_show_inputs //+——————————————————————————————————————————————————————————————————————+ #include "UGAlib.mqh" #include "Skin.mqh"//testing function //+——————————————————————————————————————————————————————————————————————+ //———————————————————————————————————————————————————————————————————————— //----------------------Incoming parameters-------------------------------- input string GenofundParam = "----Gene pool parameter----"; input int ChromosomeCount_P = 50; //Number of chromosomes in a colony input int GeneCount_P = 2; //Number of genes input int FFNormalizeDigits_P = 4; //Number of fitness symbols input int GeneNormalizeDigits_P = 4; //Number of genes input int Epoch_P = 50; //Number of epochs without progress //--- input string GA_OperatorParam = "----Operator parameters----"; input double ReplicationPortion_P = 100.0; //Proportion of replication. input double NMutationPortion_P = 10.0; //Proportion of natural mutations. input double ArtificialMutation_P = 10.0; //Proportion of artificial mutations. input double GenoMergingPortion_P = 20.0; //Proportion of borrowed genes. input double CrossingOverPortion_P = 20.0; //Proportion of crossing-over. //--- input double ReplicationOffset_P = 0.5; //Coefficient of interval borders displacement input double NMutationProbability_P= 5.0; //Probability of mutation of each gene in % //--- input string OptimisationParam = "----Optimization parameters----"; input double RangeMinimum_P = -5.0; //Minimum range search input double RangeMaximum_P = 5.0; //Maximum range search input double Precision_P = 0.0001;//The required accuracy input int OptimizeMethod_P = 1; //Optim.:1-Min,other-Max //———————————————————————————————————————————————————————————————————————— //———————————————————————————————————————————————————————————————————————— //----------------------Global variables----------------------------- double ERROR=0.0;//Average error in gen //———————————————————————————————————————————————————————————————————————— //———————————————————————————————————————————————————————————————————————— //--------------------------The body of the program-------------------------------- int OnStart() { //-----------------------Variables------------------------------------- //Preparing global variables for UGA ChromosomeCount=ChromosomeCount_P; //Number of chromosomes in the colony GeneCount =GeneCount_P; //Number of genes RangeMinimum =RangeMinimum_P; //Minimum range search RangeMaximum =RangeMaximum_P; //Maximum range search Precision =Precision_P; //Search step OptimizeMethod =OptimizeMethod_P; //1-minimum, any other - maximum FFNormalizeDigits = FFNormalizeDigits_P; //Number of symbols in fitness GeneNormalizeDigits = GeneNormalizeDigits_P;//Number of gene symbols ArrayResize(Chromosome,GeneCount+1); ArrayInitialize(Chromosome,0); Epoch=Epoch_P; //Number of epochs without progress //---------------------------------------------------------------------- //Local variables int time_start=GetTickCount(),time_end=0; //---------------------------------------------------------------------- //Launch of the main function UGA UGA ( ReplicationPortion_P, //Proportion of replication. NMutationPortion_P, //Proportion of natural mutations. ArtificialMutation_P, //Proportion of artificial mutations. GenoMergingPortion_P, //Proportion of borrowed genes. CrossingOverPortion_P,//Proportion of crossing-over. //--- ReplicationOffset_P, //Coefficient of interval border replacement NMutationProbability_P//Probability of mutation of each gene in % ); //---------------------------------- time_end=GetTickCount(); //---------------------------------- Print(time_end-time_start," mc - Time of implementation"); //---------------------------------- return(0); } //————————————————————————————————————————————————————————————————————————
Aqui está o código inteiro do script para resolver o problema. Execute-o, obtenha a informação, fornecida pelo função Comentário ():
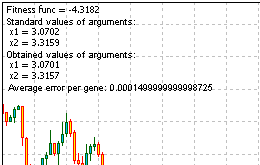
Figura 13. O resultado da solução do problema
Olhando para os resultados, vemos que o algoritmo funciona.
4.2. Exemplo 2
Acredita-se amplamente que o indicador ZZ mostra as entradas ideais de um sistema de negociação anulado. O indicador é muito popular dentre os entusiastas da "teoria da onda" e aqueles que o utilizam para determinar o tamanho das "figuras".
Problema: Determinar se há alguns outros pontos de entrada para um sistema de negociação anulado em dados históricos, diferentes dos vértices ZZ, gerando em soma mais pontos de ganho teórico?
Para os experimentos, selecionaremos um par GBPJPY para barras M1 100. Aceitar o spread de 80 pontos (quotas de cinco dígitos). Para iniciar, é necessário determinar os melhores parâmetros ZZ. Para fazer isto, utilizamos numeração simples para encontrar o melhor valor do parâmetro ExtDepth, utilizando um script simples:
#property script_show_inputs //———————————————————————————————————————————————————————————————————————— //----------------------Incoming parameters-------------------------------- input int History=100; input double Spred =80.0; input int Depth =5; //For "one-time" use input bool loop =true;//Use enumeration or not //———————————————————————————————————————————————————————————————————————— //———————————————————————————————————————————————————————————————————————— //--------------------------The body of the program-------------------------------- void OnStart() { //-----------------------Variables------------------------------------- double ZigzagBuffer [];//For storing the buffer of the ZZ indicator double PeaksOfZigzag[];//for storing the values of the ZZ extremum int Zigzag_handle; //Indicator marker ArraySetAsSeries(ZigzagBuffer,true); ArrayResize(PeaksOfZigzag,History); int depth=3; double PipsSum=0.0; int PeaksCount=0; bool flag=true; //---------------------------------------------------------------------- if(loop==true) { while(depth<200 && flag==true) { //----------------------------------------------------------- Zigzag_handle=iCustom(NULL,0,"ZigZag",depth); //--- reset the code error ResetLastError(); //--- attempt to copy the indicator values for(int i=0;i<100;i++) { if(BarsCalculated(Zigzag_handle)>0) break; Sleep(1000); } int copied=CopyBuffer(Zigzag_handle,0,0,History,ZigzagBuffer); if(copied<=0) { Print("Could not copy the indicator buffer. Error =",GetLastError()," copied=",copied); return; } //----------------------------------------------------------- PipsSum=0.0; PeaksCount=0; for(int u=0;u<history;u++) {="" if(NormalizeDouble(ZigzagBuffer[u],Digits())>0.0) { PeaksOfZigzag[PeaksCount]=NormalizeDouble(ZigzagBuffer[u],Digits()); PeaksCount++; } } //----------------------------------------------------------- for(int V=0;V<peakscount-1;V++) PipsSum+=NormalizeDouble((MathAbs(PeaksOfZigzag[V]-PeaksOfZigzag[V+1]))/Point(),Digits())-Spred; //----------------------------------------------------------- if(PeaksCount<=2) flag=false; else { Print(depth," ",PeaksCount," ",PipsSum); depth+=1; } //----------------------------------------------------------- } } else { //----------------------------------------------------------- Zigzag_handle=iCustom(NULL,0,"ZigZag",Depth); //--- reser the error code ResetLastError(); //--- attempt to copy the indicator values for(int i=0;i<history;i++) {="" if(BarsCalculated(Zigzag_handle)>0) break; Sleep(1000); } int copied=CopyBuffer(Zigzag_handle,0,0,History,ZigzagBuffer); if(copied<=0) { Print("Was not able to copy the buffer indicator. Error =",GetLastError()," copied=",copied); return; } //----------------------------------------------------------- for(int u=0;u<history;u++) {="" if(NormalizeDouble(ZigzagBuffer[u],Digits())>0.0) { PeaksOfZigzag[PeaksCount]=NormalizeDouble(ZigzagBuffer[u],Digits()); PeaksCount++; } } //----------------------------------------------------------- for(int V=0;V<peakscount-1;V++) { PipsSum+=NormalizeDouble((MathAbs(PeaksOfZigzag[V]-PeaksOfZigzag[V+1]))/Point(),Digits())-Spred; } Print(Depth," ",PeaksCount," ",PipsSum); //----------------------------------------------------------- } } //————————————————————————————————————————————————————————————————————————</peakscount-</peakscount-
Executando o script, obtemos 4077 pontos em ExtDepth = 3. Dezenove vértices de indicadores "aptos" em 100 barras. Com o aumento do ExtDepth, o número de vértices ZZ diminui assim como a lucratividade.
Agora podemos encontrar os vértices do ZZ alternativo, utilizando o UGA. Os vértices ZZ podem ter três posições para cada barra: 1) Alto, 2) Baixo, 3) Sem vértice. A presença e posição do vértice será transportada por cada gene para cada barra. Por isso, o tamanho do cromossomo - 100 genes.
De acordo com meus cálculos (e os matemáticos podem me corrigir se eu estiver errado), em 100 barras pode-se construir 3 ^ 100, ou 5.15378e47 opções alternativas "ziguezagues" . Este é o número exato de opções que precisa ser considerado, utilizando enumeração direta. Durante o cálculo com uma velocidade de 100000000 opções por segundo, precisaremos de 1.6e32 anos! Isto é mais do que a idade do universo. Aqui é quando eu começo a ter dúvidas sobre a habilidade de encontrar uma solução para este problema.
Mas, vamos começar.
Já que o UGA utiliza a representação do cromossomo por números reais, precisamos codificar, de alguma forma, a posição dos vértices. Este é precisamente o caso quando o genótipo do cromossomo não corresponde ao fenótipo. Atribua um intervalo de busca para os genes [0, 5]. Vamos concordar que o intervalo [0, 1] corresponde ao vértice de ZZ em Alto, o intervalo [4, 5] corresponde ao vértice em Baixo, e o intervalo (1, 4) corresponde à Ausência do vértice.
É necessário levar um ponto importante em consideração. Já que a proto-população é gerada de forma aleatória com genes em intervalos específicos, o primeiro espécime terá resultados bem ruins, possivelmente até com algumas centenas de pontos com um sinal de menos. Após algumas gerações (embora exista uma chance de isto acontecer na primeira geração) veremos o aparecimento do espécime, cujos genes são consistentes com a ausência de vértices em geral. Isto significaria a ausência de negociação e o inevitável pagamento do spread.
De acordo com alguns negociadores antigos: "A melhor estratégia para negociar - é não negociar". Este indivíduo será o vértice da evolução artificial. A fim de fazer com que esta evolução "artificial" dê origem a indivíduos negociadores, ou seja, fazer com que obtenha os vértices do ZZ alternativo, atribuímos a aptidão dos indivíduos, faltando vértices, o valor de "-10000000.0", deliberadamente posicionando-o no degrau mais baixo da evolução, comparado a quaisquer outros indivíduos.
Aqui está o código do script que usa o UGA para encontrar os vértices do ZZ alternativo:
#property script_show_inputs //+——————————————————————————————————————————————————————————————————————+ #include "UGAlib.mqh" //+——————————————————————————————————————————————————————————————————————+ //———————————————————————————————————————————————————————————————————————— //----------------------Incoming parameters-------------------------------- input string GenofundParam = "----Parameters of the gene pool----"; input int ChromosomeCount_P = 100; //Number of chromosomes in the colony input int GeneCount_P = 100; //Number of genes input int FFNormalizeDigits_P = 0; //Number of fitness symbols input int GeneNormalizeDigits_P= 0; //Number of gene symbols input int Epoch_P = 50; //Number of epochs without progress //--- input string GA_OperatorParam = "----Parameters of operators----"; input double ReplicationPortion_P = 100.0; //Proportion of replication. input double NMutationPortion_P = 10.0; //Proportion of natural mutations. input double ArtificialMutation_P = 10.0; //Proportion of artificial mutations. input double GenoMergingPortion_P = 20.0; //Proportion of borrowed genes. input double CrossingOverPortion_P = 20.0; //Proportion of crossing - over. input double ReplicationOffset_P = 0.5; //Coefficient of interval border displacement input double NMutationProbability_P= 5.0; //Probability of mutation of each gene in % //--- input string OptimisationParam = "----Optimization parameters----"; input double RangeMinimum_P = 0.0; //Minimum search range input double RangeMaximum_P = 5.0; //Maximum search range input double Precision_P = 1.0; //Required accuracy input int OptimizeMethod_P = 2; //Optim.:1-Min,other -Max input string Other = "----Other----"; input double Spred = 80.0; input bool Show = true; //———————————————————————————————————————————————————————————————————————— //———————————————————————————————————————————————————————————————————————— //----------------------Global variables----------------------------- double Hight []; double Low []; datetime Time []; datetime Ti []; double Peaks []; bool show; //———————————————————————————————————————————————————————————————————————— //--------------------------Body of the program-------------------------------- int OnStart() { //-----------------------Variables------------------------------------- //Preparation of global variables for UGA ChromosomeCount=ChromosomeCount_P; //Number of chromosomes in the colony GeneCount =GeneCount_P; //Number of genes RangeMinimum =RangeMinimum_P; //Minimum search range RangeMaximum =RangeMaximum_P; //Maximum search range Precision =Precision_P; //Searching step OptimizeMethod =OptimizeMethod_P; //1-minimum, any other - maximum FFNormalizeDigits = FFNormalizeDigits_P; //Number of fitness symbols GeneNormalizeDigits = GeneNormalizeDigits_P;//Number of gene symbols ArrayResize(Chromosome,GeneCount+1); ArrayInitialize(Chromosome,0); Epoch=Epoch_P; //Number of epochs without progress //---------------------------------------------------------------------- //Preparation of global variables ArraySetAsSeries(Hight,true); CopyHigh (NULL,0,0,GeneCount+1,Hight); ArraySetAsSeries(Low,true); CopyLow (NULL,0,0,GeneCount+1,Low); ArraySetAsSeries(Time,true); CopyTime (NULL,0,0,GeneCount+1,Time); ArrayResize (Ti,GeneCount+1);ArrayInitialize(Ti,0); ArrayResize(Peaks,GeneCount+1);ArrayInitialize(Peaks,0.0); show=Show; //---------------------------------------------------------------------- //local variables int time_start=GetTickCount(),time_end=0; //---------------------------------------------------------------------- //Очистим экран ObjectsDeleteAll(0,-1,-1); ChartRedraw(0); //launch of the main function UGA UGA ( ReplicationPortion_P, //Proportion of replication. NMutationPortion_P, //Proportion of replication of natural mutations. ArtificialMutation_P, //Proportion of artificial mutations. GenoMergingPortion_P, //Proportion of borrowed genes. CrossingOverPortion_P,//proportion of crossing- over. //--- ReplicationOffset_P, //Coefficient of interval border displacement NMutationProbability_P//Probability of mutation of each gene in % ); //---------------------------------- //Display the last result on the screen show=true; ServiceFunction(); //---------------------------------- time_end=GetTickCount(); //---------------------------------- Print(time_end-time_start," мс - time of execution"); //---------------------------------- return(0); } //———————————————————————————————————————————————————————————————————————— //———————————————————————————————————————————————————————————————————————— //-----------------------------------------------------------------------+ // Service function. Called up from UGA. | | //If there is no need for it, leave the function empty, like this: | // void ServiceFunction() | // { | // } | //-----------------------------------------------------------------------+ void ServiceFunction() { if(show==true) { //-----------------------Variables----------------------------------- double PipsSum=0.0; int PeaksCount=0; double temp=0.0; //-------------------------------------------------------------------- for(int u=1;u<=GeneCount;u++) { temp=Chromosome[u]; if(temp<=1.0 ) { Peaks[PeaksCount]=NormalizeDouble(Hight[u],Digits()); Ti [PeaksCount]=Time[u]; PeaksCount++; } if(temp>=4.0) { Peaks[PeaksCount]=NormalizeDouble(Low[u],Digits()); Ti [PeaksCount]=Time[u]; PeaksCount++; } } ObjectsDeleteAll(0,-1,-1); for(int V=0;V<peakscount-1;V++) { PipsSum+=NormalizeDouble((MathAbs(Peaks[V]-Peaks[V+1]))/Point(),FFNormalizeDigits)-Spred; ObjectCreate (0,"BoxBackName"+(string)V,OBJ_TREND,0,Ti[V],Peaks[V],Ti[V+1],Peaks[V+1]); ObjectSetInteger(0,"BoxBackName"+(string)V,OBJPROP_COLOR,Black); ObjectSetInteger(0,"BoxBackName"+(string)V,OBJPROP_SELECTABLE,true); } ChartRedraw(0); Comment(PipsSum); } //---------------------------------------------------------------------- else return; } //———————————————————————————————————————————————————————————————————————— //———————————————————————————————————————————————————————————————————————— //-----------------------------------------------------------------------+ // Function of determining the fitness of the individual. Called up from UGA. | //-----------------------------------------------------------------------+ void FitnessFunction(int chromos) { //-----------------------Variables------------------------------------- double PipsSum=0.0; int PeaksCount=0; double temp=0.0; //---------------------------------------------------------------------- for(int u=1;u<=GeneCount;u++) { temp=Colony[u][chromos]; if(temp<=1.0) { Peaks[PeaksCount]=NormalizeDouble(Hight[u],Digits()); PeaksCount++; } if(temp>=4.0) { Peaks[PeaksCount]=NormalizeDouble(Low[u],Digits()); PeaksCount++; } } if(PeaksCount>1) { for(int V=0;V<peakscount-1;V++) PipsSum+=NormalizeDouble((MathAbs(Peaks[V]-Peaks[V+1]))/Point(),FFNormalizeDigits)-Spred; Colony[0][chromos]=PipsSum; } else Colony[0][chromos]=-10000000.0; AmountStartsFF++; } //————————————————————————————————————————————————————————————————————————</peakscount-</peakscount-
Quando executamos o script, obtemos os vértices com um lucro total de 4939 pontos. Além do mais, levou apenas 17,929 vezes para contar os pontos, em comparação com os 3 ^ 100 necessários através da numeração direta. Em meu computador, isto são 21,7 segundos contra 1,6e32 anos!
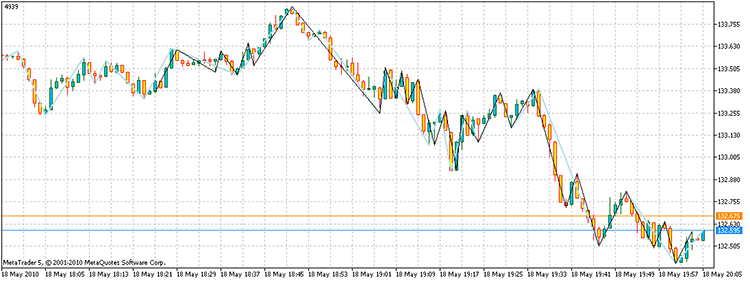
Figura 14. O resultado da solução do problema. Os segmentos coloridos em preto - um ZZ alternativo, azul-céu - indicador ZZ
Então, a resposta para a pergunta será lida conforme segue: "Existe".
5. Recomendações para trabalhar com o UGA
- Tente ajustar as condições estimadas corretamente no FF, para poder esperar um resultado adequado do algoriimo. Pense novamente no Exemplo 2. Isto é, talvez, minha principal recomendação.
- Não utilize valores pequenos demais para o parâmetro Precisão. Embora o algoritmo seja capaz de trabalhar com uma etapa 0, deve-se exigir uma exatidão razoável da solução. Este parâmetro é destinado a reduzir a dimensão do problema.
- Varie o tamanho da população e o valor limite dos números dos períodos. Uma boa solução seria atribuir uma parâmetro Período duas vezes maior do que o mostrado pelo MaxOfCurrentEpoch. Não escolha valores grandes demais, isto não acelerará o encontro de uma solução para o problema.
- Experimente com os parâmetros dos operadores genéticos. Não há parâmetros universais e você deve atribuí-los com base nas condições da tarefa perante você.
Constatações
Em conjunto com um pessoal muito poderoso, o terminal testador de estratégia, a linguagem MQL5 possibilita que você crie nada menos do que um poderoso instrumento para o negociador, permitindo que você solucione problemas verdadeiramente complexos. Obtemos um algoritmo de otimização muito flexível e escalonável. E eu estou descaradamente orgulhoso desta descoberta, mesmo que eu não tenha sido o primeiro a estabelecê-la.
Devido ao UGA ter sido inicialmente desenhado deste modo, em que ele pudesse ser facilmente modificado e ampliado com operadores adicionais e blocos de cálculo, o leitor poderá facilmente contribuir com o desenvolvimento da evolução "artificial".
Eu desejo ao leitor sucesso para encontrar soluções ótimas. Espero ter conseguido ajudar nisto. Boa sorte!
Nota. Este artigo utilizou o indicador ZigZag. Todos os códigos-fonte do UGA estão anexos.
Licenciamento: Os códigos-fonte anexos a este artigo (código UGA) são distribuídos sob as condições de licenciamento da BSD.
Traduzido do russo pela MetaQuotes Ltd.
Artigo original: https://www.mql5.com/ru/articles/55
Aviso: Todos os direitos sobre esses materiais pertencem à MetaQuotes Ltd. É proibida a reimpressão total ou parcial.
Esse artigo foi escrito por um usuário do site e reflete seu ponto de vista pessoal. A MetaQuotes Ltd. não se responsabiliza pela precisão das informações apresentadas nem pelas possíveis consequências decorrentes do uso das soluções, estratégias ou recomendações descritas.
 Um exemplo de uma estratégia de negociação baseada na diferença de fuso horário em diferentes continentes
Um exemplo de uma estratégia de negociação baseada na diferença de fuso horário em diferentes continentes
 MQL para "Principiantes": como projetar e construir classes de objeto
MQL para "Principiantes": como projetar e construir classes de objeto
 Criação e publicação de relatórios de negócios e notificação SMS
Criação e publicação de relatórios de negócios e notificação SMS
 Criando um indicador com buffers de indicador múltiplos para iniciantes
Criando um indicador com buffers de indicador múltiplos para iniciantes
- Aplicativos de negociação gratuitos
- 8 000+ sinais para cópia
- Notícias econômicas para análise dos mercados financeiros
Você concorda com a política do site e com os termos de uso
Por favor, mostre-me a lista de erros.O código é muito antigo, mas não há nada para quebrar mesmo após 14 anos.
O problema é com a variável "ArtificialMutation" e a função. Alterei a função para ArtificialMutationP e ela funciona.
Os problemas com o erro ServiceFunction e FitnessFunction estão corretos, entendo o que está acontecendo. Anexei capturas de tela.
Quando tento compilar o UGA_script ou o UGA_the_alternative_ZigZag diretamente após a extração do ZIP, recebo erros provenientes da UGAlib.
Quando alterei o nome da função (e sua chamada no caso 2) para ArtificialMutationP, ela foi compilada e funcionou.
Ainda estou analisando seu excelente trabalho para entender completamente. Obrigado, senhor
Pergunta.
os dados históricos (o número de barras) são a contagem de genes, isso está correto?
O problema é com a variável "ArtificialMutation" e a função. Alterei a função para ArtificialMutationP e ela funciona.
Os problemas com o erro ServiceFunction e FitnessFunction estão corretos, entendo o que está acontecendo. Anexei capturas de tela.
Quando tento compilar o UGA_script ou o UGA_the_alternative_ZigZag diretamente após a extração do ZIP, recebo erros provenientes da UGAlib.
Quando alterei o nome da função (e sua chamada no caso 2) para ArtificialMutationP, ela foi compilada e funcionou.
Ainda estou analisando seu excelente trabalho para entender completamente. Obrigado, senhor
Tente estearquivo
Obrigadopor suas gentis palavras .