
Algorithmes Génétiques - C'est Facile !
Introduction
L'algorithme génétique ( GA ) fait référence à l'algorithme heuristique ( EA ), qui fournit une solution acceptable au problème dans la plupart des cas pratiquement significatifs, mais la justesse des décisions n'a pas été mathématiquement prouvée, et est utilisée le plus souvent pour des problèmes, dont la solution analytique est très difficile voire impossible.
Un exemple classique d'un problème de cette classe (classe NP) est un "problème du vendeur ambulant" (c'est l'un des problèmes d'optimisation combinatoire les plus connus). Le principal défi est de trouver l'itinéraire le plus avantageux, qui traverse les villes citées au moins une fois, puis revient à la ville initiale). Mais rien n'empêche de les utiliser pour des tâches, qui cèdent à la formalisation.
Les EA sont largement utilisés pour résoudre des problèmes de complexité de calcul élevée, au lieu de passer en revue toutes les options, ce qui prend beaucoup de temps. Ils sont utilisés dans les domaines de l'intelligence artificielle, tels que la reconnaissance de formes, les logiciels antivirus, l'ingénierie, les jeux informatiques et d'autres domaines.
Il convient de mentionner que MetaQuotes Software Corp. utilise GA dans ses produits logiciels de MetaTrader4 / 5. Nous connaissons tous le testeur de stratégie et combien de temps et d'efforts peuvent être économisés en utilisant un optimiseur de stratégie intégré, dans lequel, tout comme avec l'énumération directe, il est possible d'optimiser avec l'utilisation de GA. De plus, le testeur MetaTrader 5 nous permet d'utiliser les critères d'optimisation des utilisateurs. Le lecteur sera peut-être intéressé par la lecture des articles sur l'AG et les avantages offerts par l'EA contrairement au dénombrement direct.
1. Un peu d'histoire
Il y a un peu plus d'un an, j'avais besoin d'un algorithme d'optimisation pour former des réseaux de neurones. Après m'être rapidement familiarisé avec les différents algorithmes, j'ai opté pour GA. À la suite de ma recherche d'implémentations prêtes à l'emploi, j'ai découvert que celles ouvertes au public présentaient des limitations fonctionnelles, telles que le nombre de paramètres pouvant être optimisés, ou étaient trop "étroites réglés".
J'avais besoin d'un instrument universellement flexible non seulement pour former tous les types de réseaux de neurones, mais aussi pour résoudre de manière générale tous les problèmes d'optimisation. Après une longue étude des "créations génétiques" étrangères, je n'arrivais toujours pas à comprendre leur fonctionnement. La cause en était soit un style de code élaboré, soit mon manque d'expérience en programmation, ou peut-être les deux.
Les principales difficultés se sont survenues du codage des gènes en un code binaire puis de leur utilisation sous cette forme. Quoi qu'il en soit, il a été décidé d'écrire un algorithme génétique à partir de zéro, en se concentrant sur l'extensibilité et la facilité de modification à l'avenir.
Je ne voulais pas traiter de transformation binaire, et j'ai décidé de travailler directement avec les gènes, c'est-à-dire de représenter le chromosome avec un ensemble de gènes sous forme de nombres réels. C'est ainsi qu'est apparu le code de mon algorithme génétique, avec une représentation des chromosomes par des nombres réels. Plus tard, j'ai appris que je n'avais rien découvert de nouveau et que des algorithmes génétiques analogues (on les appelle GA à code réel) existaient déjà depuis plus de 15 ans, depuis la parution des premières publications à leur sujet.
Je laisse ma vision d'aborder l’implémentation et les principes de fonctionnement de l' AG au lecteur pour juger, car elle est basée sur l'expérience personnelle de son utilisation dans des problèmes pratiques.
2. Description des GA
GA comporte les principes, empruntés à la nature elle-même. Ce sont les principes d'hérédité et de variabilité. L'hérédité est la capacité des organismes à transférer leurs traits et leurs caractéristiques évolutives à leur progéniture. Grâce à cette capacité, tous les êtres vivants laissent derrière eux les caractéristiques de leur espèce dans leur progéniture.
La variabilité des gènes dans les organismes vivants assure la diversité génétique de la population et est aléatoire, car la nature n'a aucun moyen de savoir à l'avance quelles caractéristiques seront les plus préférables à l'avenir (changement climatique, diminution/augmentation de la nourriture, l’émergence d'espèces concurrentes, etc.). Cette variabilité permet l'apparition de créatures avec de nouveaux traits, qui peuvent survivre et laisser une progéniture dans les nouvelles conditions altérées de l'habitat.
En biologie, la variabilité, qui est due à l'émergence de mutations, est appelée mutationnelle, la variabilité due à une nouvelle combinaison croisée de gènes par accouplement, est appelée combinatoire. Ces deux types de variantes sont implémentés dans l' AG. De plus, il existe une implémentation de la mutagenèse, qui imite le mécanisme naturel des mutations (modifications de la séquence nucléotidique de l'ADN) - naturelle (spontanée) et artificielle (induite).
L'unité la plus simple de transfert d'informations sur le critère de l'algorithme est le gène - unité structurelle et fonctionnelle de l'hérédité, qui contrôle le développement d'un trait ou d'une propriété particulière. Nous appellerons une variable de la fonction le gène. Le gène est représenté par un nombre réel. L'ensemble des variables génétiques de la fonction étudiée est le trait caractéristique du -chromosome
Entendons-nous pour représenter le chromosome sous la forme d'une colonne. Alors le chromosome pour la fonction f (x) = x ^ 2 ressemblerait à ceci :
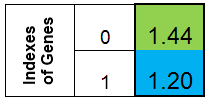
Figure 1. Chromosome pour la fonction f (x) = x ^ 2
où 0-ième indice - valeur de la fonction f (x), appelée l'adaptation des individus (nous appellerons cette fonction la fonction d’aptitude - FF , et la valeur de la fonction - VFF ). Il est pratique de stocker le chromosome dans un tableau unidimensionnel. Il s'agit du double tableau Chromosome [].
Tous les spécimens de la même ère évolutive sont combinés en une population. De plus, la population est arbitrairement divisée en deux colonies égales - la colonie mère et la colonie descendante. À la suite du croisement des espèces parentales, qui sont sélectionnées dans l'ensemble de la population, et d'autres opérateurs de l'AG, il y a une colonie de nouveaux descendants, qui est égale à la moitié de la taille de la population, qui remplace la colonie de la progéniture dans la population.
La population totale d'individus lors d'une recherche du minimum de la fonction f (x) = x ^ 2 pourrait ressembler à ceci :
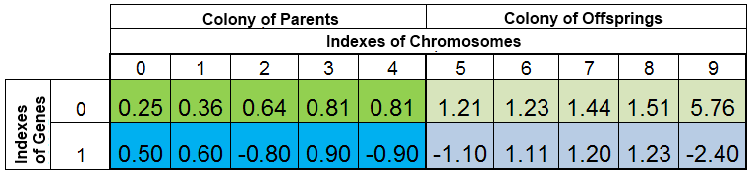
Figure 2. Population totale d'individus
La population est triée par VFF. Ici, l'indice 0-ème du chromosome est repris par le spécimen avec le plus petit VFF. La nouvelle progéniture remplace complètement uniquement les individus de la colonie du descendant, tandis que la colonie parentale reste intacte. Cependant, la colonie parentale peut ne pas toujours être complète, car les spécimens en double sont détruits, puis la nouvelle progéniture remplit les postes vacants dans la colonie parentale et le reste est placé dans la colonie descendante.
En d'autres termes, la taille de la population est rarement constante, et varie d'une époque à l'autre, presque de la même manière que dans la nature. Par exemple, la population avant la reproduction et après la reproduction peut ressembler à ceci :
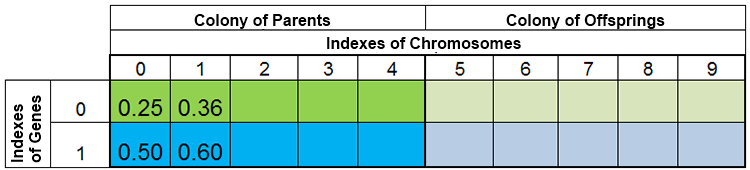
Figure 3. La population avant la reproduction
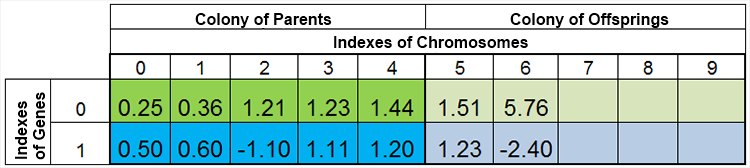
Figure 4. La population après la reproduction
Le mécanisme décrit du « demi » accomplissement de la population par les descendants, ainsi que la destruction des doublons et l'interdiction du croisement des individus avec eux-mêmes, ont un seul objectif - éviter « l'effet de goulot d'étranglement » (terme de la biologie, ce qui indique une réduction du pool génétique de la population à la suite d'une réduction critique due à un certain nombre de raisons différentes, ce qui peut conduire à une extinction totale d'une espèce, GA est confrontée à la fin des apparitions de chromosomes uniques et "se coincer" dans l'un des extrémaux locaux.)
3. Description de la fonction UGAlib
Algorithme GA :- Création d'une proto-population. Les gènes sont générés aléatoirement dans une plage donnée.
- Détermination de l'aptitude de chaque individu, ou en d'autres termes, le calcul de VFF.
- Préparation de la population à la reproduction, après élimination des doublons chromosomiques.
- Isolation et préservation du chromosome de référence (avec le meilleur VFF).
- Opérateurs d'UGA (sélection, accouplement, mutation). Pour chaque accouplement et mutation, de nouveaux parents sont sélectionnés à chaque fois. Préparer la population à l'ère suivante.
- Comparaison des gènes de la meilleure progéniture avec les gènes du chromosome de référence. Si le chromosome de la meilleure progéniture est meilleur que le chromosome de référence, remplacez le chromosome de référence.
Continuez à partir du paragraphe 5 jusqu'à ce qu'il n'y ait plus de chromosomes, meilleurs que ceux de référence apparaissant, pour un nombre spécifié d'ères.
3.1. Variables Globales Variables Globales
Annonce des variables suivantes au niveau mondial :
//----------------------Global variables----------------------------- double Chromosome[]; //A set of optimized arguments of the function - genes //(for example: the weight of the neuron network, etc.)- of the chromosome int ChromosomeCount =0; //Maximum possible amount of chromosomes in a colony int TotalOfChromosomesInHistory=0;//Total number of chromosomes in the history int ChrCountInHistory =0; //Number of unique chromosomes in the base chromosome int GeneCount =0; //Number of genes in the chromosome double RangeMinimum =0.0;//Minimum search range double RangeMaximum =0.0;//Maximum search range double Precision =0.0;//Search step int OptimizeMethod =0; //1-minimum, any other - maximum int FFNormalizeDigits =0; //Number of symbols after the comma in the fitness value int GeneNormalizeDigits =0; //Number of symbols after the comma in the genes value double Population [][1000]; //Population double Colony [][500]; //Offspring colony int PopulChromosCount =0; //The current number of chromosomes in a population int Epoch =0; //Number of epochs without progress int AmountStartsFF=0; //Number of launches of the fitness function //————————————————————————————————————————————————————————————————————————
3.2. UGA. La fonction principale de l' AG
En fait, la fonction principale de GA, appelée depuis le corps du programme pour effectuer les étapes, énumérées ci-dessus, nous n'entrerons donc pas dans les détails.
À la fin de l'algorithme, enregistré les informations suivantes dans le journal :
- Combien d'époques y avait-il au total (générations).
- Combien de fautes totales.
- Le nombre de chromosomes uniques.
- Le nombre total de lancements de FF.
- Le nombre total de chromosomes dans l'histoire.
- Rapport en pourcentage des doublons par rapport au nombre total de chromosomes dans l'historique.
- Meilleur résultat.
"Le nombre de chromosomes uniques" et le "Nombre total de lancements de FF" - les mêmes tailles, mais sont calculés différemment. Utiliser pour le contrôle.
//———————————————————————————————————————————————————————————————————————— //Basic function UGA void UGA ( double ReplicationPortion, // Proportion of replication. double NMutationPortion, // Proportion of natural mutations. double ArtificialMutation, // Proportion of artificial mutations. double GenoMergingPortion, // Proportion of borrowed genes. double CrossingOverPortion, // Proportion of cross -over. //--- double ReplicationOffset, // Coefficient of displacement of interval borders double NMutationProbability // Probability of mutation of each gene in% ) { //generator reset takes place only once MathSrand((int)TimeLocal()); //-----------------------Variables------------------------------------- int chromos=0, gene =0;//indexes of chromosomes and genes int resetCounterFF =0;//counter of resets of "Epochs without progress" int currentEpoch =1;//number of the current epoch int SumOfCurrentEpoch=0;//sum of "Epochs without progress" int MinOfCurrentEpoch=Epoch;//minimum of "epochs without progress" int MaxOfCurrentEpoch=0;//maximum of "Epochs without progress" int epochGlob =0;//total number of epochs // Colony [number of traits(genes)][number of individuals in a colony] ArrayResize(Population,GeneCount+1); ArrayInitialize(Population,0.0); // Colony of offspring [number of traits(genes)][number of individuals in a colony] ArrayResize(Colony,GeneCount+1); ArrayInitialize(Colony,0.0); // Chromosome bank // [number of traits (genes)][number of chromosomes in the bank] double historyHromosomes[][100000]; ArrayResize(historyHromosomes,GeneCount+1); ArrayInitialize(historyHromosomes,0.0); //---------------------------------------------------------------------- //--------------Verification of the correctness of incoming parameters---------------- //...the number of chromosomes mus be less than 2 if(ChromosomeCount<=1) ChromosomeCount=2; if(ChromosomeCount>500) ChromosomeCount=500; //---------------------------------------------------------------------- //====================================================================== // 1) Create a proto- population —————1) ProtopopulationBuilding(); //====================================================================== // 2) Determine the fitness of each individual —————2) //For the 1st colony for(chromos=0;chromos<ChromosomeCount;chromos++) for(gene=1;gene<=GeneCount;gene++) Colony[gene][chromos]=Population[gene][chromos]; GetFitness(historyHromosomes); for(chromos=0;chromos<ChromosomeCount;chromos++) Population[0][chromos]=Colony[0][chromos]; //For the 2nd colony for(chromos=ChromosomeCount;chromos<ChromosomeCount*2;chromos++) for(gene=1;gene<=GeneCount;gene++) Colony[gene][chromos-ChromosomeCount]=Population[gene][chromos]; GetFitness(historyHromosomes); for(chromos=ChromosomeCount;chromos<ChromosomeCount*2;chromos++) Population[0][chromos]=Colony[0][chromos-ChromosomeCount]; //====================================================================== // 3) Prepare the population for reproduction ————3) RemovalDuplicates(); //====================================================================== // 4) Extract the reference chromosome —————4) for(gene=0;gene<=GeneCount;gene++) Chromosome[gene]=Population[gene][0]; //====================================================================== ServiceFunction(); //The main cycle The main cycle of the genetic algorithm from 5 to 6 while(currentEpoch<=Epoch) { //==================================================================== // 5) Operators of UGA —————5) CycleOfOperators ( historyHromosomes, //--- ReplicationPortion, //Proportion of replication. NMutationPortion, //Proportion of natural mutation. ArtificialMutation, //Proportion of artificial mutation. GenoMergingPortion, //Proportion of borrowed genes. CrossingOverPortion,//Proportion of cross- over. //--- ReplicationOffset, //Coefficient of displacement of interval borders NMutationProbability//Probability of mutation of each gene in % ); //==================================================================== // 6) Compare the genes of the best offspring with the genes of the reference chromosome. // If the chromosome of the best offspring is better that the reference chromosome, // replace the reference. —————6) //If the optimization mode is - minimization if(OptimizeMethod==1) { //If the best chromosome of the population is better than the reference chromosome if(Population[0][0]<Chromosome[0]) { //Replace the reference chromosome for(gene=0;gene<=GeneCount;gene++) Chromosome[gene]=Population[gene][0]; ServiceFunction(); //Rest the counter of "epochs without progress" if(currentEpoch<MinOfCurrentEpoch) MinOfCurrentEpoch=currentEpoch; if(currentEpoch>MaxOfCurrentEpoch) MaxOfCurrentEpoch=currentEpoch; SumOfCurrentEpoch+=currentEpoch; currentEpoch=1; resetCounterFF++; } else currentEpoch++; } //If the optimization mode is - minimization else { //If the best chromosome of the population is better than the reference chromosome if(Population[0][0]>Chromosome[0]) { //Replace the reference chromosome for(gene=0;gene<=GeneCount;gene++) Chromosome[gene]=Population[gene][0]; ServiceFunction(); //Reset the counter of "epochs without progress" if(currentEpoch<MinOfCurrentEpoch) MinOfCurrentEpoch=currentEpoch; if(currentEpoch>MaxOfCurrentEpoch) MaxOfCurrentEpoch=currentEpoch; SumOfCurrentEpoch+=currentEpoch; currentEpoch=1; resetCounterFF++; } else currentEpoch++; } //==================================================================== //Another epoch went by.... epochGlob++; } Print("Epochs went by=",epochGlob," Number of resets=",resetCounterFF); Print("MinOfCurrentEpoch",MinOfCurrentEpoch, " AverageOfCurrentEpoch",NormalizeDouble((double)SumOfCurrentEpoch/(double)resetCounterFF,2), " MaxOfCurrentEpoch",MaxOfCurrentEpoch); Print(ChrCountInHistory," - Unique chromosome"); Print(AmountStartsFF," - Total number of launches of FF"); Print(TotalOfChromosomesInHistory," - Total number of chromosomes in the history"); Print(NormalizeDouble(100.0-((double)ChrCountInHistory*100.0/(double)TotalOfChromosomesInHistory),2),"% of duplicates"); Print(Chromosome[0]," - best result"); } //————————————————————————————————————————————————————————————————————————
3.3. Création d'une Proto-population. Création d'une proto-population.
Étant donné que dans la plupart des problèmes d'optimisation, il n'y a aucun moyen de savoir à l'avance où se trouvent les arguments de la fonction sur la droite numérique, la meilleure option optimale est la génération aléatoire dans une plage donnée.
//———————————————————————————————————————————————————————————————————————— //Creating a proto- population void ProtopopulationBuilding() { PopulChromosCount=ChromosomeCount*2; //Fill up the population with chromosomes with random //...genes in the range between RangeMinimum...RangeMaximum for(int chromos=0;chromos<PopulChromosCount;chromos++) { //beginning with the 1st indexes (the 0 index is reserved for VFF) for(int gene=1;gene<=GeneCount;gene++) Population[gene][chromos]= NormalizeDouble(SelectInDiscreteSpace(RNDfromCI(RangeMinimum,RangeMaximum),RangeMinimum,RangeMaximum,Precision,3),GeneNormalizeDigits); TotalOfChromosomesInHistory++; } } //————————————————————————————————————————————————————————————————————————
3.4. Obtenir une aptitude Obtenir l’aptitude
Effectue la fonction optimisée pour chaque chromosome dans l'ordre.
//------------------------------------------------ ------------------------ // Getting the fitness for each individual. void GetFitness ( double &historyHromosomes[][100000] ) { for(int chromos=0;chromos<ChromosomeCount;chromos++) CheckHistoryChromosomes(chromos,historyHromosomes); } //————————————————————————————————————————————————————————————————————————
3.5. CheckHistoryChromosomes. Vérification du chromosome à travers la base chromosomique
Le chromosome de chaque individu est vérifié à travers la base - si le FF a été calculé pour lui, et si c'était le cas, alors le VFF prêt est tiré de la base, sinon, le FF est-y appelé Ainsi, les calculs répétés à forte intensité d’utilisation de ressources de FF sont exclus.
//———————————————————————————————————————————————————————————————————————— //Verification of chromosome through the chromosome base. void CheckHistoryChromosomes ( int chromos, double &historyHromosomes[][100000] ) { //-----------------------Variables------------------------------------- int Ch1=0; //Index of the chromosome from the base int Ge =0; //Index of the gene int cnt=0; //Counter of unique genes. If at least one gene is different //- the chromosome is acknowledged unique //---------------------------------------------------------------------- //If at least one chromosome is stored in the base if(ChrCountInHistory>0) { //Enumerate the chromosomes in the base to find an identical one for(Ch1=0;Ch1<ChrCountInHistory && cnt<GeneCount;Ch1++) { cnt=0; //Compare the genes, while the gene index is less than the number of genes and while there are identical genes for(Ge=1;Ge<=GeneCount;Ge++) { if(Colony[Ge][chromos]!=historyHromosomes[Ge][Ch1]) break; cnt++; } } //If there are enough identical genes then we can use a ready- made solution from the base if(cnt==GeneCount) Colony[0][chromos]=historyHromosomes[0][Ch1-1]; //If there is no such chromosome, then we calculate the FF for it... else { FitnessFunction(chromos); //.. and if there is space in the base, then save it if(ChrCountInHistory<100000) { for(Ge=0;Ge<=GeneCount;Ge++) historyHromosomes[Ge][ChrCountInHistory]=Colony[Ge][chromos]; ChrCountInHistory++; } } } //If the base is empty, calculate the FF for it and save it in the base else { FitnessFunction(chromos); for(Ge=0;Ge<=GeneCount;Ge++) historyHromosomes[Ge][ChrCountInHistory]=Colony[Ge][chromos]; ChrCountInHistory++; } } //————————————————————————————————————————————————————————————————————————
3.6. Cycle d’Opérateurs. Cycle des Opérateurs en UGA
À ce stade, le destin littéraire de toute une époque de vie artificielle est en train de se décider - une nouvelle génération est née et meurt. Cela se passe de la manière suivante : deux parents sont sélectionnés pour l'accouplement, ou un seul pour commettre un acte de mutation sur lui. Pour chaque opérateur de GA, un paramètre approprié est déterminé. En conséquence, nous obtenons une progéniture. Ceci est répété encore et encore, jusqu'à ce que la colonie descendante soit complètement remplie. Ensuite, la colonie de descendants est libérée dans l'habitat, afin que chaque individu puisse se manifester aussi bien qu'il le pourrait, et nous calculons la VFF.
Après avoir été testée par "des tuyaux de feu, d'eau et de cuivre", la colonie de descendants s'est installée dans la population. La prochaine étape de l'évolution artificielle sera le meurtre sacré des clones, afin d'empêcher l'épuisement du « sang » dans les générations futures, et le classement ultérieur de la population renouvelée, selon le degré d'aptitude.
//———————————————————————————————————————————————————————————————————————— //Cycle of operators of UGA void CycleOfOperators ( double &historyHromosomes[][100000], //--- double ReplicationPortion, //Proportion of replications. double NMutationPortion, //Proportion of natural mutations. double ArtificialMutation, //Proportion of artificial mutations. double GenoMergingPortion, //Portion of borrowed genes. double CrossingOverPortion,//Proportion of cross-over. //--- double ReplicationOffset, //Coefficient of displacement of interval borders double NMutationProbability//Probability of mutation of each gene in % ) { //-----------------------Variables------------------------------------- double child[]; ArrayResize (child,GeneCount+1); ArrayInitialize(child,0.0); int gene=0,chromos=0, border=0; int i=0,u=0; double p=0.0,start=0.0; double fit[][2]; ArrayResize (fit,6); ArrayInitialize(fit,0.0); //Counter of planting spots in a new population. int T=0; //---------------------------------------------------------------------- //Set a portion of operators of UGA double portion[6]; portion[0]=ReplicationPortion; //Proportion of replications. portion[1]=NMutationPortion; //Proportion of natural mutations. portion[2]=ArtificialMutation; //Proportion of artificial mutations. portion[3]=GenoMergingPortion; //Proportion of borrowed genes. portion[4]=CrossingOverPortion;//Proportion of cross- overs. portion[5]=0.0; //------------------------Cycle of operators of UGA--------- //Fill up the new colony with offspring while(T<ChromosomeCount) { //============================ for(i=0;i<6;i++) { fit[i][0]=start; fit[i][1]=start+MathAbs(portion[i]-portion[5]); start=fit[i][1]; } p=RNDfromCI(fit[0][0],fit[4][1]); for(u=0;u<5;u++) { if((fit[u][0]<=p && p<fit[u][1]) || p==fit[u][1]) break; } //============================ switch(u) { //--------------------- case 0: //------------------------Replication-------------------------------- //If there is space in the new colony, create a new individual if(T<ChromosomeCount) { Replication(child,ReplicationOffset); //Settle the new individual into the new colony for(gene=1;gene<=GeneCount;gene++) Colony[gene][T]=child[gene]; //One place is occupied, fast- forward the counter T++; TotalOfChromosomesInHistory++; } //--------------------------------------------------------------- break; //--------------------- case 1: //---------------------Natural mutation------------------------- //If there is space in the new colony, create a new individual if(T<ChromosomeCount) { NaturalMutation(child,NMutationProbability); //Settle the new individual into the new colony for(gene=1;gene<=GeneCount;gene++) Colony[gene][T]=child[gene]; //One place is occupied, fast- forward the counter T++; TotalOfChromosomesInHistory++; } //--------------------------------------------------------------- break; //--------------------- case 2: //----------------------Artificial mutation----------------------- //If there is space in the new colony, create a new individual if(T<ChromosomeCount) { ArtificialMutation(child,ReplicationOffset); //Settle the new individual into the new colony for(gene=1;gene<=GeneCount;gene++) Colony[gene][T]=child[gene]; //One place is occupied, fast-forward the counter T++; TotalOfChromosomesInHistory++; } //--------------------------------------------------------------- break; //--------------------- case 3: //-------------The creation of an individual with borrowed genes----------- //If there is space in the new colony, create a new individual if(T<ChromosomeCount) { GenoMerging(child); //Settle the new individual into the new colony for(gene=1;gene<=GeneCount;gene++) Colony[gene][T]=child[gene]; //One space is occupied, fast forward the counter T++; TotalOfChromosomesInHistory++; } //--------------------------------------------------------------- break; //--------------------- default: //---------------------------Crossing-Over--------------------------- //If there is place in the new colony, create a new individual if(T<ChromosomeCount) { CrossingOver(child); //Settle the new individual into the new colony for(gene=1;gene<=GeneCount;gene++) Colony[gene][T]=child[gene]; //One place is occupied, fast forward the counter T++; TotalOfChromosomesInHistory++; } //--------------------------------------------------------------- break; //--------------------- } }//End of the cycle operators of UGA-- //Determine the fitness of each individual in the colony of offspring GetFitness(historyHromosomes); //Settle the offspring into the main population if(PopulChromosCount>=ChromosomeCount) { border=ChromosomeCount; PopulChromosCount=ChromosomeCount*2; } else { border=PopulChromosCount; PopulChromosCount+=ChromosomeCount; } for(chromos=0;chromos<ChromosomeCount;chromos++) for(gene=0;gene<=GeneCount;gene++) Population[gene][chromos+border]=Colony[gene][chromos]; //Prepare the population for the next reproduction RemovalDuplicates(); }//the end of the function //————————————————————————————————————————————————————————————————————————
3.7. Réplication Replication
L'opérateur est le plus proche du phénomène naturel, appelé en biologie - réplication de l'ADN, bien que, par essence, ce ne soit pas la même chose. Mais comme je n'ai pas trouvé d'équivalent, plus proche de cela dans la nature, j'ai décidé de garder ce titre.
La réplication est l'opérateur génétique le plus important, qui produit de nouveaux gènes, tout en transmettant les traits des chromosomes parentaux. L'opérateur principal, assurant la convergence de l'algorithme. GA ne peut fonctionner qu'avec lui sans recours à d'autres opérateurs, mais dans ce cas le nombre de lancements de FF serait bien plus important.
Examinons le principe de l'opérateur de réplication. Nous utilisons deux chromosomes parentaux. Le nouveau gène de la progéniture est un nombre aléatoire de l'intervalle
[C1-((C2-C1)*ReplicationOffset),C2+((C2-C1)*ReplicationOffset)]
où gènes parentaux C1 et C2 ReplicationOffset - Coefficient de déplacement des frontières d'intervalle [C1, C2] .
Par exemple, à partir de l'individu paternel (bleu) et de l'individu maternel (rose) on peut créer un enfant (vert) :
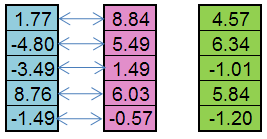
Figure 5. Le principe de l'opérateur Travail de réplication
Graphiquement, la probabilité du gène de la progéniture peut être résumée :
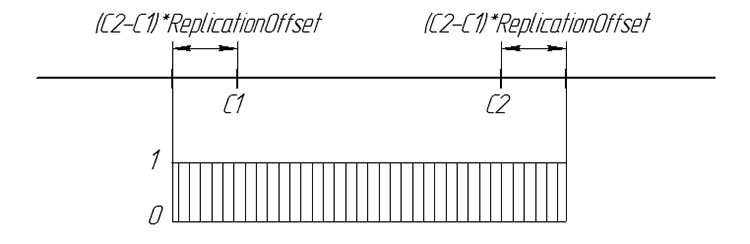
Figure 6. La probabilité de l'apparition du gène de la progéniture sur une droite numérique
Les autres gènes de la progéniture sont générés de la même manière.
//------------------------------------------------ ------------------------ // Replication void Replication ( double &child[], double ReplicationOffset ) { //-----------------------Variables------------------------------------- double C1=0.0,C2=0.0,temp=0.0,Maximum=0.0,Minimum=0.0; int address_mama=0,address_papa=0; //---------------------------------------------------------------------- SelectTwoParents(address_mama,address_papa); //-------------------Cycle of gene enumeration-------------------------------- for(int i=1;i<=GeneCount;i++) { //----figure out where the father and mother came from -------- C1 = Population[i][address_mama]; C2 = Population[i][address_papa]; //------------------------------------------ //Mandatory verification to make sure that the search had not gone over the specified range if(C1 < RangeMinimum) C1 = RangeMinimum; if(C1 > RangeMaximum) C1 = RangeMaximum; if(C2 < RangeMinimum) C2 = RangeMinimum; if(C2 > RangeMaximum) C2 = RangeMaximum; //------------------------------------------------------------------ //....determine the largest and smallest of them, //if we С1>C2, swi if(C1>C2) { temp=C1; C1=C2; C2=temp; } //-------------------------------------------- //Specify the borders of the created gene Minimum = C1-((C2-C1)*ReplicationOffset); Maximum = C2+((C2-C1)*ReplicationOffset); //-------------------------------------------- //Mandatory verification to make sure that the search has not gone over the specified range if(Minimum < RangeMinimum) Minimum = RangeMinimum; if(Maximum > RangeMaximum) Maximum = RangeMaximum; //--------------------------------------------------------------- temp=RNDfromCI(Minimum,Maximum); child[i]= NormalizeDouble(SelectInDiscreteSpace(temp,RangeMinimum,RangeMaximum,Precision,3),GeneNormalizeDigits); } } //————————————————————————————————————————————————————————————————————————3.8. NaturalMutation. Mutation naturelle
Des mutations se produisent constamment au cours des processus, se produisant dans des cellules vivantes et servent de matériau à la sélection naturelle. Ils apparaissent spontanément tout au long de la vie de l'organisme dans ses conditions normales d'habitat, avec une fréquence d'une fois par 10 ^ 10 générations cellulaires.
Nous - les chercheurs curieux, n'avons pas nécessairement besoin d'adhérer à l'ordre naturel et d'attendre si longtemps la prochaine mutation du gène. Le paramètre NMutationProbability, qui s'exprime en pourcentage et détermine la probabilité de mutation pour chaque gène du chromosome, va nous y aider.
Dans l'opérateur NaturalMutation, la mutation consiste en la génération d'un gène aléatoire dans l'intervalle [RangeMinimum, RangeMaximum] . NMutationProbability = 100 % signifierait une mutation à 100 % de tous les gènes du chromosome, et NMutationProbability = 0 % - absence totale de mutations. La dernière option est impropre à utiliser dans des problèmes pratiques.
//------------------------------------------------ ------------------------ // The natural mutation. void NaturalMutation ( double &child[], double NMutationProbability ) { //-----------------------Variables------------------------------------- int address=0; double prob=0.0; //---------------------------------------------------------------------- if(NMutationProbability<0.0) prob=0.0; if(NMutationProbability>100.0) prob=100.0; //-----------------Parent selection------------------------ SelectOneParent(address); //--------------------------------------- for(int i=1;i<=GeneCount;i++) if(RNDfromCI(0.0,100.0)<prob) child[i]=NormalizeDouble( SelectInDiscreteSpace(RNDfromCI(RangeMinimum,RangeMaximum),RangeMinimum,RangeMaximum,Precision,3),GeneNormalizeDigits ); } //————————————————————————————————————————————————————————————————————————
3.9. Mutation Artificielle Mutation artificielle
La tâche principale de l'opérateur - est la génération de sang "frais". Nous utilisons deux parents, et les gènes de la progéniture sont sélectionnés à partir des espaces, non alloués par les gènes parents, sur la droite numérique. Protège le GA de se coincer dans l'un des extrema locaux. Dans une plus grande proportion, par rapport à d'autres opérateurs, accélère la convergence, ou bien - ralentit, augmentant le nombre de lancements de FF.
Tout comme dans la Réplication, nous utilisons deux chromosomes parentaux. Mais la tâche de l'opérateur de Mutation Artificielle n'est pas de transmettre les traits parentaux à la progéniture, mais plutôt de rendre l'enfant différent d'eux. Par conséquent, étant tout le contraire, en utilisant le même coefficient de déplacement de frontière d'intervalle, mais les gènes sont générés en dehors de l'intervalle, qui aurait été pris par la Réplication. Le nouveau gène de la progéniture est un nombre aléatoire des intervalles [RangeMinimum, C1-(C2-C1) * ReplicationOffset] et [C2 + (C2-C1) * ReplicationOffset, RangeMaximum]
Graphiquement, la probabilité d'un gène dans la progéniture ReplicationOffset = 0,25 peut être représentée comme suit :
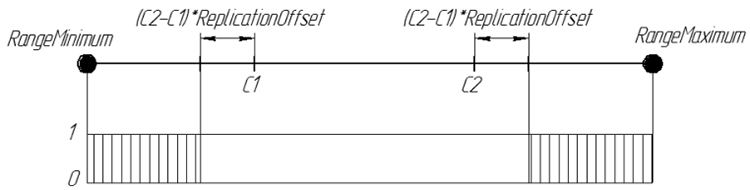
Figure 7. La probabilité d'un gène dans le descendant ReplicationOffset = 0,25 sur l'intervalle de ligne réel [RangeMinimum; RangeMaximum]
//———————————————————————————————————————————————————————————————————————— //Artificial mutation. void ArtificialMutation ( double &child[], double ReplicationOffset ) { //-----------------------Variables------------------------------------- double C1=0.0,C2=0.0,temp=0.0,Maximum=0.0,Minimum=0.0,p=0.0; int address_mama=0,address_papa=0; //---------------------------------------------------------------------- //-----------------Selecting parents------------------------ SelectTwoParents(address_mama,address_papa); //-------------------------------------------------------- //-------------------Cycle of genes enumeration------------------------------ for(int i=1;i<=GeneCount;i++) { //----determine where the mother and father are from -------- C1 = Population[i][address_mama]; C2 = Population[i][address_papa]; //------------------------------------------ //Mandatory verification to make sure that the search doesn't go beyond the specified range if(C1 < RangeMinimum) C1 = RangeMinimum; if(C1 > RangeMaximum) C1 = RangeMaximum; if(C2 < RangeMinimum) C2 = RangeMinimum; if(C2 > RangeMaximum) C2 = RangeMaximum; //------------------------------------------------------------------ //....determine the largest and smallest of them, //if С1>C2, we change their places if(C1>C2) { temp=C1; C1=C2; C2=temp; } //-------------------------------------------- //Specify the borders of creating the new gene Minimum=C1-((C2-C1)*ReplicationOffset); Maximum=C2+((C2-C1)*ReplicationOffset); //-------------------------------------------- //Mandatory verification to make sure that the search doesn't go beyond the specified range if(Minimum < RangeMinimum) Minimum = RangeMinimum; if(Maximum > RangeMaximum) Maximum = RangeMaximum; //--------------------------------------------------------------- p=MathRand(); if(p<16383.5) { temp=RNDfromCI(RangeMinimum,Minimum); child[i]= NormalizeDouble(SelectInDiscreteSpace(temp,RangeMinimum,RangeMaximum,Precision,3),GeneNormalizeDigits); } else { temp=RNDfromCI(Maximum,RangeMaximum); child[i]= NormalizeDouble(SelectInDiscreteSpace(temp,RangeMinimum,RangeMaximum,Precision,3),GeneNormalizeDigits); } } } //————————————————————————————————————————————————————————————————————————
3.10 Géno Fusion. Emprunter des gènes
L'opérateur GA donné n'a pas d'équivalent naturel. Il est en effet difficile d'imaginer comment ce merveilleux mécanisme fonctionnerait dans les organismes vivants. Cependant, il a une propriété remarquable de transférer des gènes d'un certain nombre de parents (le nombre de parents est égal au nombre de gènes) à la progéniture. L'opérateur ne génère pas de nouveaux gènes et constitue un mécanisme de recherche combinatoire.
Cela fonctionne comme ceci : pour le premier gène de la progéniture, un parent est sélectionné, et le premier gène lui est retiré, puis, pour le deuxième gène, un deuxième parent est sélectionné, et le gène lui est retiré, etc. Il est souhaitable d’appliquer si le nombre de gènes est supérieur à un. Sinon, il doit être désactivé, car l'opérateur générera des doublons des chromosomes.
//———————————————————————————————————————————————————————————————————————— //Borrowing genes. void GenoMerging ( double &child[] ) { //-----------------------Variables------------------------------------- int address=0; //---------------------------------------------------------------------- for(int i=1;i<=GeneCount;i++) { //-----------------Selecting parents------------------------ SelectOneParent(address); //-------------------------------------------------------- child[i]=Population[i][address]; } } //————————————————————————————————————————————————————————————————————————
3.11. Enjambement Enjambement
L’enjambement (également connu en biologie sous le nom de croisement) - est un phénomène d'échange de sections de chromosomes. Tout comme dans GenoMerging, il s'agit d'un mécanisme de recherche combinatoire.
Deux chromosomes parentaux sont sélectionnés. Les deux sont "coupés" dans endroit aléatoire. Le chromosome de la progéniture sera constitué de parties de chromosomes parentaux.
Il est plus simple d'illustrer ce mécanisme en image :
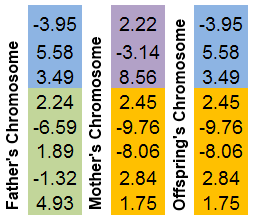
Figure 8. Le mécanisme d'échange des parties chromosomiques
Il est souhaitable d’appliquer si le nombre de gènes est supérieur à un. Sinon, il doit être désactivé, car l'opérateur générera des doublons des chromosomes.
//———————————————————————————————————————————————————————————————————————— //Crossing-over. void CrossingOver ( double &child[] ) { //-----------------------Variables------------------------------------- int address_mama=0,address_papa=0; //---------------------------------------------------------------------- //-----------------Selecting parents------------------------ SelectTwoParents(address_mama,address_papa); //-------------------------------------------------------- //Determine the breakage point int address_of_gene=(int)MathFloor((GeneCount-1)*(MathRand()/32767.5)); for(int i=1;i<=GeneCount;i++) { //----copy the mother's genes-------- if(i<=address_of_gene+1) child[i]=Population[i][address_mama]; //----copy the father's genes-------- else child[i]=Population[i][address_papa]; } } //————————————————————————————————————————————————————————————————————————
3.12. SelectionnerDeuxParents La sélection de deux parents
Pour éviter l'épuisement du patrimoine génétique, il est interdit de se croiser avec soi-même. Dix tentatives sont faites pour trouver des parents différents, et si nous ne parvenons pas à trouver un couple, nous permettons le croisement avec soi-même. En gros, on obtient une copie du même spécimen.
D'une part, la probabilité de clonage d'individus diminue, d'autre part - la circularité de la recherche est empêchée, car une situation peut survenir, dans laquelle il serait quasiment impossible de le faire (choisir des parents différents) dans un nombre raisonnable de pas.
Utilisé dans les opérateurs Replication, ArtificialMutation et CrossingOver .
//———————————————————————————————————————————————————————————————————————— //Selection of two parents. void SelectTwoParents ( int &address_mama, int &address_papa ) { //-----------------------Variables------------------------------------- int cnt=1; address_mama=0;//address of the mother individual in a population address_papa=0;//address of the father individual in a population //---------------------------------------------------------------------- //----------------------------Selection of parents-------------------------- //Ten attempts to chose different parents. while(cnt<=10) { //For the mother individual address_mama=NaturalSelection(); //For the father individual address_papa=NaturalSelection(); if(address_mama!=address_papa) break; } //--------------------------------------------------------------------- } //————————————————————————————————————————————————————————————————————————
3.13. SelectionnerUnParent La sélection d'un parent
Ici, tout est simple - un parent est sélectionné dans la population.
Utilisé dans les opérateurs NaturalMutation et GenoMerging.
//———————————————————————————————————————————————————————————————————————— //Selection of one parent. void SelectOneParent ( int &address//address of the parent individual in the population ) { //-----------------------Variables------------------------------------- address=0; //---------------------------------------------------------------------- //----------------------------Selecting a parent-------------------------- address=NaturalSelection(); //--------------------------------------------------------------------- } //————————————————————————————————————————————————————————————————————————
3.14. Sélection Naturelle Sélection naturelle
Sélection naturelle - le processus qui conduit à la survie et à la reproduction préférentielle d'individus, mieux adaptés à ces conditions environnementales, possédant des traits héréditaires utiles.
L'opérateur est similaire à l'opérateur traditionnel "Roulette" (Sélection roue de roulette - Sélection d'individus avec n "lancements" de la roulette. La roulette comporte un secteur pour chaque membre de la population. La taille du i-ième secteur est proportionnelle à la valeur correspondante d’aptitude), mais présente des différences considérables. Elle prend en compte la position des individus en rapport avec les plus et moins adaptés. De plus, même un individu qui a les pires gènes a une chance de laisser derrière lui une progéniture. C'est juste, n'est-ce pas ? Bien qu'il ne s'agisse pas de justice, mais du fait que dans la nature, tous les individus ont la chance de laisser une progéniture derrière eux.
Par exemple, prenons 10 individus, ayant le VFF suivant dans le problème de maximisation : 256, 128, 64, 32, 16, 8, 4, 2, 0, -1 - où les valeurs les plus élevées correspondent à une meilleure aptitude. Cet exemple est pris pour que l'on puisse voir que la "distance" entre individus voisins est 2 fois plus grande qu'entre les deux individus précédents. Cependant, sur le diagramme à secteurs, la probabilité que chaque individu laisse une progéniture est la suivante :
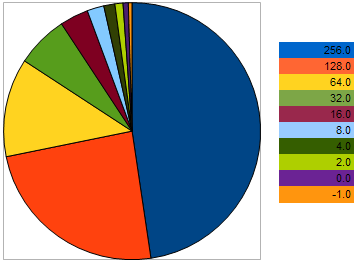
Figure 9. Le tableau de probabilité de sélection des individus parents
il démontre qu'avec l'approche des individus au pire, leurs chances s'empirent. Inversement, plus l'individu se rapproche du meilleur spécimen, meilleures sont ses chances de reproduction.

Figure 10. Le tableau de probabilité de sélection des individus parents
//———————————————————————————————————————————————————————————————————————— //Natural selection. int NaturalSelection() { //-----------------------Variables------------------------------------- int i=0,u=0; double p=0.0,start=0.0; double fit[][2]; ArrayResize (fit,PopulChromosCount); ArrayInitialize(fit,0.0); double delta=(Population[0][0]-Population[0][PopulChromosCount-1])*0.01-Population[0][PopulChromosCount-1]; //---------------------------------------------------------------------- for(i=0;i<PopulChromosCount;i++) { fit[i][0]=start; fit[i][1]=start+MathAbs(Population[0][i]+delta); start=fit[i][1]; } p=RNDfromCI(fit[0][0],fit[PopulChromosCount-1][1]); for(u=0;u<PopulChromosCount;u++) if((fit[u][0]<=p && p<fit[u][1]) || p==fit[u][1]) break; return(u); } //————————————————————————————————————————————————————————————————————————
3.15. RemovalDuplicates. Supprimer les doublons
La fonction supprime les chromosomes dupliqués dans la population, et les chromosomes uniques restants (uniques à la population de l'époque actuelle) sont triés dans l'ordre par le VFF, déterminé par le type d'optimisation, c'est-à-dire décroissant ou croissant.
//———————————————————————————————————————————————————————————————————————— //Removing duplicates sorted by VFF void RemovalDuplicates() { //-----------------------Variables------------------------------------- int chromosomeUnique[1000];//Array stores the unique trait //of each chromosome: 0-duplicate, 1-unique ArrayInitialize(chromosomeUnique,1); //Assume that there are no duplicates double PopulationTemp[][1000]; ArrayResize (PopulationTemp,GeneCount+1); ArrayInitialize(PopulationTemp,0.0); int Ge =0; //Index of the gene int Ch =0; //Index of the chromosome int Ch2=0; //Index of the second chromosome int cnt=0; //Counter //---------------------------------------------------------------------- //----------------------Remove duplicates---------------------------1 //Chose the first from the pair for comparison... for(Ch=0;Ch<PopulChromosCount;Ch++) { //If it's not a duplicate... if(chromosomeUnique[Ch]!=0) { //Chose the second from the pair... for(Ch2=0;Ch2<PopulChromosCount;Ch2++) { if(Ch!=Ch2 && chromosomeUnique[Ch2]!=0) { //Zeroize the counter of identical genes cnt=0; //Compare the genes. while there are identical genes present for(Ge=1;Ge<=GeneCount;Ge++) { if(Population[Ge][Ch]!=Population[Ge][Ch2]) break; else cnt++; } //If there are the same amount of identical genes as total genes //..the chromosome is considered a duplicate if(cnt==GeneCount) chromosomeUnique[Ch2]=0; } } } } //The counter calculates the number of unique chromosomes cnt=0; //Copy the unique chromosomes into a temporary array for(Ch=0;Ch<PopulChromosCount;Ch++) { //If the chromosome is unique, copy it, if not, go to the next if(chromosomeUnique[Ch]==1) { for(Ge=0;Ge<=GeneCount;Ge++) PopulationTemp[Ge][cnt]=Population[Ge][Ch]; cnt++; } } //Assigning the variable "All chromosomes" the value of counter of unique chromosomes PopulChromosCount=cnt; //Return unique chromosomes back to the array for temporary storage //..of combined populations for(Ch=0;Ch<PopulChromosCount;Ch++) for(Ge=0;Ge<=GeneCount;Ge++) Population[Ge][Ch]=PopulationTemp[Ge][Ch]; //=================================================================1 //----------------Ranking the population---------------------------2 PopulationRanking(); //=================================================================2 } //————————————————————————————————————————————————————————————————————————
3.16. Classement de la population. Classement de la population
Le tri est effectué par la VFF. La méthode est similaire à 'bubbly (L'algorithme consiste en des passages répétés à travers le tableau trié. Pour chaque passe, les éléments sont comparés successivement par paires, et si l'ordre d'une paire est erroné, un échange d'éléments a lieu. Les passages à travers le tableau sont répétés jusqu'à ce que l'un des passages montre que les échanges ne sont plus nécessaires, ce qui indique - le tableau a été trié.
Lors du passage dans l'algorithme, un élément qui n'est pas à sa place, "apparaît" à la position souhaitée, tout comme une bulle dans l'eau, d'où le nom de l'algorithme, mais il y a une différence - seuls les index du tableau sont triés, pas le contenu du tableau. Cette méthode est plus rapide et légèrement différente de la simple copie d'un tableau dans un autre. Et plus la taille du tableau trié est grande, plus la différence est petite.
//———————————————————————————————————————————————————————————————————————— //Population ranking. void PopulationRanking() { //-----------------------Variables------------------------------------- int cnt=1, i = 0, u = 0; double PopulationTemp[][1000]; //Temporary population ArrayResize (PopulationTemp,GeneCount+1); ArrayInitialize(PopulationTemp,0.0); int Indexes[]; //Indexes of chromosomes ArrayResize (Indexes,PopulChromosCount); ArrayInitialize(Indexes,0); int t0=0; double ValueOnIndexes[]; //VFF of corresponding //..chromosome indexes ArrayResize (ValueOnIndexes,PopulChromosCount); ArrayInitialize(ValueOnIndexes,0.0); double t1=0.0; //---------------------------------------------------------------------- //Fill in the indexes in the temporary array temp2 and //...copy the first line from the sorted array for(i=0;i<PopulChromosCount;i++) { Indexes[i] = i; ValueOnIndexes[i] = Population[0][i]; } if(OptimizeMethod==1) { while(cnt>0) { cnt=0; for(i=0;i<PopulChromosCount-1;i++) { if(ValueOnIndexes[i]>ValueOnIndexes[i+1]) { //----------------------- t0 = Indexes[i+1]; t1 = ValueOnIndexes[i+1]; Indexes [i+1] = Indexes[i]; ValueOnIndexes [i+1] = ValueOnIndexes[i]; Indexes [i] = t0; ValueOnIndexes [i] = t1; //----------------------- cnt++; } } } } else { while(cnt>0) { cnt=0; for(i=0;i<PopulChromosCount-1;i++) { if(ValueOnIndexes[i]<ValueOnIndexes[i+1]) { //----------------------- t0 = Indexes[i+1]; t1 = ValueOnIndexes[i+1]; Indexes [i+1] = Indexes[i]; ValueOnIndexes [i+1] = ValueOnIndexes[i]; Indexes [i] = t0; ValueOnIndexes [i] = t1; //----------------------- cnt++; } } } } //Create a sorted-out array based on the obtained indexes for(i=0;i<GeneCount+1;i++) for(u=0;u<PopulChromosCount;u++) PopulationTemp[i][u]=Population[i][Indexes[u]]; //Copy the sorted-out array back for(i=0;i<GeneCount+1;i++) for(u=0;u<PopulChromosCount;u++) Population[i][u]=PopulationTemp[i][u]; } //————————————————————————————————————————————————————————————————————————
3.17. RNDfromCustomInterval. Le générateur de nombres aléatoires à partir d'un intervalle donné
Juste une fonctionnalité pratique. Est pratique dans UGA.
//———————————————————————————————————————————————————————————————————————— //Generator of random numbers from the selected interval. double RNDfromCI(double RangeMinimum,double RangeMaximum) { return(RangeMinimum+((RangeMaximum-RangeMinimum)*MathRand()/32767.5));} //————————————————————————————————————————————————————————————————————————
3.18. SelectInDiscreteSpace. La sélection dans l'espace discret
Est utilisé pour réduire l'espace de recherche. Avec le paramètre step = 0.0 la recherche s'effectue dans un espace continu (restreint aux limitations de langue, en MQL jusqu'au 15ème signe significatif inclus). Pour utiliser l'algorithme GA avec une plus grande précision, vous devez écrire une bibliothèque supplémentaire pour travailler avec des nombres longs.
Le travail de la fonction à RoundMode = 1 peut être illustré par la figure suivante :
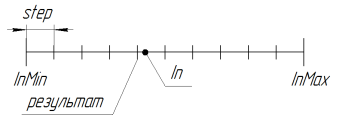
Figure 11. le travail de la fonction SelectInDiscreteSpace à RoundMode = 1
//———————————————————————————————————————————————————————————————————————— //Selection in discrete space. //Modes: //1-closest below //2-closest above //any closest double SelectInDiscreteSpace ( double In, double InMin, double InMax, double step, int RoundMode ) { if(step==0.0) return(In); // secure the correctness of borders if( InMax < InMin ) { double temp = InMax; InMax = InMin; InMin = temp; } // during a breach - return the breached border if( In < InMin ) return( InMin ); if( In > InMax ) return( InMax ); if( InMax == InMin || step <= 0.0 ) return( InMin ); // bring to the specified scale step = (InMax - InMin) / MathCeil ( (InMax - InMin) / step ); switch( RoundMode ) { case 1: return( InMin + step * MathFloor ( ( In - InMin ) / step ) ); case 2: return( InMin + step * MathCeil ( ( In - InMin ) / step ) ); default: return( InMin + step * MathRound ( ( In - InMin ) / step ) ); } } //————————————————————————————————————————————————————————————————————————
3.19. Fonction de remise en forme. Fonction de remise en forme
Ne fait pas partie de l'AG. La fonction reçoit l'indice du chromosome dans la population, pour lequel le VFF sera calculé. VFF est écrit dans l'index zéro du chromosome transmis. Le code de cette fonction est unique pour chaque tâche.
3.20. ServiceFunction. Fonction de service
Ne fait pas partie de l'AG. Le code de cette fonction est unique pour chaque tâche spécifique. Il peut être utilisé pour implémenter un contrôle sur les époques. Par exemple, afin d'afficher le meilleur VFF pour l'époque actuelle.
4. Exemples de travaux UGA
Tous les problèmes d'optimisation sont résolus à l’aide d' EA, et sont divisés en deux types :
- Le génotype correspond à un phénotype. Les valeurs des gènes chromosomiques sont directement désignées par les arguments d'une fonction d'optimisation. Exemple 1
- Le génotype ne correspond pas au phénotype. L'interprétation de la signification des gènes chromosomiques est nécessaire pour calculer la fonction optimisée. Exemple 2
4.1. Exemple 1
Examinons le problème avec une réponse connue, pour s’assurer que l'algorithme fonctionne et puis passez à la résolution du problème, dont la solution intéresse de nombreux traders.
Problème: Trouvez la fonction minimum et maximum "Skin":
sur le segment [-5, 5].
Réponse : fmin (3.07021,3.315935) = -4.3182, fmax (-3.315699 ; -3.072485) = 14.0606.
Figure 12. Le graphique de "Skin" sur le segment [-5, 5]
Pour résoudre le problème, nous écrivons le script suivant :
#property script_show_inputs //+——————————————————————————————————————————————————————————————————————+ #include "UGAlib.mqh" #include "Skin.mqh"//testing function //+——————————————————————————————————————————————————————————————————————+ //———————————————————————————————————————————————————————————————————————— //----------------------Incoming parameters-------------------------------- input string GenofundParam = "----Gene pool parameter----"; input int ChromosomeCount_P = 50; //Number of chromosomes in a colony input int GeneCount_P = 2; //Number of genes input int FFNormalizeDigits_P = 4; //Number of fitness symbols input int GeneNormalizeDigits_P = 4; //Number of genes input int Epoch_P = 50; //Number of epochs without progress //--- input string GA_OperatorParam = "----Operator parameters----"; input double ReplicationPortion_P = 100.0; //Proportion of replication. input double NMutationPortion_P = 10.0; //Proportion of natural mutations. input double ArtificialMutation_P = 10.0; //Proportion of artificial mutations. input double GenoMergingPortion_P = 20.0; //Proportion of borrowed genes. input double CrossingOverPortion_P = 20.0; //Proportion of crossing-over. //--- input double ReplicationOffset_P = 0.5; //Coefficient of interval borders displacement input double NMutationProbability_P= 5.0; //Probability of mutation of each gene in % //--- input string OptimisationParam = "----Optimization parameters----"; input double RangeMinimum_P = -5.0; //Minimum range search input double RangeMaximum_P = 5.0; //Maximum range search input double Precision_P = 0.0001;//The required accuracy input int OptimizeMethod_P = 1; //Optim.:1-Min,other-Max //———————————————————————————————————————————————————————————————————————— //———————————————————————————————————————————————————————————————————————— //----------------------Global variables----------------------------- double ERROR=0.0;//Average error in gen //———————————————————————————————————————————————————————————————————————— //———————————————————————————————————————————————————————————————————————— //--------------------------The body of the program-------------------------------- int OnStart() { //-----------------------Variables------------------------------------- //Preparing global variables for UGA ChromosomeCount=ChromosomeCount_P; //Number of chromosomes in the colony GeneCount =GeneCount_P; //Number of genes RangeMinimum =RangeMinimum_P; //Minimum range search RangeMaximum =RangeMaximum_P; //Maximum range search Precision =Precision_P; //Search step OptimizeMethod =OptimizeMethod_P; //1-minimum, any other - maximum FFNormalizeDigits = FFNormalizeDigits_P; //Number of symbols in fitness GeneNormalizeDigits = GeneNormalizeDigits_P;//Number of gene symbols ArrayResize(Chromosome,GeneCount+1); ArrayInitialize(Chromosome,0); Epoch=Epoch_P; //Number of epochs without progress //---------------------------------------------------------------------- //Local variables int time_start=GetTickCount(),time_end=0; //---------------------------------------------------------------------- //Launch of the main function UGA UGA ( ReplicationPortion_P, //Proportion of replication. NMutationPortion_P, //Proportion of natural mutations. ArtificialMutation_P, //Proportion of artificial mutations. GenoMergingPortion_P, //Proportion of borrowed genes. CrossingOverPortion_P,//Proportion of crossing-over. //--- ReplicationOffset_P, //Coefficient of interval border replacement NMutationProbability_P//Probability of mutation of each gene in % ); //---------------------------------- time_end=GetTickCount(); //---------------------------------- Print(time_end-time_start," mc - Time of implementation"); //---------------------------------- return(0); } //————————————————————————————————————————————————————————————————————————
Voici le code complet du script pour résoudre le problème. Exécutez-le, récupérez les informations, fournies par la fonction Comment () :
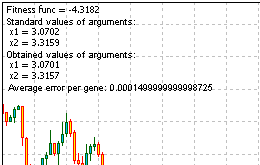
Figure 13. Le résultat de la résolution du problème
En regardant les résultats, nous voyons que l'algorithme fonctionne.
4.2. Exemple 2
Il est largement admis que l'indicateur ZZ montre les entrées idéales d'un système de trading inversé. L'indicateur est très populaire parmi les partisans de théorie la "Wave Theory" et ceux qui l'utilisent pour déterminer la taille des "chiffres".
Problème : Déterminez s'il existe d'autres points d'entrée pour un système de trading de inversé sur des données historiques, différentes des sommets ZZ, donnant en somme plus de points de gain théorique ?
Pour les expérimentations nous sélectionnerons une paire GBPJPY pour M1 100 barres. Acceptez l'écart de 80 points (cotations à cinq chiffres). Pour commencer, vous devez déterminer les meilleurs paramètres ZZ. Pour se faire, nous utilisons une énumération simple pour trouver la meilleure valeur du paramètre ExtDepth, à l'aide d'un script simple :
#property script_show_inputs //———————————————————————————————————————————————————————————————————————— //----------------------Incoming parameters-------------------------------- input int History=100; input double Spred =80.0; input int Depth =5; //For "one-time" use input bool loop =true;//Use enumeration or not //———————————————————————————————————————————————————————————————————————— //———————————————————————————————————————————————————————————————————————— //--------------------------The body of the program-------------------------------- void OnStart() { //-----------------------Variables------------------------------------- double ZigzagBuffer [];//For storing the buffer of the ZZ indicator double PeaksOfZigzag[];//for storing the values of the ZZ extremum int Zigzag_handle; //Indicator marker ArraySetAsSeries(ZigzagBuffer,true); ArrayResize(PeaksOfZigzag,History); int depth=3; double PipsSum=0.0; int PeaksCount=0; bool flag=true; //---------------------------------------------------------------------- if(loop==true) { while(depth<200 && flag==true) { //----------------------------------------------------------- Zigzag_handle=iCustom(NULL,0,"ZigZag",depth); //--- reset the code error ResetLastError(); //--- attempt to copy the indicator values for(int i=0;i<100;i++) { if(BarsCalculated(Zigzag_handle)>0) break; Sleep(1000); } int copied=CopyBuffer(Zigzag_handle,0,0,History,ZigzagBuffer); if(copied<=0) { Print("Could not copy the indicator buffer. Error =",GetLastError()," copied=",copied); return; } //----------------------------------------------------------- PipsSum=0.0; PeaksCount=0; for(int u=0;u<History;u++) { if(NormalizeDouble(ZigzagBuffer[u],Digits())>0.0) { PeaksOfZigzag[PeaksCount]=NormalizeDouble(ZigzagBuffer[u],Digits()); PeaksCount++; } } //----------------------------------------------------------- for(int V=0;V<PeaksCount-1;V++) PipsSum+=NormalizeDouble((MathAbs(PeaksOfZigzag[V]-PeaksOfZigzag[V+1]))/Point(),Digits())-Spred; //----------------------------------------------------------- if(PeaksCount<=2) flag=false; else { Print(depth," ",PeaksCount," ",PipsSum); depth+=1; } //----------------------------------------------------------- } } else { //----------------------------------------------------------- Zigzag_handle=iCustom(NULL,0,"ZigZag",Depth); //--- reser the error code ResetLastError(); //--- attempt to copy the indicator values for(int i=0;i<History;i++) { if(BarsCalculated(Zigzag_handle)>0) break; Sleep(1000); } int copied=CopyBuffer(Zigzag_handle,0,0,History,ZigzagBuffer); if(copied<=0) { Print("Was not able to copy the buffer indicator. Error =",GetLastError()," copied=",copied); return; } //----------------------------------------------------------- for(int u=0;u<History;u++) { if(NormalizeDouble(ZigzagBuffer[u],Digits())>0.0) { PeaksOfZigzag[PeaksCount]=NormalizeDouble(ZigzagBuffer[u],Digits()); PeaksCount++; } } //----------------------------------------------------------- for(int V=0;V<PeaksCount-1;V++) { PipsSum+=NormalizeDouble((MathAbs(PeaksOfZigzag[V]-PeaksOfZigzag[V+1]))/Point(),Digits())-Spred; } Print(Depth," ",PeaksCount," ",PipsSum); //----------------------------------------------------------- } } //————————————————————————————————————————————————————————————————————————
En exécutant le script, nous obtenons 4077 points dans ExtDepth = 3. Dix-neuf sommets indicateurs "s'ajustent" sur 100 barres. Avec l'augmentation de ExtDepth, le nombre de sommets ZZ diminue, tout comme la rentabilité.
Maintenant, nous pouvons trouver les sommets de l'alternative ZZ, en utilisant UGA. Les sommets ZZ peuvent avoir trois positions pour chaque barre : 1) Haut, 2) Bas, 3) Pas de sommet. La présence et la position du vertex seront portées par chaque gène pour chaque barre. Ainsi, la taille du chromosome - 100 gènes.
D'après mes calculs (et les mathématiciens peuvent me corriger si je me trompe), sur 100 barres vous pouvez construire 3 ^ 100, soit 5.15378e47 options alternatives "zigzags" . Il s'agit du nombre exact d'options qui doivent être prises en compte, en utilisant l'énumération directe. Lors du calcul avec une vitesse de 100000000 options par seconde, il nous faudra 1,6e32 ans ! C'est plus que l'âge de l'univers. C'est là où j'ai commencé à avoir des doutes sur la capacité de trouver une solution à ce problème.
Mais commençons.
Puisque UGA utilise la représentation du chromosome par des nombres réels, nous devons en quelque sorte coder la position des sommets. C'est précisément le cas lorsque le génotype du chromosome ne correspond pas au phénotype. Attribuez un intervalle de recherche pour les gènes [0, 5]. Convenons à ca que l'intervalle [0, 1] correspond au sommet de ZZ sur High, l'intervalle [4, 5] correspond au sommet sur Low, et l'intervalle (1, 4) correspond à l'absence du sommet.
Il est nécessaire de tenir compte d’ un point important. Étant donné que la proto-population est générée aléatoirement avec des gènes dans l'intervalle indiqué, le premier spécimen présentera des résultats très médiocres, peut-être même avec quelques centaines de points avec un signe moins. Après quelques générations (bien qu'il y ait une chance que cela se produise dans la première génération), nous verrons l'apparition de spécimens, dont les gènes sont cohérents avec l'absence de sommets en général. Cela indiquerait l'absence de trade et le paiement de l'inévitable spread.
Selon certains anciens traders : "La meilleure stratégie pour le trade - pas de trade". Cet individu sera le sommet de l'évolution artificielle. Afin que cette évolution "artificielle" donne naissance à des individus marchands, c'est-à-dire qu'elle arrange les sommets de l'alternative ZZ, on attribue à l’aptitude des individus, dépourvus de sommets, la valeur de "-10000000.0", en la plaçant volontairement sur le plus bas échelon de l'évolution, par rapport à tout autre individu.
Voici le code de script qui utilise UGA pour trouver les sommets de l'alternative ZZ :
#property script_show_inputs //+——————————————————————————————————————————————————————————————————————+ #include "UGAlib.mqh" //+——————————————————————————————————————————————————————————————————————+ //———————————————————————————————————————————————————————————————————————— //----------------------Incoming parameters-------------------------------- input string GenofundParam = "----Parameters of the gene pool----"; input int ChromosomeCount_P = 100; //Number of chromosomes in the colony input int GeneCount_P = 100; //Number of genes input int FFNormalizeDigits_P = 0; //Number of fitness symbols input int GeneNormalizeDigits_P= 0; //Number of gene symbols input int Epoch_P = 50; //Number of epochs without progress //--- input string GA_OperatorParam = "----Parameters of operators----"; input double ReplicationPortion_P = 100.0; //Proportion of replication. input double NMutationPortion_P = 10.0; //Proportion of natural mutations. input double ArtificialMutation_P = 10.0; //Proportion of artificial mutations. input double GenoMergingPortion_P = 20.0; //Proportion of borrowed genes. input double CrossingOverPortion_P = 20.0; //Proportion of crossing - over. input double ReplicationOffset_P = 0.5; //Coefficient of interval border displacement input double NMutationProbability_P= 5.0; //Probability of mutation of each gene in % //--- input string OptimisationParam = "----Optimization parameters----"; input double RangeMinimum_P = 0.0; //Minimum search range input double RangeMaximum_P = 5.0; //Maximum search range input double Precision_P = 1.0; //Required accuracy input int OptimizeMethod_P = 2; //Optim.:1-Min,other -Max input string Other = "----Other----"; input double Spred = 80.0; input bool Show = true; //———————————————————————————————————————————————————————————————————————— //———————————————————————————————————————————————————————————————————————— //----------------------Global variables----------------------------- double Hight []; double Low []; datetime Time []; datetime Ti []; double Peaks []; bool show; //———————————————————————————————————————————————————————————————————————— //--------------------------Body of the program-------------------------------- int OnStart() { //-----------------------Variables------------------------------------- //Preparation of global variables for UGA ChromosomeCount=ChromosomeCount_P; //Number of chromosomes in the colony GeneCount =GeneCount_P; //Number of genes RangeMinimum =RangeMinimum_P; //Minimum search range RangeMaximum =RangeMaximum_P; //Maximum search range Precision =Precision_P; //Searching step OptimizeMethod =OptimizeMethod_P; //1-minimum, any other - maximum FFNormalizeDigits = FFNormalizeDigits_P; //Number of fitness symbols GeneNormalizeDigits = GeneNormalizeDigits_P;//Number of gene symbols ArrayResize(Chromosome,GeneCount+1); ArrayInitialize(Chromosome,0); Epoch=Epoch_P; //Number of epochs without progress //---------------------------------------------------------------------- //Preparation of global variables ArraySetAsSeries(Hight,true); CopyHigh (NULL,0,0,GeneCount+1,Hight); ArraySetAsSeries(Low,true); CopyLow (NULL,0,0,GeneCount+1,Low); ArraySetAsSeries(Time,true); CopyTime (NULL,0,0,GeneCount+1,Time); ArrayResize (Ti,GeneCount+1);ArrayInitialize(Ti,0); ArrayResize(Peaks,GeneCount+1);ArrayInitialize(Peaks,0.0); show=Show; //---------------------------------------------------------------------- //local variables int time_start=GetTickCount(),time_end=0; //---------------------------------------------------------------------- //Очистим экран ObjectsDeleteAll(0,-1,-1); ChartRedraw(0); //launch of the main function UGA UGA ( ReplicationPortion_P, //Proportion of replication. NMutationPortion_P, //Proportion of replication of natural mutations. ArtificialMutation_P, //Proportion of artificial mutations. GenoMergingPortion_P, //Proportion of borrowed genes. CrossingOverPortion_P,//proportion of crossing- over. //--- ReplicationOffset_P, //Coefficient of interval border displacement NMutationProbability_P//Probability of mutation of each gene in % ); //---------------------------------- //Display the last result on the screen show=true; ServiceFunction(); //---------------------------------- time_end=GetTickCount(); //---------------------------------- Print(time_end-time_start," мс - time of execution"); //---------------------------------- return(0); } //———————————————————————————————————————————————————————————————————————— //———————————————————————————————————————————————————————————————————————— //-----------------------------------------------------------------------+ // Service function. Called up from UGA. | | //If there is no need for it, leave the function empty, like this: | // void ServiceFunction() | // { | // } | //-----------------------------------------------------------------------+ void ServiceFunction() { if(show==true) { //-----------------------Variables----------------------------------- double PipsSum=0.0; int PeaksCount=0; double temp=0.0; //-------------------------------------------------------------------- for(int u=1;u<=GeneCount;u++) { temp=Chromosome[u]; if(temp<=1.0 ) { Peaks[PeaksCount]=NormalizeDouble(Hight[u],Digits()); Ti [PeaksCount]=Time[u]; PeaksCount++; } if(temp>=4.0) { Peaks[PeaksCount]=NormalizeDouble(Low[u],Digits()); Ti [PeaksCount]=Time[u]; PeaksCount++; } } ObjectsDeleteAll(0,-1,-1); for(int V=0;V<PeaksCount-1;V++) { PipsSum+=NormalizeDouble((MathAbs(Peaks[V]-Peaks[V+1]))/Point(),FFNormalizeDigits)-Spred; ObjectCreate (0,"BoxBackName"+(string)V,OBJ_TREND,0,Ti[V],Peaks[V],Ti[V+1],Peaks[V+1]); ObjectSetInteger(0,"BoxBackName"+(string)V,OBJPROP_COLOR,Black); ObjectSetInteger(0,"BoxBackName"+(string)V,OBJPROP_SELECTABLE,true); } ChartRedraw(0); Comment(PipsSum); } //---------------------------------------------------------------------- else return; } //———————————————————————————————————————————————————————————————————————— //———————————————————————————————————————————————————————————————————————— //-----------------------------------------------------------------------+ // Function of determining the fitness of the individual. Called up from UGA. | //-----------------------------------------------------------------------+ void FitnessFunction(int chromos) { //-----------------------Variables------------------------------------- double PipsSum=0.0; int PeaksCount=0; double temp=0.0; //---------------------------------------------------------------------- for(int u=1;u<=GeneCount;u++) { temp=Colony[u][chromos]; if(temp<=1.0) { Peaks[PeaksCount]=NormalizeDouble(Hight[u],Digits()); PeaksCount++; } if(temp>=4.0) { Peaks[PeaksCount]=NormalizeDouble(Low[u],Digits()); PeaksCount++; } } if(PeaksCount>1) { for(int V=0;V<PeaksCount-1;V++) PipsSum+=NormalizeDouble((MathAbs(Peaks[V]-Peaks[V+1]))/Point(),FFNormalizeDigits)-Spred; Colony[0][chromos]=PipsSum; } else Colony[0][chromos]=-10000000.0; AmountStartsFF++; } //————————————————————————————————————————————————————————————————————————
Lorsque nous exécutons le script, nous obtenons les sommets avec un gain total de 4939 points. De plus, il n'a fallu que 17 929 fois pour compter les points, contre 3 ^ 100 nécessaires par dénombrement direct. Sur mon ordinateur, cela fait 21,7 secondes contre 1,6e32 ans !
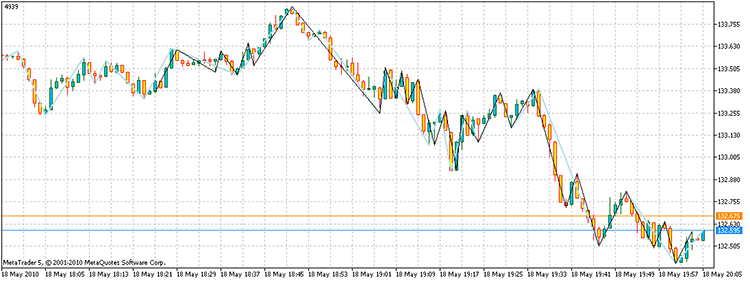
Figure 14. Le résultat de la solution du problème. Les segments de couleur noire - une alternative ZZ, bleu ciel - indicateur ZZ
Ainsi, la réponse à la question sera la suivante : Existe
5. Recommandations pour travailler avec UGA
- Essayez de définir correctement les conditions estimées dans FF, pour pouvoir espérer un résultat adéquat de l'algorithme. Repensez à l'exemple 2. C'est peut-être là ma principale recommandation.
- N'utilisez pas une valeur trop petite pour le paramètre Précision. Bien que l'algorithme puisse fonctionner avec une étape 0, vous devez exiger une précision raisonnable de la solution. Ce paramètre est destiné à réduire la dimension du problème.
- Variez la taille de la population et la valeur seuil du nombre d'époques. Une solution idéale serait d'attribuer un paramètre Epoch deux fois plus grand que celui indiqué par MaxOfCurrentEpoch. Ne choisissez pas une valeur trop élevée, cela n'accélérera pas la recherche d'une solution au problème.
- Expérimentez avec les paramètres des opérateurs génétiques. Il n'y a pas de paramètres universels et vous devez les attribuer en fonction des conditions de la tâche qui vous attend.
Conclusions
Avec un testeur de stratégie de terminal de dotation très puissant, le langage MQL5 vous permet de créer un instrument non moins puissant pour le trader, vous permettant de résoudre des problèmes vraiment complexes. On obtient un algorithme d'optimisation très flexible et extensible. Et je suis véritablement fière de cette découverte, même si je n'ai pas été le premier à l'établir.
Parce que l 'UGA a été initialement conçu de manière à pouvoir être facilement modifié et étendu avec des opérateurs et des blocs de calcul supplémentaires, le lecteur sera facilement en mesure de contribuer au développement de l'évolution "artificielle".
Je souhaite au lecteur de réussir à trouver des solutions optimales. J'espère avoir pu l'aider dans cette tâche. Bonne chance!
Note. L'article utilisait l'indicateur ZigZag. Tous les codes sources de l' UGA sont joints.
Licences : Les codes sources joints à l'article (code UGA) sont distribués sous les conditions de licence BSD.
Traduit du russe par MetaQuotes Ltd.
Article original : https://www.mql5.com/ru/articles/55
Avertissement: Tous les droits sur ces documents sont réservés par MetaQuotes Ltd. La copie ou la réimpression de ces documents, en tout ou en partie, est interdite.
Cet article a été rédigé par un utilisateur du site et reflète ses opinions personnelles. MetaQuotes Ltd n'est pas responsable de l'exactitude des informations présentées, ni des conséquences découlant de l'utilisation des solutions, stratégies ou recommandations décrites.
 Un Exemple de Stratégie de Trading Axée sur les Différences de Fuseau Horaire sur Différents Continents
Un Exemple de Stratégie de Trading Axée sur les Différences de Fuseau Horaire sur Différents Continents
 MQL pour "Nuls" : Comment Concevoir et Construire des Classes d'Objets
MQL pour "Nuls" : Comment Concevoir et Construire des Classes d'Objets
 Création et publication des rapports de trade et de notifications par SMS
Création et publication des rapports de trade et de notifications par SMS
 Création d'un indicateur avec plusieurs tampons d'indicateurs pour les débutants
Création d'un indicateur avec plusieurs tampons d'indicateurs pour les débutants
- Applications de trading gratuites
- Plus de 8 000 signaux à copier
- Actualités économiques pour explorer les marchés financiers
Vous acceptez la politique du site Web et les conditions d'utilisation
Veuillez me montrer la liste des erreurs. Le code est très vieux, mais il n'y a rien à casser même après 14 ans.
Le problème concerne la variable "ArtificialMutation" et la fonction. J'ai remplacé la fonction par ArtificialMutationP et cela fonctionne.
Les problèmes liés à l'erreur ServiceFunction et FitnessFunction sont corrects, je comprends ce qui se passe. J'ai joint des captures d'écran.
Lorsque j'essaie de compiler UGA_script ou UGA_the_alternative_ZigZag directement après l'extraction du ZIP, j'obtiens des erreurs qui proviennent de l'UGAlib.
Une fois que j'ai changé le nom de la fonction (et son appel dans le cas 2) en ArtificialMutationP, cela a compilé et fonctionné.
Je suis toujours en train de travailler sur votre excellent travail pour bien comprendre. Je vous remercie de votre attention.
Question.
Les données historiques (le nombre de barres) sont le nombre de gènes, est-ce correct ?
Le problème est lié à la variable "ArtificialMutation" et à la fonction. J'ai remplacé la fonction par "ArtificialMutationP" et cela fonctionne.
Les problèmes liés à l'erreur ServiceFunction et FitnessFunction sont corrects, je comprends ce qui se passe. J'ai joint des captures d'écran.
Lorsque j'essaie de compiler UGA_script ou UGA_the_alternative_ZigZag directement après l'extraction du ZIP, j'obtiens des erreurs qui proviennent de l'UGAlib.
Une fois que j'ai changé le nom de la fonction (et son appel dans le cas 2) en ArtificialMutationP, cela a compilé et fonctionné.
Je suis toujours en train de travailler sur votre excellent travail pour bien comprendre. Je vous remercie de votre attention.
Essayez cefichier
Merci pourvos aimables paroles .