
유전 알고리즘-쉬워요
개요
유전 알고리즘 ( GA )은 경험적 알고리즘 ( EA )을 의미하며, 실질적으로 중요한 대부분의 경우 문제에 대해 허용 가능한 솔루션을 제공하지만 결정의 정확성 수학적으로 입증되지 않았으며 분석 솔루션이 매우 어렵거나 불가능한 문제에 가장 자주 사용됩니다.
이 클래스(클래스 NP) 문제의 전형적인 예는 "여행하는 세일즈맨 문제"(가장 유명한 조합 최적화 문제 중 하나)입니다. 주된 과제는 주어진 도시를 한 번 이상 통과 한 다음 초기 도시로 돌아가는 가장 유리한 경로를 찾는 것입니다. 그러나 형식화로 이어지는 작업에 사용하는 것을 방해하는 것은 없습니다.
EA는 많은 시간이 소요되는 모든 옵션을 거치지 않고 계산 복잡성이 높은 문제를 해결하는 데 널리 사용됩니다. 패턴 인식과 같은 인공 지능 분야, 바이러스 백신 소프트웨어, 엔지니어링, 컴퓨터 게임 및 기타 분야에서 사용됩니다.
MetaTrader사의 MetaTrader4와 5에서는 GA가 사용됩니다. 우리 모두는 전략 테스터에 대해 알고 있으며 내장된 전략 최적화 프로그램을 사용하여 얼마나 많은 시간과 노력을 절약할 수 있는지 알고 있습니다. 직접 열거와 마찬가지로 GA를 사용하여 최적화할 수 있습니다. MetaTrader5 테스터는 사용자 설정 최적화 기준을 사용할 수 있게 해 주죠. 독자 여러분이 직접 열거보다는 GA 및 EA가 제공하는 장점에 대한 글을 더 읽고 싶어할지도 모르겠네요.
1. 약간의 이야기
약 1년 전, 신경망 훈련을 위한 최적화 알고리즘이 필요했습니다. 여러 알고리즘을 살펴본 후 GA를 선택했죠. 기성 구현을 검색 한 결과, 공개 액세스를 위해 개방 된 것이 최적화 할 수있는 매개 변수의 수와 같은 기능적 제한이 있거나 너무 "좁게 조정"되어 있음을 발견했습니다.
저는 모든 유형의 신경망을 훈련시킬뿐만 아니라 일반적으로 최적화 문제를 해결하기 위해 보편적으로 유연한 도구가 필요했습니다. 외국의 "유전 적 창조물"에 대한 오랜 연구 끝에 나는 그것이 어떻게 작동하는지 여전히 이해할 수 없었습니다. 그 원인은 정교한 코드 스타일이거나 프로그래밍 경험이 부족하거나 둘 다 때문일 수 있습니다.
주요 어려움은 유전자를 이진 코드로 코딩 한 다음이 형식으로 작업하는 것에서 발생했습니다. 어느 쪽이든, 미래의 확장 성과 쉬운 수정에 초점을 맞춘 유전 알고리즘을 처음부터 작성하기로 결정했습니다.
저는 이진 변환을 다루고 싶지 않았고 유전자를 직접 다루기로 결정했습니다. 즉, 일련의 유전자로 염색체를 실수 형태로 표현하는 것입니다. 이것이 염색체를 실수로 표현한 유전 알고리즘의 코드가 나타난 방식입니다. 나중에 나는 새로운 것을 발견하지 못했고 유사한 유전 알고리즘( 실제 코딩 된 GA </ b0>라고 함)이 이미 15 년 이상 존재했다는 것을 알게되었습니다.
실제 문제에서 사용하는 개인적인 경험을 기반으로하므로 독자가 판단 할 수 있도록 GA 기능의 구현 및 원칙에 접근하는 비전을 남겨 둡니다.
2. GA 설명
GA는 자연 자체에서 빌린 원칙을 포함합니다. 유전과 가변성의 원리이죠. 유전은 유기체가 자신의 특성과 진화적 특성을 자손에게 전이하는 능력입니다. 이 능력 덕분에 모든 생물은 자손에게 종의 특성을 남깁니다.
살아있는 유기체에서 유전자의 다양성은 개체군의 유전적 다양성을 보장하며 무작위로 나타납니다. 자연은 미래를 예측하지 못하기 때문입니다(기후 변화, 식량 감소/증가, 경쟁 종의 출현 등). 이 가변성은 새로운 특성을 가진 생물의 출현을 가능하게 하여 새롭고 변경된 서식지 조건에서 생존하고 자손을 남기게 도와줍니다.
생물학에서는 돌연변이의 출현으로 인해 발생하는 가변성을 돌연변이라고 하고 교배에 의한 추가 유전자 교차 조합으로 인한 가변성을 조합이라고 합니다. 이러한 변형은 모두 GA에서 구현됩니다. 또한 자연적(자발적) 및 인공적(유도적) 돌연변이(DNA의 뉴클레오타이드 서열 변화)의 자연적 메커니즘을 모방하는 돌연변이 유발에 대한 구현도 이루어집니다.
알고리즘의 기준에서 가장 간단한 정보 전달 단위는 유전자</ b0>입니다. 유전의 구조적 및 기능적 단위로, 특정 특성 또는 속성의 개발을 제어하죠. 우리는 함수의 각 변수를 유전자라고 부를 것입니다. 유전자는 숫자로 나타나죠. 함수의 유전자 변수 세트는 염색체</ b1>의 특징입니다.
열의 형태로 염색체를 표현하기로 합시다. 그러면 함수 f(x)=x^2의 염색체는 다음과 같습니다.
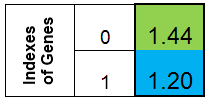
그림 1. 함수 f(x)=x^2의 염색체
f(x) 함수의 0번째 인덱스 값이 개인의 적응으로 불리는(해당 함수를 적합성 함수 'FF</ b0>'라고 부르고 함수 값은 'VFF</ b1>'로 표시). 염색체는 1차원 배열에 저장하는 것이 편합니다. 이건 double Chromosome [] 배열이죠.
동일한 진화 시기의 모든 종은 인구 단위로 합쳐집니다. 더욱이, 인구는 임의적으로 두 개의 동등한 식민지</ b1>(부모 및 후손 식민지)로 나뉩니다. 전체 개체군에서 선택된 부모 종과 GA의 다른 연산자를 교배한 결과, 개체군의 절반 크기에 해당하는 새로운 자손 식민지가 발생해 기존 식민지를 대체합니다.
함수의 최솟값 f(x)=x^2를 검색하는 동안 개인 총 인구는 다음과 같을 수 있습니다.
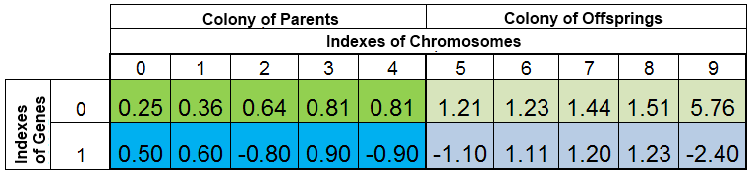
그림 2. 개체 총 인구
인구는 VFF로 분류됩니다. 여기서 염색체의 0 번째 인덱스는 VFF가 가장 작은 표본이 차지합니다. 새로운 자손은 자손의 식민지에 있는 개체만 완전히 대체하는 반면 부모 식민지는 그대로 유지됩니다. 그러나 복제 표본이 파괴되고 새로운 자손이 부모 식민지의 공석을 채우고 나머지는 자손의 식민지에 배치되기 때문에 부모 식민지가 항상 완전하지는 않을 수 있습니다.
즉, 인구 규모는 거의 일정하지 않으며 자연과 거의 같은 방식으로 시대에 따라 다릅니다. 다음은 번식 전후 인구에 대한 예시입니다.
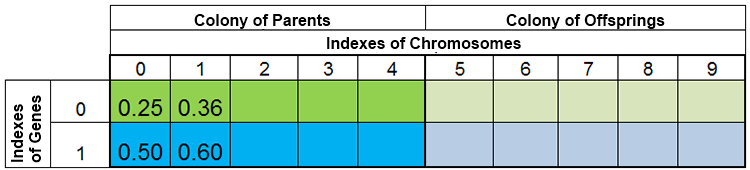
그림 3. 번식 전 인구
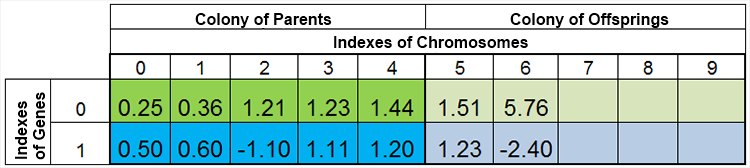
그림 4. 번식 후 인구
후손에 의한 인구의 "반"성취에 대한 설명 된 메커니즘과 중복의 파괴 및 개인과의 교배 금지는 "병목 효과"(용어)를 피하는 단일 목표를 가지고 있습니다. 생물학에서, 이는 종의 완전한 멸종으로 이어질 수있는 여러 가지 이유로 인해 결정적인 감소의 결과로 집단의 유전자 풀의 감소를 의미하며, GA는 독특한 염색체의 출현의 끝을 직면하고 있습니다. 지역 극단 중 하나에서 "막힘").
3. UGAlib 함수 설명
GA 알고리즘- 원형 인구 만들기 유전자는 주어진 범위 내에서 무작위로 생성됩니다.
- 각 개인의 적합성 또는 VFF 계산을 결정합니다.
- 염색체 중복을 제거한 후 번식을 위해 인구를 준비합니다.
- 참조 염색체를 분리 및 보존(최상의 VFF 사용)합니다.
- UGA 연산자(선택, 교배, 돌연변이) 각 짝짓기와 돌연변이에 대해 매번 새로운 부모가 선택됩니다. 다음 시대를 위해 인구를 준비합니다.
- 가장 좋은 자손의 유전자와 참조 염색체의 유전자를 비교합니다. 가장 좋은 자손의 염색체가 참조 염색체보다 나은 경우 참조 염색체를 교체합니다.
지정된 수 만큼의 시대에 대해 나타나는 참조 염색체보다 더 나은 염색체가 더 이상 없을 때까지 단락5부터 반복합니다.
3.1. 전역 변수 전역 변수
다음 변수를 전역 수준에서 선언합니다.
//----------------------Global variables----------------------------- double Chromosome[]; //A set of optimized arguments of the function - genes //(for example: the weight of the neuron network, etc.)- of the chromosome int ChromosomeCount =0; //Maximum possible amount of chromosomes in a colony int TotalOfChromosomesInHistory=0;//Total number of chromosomes in the history int ChrCountInHistory =0; //Number of unique chromosomes in the base chromosome int GeneCount =0; //Number of genes in the chromosome double RangeMinimum =0.0;//Minimum search range double RangeMaximum =0.0;//Maximum search range double Precision =0.0;//Search step int OptimizeMethod =0; //1-minimum, any other - maximum int FFNormalizeDigits =0; //Number of symbols after the comma in the fitness value int GeneNormalizeDigits =0; //Number of symbols after the comma in the genes value double Population [][1000]; //Population double Colony [][500]; //Offspring colony int PopulChromosCount =0; //The current number of chromosomes in a population int Epoch =0; //Number of epochs without progress int AmountStartsFF=0; //Number of launches of the fitness function //————————————————————————————————————————————————————————————————————————
3.2. UGA GA 메인 함수
사실 GA의 주요 함수는 위에 나열된 단계를 수행하기 위해 프로그램 본문에서 호출되므로 자세한 내용은 다루지 않겠습니다.
알고리즘이 완료된 후 다음 정보가 로그에 기록되었습니다.
- 총 몇 세대가 있었는지
- 총 몇 개의 실수가 발생했는지
- 유일 염색체의 개수는 얼마인지
- FF 런치 횟수는 몇 번인지
- 총 몇 개의 염색체가 기록되었는지
- 기록된 총 염색체 수에 대한 중복 비율은 얼마인지
- 최적의 결과
'유일 염색체의 개수'와 'FF 총 런치 횟수'는 크기가 같지만 따로 연산됩니다. 컨트롤 사용
//———————————————————————————————————————————————————————————————————————— //Basic function UGA void UGA ( double ReplicationPortion, // Proportion of replication. double NMutationPortion, // Proportion of natural mutations. double ArtificialMutation, // Proportion of artificial mutations. double GenoMergingPortion, // Proportion of borrowed genes. double CrossingOverPortion, // Proportion of cross -over. //--- double ReplicationOffset, // Coefficient of displacement of interval borders double NMutationProbability // Probability of mutation of each gene in% ) { //generator reset takes place only once MathSrand((int)TimeLocal()); //-----------------------Variables------------------------------------- int chromos=0, gene =0;//indexes of chromosomes and genes int resetCounterFF =0;//counter of resets of "Epochs without progress" int currentEpoch =1;//number of the current epoch int SumOfCurrentEpoch=0;//sum of "Epochs without progress" int MinOfCurrentEpoch=Epoch;//minimum of "epochs without progress" int MaxOfCurrentEpoch=0;//maximum of "Epochs without progress" int epochGlob =0;//total number of epochs // Colony [number of traits(genes)][number of individuals in a colony] ArrayResize(Population,GeneCount+1); ArrayInitialize(Population,0.0); // Colony of offspring [number of traits(genes)][number of individuals in a colony] ArrayResize(Colony,GeneCount+1); ArrayInitialize(Colony,0.0); // Chromosome bank // [number of traits (genes)][number of chromosomes in the bank] double historyHromosomes[][100000]; ArrayResize(historyHromosomes,GeneCount+1); ArrayInitialize(historyHromosomes,0.0); //---------------------------------------------------------------------- //--------------Verification of the correctness of incoming parameters---------------- //...the number of chromosomes mus be less than 2 if(ChromosomeCount<=1) ChromosomeCount=2; if(ChromosomeCount>500) ChromosomeCount=500; //---------------------------------------------------------------------- //====================================================================== // 1) Create a proto- population —————1) ProtopopulationBuilding(); //====================================================================== // 2) Determine the fitness of each individual —————2) //For the 1st colony for(chromos=0;chromos<ChromosomeCount;chromos++) for(gene=1;gene<=GeneCount;gene++) Colony[gene][chromos]=Population[gene][chromos]; GetFitness(historyHromosomes); for(chromos=0;chromos<ChromosomeCount;chromos++) Population[0][chromos]=Colony[0][chromos]; //For the 2nd colony for(chromos=ChromosomeCount;chromos<ChromosomeCount*2;chromos++) for(gene=1;gene<=GeneCount;gene++) Colony[gene][chromos-ChromosomeCount]=Population[gene][chromos]; GetFitness(historyHromosomes); for(chromos=ChromosomeCount;chromos<ChromosomeCount*2;chromos++) Population[0][chromos]=Colony[0][chromos-ChromosomeCount]; //====================================================================== // 3) Prepare the population for reproduction ————3) RemovalDuplicates(); //====================================================================== // 4) Extract the reference chromosome —————4) for(gene=0;gene<=GeneCount;gene++) Chromosome[gene]=Population[gene][0]; //====================================================================== ServiceFunction(); //The main cycle The main cycle of the genetic algorithm from 5 to 6 while(currentEpoch<=Epoch) { //==================================================================== // 5) Operators of UGA —————5) CycleOfOperators ( historyHromosomes, //--- ReplicationPortion, //Proportion of replication. NMutationPortion, //Proportion of natural mutation. ArtificialMutation, //Proportion of artificial mutation. GenoMergingPortion, //Proportion of borrowed genes. CrossingOverPortion,//Proportion of cross- over. //--- ReplicationOffset, //Coefficient of displacement of interval borders NMutationProbability//Probability of mutation of each gene in % ); //==================================================================== // 6) Compare the genes of the best offspring with the genes of the reference chromosome. // If the chromosome of the best offspring is better that the reference chromosome, // replace the reference. —————6) //If the optimization mode is - minimization if(OptimizeMethod==1) { //If the best chromosome of the population is better than the reference chromosome if(Population[0][0]<Chromosome[0]) { //Replace the reference chromosome for(gene=0;gene<=GeneCount;gene++) Chromosome[gene]=Population[gene][0]; ServiceFunction(); //Rest the counter of "epochs without progress" if(currentEpoch<MinOfCurrentEpoch) MinOfCurrentEpoch=currentEpoch; if(currentEpoch>MaxOfCurrentEpoch) MaxOfCurrentEpoch=currentEpoch; SumOfCurrentEpoch+=currentEpoch; currentEpoch=1; resetCounterFF++; } else currentEpoch++; } //If the optimization mode is - minimization else { //If the best chromosome of the population is better than the reference chromosome if(Population[0][0]>Chromosome[0]) { //Replace the reference chromosome for(gene=0;gene<=GeneCount;gene++) Chromosome[gene]=Population[gene][0]; ServiceFunction(); //Reset the counter of "epochs without progress" if(currentEpoch<MinOfCurrentEpoch) MinOfCurrentEpoch=currentEpoch; if(currentEpoch>MaxOfCurrentEpoch) MaxOfCurrentEpoch=currentEpoch; SumOfCurrentEpoch+=currentEpoch; currentEpoch=1; resetCounterFF++; } else currentEpoch++; } //==================================================================== //Another epoch went by.... epochGlob++; } Print("Epochs went by=",epochGlob," Number of resets=",resetCounterFF); Print("MinOfCurrentEpoch",MinOfCurrentEpoch, " AverageOfCurrentEpoch",NormalizeDouble((double)SumOfCurrentEpoch/(double)resetCounterFF,2), " MaxOfCurrentEpoch",MaxOfCurrentEpoch); Print(ChrCountInHistory," - Unique chromosome"); Print(AmountStartsFF," - Total number of launches of FF"); Print(TotalOfChromosomesInHistory," - Total number of chromosomes in the history"); Print(NormalizeDouble(100.0-((double)ChrCountInHistory*100.0/(double)TotalOfChromosomesInHistory),2),"% of duplicates"); Print(Chromosome[0]," - best result"); } //————————————————————————————————————————————————————————————————————————
3.3. 원형 인구 만들기 원형 인구 만들기
대부분의 최적화 문제에서 함수 전달 인자의 숫자 선상 위치를 미리 알 수있는 방법이 없기 때문에 최상의 최적 옵션은 주어진 범위 내에서 임의 생성이 됩니다.
//———————————————————————————————————————————————————————————————————————— //Creating a proto- population void ProtopopulationBuilding() { PopulChromosCount=ChromosomeCount*2; //Fill up the population with chromosomes with random //...genes in the range between RangeMinimum...RangeMaximum for(int chromos=0;chromos<PopulChromosCount;chromos++) { //beginning with the 1st indexes (the 0 index is reserved for VFF) for(int gene=1;gene<=GeneCount;gene++) Population[gene][chromos]= NormalizeDouble(SelectInDiscreteSpace(RNDfromCI(RangeMinimum,RangeMaximum),RangeMinimum,RangeMaximum,Precision,3),GeneNormalizeDigits); TotalOfChromosomesInHistory++; } } //————————————————————————————————————————————————————————————————————————
3.4. GetFitness. 적합성 얻기
각 염색체의 순서에 따른 최적화 함수 실행
//------------------------------------------------ ------------------------ // Getting the fitness for each individual. void GetFitness ( double &historyHromosomes[][100000] ) { for(int chromos=0;chromos<ChromosomeCount;chromos++) CheckHistoryChromosomes(chromos,historyHromosomes); } //————————————————————————————————————————————————————————————————————————
3.5. CheckHistoryChromosomes. 염색체 염기를 통한 염색체 확인
각 개인의 염색체는 염기를 통해 확인됩니다. FF가 계산이 되었는지, 계산이 된 경우 준비된 VFF가 가져오기 되고 그렇지 않은 경우 FF가 호출됩니다. 따라서 반복되는 리소스, 즉 FF에 대한 집중 계산은 제외됩니다.
//———————————————————————————————————————————————————————————————————————— //Verification of chromosome through the chromosome base. void CheckHistoryChromosomes ( int chromos, double &historyHromosomes[][100000] ) { //-----------------------Variables------------------------------------- int Ch1=0; //Index of the chromosome from the base int Ge =0; //Index of the gene int cnt=0; //Counter of unique genes. If at least one gene is different //- the chromosome is acknowledged unique //---------------------------------------------------------------------- //If at least one chromosome is stored in the base if(ChrCountInHistory>0) { //Enumerate the chromosomes in the base to find an identical one for(Ch1=0;Ch1<ChrCountInHistory && cnt<GeneCount;Ch1++) { cnt=0; //Compare the genes, while the gene index is less than the number of genes and while there are identical genes for(Ge=1;Ge<=GeneCount;Ge++) { if(Colony[Ge][chromos]!=historyHromosomes[Ge][Ch1]) break; cnt++; } } //If there are enough identical genes then we can use a ready- made solution from the base if(cnt==GeneCount) Colony[0][chromos]=historyHromosomes[0][Ch1-1]; //If there is no such chromosome, then we calculate the FF for it... else { FitnessFunction(chromos); //.. and if there is space in the base, then save it if(ChrCountInHistory<100000) { for(Ge=0;Ge<=GeneCount;Ge++) historyHromosomes[Ge][ChrCountInHistory]=Colony[Ge][chromos]; ChrCountInHistory++; } } } //If the base is empty, calculate the FF for it and save it in the base else { FitnessFunction(chromos); for(Ge=0;Ge<=GeneCount;Ge++) historyHromosomes[Ge][ChrCountInHistory]=Colony[Ge][chromos]; ChrCountInHistory++; } } //————————————————————————————————————————————————————————————————————————
3.6. CycleOfOperators. UGA 연산자 주기
이 단계에서는 인공 생명의 시대 전체의 운명이 결정됩니다. 새로운 세대가 태어나고 죽죠. 이것은 다음과 같은 방식으로 발생합니다. 두 부모가 짝짓기를 위해 선택되거나 한 부모가 자신을 돌연변이로 만들죠. 각각의 GA 연산자에게 알맞은 매개 변수가 지정됩니다. 그 결과 하나의 후손을 갖게 됩니다. 이는 후손 식민지가 완전히 찰 때까지 반복됩니다. 그런 다음 후손 식민지는 서식지로 내보내져 각 개인이 잘 보이게 됩니다. 우리는 VFF를 계산하죠.
'불, 물 및 구리 파이프'에 의한 테스트 후 후손 식민지는 인구로 정착합니다. 인공 진화의 다음 단계는 미래 세대에서 '혈액'의 고갈을 방지하기 위한 클론의 신성한 살인 및 적합성에 따라 갱신된 인구의 순위를 매기는 것입니다.
//———————————————————————————————————————————————————————————————————————— //Cycle of operators of UGA void CycleOfOperators ( double &historyHromosomes[][100000], //--- double ReplicationPortion, //Proportion of replications. double NMutationPortion, //Proportion of natural mutations. double ArtificialMutation, //Proportion of artificial mutations. double GenoMergingPortion, //Portion of borrowed genes. double CrossingOverPortion,//Proportion of cross-over. //--- double ReplicationOffset, //Coefficient of displacement of interval borders double NMutationProbability//Probability of mutation of each gene in % ) { //-----------------------Variables------------------------------------- double child[]; ArrayResize (child,GeneCount+1); ArrayInitialize(child,0.0); int gene=0,chromos=0, border=0; int i=0,u=0; double p=0.0,start=0.0; double fit[][2]; ArrayResize (fit,6); ArrayInitialize(fit,0.0); //Counter of planting spots in a new population. int T=0; //---------------------------------------------------------------------- //Set a portion of operators of UGA double portion[6]; portion[0]=ReplicationPortion; //Proportion of replications. portion[1]=NMutationPortion; //Proportion of natural mutations. portion[2]=ArtificialMutation; //Proportion of artificial mutations. portion[3]=GenoMergingPortion; //Proportion of borrowed genes. portion[4]=CrossingOverPortion;//Proportion of cross- overs. portion[5]=0.0; //------------------------Cycle of operators of UGA--------- //Fill up the new colony with offspring while(T<ChromosomeCount) { //============================ for(i=0;i<6;i++) { fit[i][0]=start; fit[i][1]=start+MathAbs(portion[i]-portion[5]); start=fit[i][1]; } p=RNDfromCI(fit[0][0],fit[4][1]); for(u=0;u<5;u++) { if((fit[u][0]<=p && p<fit[u][1]) || p==fit[u][1]) break; } //============================ switch(u) { //--------------------- case 0: //------------------------Replication-------------------------------- //If there is space in the new colony, create a new individual if(T<ChromosomeCount) { Replication(child,ReplicationOffset); //Settle the new individual into the new colony for(gene=1;gene<=GeneCount;gene++) Colony[gene][T]=child[gene]; //One place is occupied, fast- forward the counter T++; TotalOfChromosomesInHistory++; } //--------------------------------------------------------------- break; //--------------------- case 1: //---------------------Natural mutation------------------------- //If there is space in the new colony, create a new individual if(T<ChromosomeCount) { NaturalMutation(child,NMutationProbability); //Settle the new individual into the new colony for(gene=1;gene<=GeneCount;gene++) Colony[gene][T]=child[gene]; //One place is occupied, fast- forward the counter T++; TotalOfChromosomesInHistory++; } //--------------------------------------------------------------- break; //--------------------- case 2: //----------------------Artificial mutation----------------------- //If there is space in the new colony, create a new individual if(T<ChromosomeCount) { ArtificialMutation(child,ReplicationOffset); //Settle the new individual into the new colony for(gene=1;gene<=GeneCount;gene++) Colony[gene][T]=child[gene]; //One place is occupied, fast-forward the counter T++; TotalOfChromosomesInHistory++; } //--------------------------------------------------------------- break; //--------------------- case 3: //-------------The creation of an individual with borrowed genes----------- //If there is space in the new colony, create a new individual if(T<ChromosomeCount) { GenoMerging(child); //Settle the new individual into the new colony for(gene=1;gene<=GeneCount;gene++) Colony[gene][T]=child[gene]; //One space is occupied, fast forward the counter T++; TotalOfChromosomesInHistory++; } //--------------------------------------------------------------- break; //--------------------- default: //---------------------------Crossing-Over--------------------------- //If there is place in the new colony, create a new individual if(T<ChromosomeCount) { CrossingOver(child); //Settle the new individual into the new colony for(gene=1;gene<=GeneCount;gene++) Colony[gene][T]=child[gene]; //One place is occupied, fast forward the counter T++; TotalOfChromosomesInHistory++; } //--------------------------------------------------------------- break; //--------------------- } }//End of the cycle operators of UGA-- //Determine the fitness of each individual in the colony of offspring GetFitness(historyHromosomes); //Settle the offspring into the main population if(PopulChromosCount>=ChromosomeCount) { border=ChromosomeCount; PopulChromosCount=ChromosomeCount*2; } else { border=PopulChromosCount; PopulChromosCount+=ChromosomeCount; } for(chromos=0;chromos<ChromosomeCount;chromos++) for(gene=0;gene<=GeneCount;gene++) Population[gene][chromos+border]=Colony[gene][chromos]; //Prepare the population for the next reproduction RemovalDuplicates(); }//the end of the function //————————————————————————————————————————————————————————————————————————
3.7. Replication. 복제
연산자는 생물학에서 DNA 복제라고 불리는 자연 현상에 가장 가깝지만 본질적으로는 같은 것은 아닙니다. 그러나 본질적으로 동등한 것을 찾지 못했기 때문에 이 제목을 유지하기로 했죠.
복제는 부모 염색체의 특성을 전달하면서 새로운 유전자를 생성하는 가장 중요한 유전 연산자입니다. 알고리즘의 수렴을 감시하는 메인 연산자죠. GA는 다른 연산자를 사용하지 않고서도 작동할 수 있지만 이 경우 FF 런치 횟수가 훨씬 많아집니다.
복제 연산자 원리를 생각해 보세요. 두 개의 부모 염색체가 있습니다. 후손 유전자는 인터벌 내에서 선택된 임의의 숫자죠.
[C1-((C2-C1)*ReplicationOffset),C2+((C2-C1)*ReplicationOffset)]
C1과 C2 부모 유전자의 ReplicationOffset -[C1, C2] 인터벌 이동 계수
예를 들어 부계 개인(파란색)과 모계 개인(분홍색)에서부터 아이(녹색)가 탄생할 수 있습니다.
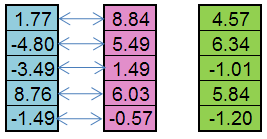
그림 5. 복제 연산자 작동 원리
다음은 그래픽으로 요약한 자손 발생 확률입니다.
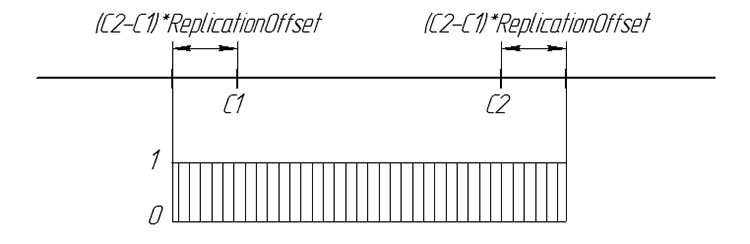
그림 6. 숫자 라인상 자손 유전자 출현 확률
나머지 자손 유전자도 동일한 방법으로 생성됩니다.
//------------------------------------------------ ------------------------ // Replication void Replication ( double &child[], double ReplicationOffset ) { //-----------------------Variables------------------------------------- double C1=0.0,C2=0.0,temp=0.0,Maximum=0.0,Minimum=0.0; int address_mama=0,address_papa=0; //---------------------------------------------------------------------- SelectTwoParents(address_mama,address_papa); //-------------------Cycle of gene enumeration-------------------------------- for(int i=1;i<=GeneCount;i++) { //----figure out where the father and mother came from -------- C1 = Population[i][address_mama]; C2 = Population[i][address_papa]; //------------------------------------------ //Mandatory verification to make sure that the search had not gone over the specified range if(C1 < RangeMinimum) C1 = RangeMinimum; if(C1 > RangeMaximum) C1 = RangeMaximum; if(C2 < RangeMinimum) C2 = RangeMinimum; if(C2 > RangeMaximum) C2 = RangeMaximum; //------------------------------------------------------------------ //....determine the largest and smallest of them, //if we С1>C2, swi if(C1>C2) { temp=C1; C1=C2; C2=temp; } //-------------------------------------------- //Specify the borders of the created gene Minimum = C1-((C2-C1)*ReplicationOffset); Maximum = C2+((C2-C1)*ReplicationOffset); //-------------------------------------------- //Mandatory verification to make sure that the search has not gone over the specified range if(Minimum < RangeMinimum) Minimum = RangeMinimum; if(Maximum > RangeMaximum) Maximum = RangeMaximum; //--------------------------------------------------------------- temp=RNDfromCI(Minimum,Maximum); child[i]= NormalizeDouble(SelectInDiscreteSpace(temp,RangeMinimum,RangeMaximum,Precision,3),GeneNormalizeDigits); } } //————————————————————————————————————————————————————————————————————————3.8. NaturalMutation. 자연 변이
돌연변이는 살아있는 세포의 발생 과정 전반에 걸쳐 지속적으로 발생하며 자연 선택의 대상이 됩니다. 정상적인 서식지 조건에서 유기체의 생애 동안 자발적으로 발생하며 10^10 세포 세대당 한 번의 빈도로 발생합니다.
우리처럼 호기심이 많은 연구자들은 자연적 순서를 지키면서 다음 변이를 기다릴 여유가 없죠. 백분율로 표시되며 염색체의 각 유전자에 대한 돌연변이 확률을 결정하는 NMutationProbability 매개 변수가 우리를 도와줄 겁니다.
NaturalMutation 연산자의 경우, 돌연변이는 [RangeMinimum, RangeMaximum] 인터벌에서 임의의 유전자 생성으로 구성됩니다. NMutationProbability=100%인 경우 염색체 내 유전자의 100% 변이를, NMutationProbability=0%인 경우 변이가 전혀 일어나지 않음을 의미합니다. 실제 문제 해결에 마지막 옵션은 거의 사용되지 않겠죠.
//------------------------------------------------ ------------------------ // The natural mutation. void NaturalMutation ( double &child[], double NMutationProbability ) { //-----------------------Variables------------------------------------- int address=0; double prob=0.0; //---------------------------------------------------------------------- if(NMutationProbability<0.0) prob=0.0; if(NMutationProbability>100.0) prob=100.0; //-----------------Parent selection------------------------ SelectOneParent(address); //--------------------------------------- for(int i=1;i<=GeneCount;i++) if(RNDfromCI(0.0,100.0)<prob) child[i]=NormalizeDouble( SelectInDiscreteSpace(RNDfromCI(RangeMinimum,RangeMaximum),RangeMinimum,RangeMaximum,Precision,3),GeneNormalizeDigits ); } //————————————————————————————————————————————————————————————————————————
3.9. ArtificialMutation. 인공 변이
연산자의 주요 임무는 '신선한' 피의 생성입니다. 두 개의 부모를 이용하며, 자손의 유전자는 부모 유전자에 의해 할당되지 않은 숫자 선상의 공백에서 선택됩니다. GA가 국소 극단에 갇히지 않도록 보호합니다. 다른 연산자에 비해 더 큰 비율로 수렴을 가속화하거나 속도를 늦추어 FF 런치 횟수를 늘립니다.
복제와 같이 두 개의 부모 염색체를 사용합니다. 그러나 ArtificialMutation 연산자의 임무는 부모의 특성을 자손에게 전달하는 것이 아니라 자식을 그들과 다르게 만드는 것입니다. 따라서 동일한 간격 경계 변위 계수를 사용하여 완전히 반대이지만 유전자는 간격 밖에서 생성되며 이는 복제가 되었을 것입니다. 후손의 새로운 유전자는 [RangeMinimum, C1-(C2-C1) * ReplicationOffset] 및 [C2 + (C2-C1) * ReplicationOffset, RangeMaximum] 인터벌에서 무작위로 선정됩니다.
다음은 그래픽으로 후손의 유전자 확률 ReplicationOffset=0.25를 나타낸 것입니다.
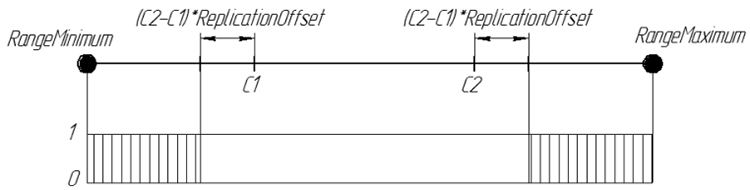
그림 7. 실선 인터벌 [RangeMinimum; RangeMaximum]에 나타난 후손 유전자 확률 ReplicationOffset=0.25
//———————————————————————————————————————————————————————————————————————— //Artificial mutation. void ArtificialMutation ( double &child[], double ReplicationOffset ) { //-----------------------Variables------------------------------------- double C1=0.0,C2=0.0,temp=0.0,Maximum=0.0,Minimum=0.0,p=0.0; int address_mama=0,address_papa=0; //---------------------------------------------------------------------- //-----------------Selecting parents------------------------ SelectTwoParents(address_mama,address_papa); //-------------------------------------------------------- //-------------------Cycle of genes enumeration------------------------------ for(int i=1;i<=GeneCount;i++) { //----determine where the mother and father are from -------- C1 = Population[i][address_mama]; C2 = Population[i][address_papa]; //------------------------------------------ //Mandatory verification to make sure that the search doesn't go beyond the specified range if(C1 < RangeMinimum) C1 = RangeMinimum; if(C1 > RangeMaximum) C1 = RangeMaximum; if(C2 < RangeMinimum) C2 = RangeMinimum; if(C2 > RangeMaximum) C2 = RangeMaximum; //------------------------------------------------------------------ //....determine the largest and smallest of them, //if С1>C2, we change their places if(C1>C2) { temp=C1; C1=C2; C2=temp; } //-------------------------------------------- //Specify the borders of creating the new gene Minimum=C1-((C2-C1)*ReplicationOffset); Maximum=C2+((C2-C1)*ReplicationOffset); //-------------------------------------------- //Mandatory verification to make sure that the search doesn't go beyond the specified range if(Minimum < RangeMinimum) Minimum = RangeMinimum; if(Maximum > RangeMaximum) Maximum = RangeMaximum; //--------------------------------------------------------------- p=MathRand(); if(p<16383.5) { temp=RNDfromCI(RangeMinimum,Minimum); child[i]= NormalizeDouble(SelectInDiscreteSpace(temp,RangeMinimum,RangeMaximum,Precision,3),GeneNormalizeDigits); } else { temp=RNDfromCI(Maximum,RangeMaximum); child[i]= NormalizeDouble(SelectInDiscreteSpace(temp,RangeMinimum,RangeMaximum,Precision,3),GeneNormalizeDigits); } } } //————————————————————————————————————————————————————————————————————————
3.10 GenoMerging. 유전자 빌리기
주어진 GA 연산자는 자연에서 동등한 상대를 갖지 않습니다. 사실이 놀라운 메커니즘이 살아있는 유기체에서 어떻게 기능하는지 상상하기는 어렵습니다. 그러나 많은 부모(부모의 수는 유전자의 수와 동일)로부터 자손에게 유전자를 전달하는 놀라운 특성이 있습니다. 연산자는 새로운 유전자를 생성하지 않으며 조합 검색 메커니즘에 해당합니다.
첫 번째 자손 유전자의 경우 부모가 선택되고 첫 번째 유전자가 그에게서 취해진 다음 두 번째 유전자의 경우 두 번째 부모가 선택되고 유전자가 그에게서 취해집니다. 유전자 수가 1 이상일 때 적용이 권장됩니다. 그렇지 않으면 운영자가 염색체의 복제를 생성하므로 비활성화해야 합니다.
//———————————————————————————————————————————————————————————————————————— //Borrowing genes. void GenoMerging ( double &child[] ) { //-----------------------Variables------------------------------------- int address=0; //---------------------------------------------------------------------- for(int i=1;i<=GeneCount;i++) { //-----------------Selecting parents------------------------ SelectOneParent(address); //-------------------------------------------------------- child[i]=Population[i][address]; } } //————————————————————————————————————————————————————————————————————————
3.11. CrossingOver. 교차
교차는 염색체 일부분을 교환하는 현상입니다. GenoMerging과 마찬가지로 조합 검색 메커니즘이죠.
두 개의 부모 염색체가 선택됩니다. 둘 모두 무작위로 '잘리게' 됩니다. 후손의 염색체는 부모 염색체의 여러 부분으로 이루어집니다.
그림으로 설명하는 게 제일 쉽죠.
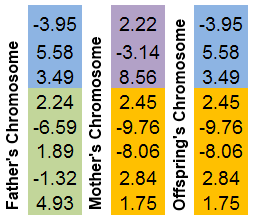
그림 8. 부분 염색체 교환 메커니즘
유전자 수가 1 이상일 때 적용이 권장됩니다. 그렇지 않으면 운영자가 염색체의 복제를 생성하므로 비활성화해야 합니다.
//———————————————————————————————————————————————————————————————————————— //Crossing-over. void CrossingOver ( double &child[] ) { //-----------------------Variables------------------------------------- int address_mama=0,address_papa=0; //---------------------------------------------------------------------- //-----------------Selecting parents------------------------ SelectTwoParents(address_mama,address_papa); //-------------------------------------------------------- //Determine the breakage point int address_of_gene=(int)MathFloor((GeneCount-1)*(MathRand()/32767.5)); for(int i=1;i<=GeneCount;i++) { //----copy the mother's genes-------- if(i<=address_of_gene+1) child[i]=Population[i][address_mama]; //----copy the father's genes-------- else child[i]=Population[i][address_papa]; } } //————————————————————————————————————————————————————————————————————————
3.12. SelectTwoParents. 두 부모 선택
유전자 풀의 고갈을 막기 위해 자기 교배를 금지하고 있습니다. 다른 부모를 찾기 위해 10번의 시도 후에도 쌍을 이루지 못하면 자기 교배가 허용됩니다. 동일한 종이 복사되는 것과 마찬가지죠.
한편으로는 자가 복제 확률이 감소하며, 다른 한편으로는 검색의 순환성이 방지됩니다. 적당한 횟수의 시도로도 다른 부모를 선택하지 못하는 상황이 발생할 수 있기 때문입니다.
Replication, ArtificialMutation 및 CrossingOver 연산자에서 사용됩니다.
//———————————————————————————————————————————————————————————————————————— //Selection of two parents. void SelectTwoParents ( int &address_mama, int &address_papa ) { //-----------------------Variables------------------------------------- int cnt=1; address_mama=0;//address of the mother individual in a population address_papa=0;//address of the father individual in a population //---------------------------------------------------------------------- //----------------------------Selection of parents-------------------------- //Ten attempts to chose different parents. while(cnt<=10) { //For the mother individual address_mama=NaturalSelection(); //For the father individual address_papa=NaturalSelection(); if(address_mama!=address_papa) break; } //--------------------------------------------------------------------- } //————————————————————————————————————————————————————————————————————————
3.13. SelectOneParent. 한 부모 선택
간단합니다. 인구에서 한 부모가 선택되는 거죠.
NaturalMutation 및 GenoMerging 연산자에서 사용됩니다.
//———————————————————————————————————————————————————————————————————————— //Selection of one parent. void SelectOneParent ( int &address//address of the parent individual in the population ) { //-----------------------Variables------------------------------------- address=0; //---------------------------------------------------------------------- //----------------------------Selecting a parent-------------------------- address=NaturalSelection(); //--------------------------------------------------------------------- } //————————————————————————————————————————————————————————————————————————
3.14. NaturalSelection. 자연 선택
자연 선택-환경 조건에 더 잘 적응하는 유용한 유전적 특성을 지닌 개인의 생존 및 우선 번식으로 이어지는 과정.
연산자는 기존 연산자 "룰렛"과 유사합니다 (룰렛 휠 선택-룰렛을 n 번 "실행"하는 개인 선택). 룰렛 휠에는 인구의 각 구성원에 대해 하나의 섹터가 있습니다. i번째 섹터의 크기는 해당 피트니스 값에 비례하지만 상당한 차이가 있습니다. 가장 적합한 사람과 가장 적합한 사람에 비해 개인의 위치를 고려합니다. 더욱이 최악의 유전자를 가진 개체조차도 자손을 남길 기회가 있습니다. 공정하죠. 아닌가요? 공정성에 관한 것이 아니라 사실상 모든 개인이 자손을 떠날 기회가 있다는 사실에 관한 것입니다.
예를 들어, 최대화 문제에서 다음 VFF가있는 10 명의 개인을 취하십시오 : 256, 128, 64, 32, 16, 8, 4, 2, 0, -1-여기서 큰 값은 더 나은 체력에 해당합니다. 이 예는 이웃 개인 간의 "거리"가 이전 두 개인 간의 "거리"보다 2 배 더 큼을 알 수 있도록 취해졌습니다. 그러나 원형 차트에서 각 개인이 자손을 떠날 확률은 다음과 같습니다.
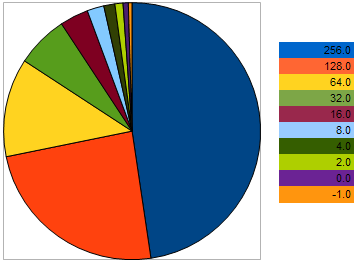
그림 9. 부모 선택 확률 차트
개인이 최악의 상황에 접근하면 기회가 더 나빠진다는 것을 보여줍니다. 반대로, 개인이 더 나은 표본에 가까울수록 번식 가능성이 높아집니다.

그림 10. 부모 선택 확률 차트
//———————————————————————————————————————————————————————————————————————— //Natural selection. int NaturalSelection() { //-----------------------Variables------------------------------------- int i=0,u=0; double p=0.0,start=0.0; double fit[][2]; ArrayResize (fit,PopulChromosCount); ArrayInitialize(fit,0.0); double delta=(Population[0][0]-Population[0][PopulChromosCount-1])*0.01-Population[0][PopulChromosCount-1]; //---------------------------------------------------------------------- for(i=0;i<PopulChromosCount;i++) { fit[i][0]=start; fit[i][1]=start+MathAbs(Population[0][i]+delta); start=fit[i][1]; } p=RNDfromCI(fit[0][0],fit[PopulChromosCount-1][1]); for(u=0;u<PopulChromosCount;u++) if((fit[u][0]<=p && p<fit[u][1]) || p==fit[u][1]) break; return(u); } //————————————————————————————————————————————————————————————————————————
3.15. RemovalDuplicates. 사본 삭제
이 기능은 모집단에서 중복 염색체를 제거하고 나머지 고유 염색체 (현재 시대의 모집단에 고유)는 최적화 유형 (예 : 감소 또는 증가)에 의해 결정된 VFF에 따라 순서대로 정렬됩니다.
//———————————————————————————————————————————————————————————————————————— //Removing duplicates sorted by VFF void RemovalDuplicates() { //-----------------------Variables------------------------------------- int chromosomeUnique[1000];//Array stores the unique trait //of each chromosome: 0-duplicate, 1-unique ArrayInitialize(chromosomeUnique,1); //Assume that there are no duplicates double PopulationTemp[][1000]; ArrayResize (PopulationTemp,GeneCount+1); ArrayInitialize(PopulationTemp,0.0); int Ge =0; //Index of the gene int Ch =0; //Index of the chromosome int Ch2=0; //Index of the second chromosome int cnt=0; //Counter //---------------------------------------------------------------------- //----------------------Remove duplicates---------------------------1 //Chose the first from the pair for comparison... for(Ch=0;Ch<PopulChromosCount;Ch++) { //If it's not a duplicate... if(chromosomeUnique[Ch]!=0) { //Chose the second from the pair... for(Ch2=0;Ch2<PopulChromosCount;Ch2++) { if(Ch!=Ch2 && chromosomeUnique[Ch2]!=0) { //Zeroize the counter of identical genes cnt=0; //Compare the genes. while there are identical genes present for(Ge=1;Ge<=GeneCount;Ge++) { if(Population[Ge][Ch]!=Population[Ge][Ch2]) break; else cnt++; } //If there are the same amount of identical genes as total genes //..the chromosome is considered a duplicate if(cnt==GeneCount) chromosomeUnique[Ch2]=0; } } } } //The counter calculates the number of unique chromosomes cnt=0; //Copy the unique chromosomes into a temporary array for(Ch=0;Ch<PopulChromosCount;Ch++) { //If the chromosome is unique, copy it, if not, go to the next if(chromosomeUnique[Ch]==1) { for(Ge=0;Ge<=GeneCount;Ge++) PopulationTemp[Ge][cnt]=Population[Ge][Ch]; cnt++; } } //Assigning the variable "All chromosomes" the value of counter of unique chromosomes PopulChromosCount=cnt; //Return unique chromosomes back to the array for temporary storage //..of combined populations for(Ch=0;Ch<PopulChromosCount;Ch++) for(Ge=0;Ge<=GeneCount;Ge++) Population[Ge][Ch]=PopulationTemp[Ge][Ch]; //=================================================================1 //----------------Ranking the population---------------------------2 PopulationRanking(); //=================================================================2 } //————————————————————————————————————————————————————————————————————————
3.16. PopulationRanking. 인구 순위 부여
VFF에 의해 분류됩니다. 이 방법은 'bubbly'와 유사합니다(알고리즘은 정렬된 배열을 통해 반복되는 구절로 구성). 모든 패스에 대해 요소는 연속적으로 쌍으로 비교되며 쌍의 순서가 잘못된 경우 요소 교환이 발생합니다. 배열을 통과하는 통로는 통로 중 하나가 교환이 더 이상 필요하지 않음을 보여줄 때까지 반복됩니다. 즉, 배열이 정렬됩니다.
알고리즘을 통과 할 때 눈에 띄는 요소가 물 속의 거품처럼 원하는 위치로 "팝업"하므로 알고리즘의 이름이 있지만 차이가 있습니다. 배열의 인덱스 만 배열의 내용이 아니라 정렬됩니다. 이 방법은 단순히 한 배열을 다른 배열로 복사하는 것과 속도면에서 더 빠르고 약간 다릅니다. 정렬된 배열의 크기가 클수록 차이가 작아집니다.
//———————————————————————————————————————————————————————————————————————— //Population ranking. void PopulationRanking() { //-----------------------Variables------------------------------------- int cnt=1, i = 0, u = 0; double PopulationTemp[][1000]; //Temporary population ArrayResize (PopulationTemp,GeneCount+1); ArrayInitialize(PopulationTemp,0.0); int Indexes[]; //Indexes of chromosomes ArrayResize (Indexes,PopulChromosCount); ArrayInitialize(Indexes,0); int t0=0; double ValueOnIndexes[]; //VFF of corresponding //..chromosome indexes ArrayResize (ValueOnIndexes,PopulChromosCount); ArrayInitialize(ValueOnIndexes,0.0); double t1=0.0; //---------------------------------------------------------------------- //Fill in the indexes in the temporary array temp2 and //...copy the first line from the sorted array for(i=0;i<PopulChromosCount;i++) { Indexes[i] = i; ValueOnIndexes[i] = Population[0][i]; } if(OptimizeMethod==1) { while(cnt>0) { cnt=0; for(i=0;i<PopulChromosCount-1;i++) { if(ValueOnIndexes[i]>ValueOnIndexes[i+1]) { //----------------------- t0 = Indexes[i+1]; t1 = ValueOnIndexes[i+1]; Indexes [i+1] = Indexes[i]; ValueOnIndexes [i+1] = ValueOnIndexes[i]; Indexes [i] = t0; ValueOnIndexes [i] = t1; //----------------------- cnt++; } } } } else { while(cnt>0) { cnt=0; for(i=0;i<PopulChromosCount-1;i++) { if(ValueOnIndexes[i]<ValueOnIndexes[i+1]) { //----------------------- t0 = Indexes[i+1]; t1 = ValueOnIndexes[i+1]; Indexes [i+1] = Indexes[i]; ValueOnIndexes [i+1] = ValueOnIndexes[i]; Indexes [i] = t0; ValueOnIndexes [i] = t1; //----------------------- cnt++; } } } } //Create a sorted-out array based on the obtained indexes for(i=0;i<GeneCount+1;i++) for(u=0;u<PopulChromosCount;u++) PopulationTemp[i][u]=Population[i][Indexes[u]]; //Copy the sorted-out array back for(i=0;i<GeneCount+1;i++) for(u=0;u<PopulChromosCount;u++) Population[i][u]=PopulationTemp[i][u]; } //————————————————————————————————————————————————————————————————————————
3.17. RNDfromCustomInterval. 인터벌별 랜덤 넘버 생성기
편리한 기능이죠. UGA에서 유용하게 쓰입니다.
//———————————————————————————————————————————————————————————————————————— //Generator of random numbers from the selected interval. double RNDfromCI(double RangeMinimum,double RangeMaximum) { return(RangeMinimum+((RangeMaximum-RangeMinimum)*MathRand()/32767.5));} //————————————————————————————————————————————————————————————————————————
3.18. SelectInDiscreteSpace. 개별 공간 내 선택
검색 공간을 줄이기 위해 사용됩니다. 매개 변수 step=0.0인 경우 검색은 연속되는 공간에서 실행됩니다(언어 제한, MQL에서 15번째 의미 기호 포함). GA 알고리즘을 더 정확하게 사용하려면 긴 숫자 작업을위한 추가 라이브러리를 작성해야합니다.
RoundMode=1인 경우 함수 작동은 다음과 같이 이루어집니다.
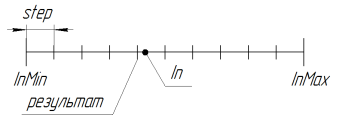
그림 11. RoundMode=1일 때 SelectInDiscreteSpace 함수 실행 과정
//———————————————————————————————————————————————————————————————————————— //Selection in discrete space. //Modes: //1-closest below //2-closest above //any closest double SelectInDiscreteSpace ( double In, double InMin, double InMax, double step, int RoundMode ) { if(step==0.0) return(In); // secure the correctness of borders if( InMax < InMin ) { double temp = InMax; InMax = InMin; InMin = temp; } // during a breach - return the breached border if( In < InMin ) return( InMin ); if( In > InMax ) return( InMax ); if( InMax == InMin || step <= 0.0 ) return( InMin ); // bring to the specified scale step = (InMax - InMin) / MathCeil ( (InMax - InMin) / step ); switch( RoundMode ) { case 1: return( InMin + step * MathFloor ( ( In - InMin ) / step ) ); case 2: return( InMin + step * MathCeil ( ( In - InMin ) / step ) ); default: return( InMin + step * MathRound ( ( In - InMin ) / step ) ); } } //————————————————————————————————————————————————————————————————————————
3.19. FitnessFunction. 적합성 함수
는 GA에 포함되지 않습니다. 이 함수는 VFF가 계산될 인구의 염색체 인덱스를 받습니다. VFF는 전달된 염색체의 제로 인덱스에 쓰여 있습니다. 각 연산 별로 함수 코드가 상이합니다.
3.20. ServiceFunction. 서비스 함수
는 GA에 포함되지 않습니다. 따라서 각 태스크별 함수 코드가 상이합니다. 세대에 대한 통제를 실행할 수 있습니다. 예를 들어 현재 세대의 최상 VFF를 표시할 수 있습니다.
4. UGA 예제
모든 최적화 문제는 EA로 해결되며 두 가지 형식으로 분류됩니다.
- 유전자형과 표현형이 일치하는 경우 염색체 유전자의 값은 최적화 함수의 인수에 의해 직접 지정됩니다. 예제 1.
- 유전자형과 표현형이 일치하지 않는 경우 최적화된 기능을 계산하려면 염색체 유전자의 의미에 대한 해석이 필요합니다. 예제 2.
4.1. 예제 1
알고리즘이 작동하는지 확인하기 위해 알려진 답변이있는 문제를 고려한 다음 문제 해결로 이동합니다. 그 해결책은 많은 거래자들에게 관심이 있습니다.
문제: 'skin' 함수의 최솟값 및 최대값 찾기
인터벌 [-5, 5]
정답: fmin (3.07021,3.315935) = -4.3182, fmax (-3.315699; -3.072485) = 14.0606.
그림 12. 인터벌 [-5, 5]에 대한 'skin' 그래프
문제 해결을 위해 다음의 스크립트를 작성합니다.
#property script_show_inputs //+——————————————————————————————————————————————————————————————————————+ #include "UGAlib.mqh" #include "Skin.mqh"//testing function //+——————————————————————————————————————————————————————————————————————+ //———————————————————————————————————————————————————————————————————————— //----------------------Incoming parameters-------------------------------- input string GenofundParam = "----Gene pool parameter----"; input int ChromosomeCount_P = 50; //Number of chromosomes in a colony input int GeneCount_P = 2; //Number of genes input int FFNormalizeDigits_P = 4; //Number of fitness symbols input int GeneNormalizeDigits_P = 4; //Number of genes input int Epoch_P = 50; //Number of epochs without progress //--- input string GA_OperatorParam = "----Operator parameters----"; input double ReplicationPortion_P = 100.0; //Proportion of replication. input double NMutationPortion_P = 10.0; //Proportion of natural mutations. input double ArtificialMutation_P = 10.0; //Proportion of artificial mutations. input double GenoMergingPortion_P = 20.0; //Proportion of borrowed genes. input double CrossingOverPortion_P = 20.0; //Proportion of crossing-over. //--- input double ReplicationOffset_P = 0.5; //Coefficient of interval borders displacement input double NMutationProbability_P= 5.0; //Probability of mutation of each gene in % //--- input string OptimisationParam = "----Optimization parameters----"; input double RangeMinimum_P = -5.0; //Minimum range search input double RangeMaximum_P = 5.0; //Maximum range search input double Precision_P = 0.0001;//The required accuracy input int OptimizeMethod_P = 1; //Optim.:1-Min,other-Max //———————————————————————————————————————————————————————————————————————— //———————————————————————————————————————————————————————————————————————— //----------------------Global variables----------------------------- double ERROR=0.0;//Average error in gen //———————————————————————————————————————————————————————————————————————— //———————————————————————————————————————————————————————————————————————— //--------------------------The body of the program-------------------------------- int OnStart() { //-----------------------Variables------------------------------------- //Preparing global variables for UGA ChromosomeCount=ChromosomeCount_P; //Number of chromosomes in the colony GeneCount =GeneCount_P; //Number of genes RangeMinimum =RangeMinimum_P; //Minimum range search RangeMaximum =RangeMaximum_P; //Maximum range search Precision =Precision_P; //Search step OptimizeMethod =OptimizeMethod_P; //1-minimum, any other - maximum FFNormalizeDigits = FFNormalizeDigits_P; //Number of symbols in fitness GeneNormalizeDigits = GeneNormalizeDigits_P;//Number of gene symbols ArrayResize(Chromosome,GeneCount+1); ArrayInitialize(Chromosome,0); Epoch=Epoch_P; //Number of epochs without progress //---------------------------------------------------------------------- //Local variables int time_start=GetTickCount(),time_end=0; //---------------------------------------------------------------------- //Launch of the main function UGA UGA ( ReplicationPortion_P, //Proportion of replication. NMutationPortion_P, //Proportion of natural mutations. ArtificialMutation_P, //Proportion of artificial mutations. GenoMergingPortion_P, //Proportion of borrowed genes. CrossingOverPortion_P,//Proportion of crossing-over. //--- ReplicationOffset_P, //Coefficient of interval border replacement NMutationProbability_P//Probability of mutation of each gene in % ); //---------------------------------- time_end=GetTickCount(); //---------------------------------- Print(time_end-time_start," mc - Time of implementation"); //---------------------------------- return(0); } //————————————————————————————————————————————————————————————————————————
다음은 문제 해결 스크립트의 전체 코드입니다. 실행하여 Comment () 함수가 제공하는 정보를 획득하세요.
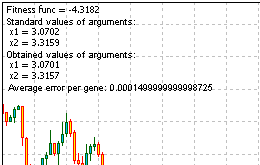
그림 13. 문제 해결 결과
결과를 보니 알고리즘이 제대로 작동한 것 같군요.
4.2. 예제 2
지표 ZZ는 전복 거래 시스템의 이상적인 입력을 보여주는 것으로 널리 알려져 있습니다. 이 지표는 "파동 이론"지지자들과 "모형"의 크기를 결정하는 데 사용하는 사람들 사이에서 매우 인기가 있습니다.
문제: ZZ 정점과 다른 과거 데이터에 대한 전복 거래 시스템에 대한 더 많은 이론적 이득 포인트를 제공하는 다른 진입점이 있나?
실험을 위해 M1 100 바에 GBPJPY 쌍을 선택합니다. 80포인트(5자리 값)의 분산을 수락합니다. 시작에 앞서 ZZ 변수를 정해야 합니다. 이를 위해 간단한 스크립트를 사용하여 ExtDepth 매개 변수의 최상의 값을 찾기 위해 간단한 열거를 사용합니다.
#property script_show_inputs //———————————————————————————————————————————————————————————————————————— //----------------------Incoming parameters-------------------------------- input int History=100; input double Spred =80.0; input int Depth =5; //For "one-time" use input bool loop =true;//Use enumeration or not //———————————————————————————————————————————————————————————————————————— //———————————————————————————————————————————————————————————————————————— //--------------------------The body of the program-------------------------------- void OnStart() { //-----------------------Variables------------------------------------- double ZigzagBuffer [];//For storing the buffer of the ZZ indicator double PeaksOfZigzag[];//for storing the values of the ZZ extremum int Zigzag_handle; //Indicator marker ArraySetAsSeries(ZigzagBuffer,true); ArrayResize(PeaksOfZigzag,History); int depth=3; double PipsSum=0.0; int PeaksCount=0; bool flag=true; //---------------------------------------------------------------------- if(loop==true) { while(depth<200 && flag==true) { //----------------------------------------------------------- Zigzag_handle=iCustom(NULL,0,"ZigZag",depth); //--- reset the code error ResetLastError(); //--- attempt to copy the indicator values for(int i=0;i<100;i++) { if(BarsCalculated(Zigzag_handle)>0) break; Sleep(1000); } int copied=CopyBuffer(Zigzag_handle,0,0,History,ZigzagBuffer); if(copied<=0) { Print("Could not copy the indicator buffer. Error =",GetLastError()," copied=",copied); return; } //----------------------------------------------------------- PipsSum=0.0; PeaksCount=0; for(int u=0;u<History;u++) { if(NormalizeDouble(ZigzagBuffer[u],Digits())>0.0) { PeaksOfZigzag[PeaksCount]=NormalizeDouble(ZigzagBuffer[u],Digits()); PeaksCount++; } } //----------------------------------------------------------- for(int V=0;V<PeaksCount-1;V++) PipsSum+=NormalizeDouble((MathAbs(PeaksOfZigzag[V]-PeaksOfZigzag[V+1]))/Point(),Digits())-Spred; //----------------------------------------------------------- if(PeaksCount<=2) flag=false; else { Print(depth," ",PeaksCount," ",PipsSum); depth+=1; } //----------------------------------------------------------- } } else { //----------------------------------------------------------- Zigzag_handle=iCustom(NULL,0,"ZigZag",Depth); //--- reser the error code ResetLastError(); //--- attempt to copy the indicator values for(int i=0;i<History;i++) { if(BarsCalculated(Zigzag_handle)>0) break; Sleep(1000); } int copied=CopyBuffer(Zigzag_handle,0,0,History,ZigzagBuffer); if(copied<=0) { Print("Was not able to copy the buffer indicator. Error =",GetLastError()," copied=",copied); return; } //----------------------------------------------------------- for(int u=0;u<History;u++) { if(NormalizeDouble(ZigzagBuffer[u],Digits())>0.0) { PeaksOfZigzag[PeaksCount]=NormalizeDouble(ZigzagBuffer[u],Digits()); PeaksCount++; } } //----------------------------------------------------------- for(int V=0;V<PeaksCount-1;V++) { PipsSum+=NormalizeDouble((MathAbs(PeaksOfZigzag[V]-PeaksOfZigzag[V+1]))/Point(),Digits())-Spred; } Print(Depth," ",PeaksCount," ",PipsSum); //----------------------------------------------------------- } } //————————————————————————————————————————————————————————————————————————
스크립트를 실행해 ExtDepth=3인 4077 포인트를 얻습니다. 19개의 지표 꼭지점이 100개의 바에 '맞습니다'. ExtDepth가 증가하면 ZZ 정점의 수가 감소하고 수익성도 감소합니다.
이제 UGA를 사용하여 대체 ZZ의 정점을 찾을 수 있습니다. ZZ 정점은 각 막대에 대해 1) 높음, 2) 낮음, 3) 정점 없음의 세 위치를 가질 수 있습니다. 정점의 존재와 위치는 각 막대에 대한 모든 유전자에 의해 전달됩니다. 따라서 염색체의 크기는 유전자 100개가 됩니다.
내 계산에 따르면 (그리고 내가 틀렸다면 수학자가 나를 고칠 수 있음), 100 개의 막대에서 3 ^ 100 또는 5.15378e47 대체 옵션 "지그재그"</ b0>를 만들 수 있습니다. 이것은 직접 열거를 사용하여 고려해야 할 정확한 옵션 수입니다. 초당 100000000 옵션의 속도로 계산하는 동안 1.6e32 년 </ b1>이 필요합니다! 우주의 나이보다도 큰 숫자입니다. 이 문제에 대한 해결책을 찾는 능력에 의문이 생기기 시작했을 때입니다.
하지만 계속하죠.
UGA는 염색체를 실수로 표현하기 때문에 꼭지점의 위치를 인코딩해야합니다. 이것은 염색체의 유전자형이 표현형과 일치하지 않는 경우입니다. 유전자 검색 인터벌을 [0, 5]로 설정합니다. 간격 [0, 1]은 높음의 ZZ 정점에 해당하고, 간격 [4, 5]는 낮음의 정점에 해당하고, 간격 (1, 4)는 꼭지점이 없음에 해당합니다.
한 가지 중요한 점을 고려할 필요가 있습니다. 원형 집단은 지정된 간격의 유전자로 무작위로 생성되기 때문에 첫 번째 표본은 마이너스 기호가있는 수백 점에서도 매우 열악한 결과를 나타냅니다. 몇 세대 후에 (1 세대에서 일어날 가능성이 있지만) 우리는 일반적으로 꼭지점이없는 것과 유전자가 일치하는 표본의 출현을 보게 될 것입니다. 이것은 거래의 부재와 불가피한 스프레드의 지불을 의미합니다.
일부 전직 투자자에 따르면 "투자를 위한 최고의 전략은 투자를 하지 않는 것입니다". 이 개인은 인공 진화의 정점이 될 것입니다. 이 "인공적인" 진화가 투자 개인을 낳도록 만들기 위해, 즉 대체 ZZ의 꼭지점을 배열하도록 하기 위해, 우리는 꼭지점이 없는 개인의 적합성, "-10000000.0"의 값을 할당하여 의도적으로 가장 낮은 위치에 배치합니다.
다음은 대체 ZZ의 정점을 찾기 위해 UGA를 사용하는 스크립트 코드입니다.
#property script_show_inputs //+——————————————————————————————————————————————————————————————————————+ #include "UGAlib.mqh" //+——————————————————————————————————————————————————————————————————————+ //———————————————————————————————————————————————————————————————————————— //----------------------Incoming parameters-------------------------------- input string GenofundParam = "----Parameters of the gene pool----"; input int ChromosomeCount_P = 100; //Number of chromosomes in the colony input int GeneCount_P = 100; //Number of genes input int FFNormalizeDigits_P = 0; //Number of fitness symbols input int GeneNormalizeDigits_P= 0; //Number of gene symbols input int Epoch_P = 50; //Number of epochs without progress //--- input string GA_OperatorParam = "----Parameters of operators----"; input double ReplicationPortion_P = 100.0; //Proportion of replication. input double NMutationPortion_P = 10.0; //Proportion of natural mutations. input double ArtificialMutation_P = 10.0; //Proportion of artificial mutations. input double GenoMergingPortion_P = 20.0; //Proportion of borrowed genes. input double CrossingOverPortion_P = 20.0; //Proportion of crossing - over. input double ReplicationOffset_P = 0.5; //Coefficient of interval border displacement input double NMutationProbability_P= 5.0; //Probability of mutation of each gene in % //--- input string OptimisationParam = "----Optimization parameters----"; input double RangeMinimum_P = 0.0; //Minimum search range input double RangeMaximum_P = 5.0; //Maximum search range input double Precision_P = 1.0; //Required accuracy input int OptimizeMethod_P = 2; //Optim.:1-Min,other -Max input string Other = "----Other----"; input double Spred = 80.0; input bool Show = true; //———————————————————————————————————————————————————————————————————————— //———————————————————————————————————————————————————————————————————————— //----------------------Global variables----------------------------- double Hight []; double Low []; datetime Time []; datetime Ti []; double Peaks []; bool show; //———————————————————————————————————————————————————————————————————————— //--------------------------Body of the program-------------------------------- int OnStart() { //-----------------------Variables------------------------------------- //Preparation of global variables for UGA ChromosomeCount=ChromosomeCount_P; //Number of chromosomes in the colony GeneCount =GeneCount_P; //Number of genes RangeMinimum =RangeMinimum_P; //Minimum search range RangeMaximum =RangeMaximum_P; //Maximum search range Precision =Precision_P; //Searching step OptimizeMethod =OptimizeMethod_P; //1-minimum, any other - maximum FFNormalizeDigits = FFNormalizeDigits_P; //Number of fitness symbols GeneNormalizeDigits = GeneNormalizeDigits_P;//Number of gene symbols ArrayResize(Chromosome,GeneCount+1); ArrayInitialize(Chromosome,0); Epoch=Epoch_P; //Number of epochs without progress //---------------------------------------------------------------------- //Preparation of global variables ArraySetAsSeries(Hight,true); CopyHigh (NULL,0,0,GeneCount+1,Hight); ArraySetAsSeries(Low,true); CopyLow (NULL,0,0,GeneCount+1,Low); ArraySetAsSeries(Time,true); CopyTime (NULL,0,0,GeneCount+1,Time); ArrayResize (Ti,GeneCount+1);ArrayInitialize(Ti,0); ArrayResize(Peaks,GeneCount+1);ArrayInitialize(Peaks,0.0); show=Show; //---------------------------------------------------------------------- //local variables int time_start=GetTickCount(),time_end=0; //---------------------------------------------------------------------- //Очистим экран ObjectsDeleteAll(0,-1,-1); ChartRedraw(0); //launch of the main function UGA UGA ( ReplicationPortion_P, //Proportion of replication. NMutationPortion_P, //Proportion of replication of natural mutations. ArtificialMutation_P, //Proportion of artificial mutations. GenoMergingPortion_P, //Proportion of borrowed genes. CrossingOverPortion_P,//proportion of crossing- over. //--- ReplicationOffset_P, //Coefficient of interval border displacement NMutationProbability_P//Probability of mutation of each gene in % ); //---------------------------------- //Display the last result on the screen show=true; ServiceFunction(); //---------------------------------- time_end=GetTickCount(); //---------------------------------- Print(time_end-time_start," мс - time of execution"); //---------------------------------- return(0); } //———————————————————————————————————————————————————————————————————————— //———————————————————————————————————————————————————————————————————————— //-----------------------------------------------------------------------+ // Service function. Called up from UGA. | | //If there is no need for it, leave the function empty, like this: | // void ServiceFunction() | // { | // } | //-----------------------------------------------------------------------+ void ServiceFunction() { if(show==true) { //-----------------------Variables----------------------------------- double PipsSum=0.0; int PeaksCount=0; double temp=0.0; //-------------------------------------------------------------------- for(int u=1;u<=GeneCount;u++) { temp=Chromosome[u]; if(temp<=1.0 ) { Peaks[PeaksCount]=NormalizeDouble(Hight[u],Digits()); Ti [PeaksCount]=Time[u]; PeaksCount++; } if(temp>=4.0) { Peaks[PeaksCount]=NormalizeDouble(Low[u],Digits()); Ti [PeaksCount]=Time[u]; PeaksCount++; } } ObjectsDeleteAll(0,-1,-1); for(int V=0;V<PeaksCount-1;V++) { PipsSum+=NormalizeDouble((MathAbs(Peaks[V]-Peaks[V+1]))/Point(),FFNormalizeDigits)-Spred; ObjectCreate (0,"BoxBackName"+(string)V,OBJ_TREND,0,Ti[V],Peaks[V],Ti[V+1],Peaks[V+1]); ObjectSetInteger(0,"BoxBackName"+(string)V,OBJPROP_COLOR,Black); ObjectSetInteger(0,"BoxBackName"+(string)V,OBJPROP_SELECTABLE,true); } ChartRedraw(0); Comment(PipsSum); } //---------------------------------------------------------------------- else return; } //———————————————————————————————————————————————————————————————————————— //———————————————————————————————————————————————————————————————————————— //-----------------------------------------------------------------------+ // Function of determining the fitness of the individual. Called up from UGA. | //-----------------------------------------------------------------------+ void FitnessFunction(int chromos) { //-----------------------Variables------------------------------------- double PipsSum=0.0; int PeaksCount=0; double temp=0.0; //---------------------------------------------------------------------- for(int u=1;u<=GeneCount;u++) { temp=Colony[u][chromos]; if(temp<=1.0) { Peaks[PeaksCount]=NormalizeDouble(Hight[u],Digits()); PeaksCount++; } if(temp>=4.0) { Peaks[PeaksCount]=NormalizeDouble(Low[u],Digits()); PeaksCount++; } } if(PeaksCount>1) { for(int V=0;V<PeaksCount-1;V++) PipsSum+=NormalizeDouble((MathAbs(Peaks[V]-Peaks[V+1]))/Point(),FFNormalizeDigits)-Spred; Colony[0][chromos]=PipsSum; } else Colony[0][chromos]=-10000000.0; AmountStartsFF++; } //————————————————————————————————————————————————————————————————————————
스크립트를 실행하면 총 수익이 4939포인트인 꼭지점을 얻습니다. 또한 직접 열거를 통해 필요한 3^100에 비해 점수를 계산하는 데 17,929번 밖에 걸리지 않았습니다. 제 컴퓨터로 보면 1.6e32년 대비 21.7초입니다!
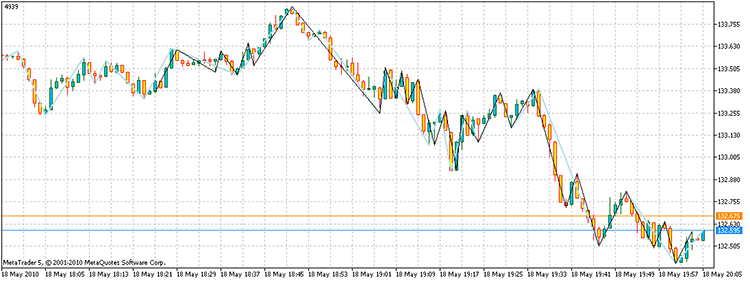
그림 14. 문제 해결 결과 검은색 세그먼트-대체 ZZ, 하늘색-ZZ 인디케이터
따라서 질문에 대한 답은 다음과 같습니다. "존재합니다."
5. UGA 사용 관련 조언
- 알고리즘에서 적절한 결과를 기대할 수 있도록 FF에서 예상 조건을 올바르게 설정하세요. 예제 2를 다시 생각해 보시고요. 아마 이게 가장 중요한 조언일 거예요.
- Precision 매개 변수에 너무 작은 값을 사용하지 마세요. 알고리즘이 0단계로 작동할 수도 있지만 솔루션의 적절한 정확도를 요청해야합니다. 이 매개 변수는 문제의 규모를 줄이는 역할을 합니다.
- 모집단의 크기와 세대 수의 임계 값을 변경합니다. 좋은 해결책은 MaxOfCurrentEpoch에 표시된 것보다 두 배 더 큰 세대 매개 변수를 할당하는 것입니다. 그렇다고 숫자가 클수록 더 빠르게 해결책을 찾을 수 있다는 건 아닙니다.
- 매개 변수 및 유전 연산자와 이것 저것 실험해 보세요. 보편적인 매개 변수는 없으며 작업 조건에 따라 정해야 합니다.
결론
강력한 터미널 전략 테스터와 MQL5 언어를 사용하면 투자자를 위한 뛰어난 도구를 만들어 복잡한 문제를 해결할 수 있습니다. 굉장히 유연하고 가늠 가능한 최적화 알고리즘을 얻게 되죠. 제가 처음 발견한 건 아니지만 제 자신이 자랑스럽네요.
UGA는 처음에 이러한 방식으로 설계되었기 때문에 추가 연산자 및 계산 블록을 사용하여 쉽게 수정하고 확장할 수 있으므로 독자는 "인공" 진화의 개발에 쉽게 기여할 수 있습니다.
독자분들이 최상의 해결책을 찾으시면 좋겠습니다. 제가 도움이 됐으면 좋겠네요. 행운을 빕니다!
노트 이 글에서는 ZigZag 인디케이터가 사용되었습니다. 모든 UGA 소스 코드가 첨부되어 있습니다.
라이센스: 본문에 포함된 소스 코드(UGA 코드)는 BSD 라이센스 조건을 따라 첨부되었습니다.
MetaQuotes 소프트웨어 사를 통해 러시아어가 번역됨.
원본 기고글: https://www.mql5.com/ru/articles/55
경고: 이 자료들에 대한 모든 권한은 MetaQuotes(MetaQuotes Ltd.)에 있습니다. 이 자료들의 전부 또는 일부에 대한 복제 및 재출력은 금지됩니다.
이 글은 사이트 사용자가 작성했으며 개인의 견해를 반영합니다. Metaquotes Ltd는 제시된 정보의 정확성 또는 설명 된 솔루션, 전략 또는 권장 사항의 사용으로 인한 결과에 대해 책임을 지지 않습니다.
 다양한 대륙과 시간대에 맞춘 거래 전략의 예시
다양한 대륙과 시간대에 맞춘 거래 전략의 예시
 바보도 할 수 있는 MQL: 객체 클래스 디자인 및 생성 방법
바보도 할 수 있는 MQL: 객체 클래스 디자인 및 생성 방법
 문자 알림과 트레이드 리포트 생성 및 발간
문자 알림과 트레이드 리포트 생성 및 발간
 뉴비들을 위한 복합 인디케이터 버퍼 만들기
뉴비들을 위한 복합 인디케이터 버퍼 만들기
제발 오류목록을 보여주세요 .코드는매우 오래되었지만 그러나 14 년후에도 심지어 깨뜨릴 것이 없습니다.
문제는 "ArtificialMutation" 변수와 함수에 있습니다. 함수를 ArtificialMutationP로 변경했는데 작동합니다.
서비스 함수 및 피트니스 함수 오류 문제는 괜찮으며 무슨 일이 일어나고 있는지 이해합니다. 스크린샷을 첨부했습니다.
ZIP에서 추출한 후 UGA_script 또는 UGA_the_alternative_ZigZag를 직접 컴파일하려고 할 때 UGAlib에서 발생하는 오류가 발생합니다.
함수 이름(2번의 경우 호출)을 ArtificialMutationP로 변경하면 컴파일되고 작동했습니다.
나는 여전히 완전히 이해하기 위해 당신의 훌륭한 작업을 통해 작업하고 있습니다. 고마워요
질문.
과거 데이터(막대 수)가 유전자 수인데 이것이 맞나요?
변수와 함수에 문제가 있는 것 같습니다. 함수를 ArtificialMutationP로 변경했는데 작동합니다.
ServiceFunction 및 FitnessFunction 오류 문제는 괜찮으며 무슨 일이 일어나고 있는지 이해합니다. 스크린샷을 첨부했습니다.
ZIP에서 추출한 후 UGA_script 또는 UGA_the_alternative_ZigZag를 직접 컴파일하려고 할 때 UGAlib에서 발생하는 오류가 발생합니다.
함수 이름(2번의 경우 호출)을 ArtificialMutationP로 변경하자 컴파일이 되고 작동했습니다.
나는 여전히 완전히 이해하기 위해 당신의 훌륭한 작업을 통해 작업하고 있습니다. 고마워요
시도 이 파일
친절한 말씀 감사합니다 .