
Neuroredes profundas (Parte V). Optimización bayesiana de los hiperparámetros de las DNN
Contenido
- Introducción
- 1 Definiendo los hiperparámetros óptimos de la DNN (darch)
- Formando los conjuntos iniciales de datos
- Eliminando los predictores estadísticamente irrelevantes
- Definiendo los hiperparámetros de la DNN para optimizar y determinar su límite de cambio
- Definiendo las funciones de preentrenamiento y ajuste preciso DNN
- Definiendo las funciones de adecuación para la optimización
- Calculando los parámetros óptimos de la DNN
- 2. Entrenamiento y simulación de la DNN con los parámetros óptimos
- 3. Análisis de los resultados de la simulación de la DNN con los parámetros óptimos
- 4. Forward test de los modelos con los parámetros óptimos
- Conclusión
- Anexos
Introducción
En el artículo anterior de este ciclo entrenamos un modelo básico y un modelo de DNN con parámetros por defecto. La calidad de la clasificación de nuestro modelo ha resultado insatisfactoria. ¿Qué podemos hacer para aumentar la calidad de la clasificación?
- Optimizar los hiperparémtros de la DNN
- Aumentar la regularización de la DNN
- Aumentar el número de ejemplos para el entrenamiento
- Modificar la estructura de la neurored
En este y los siguientes artículos proponemos analizar todas las posibilidades enumeradas para el refuerzo de las DNN disponibles. Vamos a comenzar con los hiperparámetros de la red.
1 Definiendo los hiperparámetros óptimos de la DNN
Los hiperparámetros de la neurored en general se pueden dividir en dos grupos: globales y locales (de nodo). En los hiperparámetros globales se incluye el número de capas ocultas, el número de neuronas en cada capa, el nivel de entrenamiento y tiempo para inicializar los pesos de las neuronas. Los hiperparámetros locales son: el tipo de capa, la función de activación, dropout/dropconect y otros parámetros de regularización.
La estructura de optimización de los hiperparámetros se presenta a continuación:
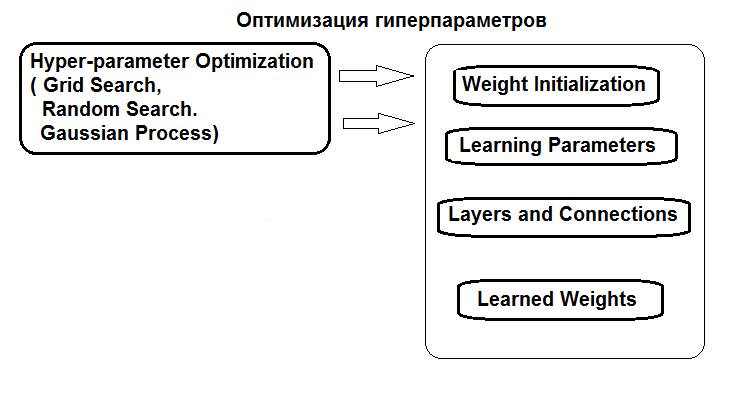
Fig. 1. Estructura de los hiperparámetros de la red y métodos de optimización
Es posible optimizar los hiperparámetros tres maneras:
- Búsqueda en cuadrícula: para cada hiperparámetro se establece un vector con varios valores fijos. Luego, utilizando la función caret::train() o bien un script propio, se entrena el modelo con todas las combinaciones de valores de los hiperparámetros. Después de eso, elegimos el modelo con los mejores índices de calidad. Precisamente sus parámetros se tomarán como óptimos. La desventaja de este método es que al establecer una cuadrícula de valores, es probable que se pierda el óptimo.
- Optimización genética: búsqueda estocástica de los mejores parámetros mediante el uso de algoritmos genéticos. Ya hemos analizado anteriormente con bastante detalle varios algoritmos de optimización genética. Por no vamos a hacerlo de nuevo.
- Y por último, la optimización bayesiana. La vamos a utilizar en este artículo.
El enfoque bayesiano implica procesos gaussianos y MCMC. Usaremos el paquete rBayesianOptimization (version 1.1.0). La teoría de los métodos utilizados se muestra ampliamente en la literatura sobre el tema y se presenta, por ejemplo, en este artículo.
Para llevar a cabo la optimización bayesiana, tenemos que:
- determinar la función de adecuación;
- determinar la lista y los límites de los cambios de los hiperparámetros.
la función de adecuación (FA) deberá retornar el índice de calidad (criterio de optimización, magnitud escalar) que debe ser maximizado durante la optimización, así como los valores predichos para la variable meta. La FA retornará el valor mean(F1), es decir, el valor medio F1 para las dos clases. Vamos a entrenar el modelo de la DNN con preentrenamiento.
Formando los conjuntos iniciales de datos
Para los experimentos usaremos una nueva versión de MRO 3.4.2. En ella se implementan varios paquetes nuevos, que no hemos utilizado antes.
Iniciamos RStudio, cargamos desde GitHub/Part_I el archivo Cotir.RData, que contiene las cotizaciones obtenidas del terminal, y el archivo FunPrepareData.R con las funciones de preparación de datos de GitHub/Part_IV.
Ya hemos determinado anteriormente que un conjunto de datos con las muestras imputadas y los datos normalizados permite obtener mejores resultados cuando se entrena con preentrenamiento. Usted también podrá comprobar las otras variantes de preprocesamiento que hemos analizado con anterioridad.
Al dividir los apartados en pretrain/train/val/test, usamos la primera posibilidad de mejora de la calidad de la clasificación, ya que estamos aumentando el número de ejemplos para el entrenamiento. En el apartado pretrain, aumentamos el número de ejemplos hasta 4000.
#----Prepare------------- library(anytime) library(rowr) library(darch) library(rBayesianOptimization) library(foreach) library(magrittr) #source(file = "FunPrepareData.R") #source(file = "FUN_Optim.R") #---prepare---- evalq({ dt <- PrepareData(Data, Open, High, Low, Close, Volume) DT <- SplitData(dt, 4000, 1000, 500, 100, start = 1) pre.outl <- PreOutlier(DT$pretrain) DTcap <- CappingData(DT, impute = T, fill = T, dither = F, pre.outl = pre.outl) preproc <- PreNorm(DTcap, meth = meth) DTcap.n <- NormData(DTcap, preproc = preproc) }, env)
Modificando el parámetro start en la función SplitData(), podemos obtener conjuntos desplazados a la derecha en la magnitud start. Esto nos permitirá comprobar la calidad en diferentes partes del rango de precios, y determinar cómo cambia en la historia.
Eliminando los predictores estadísticamente irrelevantes
Quitamos las dos variables c(v.rstl, v.pcci)., estadísticamente irrelevantes. Las hemos definido en el anterior artículo de la serie.
##---Data DT-------------- require(foreach) evalq({ foreach(i = 1:4) %do% { DTcap.n[[i]] %>% dplyr::select(-c(v.rstl, v.pcci)) } -> DT list(pretrain = DT[[1]], train = DT[[2]], val = DT[[3]], test = DT[[4]]) -> DT }, env)
Creamos los conjuntos de datos (pretrain/train/test/test1) para el preentrenamiento, el ajuste preciso y la simulación, reunidos en la lista X.
#-----Data X------------------
evalq({
list(
pretrain = list(
x = DT$pretrain %>% dplyr::select(-c(Data, Class)) %>% as.data.frame(),
y = DT$pretrain$Class %>% as.data.frame()
),
train = list(
x = DT$train %>% dplyr::select(-c(Data, Class)) %>% as.data.frame(),
y = DT$train$Class %>% as.data.frame()
),
test = list(
x = DT$val %>% dplyr::select(-c(Data, Class)) %>% as.data.frame(),
y = DT$val$Class %>% as.data.frame()
),
test1 = list(
x = DT$test %>% dplyr::select(-c(Data, Class)) %>% as.data.frame(),
y = DT$test$Class %>% as.vector()
)
) -> X
}, env)
Los conjuntos para la realización de experimentos ya están listos.
Necesitamos una función que calcule la métrica según los resultados de la simulación. El valor mean(F1) lo usaremos como criterio de optimización (maximización). Cargamos esta función en el entorno env.
evalq( #input actual & predicted vectors or actual vs predicted confusion matrix # https://github.com/saidbleik/Evaluation/blob/master/eval.R Evaluate <- function(actual=NULL, predicted=NULL, cm=NULL){ if (is.null(cm)) { actual = actual[!is.na(actual)] predicted = predicted[!is.na(predicted)] f = factor(union(unique(actual), unique(predicted))) actual = factor(actual, levels = levels(f)) predicted = factor(predicted, levels = levels(f)) cm = as.matrix(table(Actual = actual, Predicted = predicted)) } n = sum(cm) # number of instances nc = nrow(cm) # number of classes diag = diag(cm) # number of correctly classified instances per class rowsums = apply(cm, 1, sum) # number of instances per class colsums = apply(cm, 2, sum) # number of predictions per class p = rowsums / n # distribution of instances over the classes q = colsums / n # distribution of instances over the predicted classes #accuracy accuracy = sum(diag) / n #per class recall = diag / rowsums precision = diag / colsums f1 = 2 * precision * recall / (precision + recall) #macro macroPrecision = mean(precision) macroRecall = mean(recall) macroF1 = mean(f1) #1-vs-all matrix oneVsAll = lapply(1:nc, function(i){ v = c(cm[i,i], rowsums[i] - cm[i,i], colsums[i] - cm[i,i], n - rowsums[i] - colsums[i] + cm[i,i]); return(matrix(v, nrow = 2, byrow = T))}) s = matrix(0, nrow = 2, ncol = 2) for (i in 1:nc) {s = s + oneVsAll[[i]]} #avg accuracy avgAccuracy = sum(diag(s))/sum(s) #micro microPrf = (diag(s) / apply(s,1, sum))[1]; #majority class mcIndex = which(rowsums == max(rowsums))[1] # majority-class index mcAccuracy = as.numeric(p[mcIndex]) mcRecall = 0*p; mcRecall[mcIndex] = 1 mcPrecision = 0*p; mcPrecision[mcIndex] = p[mcIndex] mcF1 = 0*p; mcF1[mcIndex] = 2 * mcPrecision[mcIndex] / (mcPrecision[mcIndex] + 1) #random accuracy expAccuracy = sum(p*q) #kappa kappa = (accuracy - expAccuracy) / (1 - expAccuracy) #random guess rgAccuracy = 1 / nc rgPrecision = p rgRecall = 0*p + 1 / nc rgF1 = 2 * p / (nc * p + 1) #rnd weighted rwgAccurcy = sum(p^2) rwgPrecision = p rwgRecall = p rwgF1 = p classNames = names(diag) if (is.null(classNames)) classNames = paste("C",(1:nc),sep = "") return(list( ConfusionMatrix = cm, Metrics = data.frame( Class = classNames, Accuracy = accuracy, Precision = precision, Recall = recall, F1 = f1, MacroAvgPrecision = macroPrecision, MacroAvgRecall = macroRecall, MacroAvgF1 = macroF1, AvgAccuracy = avgAccuracy, MicroAvgPrecision = microPrf, MicroAvgRecall = microPrf, MicroAvgF1 = microPrf, MajorityClassAccuracy = mcAccuracy, MajorityClassPrecision = mcPrecision, MajorityClassRecall = mcRecall, MajorityClassF1 = mcF1, Kappa = kappa, RandomGuessAccuracy = rgAccuracy, RandomGuessPrecision = rgPrecision, RandomGuessRecall = rgRecall, RandomGuessF1 = rgF1, RandomWeightedGuessAccurcy = rwgAccurcy, RandomWeightedGuessPrecision = rwgPrecision, RandomWeightedGuessRecall = rwgRecall, RandomWeightedGuessWeightedF1 = rwgF1))) }, env) #-------------------------
La función retorna un amplio espectro de métricas, de las cuales necesitaremos ahora el valor F1.
Vamos a utilizar una neurored con dos capas ocultas, como en la parte anterior del artículo. El entrenamiento de la DNN se realizará en dos etapas, con preentrenamiento. Las variantes posibles son:
- Preentrenamiento:
- solo entrenamos SRBM;
- entrenamos SRBM + la capa superior de la neurored.
- Entrenamiento preciso:
- usamos el método de entrenamiento backpropagation;
- usamos el método de entrenamiento rpropagation.
Cada una de las cuatro variantes de entrenamiento tiene un conjunto distinto de hiperparámetros para la optimización.
Definiendo los hiperparámetros para la optimización
Vamos a definir la lista de parámetros cuyos valores se supone que vamos a optimizar, y también sus límites de cambio:
- n1, n2 — número de neuronas en cada capa oculta. Rango de cambio: de 1 a 25. Antes de ser introducido en el modelo, el parámetro se multiplica por 2, puesto que necesitamos un número múltiple de 2 (poolSize). Esto será necesario para la función de activación maxout.
- fact1, fact2 son los índices de la función de activación para cada capa oculta, elegidos de la lista de funciones de activación establecidas por el vector Fact <- c("tanhUnit","maxoutUnit","softplusUnit", "sigmoidUnit"). Usted también puede añadir otras funciones.
- dr1, dr2 — magnitud del cribado en cada capa, rango: de 0 a 0.5.
- Lr.rbm — nivel de entrenamiento de StackedRBM, rango: de 0.01 a 1.0 en la etapa de preentrenamiento.
- Lr.top — nivel de entrenamiento de la capa superior de la neurored en la etapa de preentrenamiento, rango de 0.01 a 1.0. En el caso del preentrenamiento no supervisado de la capa superior de la neurored, este parámetro no será necesario.
- Lr.fine — nivel de entrenamiento de la neurored en la etapa de ajuste preciso al usar backpropagation, rango de 0.01 a 1.0. Al usar rpropagation, este parámetro no será necesario.
En el anterior artículo se muestra una descripción detallada de todos los parámetros, y también en la descripción del paquete. Recordemos que todos los parámetros de la función darch() tienen los valores por defecto. Se los puede dividir en varios grupos.
- Parámetros globales. Se usan tanto para el preentrenamiento, como para el ajuste preciso.
- Parámetros de preprocesamiento de datos. Se usan las capacidades de caret::preProcess().
- Parámetros para SRBM. Solo se usan al realizar el preentrenamiento.
- Parámetros NN. Se usan tanto para el preentrenamiento como para el ajuste preciso, pero pueden tener diferentes valores para cada una de las etapas.
El valor de los parámetros por defecto se puede modificar creando una lista con sus nuevos valores o inscribiéndolos en la función darch() explícitamente. Vamos a aclarar brevemente los hiperparámetros que optimizaremos.
Primero indicamos los parámetros de la DNN comunes para las etapas pretrain/train.
Ln <- c(0, 2*n1, 2*n2, 0) — vector que indica que estamos creando una red de 4 capas con dos capas ocultas. El número de neuronas en las capas de entrada y de salida se determina a partir de los datos de entrada, no se puede especificar de forma explícita. En las capas ocultas, el número de neuronas es 2*n1 y 2*n2, respectivamente.
A continuación, definimos los niveles de entrenamiento para RBM (Lr.rbm), para la capa superior de la DNN en el preentrenamiento Lr.top. y para todas las capas en el ajuste preciso Lr.fine.
fact1/fact2 — indican el índice de la función de activación para cada capa oculta de la lista de funciones de activación establecida por el vector Fact. En la capa de salida, aplicamos la función softmax.
dr1/dr2 — nivel de cribado en cada capa oculta.
darch.trainLayers — indica qué capas se deben entrenar en el preentrenamiento y qué capas durante el ajuste preciso.
Inscribimos los hiperparámetros y los límites de su cambio para cada una de las 4 variantes de aprendizaje/optimización. Aparte, sacamos los parámatros Bs.rbm = 100 (rbm.batchSize) y Bs.nn = 50 (darch.batchSize) para mayor comodidad a la hora de buscar las mejores variantes de aprendizaje. Al reducirlos, la calidad de la clasificación mejora, pero crece significativamente el tiempo de optimización.
#-2---------------------- evalq({ #--InitParams--------------------- Fact <- c("tanhUnit","maxoutUnit","softplusUnit", "sigmoidUnit") wUpd <- c("weightDecayWeightUpdate", "maxoutWeightUpdate", "weightDecayWeightUpdate", "weightDecayWeightUpdate") #---SRBM + RP---------------- bonds1 <- list( #n1, n2, fact1, fact2, dr1, dr2, Lr.rbm n1 = c(1L, 25L), n2 = c(1L, 25L), fact1 = c(1L, 4L), fact2 = c(1L, 4L), dr1 = c(0, 0.5), dr2 = c(0, 0.5), Lr.rbm = c(0.01, 1.0)#, ) #---SRBM + BP---------------- bonds2 <- list( #n1, n2, fact1, fact2, dr1, dr2, Lr.rbm, Lr.fine n1 = c(1L, 25L), n2 = c(1L, 25L), fact1 = c(1L, 4L), fact2 = c(1L, 4L), dr1 = c(0, 0.5), dr2 = c(0, 0.5), Lr.rbm = c(0.01, 1.0), Lr.fine = c(0.01, 1.0) ) #---SRBM + upperLayer + BP---- bonds3 <- list( #n1, n2, fact1, fact2, dr1, dr2, Lr.rbm , Lr.top, Lr.fine n1 = c(1L, 25L), n2 = c(1L, 25L), fact1 = c(1L, 4L), fact2 = c(1L, 4L), dr1 = c(0, 0.5), dr2 = c(0, 0.5), Lr.rbm = c(0.01, 1.0), Lr.top = c(0.01, 1.0), Lr.fine = c(0.01, 1.0) ) #---SRBM + upperLayer + RP----- bonds4 <- list( #n1, n2, fact1, fact2, dr1, dr2, Lr.rbm, Lr.top n1 = c(1L, 25L), n2 = c(1L, 25L), fact1 = c(1L, 4L), fact2 = c(1L, 4L), dr1 = c(0, 0.5), dr2 = c(0, 0.5), Lr.rbm = c(0.01, 1.0), Lr.top = c(0.01, 1.0) ) Bs.rbm <- 100L Bs.nn <- 50L },envir = env)
Definimos las funciones del preentrenemiento y el ajuste preciso
Entrenaremos las DNN con las cuatro variantes. Todas las funciones necesarias para ello están en el script FUN_Optim.R, que se debe cargar antes de calcular Git/PartV.
Funciones de preentrenamiento y ajuste preciso para cada una de las variantes:
- pretrainSRBM(Ln, fact1, fact2, dr1, dr2, Lr.rbm ) — preentrenamiento solo SRBM
- pretrainSRBM_topLayer(Ln, fact1, fact2, dr1, dr2, Lr.rbm, Lr.top) — preentrenamiento SRBM + upper Layer (backpropagation)
- fineTuneRP(Ln, fact1, fact2, dr1, dr2, Dnn) — ajuste preciso de la DNN usando rpropagation
- fineTuneBP(Ln, fact1, fact2, dr1, dr2, Dnn, Lr.fine) — ajuste preciso de la DNN usando backpropagation
Para no saturar el artículo con listas de funciones monótonas, analizaremos con detalle solo la variante con preentrenamiento (SRBM + topLayer) + RP(ajuste preciso rpropagation). Esta variante ha demostrado el mejor resultado en la mayoría de los casos en nuestros numerosos experimentos. Las funciones para el resto de variantes son análogas.
# SRBM + upper Layer (backpropagation) pretrainSRBM_topLayer <- function(Ln, fact1, fact2, dr1, dr2, Lr.rbm, Lr.top) # SRBM + upper Layer (backpropagation) { darch( x = X$pretrain$x, y = X$pretrain$y, xValid = X$train$x, yValid = X$train$y, #=====constant====================================== layers = Ln, paramsList = list(), darch = NULL, shuffleTrainData = T, seed = 54321, logLevel = "WARN", #FATAL, ERROR, WARN, DEBUG, and TRACE. #--optimization parameters---------------------------------- darch.unitFunction = c(Fact[fact1], Fact[fact2], "softmaxUnit"), darch.weightUpdateFunction = c(wUpd[fact1], wUpd[fact2], "weightDecayWeightUpdate"), rbm.learnRate = Lr.rbm, bp.learnRate = Lr.top, darch.dropout = c(0, dr1, dr2), #=== params RBM ============== rbm.numEpochs = 30L, rbm.allData = T, rbm.batchSize = Bs.rbm, rbm.consecutive = F, rbm.errorFunction = mseError, #rmseError rbm.finalMomentum = 0.9, rbm.initialMomentum = 0.5, rbm.momentumRampLength = 1, rbm.lastLayer = -1, rbm.learnRateScale = 1, rbm.numCD = 1L, rbm.unitFunction = tanhUnitRbm, rbm.updateFunction = rbmUpdate, rbm.weightDecay = 2e-04, #=== parameters NN ======================== darch.numEpochs = 30L, darch.batchSize = Bs.nn, darch.trainLayers = c(FALSE, FALSE,TRUE ), darch.fineTuneFunction = "backpropagation", #rpropagation bp.learnRateScale = 1, #0.99 #--weight----------------- generateWeightsFunction = generateWeightsGlorotUniform, # generateWeightsUniform (default), # generateWeightsGlorotUniform, # generateWeightsHeUniform. # generateWeightsNormal, # generateWeightsGlorotNormal, # generateWeightsHeNormal, darch.weightDecay = 2e-04, normalizeWeights = T, normalizeWeightsBound = 15, #--parameters regularization----------- darch.dither = F, darch.dropout.dropConnect = F, darch.dropout.oneMaskPerEpoch = T, darch.maxout.poolSize = 2L, darch.maxout.unitFunction = "exponentialLinearUnit", darch.elu.alpha = 2, darch.returnBestModel = T #darch.returnBestModel.validationErrorFactor = 0, ) }
Como conjunto de validación, en esta función usaremos los valores del conjunto X$train.
Función de ajuste preciso con uso de rpropagation. De esta función, aparte de los parámtros, transmitiremos la estructura de la Dnn entrenada de forma preliminar con preentrenamiento.
fineTuneRP <- function(Ln, fact1, fact2, dr1, dr2, Dnn) # rpropagation { darch( x = X$train$x, y = X$train$y, #xValid = X$test$x, yValid = X$test$y, xValid = X$test$x %>% head(250), yValid = X$test$y %>% head(250), #=====constant====================================== layers = Ln, paramsList = list(), darch = Dnn, shuffleTrainData = T, seed = 54321, logLevel = "WARN", #FATAL, ERROR, WARN, DEBUG, and TRACE. rbm.numEpochs = 0L, #--optimization parameters---------------------------------- darch.unitFunction = c(Fact[fact1], Fact[fact2], "softmaxUnit"), darch.weightUpdateFunction = c(wUpd[fact1], wUpd[fact2], "weightDecayWeightUpdate"), darch.dropout = c(0, dr1, dr2), #=== parameters NN ======================== darch.numEpochs = 50L, darch.batchSize = Bs.nn, darch.trainLayers = c(TRUE,TRUE, TRUE), darch.fineTuneFunction = "rpropagation", #"rpropagation" "backpropagation" #=== params RPROP ====== rprop.decFact = 0.5, rprop.incFact = 1.2, rprop.initDelta = 1/80, rprop.maxDelta = 50, rprop.method = "iRprop+", rprop.minDelta = 1e-06, #--weight----------------- darch.weightDecay = 2e-04, normalizeWeights = T, normalizeWeightsBound = 15, #--parameters regularization----------- darch.dither = F, darch.dropout.dropConnect = F, darch.dropout.oneMaskPerEpoch = T, darch.maxout.poolSize = 2L, darch.maxout.unitFunction = "exponentialLinearUnit", darch.elu.alpha = 2, darch.returnBestModel = T #darch.returnBestModel.validationErrorFactor = 0, ) }
Aquí usaremos como conjunto de validación los primeros 250 valores de X$test. Todas las funciones para todas las variantes de entrenamiento deberán cargarse en el entorno env.
Definiendo las funciones de adecuación
Usando estas dos funciones, escribiremos la función de adecuación, necesaria para optimizar los hiperparámetros. Esta retorna el valor del criterio de optimización Score = mean(F1), que se debe maximizar, y los valores predichos de la variable meta Ypred.
#---SRBM + upperLayer + RP---- fitnes4.DNN <- function(n1, n2, fact1, fact2, dr1, dr2, Lr.rbm, Lr.top) { Ln <- c(0, 2*n1, 2*n2, 0) #-- pretrainSRBM_topLayer(Ln, fact1, fact2, dr1, dr2, Lr.rbm, Lr.top) -> Dnn fineTuneRP(Ln, fact1, fact2, dr1, dr2, Dnn) -> Dnn predict(Dnn, newdata = X$test$x %>% tail(250) , type = "class") -> Ypred yTest <- X$test$y[ ,1] %>% tail(250) #numIncorrect <- sum(Ypred != yTest) #Score <- 1 - round(numIncorrect/nrow(xTest), 2) Score <- Evaluate(actual = yTest, predicted = Ypred)$Metrics$F1 %>% mean() return(list(Score = Score, Pred = Ypred) }
Definiendo los parámetros óptimos de la DNN
Iniciamos la función de optimización BayesianOptimization() con el uso de 10 puntos iniciales en el espacio de los hiperparámetros obtenidos de forma aleatoria. A pesar de que el cálculo se paraleliza en todos los núcleos del procesador (Intel MKL), ocupa de todas formas un tiempo considerable, que depende del número de iteraciones y el tamaño de batchsize. Para ahorrar tiempo, comenzaremos con 10 iteraciones. Posteriormente, si los resultados no son satisfactorios, podremos continuar la optimización, utilizando como valores inicales los valores óptimos del anterior ciclo de optimización.
Variante de entrenamiento SRBM + RP
#---SRBM + RP---------------- evalq( OPT_Res1 <- BayesianOptimization(fitnes1.DNN, bounds = bonds1, init_grid_dt = NULL, init_points = 10, n_iter = 10, acq = "ucb", kappa = 2.576, eps = 0.0, verbose = TRUE) , envir = env) Best Parameters Found: Round = 7 n1 = 22.0000 n2 = 2.0000 fact1 = 3.0000 fact2 = 2.0000 dr1 = 0.4114 dr2 = 0.4818 Lr.rbm = 0.7889 Value = 0.7531
Vamos a ver qué variantes de los parámetros óptimos y F1 hemos obtenido. La función BayesianOptimization() retorna una serie de valores: los mejores valores de los parámetros son Best_Par, el mejor valor del criterio de optimización con estos parámetros óptimos es Best_Value, la historia de optimización es History, y las predicciones obtenidas después de todas las iteraciones son Pred. Vamos a ver la historia de optimización, ordenándola preliminarmente de forma descendente conforme al valor Value.
evalq({
OPT_Res1 %$% History %>% dplyr::arrange(desc(Value)) %>% head(10) %>%
dplyr::select(-Round) -> best.init1
best.init1
}, env)
n1 n2 fact1 fact2 dr1 dr2 Lr.rbm Value
1 22 2 3 2 0.41136623 0.48175897 0.78886312 0.7531204
2 23 8 4 2 0.16814464 0.16221565 0.08381839 0.7485614
3 19 17 3 3 0.17274258 0.46809117 0.72698789 0.7485614
4 25 25 4 2 0.30039573 0.26894463 0.11226139 0.7473266
5 11 24 3 2 0.31564303 0.11091751 0.40387209 0.7462520
6 1 6 3 4 0.36876218 0.17403265 0.90387675 0.7450260
7 25 25 3 1 0.06872059 0.42459582 0.40072731 0.7447972
8 1 25 4 1 0.24871843 0.18593687 0.31920691 0.7445628
9 18 1 4 3 0.49846810 0.38517469 0.51115471 0.7423566
10 13 25 4 1 0.37052548 0.07603925 0.87100360 0.7402597
Se trata de un buen resultado. Haremos otra pasada de la optimización, pero para inicializar los primeros 10 puntos en el espacio de los hiperparámetros utilizaremos los mejores valores de la pasada anterior best_init1.
evalq( OPT_Res1.1 <- BayesianOptimization(fitnes1.DNN, bounds = bonds1, init_grid_dt = best.init1, init_points = 10, n_iter = 10, acq = "ucb", kappa = 2.576, eps = 0.0, verbose = TRUE) , envir = env) Best Parameters Found: Round = 1 n1 = 4.0000 n2 = 1.0000 fact1 = 1.0000 fact2 = 4.0000 dr1 = 0.1870 dr2 = 0.0000 Lr.rbm = 0.9728 Value = 0.7608
Vamos a ver los 10 mejores resultados de esta pasada.
evalq({
OPT_Res1.1 %$% History %>% dplyr::arrange(desc(Value)) %>% head(10) %>%
dplyr::select(-Round) -> best.init1
best.init1
}, env)
n1 n2 fact1 fact2 dr1 dr2 Lr.rbm Value
1 4 1 1 4 0.18701522 2.220446e-16 0.9728164 0.7607811
2 3 24 1 4 0.12698982 1.024231e-01 0.5540933 0.7549180
3 5 5 1 3 0.07366640 2.630144e-01 0.2156837 0.7542661
4 1 18 1 4 0.41907554 4.641130e-02 0.6092082 0.7509800
5 1 23 1 4 0.25279461 1.365197e-01 0.2957633 0.7504026
6 4 25 4 1 0.09500347 3.083338e-01 0.2522729 0.7488496
7 17 3 3 3 0.36117416 3.162195e-01 0.4214501 0.7458489
8 13 4 3 3 0.22496776 1.481455e-01 0.4448280 0.7437376
9 21 24 1 3 0.36154287 1.335931e-01 0.6749752 0.7435897
10 5 11 3 3 0.29627244 3.425604e-01 0.1251956 0.7423566
Ha mejorado no solo el mejor resultado, sino también la calidad de los 10 mejores, y han aumentado los índices de Value. Podemos repetir la optimización varias veces, eligiendo diferentes puntos iniciales (por ejemplo, probar a optimizar con los 10 peores valores, etcétera).
Para esta variante de entrenamiento, aplicaremos los siguientes hiperparámetros:
> env$OPT_Res1.1$Best_Par %>% round(4) n1 n2 fact1 fact2 dr1 dr2 Lr.rbm 4.0000 1.0000 1.0000 4.0000 0.1870 0.0000 0.9728
Los desciframos. Obtenemos los siguientes parámetros óptimos:
- número de neuronas de la primera capa oculta - 2*n1 = 8
- número de neuronas de la segunda capa oculta - 2*n2 = 2
- función de activación de la primera capa oculta Fact[fact1] ="tanhdUnit"
- función de activación de la segunda capa oculta Fact[fact2] = "sigmoidUnit"
- nivel de cribado de la primera capa oculta dr1 = 0.187
- nivel de cribado de la segunda capa oculta dr2 = 0.0
- nivel de entrenamiento de SRBM con preentrenamiento Lr.rbm = 0.9729
Además de haber obtenido un buen resultado en general, hemos conseguido una estructura interesante de la DNN (10-8-2-2).
Variante de entrenamiento SRBM + BP
#---SRBM + BP---------------- evalq( OPT_Res2 <- BayesianOptimization(fitnes2.DNN, bounds = bonds2, init_grid_dt = NULL, init_points = 10, n_iter = 10, acq = "ucb", kappa = 2.576, eps = 0.0, verbose = TRUE) , envir = env)
Veamos los 10 mejores valores:
> evalq({
+ OPT_Res2 %$% History %>% dplyr::arrange(desc(Value)) %>% head(10) %>%
+ dplyr::select(-Round) -> best.init2
+ best.init2
+ }, env)
n1 n2 fact1 fact2 dr1 dr2 Lr.rbm Lr.fine Value
1 23 24 2 1 0.45133494 0.14589979 0.89897498 0.2325569 0.7612619
2 3 24 4 3 0.07673542 0.42267387 0.59938522 0.4376796 0.7551184
3 15 13 4 1 0.32812018 0.45708556 0.09472489 0.8220925 0.7516732
4 7 18 3 1 0.15980725 0.12045896 0.82638047 0.2752569 0.7473167
5 7 23 3 3 0.37716019 0.23287775 0.61652190 0.9749432 0.7440724
6 21 23 3 1 0.22184400 0.08634275 0.08049532 0.3349808 0.7440647
7 23 8 3 4 0.26182910 0.11339229 0.31787446 0.9639373 0.7429621
8 5 2 1 1 0.25633998 0.27587931 0.17733507 0.4987357 0.7429471
9 1 24 1 2 0.12937722 0.22952235 0.19549144 0.6538553 0.7426660
10 18 8 4 1 0.44986721 0.28928018 0.12523905 0.2441150 0.7384895
El resultado es bueno, no necesitaremos optimización adicional.
Los mejores hiperparámetros para esta variante:
> env$OPT_Res2$Best_Par %>% round(4) n1 n2 fact1 fact2 dr1 dr2 Lr.rbm Lr.fine 23.0000 24.0000 2.0000 1.0000 0.4513 0.1459 0.8990 0.2326
Variante de entrenamiento SRBM + upperLayer + BP
#---SRBM + upperLayer + BP---- evalq( OPT_Res3 <- BayesianOptimization(fitnes3.DNN, bounds = bonds3, init_grid_dt = NULL, init_points = 10, n_iter = 10, acq = "ucb", kappa = 2.576, eps = 0.0, verbose = TRUE) , envir = env)Best Parameters Found: Round = 20 n1 = 24.0000 n2 = 5.0000 fact1 = 1.0000 fact2 = 2.0000 dr1 = 0.4060 dr2 = 0.2790 Lr.rbm = 0.9586 Lr.top = 0.8047 Lr.fine = 0.8687 Value = 0.7697
Veamos los 10 mejores valores:
evalq({
OPT_Res3 %$% History %>% dplyr::arrange(desc(Value)) %>% head(10) %>%
dplyr::select(-Round) -> best.init3
best.init3
}, env)
n1 n2 fact1 fact2 dr1 dr2 Lr.rbm Lr.top Lr.fine Value
1 24 5 1 2 0.40597650 0.27897269 0.9585567 0.8046758 0.86871454 0.7696970
2 24 13 1 1 0.02456308 0.08652276 0.9807432 0.8033236 0.87293155 0.7603146
3 7 8 3 3 0.24115850 0.42538540 0.5970306 0.2897183 0.64518524 0.7543239
4 9 15 3 3 0.14951302 0.04013773 0.3734516 0.2499858 0.14993060 0.7521897
5 4 20 3 3 0.45660260 0.12858958 0.8280872 0.1998107 0.08997839 0.7505357
6 21 6 3 1 0.38742051 0.12644262 0.5145560 0.3599426 0.24159111 0.7403176
7 22 3 1 1 0.13356602 0.12940396 0.1188595 0.8979277 0.84890568 0.7369316
8 7 18 3 4 0.44786101 0.33788727 0.4302948 0.2660965 0.75709349 0.7357294
9 25 13 2 1 0.02456308 0.08652276 0.9908265 0.8065841 0.87293155 0.7353894
10 24 17 1 1 0.23273972 0.01572794 0.9193522 0.6654211 0.26861297 0.7346243
El resultado es bueno, no necesitaremos optimización adicional.
Los mejores hiperparámetros para esta variante de entrenamiento:
> env$OPT_Res3$Best_Par %>% round(4) n1 n2 fact1 fact2 dr1 dr2 Lr.rbm Lr.top Lr.fine 24.0000 5.0000 1.0000 2.0000 0.4060 0.2790 0.9586 0.8047 0.8687
Variante de entrenamiento SRBM + upperLayer + RP
#---SRBM + upperLayer + RP---- evalq( OPT_Res4 <- BayesianOptimization(fitnes4.DNN, bounds = bonds4, init_grid_dt = NULL, init_points = 10, n_iter = 10, acq = "ucb", kappa = 2.576, eps = 0.0, verbose = TRUE) , envir = env)Best Parameters Found: Round = 15 n1 = 23.0000 n2 = 7.0000 fact1 = 3.0000 fact2 = 1.0000 dr1 = 0.3482 dr2 = 0.4726 Lr.rbm = 0.0213 Lr.top = 0.5748 Value = 0.7625
Las 10 mejores variantes de los hiperparámetros:
evalq({
OPT_Res4 %$% History %>% dplyr::arrange(desc(Value)) %>% head(10) %>%
dplyr::select(-Round) -> best.init4
best.init4
}, env)
n1 n2 fact1 fact2 dr1 dr2 Lr.rbm Lr.top Value
1 23 7 3 1 0.34823851 0.4726219 0.02129964 0.57482890 0.7625131
2 24 13 3 1 0.38677878 0.1006743 0.72237324 0.42955366 0.7560023
3 1 1 4 3 0.17036760 0.1465872 0.40598393 0.06420964 0.7554773
4 23 7 3 1 0.34471936 0.4726219 0.02129964 0.57405944 0.7536946
5 19 16 3 3 0.25563914 0.1349885 0.83913339 0.77474220 0.7516732
6 8 12 3 1 0.23000115 0.2758919 0.54359416 0.46533472 0.7475112
7 10 8 3 1 0.23661048 0.4030048 0.15234740 0.27667214 0.7458489
8 6 19 1 2 0.18992796 0.4779443 0.98278107 0.84591090 0.7391758
9 11 10 1 2 0.47157135 0.2730922 0.86300945 0.80325083 0.7369316
10 18 21 2 1 0.05182149 0.3503253 0.55296502 0.86458533 0.7359324
El resultado es bueno, no necesitaremos optimización adicional.
Adoptamos los mejores hiperparámetros para esta variante de entrenamiento:
> env$OPT_Res4$Best_Par %>% round(4) n1 n2 fact1 fact2 dr1 dr2 Lr.rbm Lr.top 23.0000 7.0000 3.0000 1.0000 0.3482 0.4726 0.0213 0.5748
Entrenamiento y simulación de la DNN con los parámetros óptimos
Analizamos las métricas obtenidas al simular las variantes.
Variante SRBM + RP
Para simular las DNN con parámetros óptimos, escribiremos una función especial. Aquí la vamos a mostrar solo para esta variante de entrenamiento. Será análoga para el resto de las variantes.
#---SRBM + RP---------------- test1.DNN <- function(n1, n2, fact1, fact2, dr1, dr2, Lr.rbm) { Ln <- c(0, 2*n1, 2*n2, 0) #-- pretrainSRBM(Ln, fact1, fact2, dr1, dr2, Lr.rbm) -> Dnn fineTuneRP(Ln, fact1, fact2, dr1, dr2, Dnn) -> Dnn.opt predict(Dnn.opt, newdata = X$test$x %>% tail(250) , type = "class") -> Ypred yTest <- X$test$y[ ,1] %>% tail(250) #numIncorrect <- sum(Ypred != yTest) #Score <- 1 - round(numIncorrect/nrow(xTest), 2) Score <- Evaluate(actual = yTest, predicted = Ypred)$Metrics[ ,2:5] %>% round(3) return(list(Score = Score, Pred = Ypred, Dnn = Dnn, Dnn.opt = Dnn.opt)) }
Los parámetros de la función test1.DNN() son los hiperparámetros óptimos que hemos obtenido antes. A continuación, hacemos el preentrenamiento con la función pretrainSRBM(), obtenemos la DNN preentrenada, que luego suministramos a la función de ajuste preciso fineTuneRP() para obtener la Dnn.opt especial. Usando esta Dnn.opt y los últimos 250 valores del conjunto X$test, obtendremos los valores predichos de la variable meta Ypred. Usando los Ypred predichos y los valores reales de la variable meta yTest, calculamos una serie de métricas con la ayuda de la función Evaluate(). Todas las variantes posibles con la elección de métrica. Como resultado, la función nos dará una lista con los siguientes objetos: Score — métrica de prueba, Pred — valores predichos de la variable meta, Dnn — DNN preentrenada, Dnn.opt — DNN completamente entrenada.
Simulamos y miramos el resultado con los hiperparámetros obtenidos después de la optimización provisional:
evalq({
#--BestParams--------------------------
best.par <- OPT_Res1.1$Best_Par %>% unname
# n1, n2, fact1, fact2, dr1, dr2, Lr.rbm
n1 = best.par[1]; n2 = best.par[2]
fact1 = best.par[3]; fact2 = best.par[4]
dr1 = best.par[5]; dr2 = best.par[6]
Lr.rbm = best.par[7]
Ln <- c(0, 2*n1, 2*n2, 0)
#---train/test--------
Res1 <- test1.DNN(n1, n2, fact1, fact2, dr1, dr2, Lr.rbm)
}, env)
env$Res1$Score
Accuracy Precision Recall F1
-1 0.74 0.718 0.724 0.721
1 0.74 0.759 0.754 0.757
El resultado es peor que tras la primera optimización, existe una rasgo explícito de sobreentrenamiento. Simulamos con los valores iniciales de los hiperparámetros:
evalq({
#--BestParams--------------------------
best.par <- OPT_Res1$Best_Par %>% unname
# n1, n2, fact1, fact2, dr1, dr2, Lr.rbm
n1 = best.par[1]; n2 = best.par[2]
fact1 = best.par[3]; fact2 = best.par[4]
dr1 = best.par[5]; dr2 = best.par[6]
Lr.rbm = best.par[7]
Ln <- c(0, 2*n1, 2*n2, 0)
#---train/test--------
Res1 <- test1.DNN(n1, n2, fact1, fact2, dr1, dr2, Lr.rbm)
}, env)
env$Res1$Score
Accuracy Precision Recall F1
-1 0.756 0.757 0.698 0.726
1 0.756 0.755 0.806 0.780
El resultado es bueno. Vamos a ver el gráfico de la historia de entrenamiento:
plot(env$Res1$Dnn.opt, type = "class")
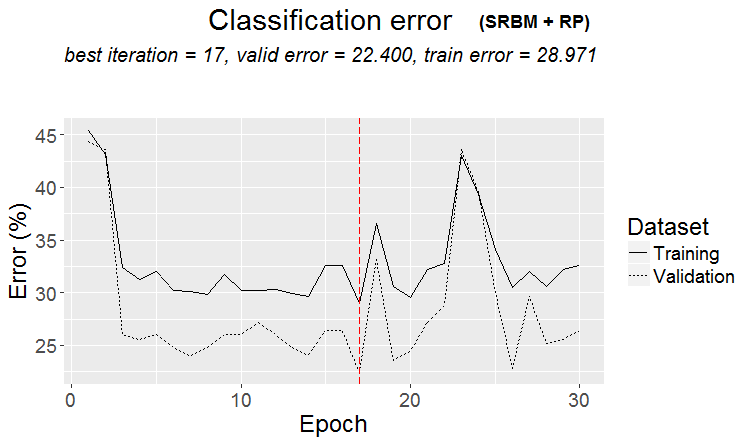
Fig.2 Historia de entrenamiento de la DNN con la variante SRBM + RP
Como podemos ver por la figura, el error en el conjunto de validación es inferior al error en el conjunto de entrenamiento. Esto indica que nuestro modelo no ha sido sobreentrenado y tiene una buena capacidad de generalización. La línea roja vertical muestra los resultados del modelo reconocido como el mejor, que hemos obtenido tras el entrenamiento.
Para las otras tres variantes de entrenamiento, mostraremos solo los resultados de los cálculos y los gráficos de la historia sin aclaraciones detalladas. Todo se calcula de forma análoga.
Variante SRBM + BP
Simulamos:
evalq({
#--BestParams--------------------------
best.par <- OPT_Res2$Best_Par %>% unname
# n1, n2, fact1, fact2, dr1, dr2, Lr.rbm, Lr.fine
n1 = best.par[1]; n2 = best.par[2]
fact1 = best.par[3]; fact2 = best.par[4]
dr1 = best.par[5]; dr2 = best.par[6]
Lr.rbm = best.par[7]; Lr.fine = best.par[8]
Ln <- c(0, 2*n1, 2*n2, 0)
#----train/test-------
Res2 <- test2.DNN(n1, n2, fact1, fact2, dr1, dr2, Lr.rbm, Lr.fine)
}, env)
env$Res2$Score
Accuracy Precision Recall F1
-1 0.768 0.815 0.647 0.721
1 0.768 0.741 0.873 0.801
Podemos decir que el resultado es excelente. Vamos a ver la historia del entrenamiento:
plot(env$Res2$Dnn.opt, type = "class")
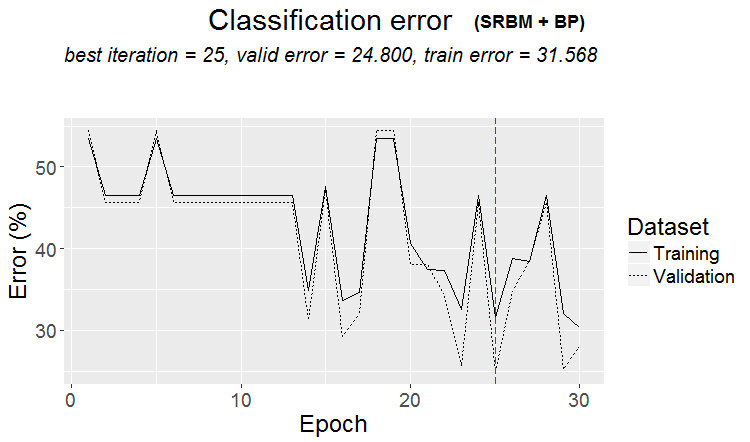
Fig.3 Historia del entrenamiento de la DNN con la variante SRBM + ВP
Variante SRBM + upperLayer + BP
Simulamos:
evalq({
#--BestParams--------------------------
best.par <- OPT_Res3$Best_Par %>% unname
# n1, n2, fact1, fact2, dr1, dr2, Lr.rbm , Lr.top, Lr.fine
n1 = best.par[1]; n2 = best.par[2]
fact1 = best.par[3]; fact2 = best.par[4]
dr1 = best.par[5]; dr2 = best.par[6]
Lr.rbm = best.par[7]
Lr.top = best.par[8]
Lr.fine = best.par[9]
Ln <- c(0, 2*n1, 2*n2, 0)
#----train/test-------
Res3 <- test3.DNN(n1, n2, fact1, fact2, dr1, dr2, Lr.rbm, Lr.top, Lr.fine)
}, env)
env$Res3$Score
Accuracy Precision Recall F1
-1 0.772 0.771 0.724 0.747
1 0.772 0.773 0.813 0.793
El resultado es magnífico. Preste atención: el valor medio F1 usado como criterio de optimización da una calidad idéntica para ambas clases, a pesar del desequilibrio existente entre ellas.
Gráfico de la historia del entrenamiento:
plot(env$Res3$Dnn.opt, type = "class")
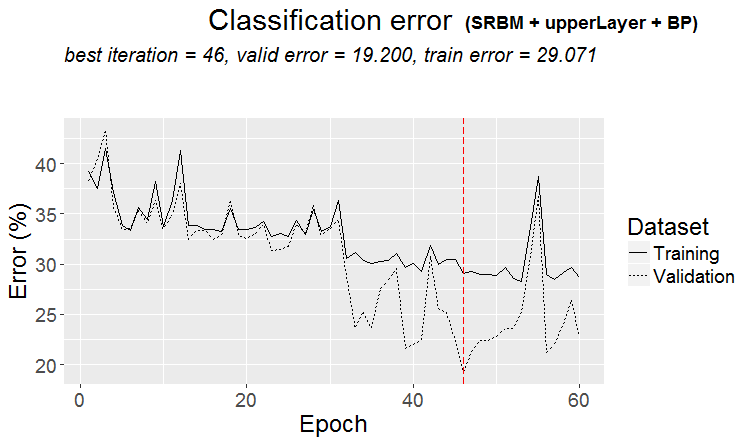
Fig. 4. Historia del entrenamiento de la DNN con la variante SRBM + upperLayer + BP
Variante SRBM + upperLayer + RP
Simulamos:
evalq({
#--BestParams--------------------------
best.par <- OPT_Res4$Best_Par %>% unname
# n1, n2, fact1, fact2, dr1, dr2, Lr.rbm, Lr.top
n1 = best.par[1]; n2 = best.par[2]
fact1 = best.par[3]; fact2 = best.par[4]
dr1 = best.par[5]; dr2 = best.par[6]
Lr.rbm = best.par[7]
Lr.top = best.par[8]
Ln <- c(0, 2*n1, 2*n2, 0)
#----train/test-------
Res4 <- test4.DNN(n1, n2, fact1, fact2, dr1, dr2, Lr.rbm, Lr.top)
}, env)
env$Res4$Score
Accuracy Precision Recall F1
-1 0.768 0.802 0.664 0.726
1 0.768 0.747 0.858 0.799
Un resultado muy bueno. Vamos a analizar el gráfico de la historia del entrenamiento:
plot(env$Res4$Dnn.opt, type = "class")
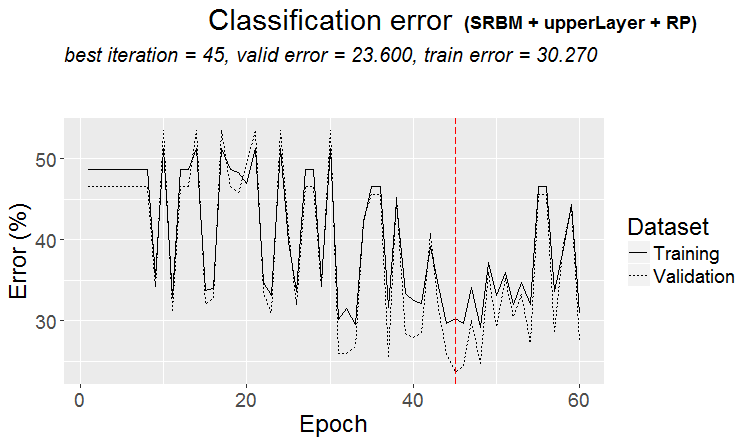
Fig. 5. Historia del entrenamiento de la DNN con la variante SRBM + upperLayer + RP
Análisis de los resultados de la simulación de la DNN con los parámetros óptimos
Los resultados del entrenamiento y la simulación de los modelos de la DNN, entrenados con los valores optimizados de los hiperparámetros, da buenos resultados con una precisión del 75 (+/-5)%. El error de clasificación se encuentra estable en el 25%, no depende del método de entrenamiento e indica que la estructura de datos no coincide con la estructura de la variable meta en una cuarta parte de los casos. Hemos observado el mismo resultado cuando investigamos la presencia de ejemplos de ruido en el conjunto de datos original. Su cantidad también era del 25% y no dependía de los métodos de transformación de los predictores. Eso es normal. Pregunta: ¿cómo mejorar el pronóstico sin reentrenar el modelo? Tenemos varias posibilidades:
- usar un conjunto de neuroredes de los mejores modelos, entrenados según las 4 variantes;
- usar un conjunto de neuroredes de los 10 mejores modelos, obtenidos al optimizar según cada variante de entrenamiento;
- reetiquetar los ejemplos de ruido en el conjunto de entrenamiento en una clase adicional "0" y entrenar el modelo de DNN según esta variable meta(con las tres clases con("-1", "0", "1"));
- reetiquetar los ejemplos clasificados de forma errónea con la clase adicional "0" y entrenar el modelo de DNN según esta variable meta(con las tres clases con("-0", "1", "1"));
Analizaremos con detalle la creación, el entrenamiento y el uso de conjuntos en el siguiente artículo de esta serie.
El experimento con el reetiquetado (relabel) de los ejemplos de ruido merece una investigación aparte y se sale del marco de este artículo. La idea es sencilla. Con la ayuda de la función ORBoostFilter::NoiseFiltersR, que ya hemos analizado en la parte anterior, definimos los parámetros de ruido en los conjuntos de validación y prueba simultáneamente. En la variable meta, cambiamos los valores de las clases ("-1"/"1") que se correspondan con estos ejemplos, por la clase "0". Es decir, en nuestra variable meta habrá tres clases. Así que tratamos de enseñar al modelo, no a clasificar los ejemplos de ruido, que, por regla general, dan error de clasificación. En este caso partiremos de la premisa de que la oportunidad perdida de conseguir beneficios no equivale a una pérdida.
Forward test de los modelos con los parámetros óptimos
Vamos a comprobar cuánto tiempo nos darán los parámetros óptimos de DNN resultados aceptables en cuanto a la calidad en los tests de los valores "futuros" de las cotizaciones. Haremos la prueba en el entorno que quede después de las anteriores optimizaciones y la simulación de la forma siguiente.
Usando una ventana móvil con un tamaño de 1350 barras, train = 1000, test = 350 (para la validación: los 250 primeros ejemplos, para la simulación: los últimos 100 ejemplos) y un salto de 100, pasaremos por los datos después de las primeras (4000 + 100) barras usadas para el preentrenamiento. Hacemos 10 saltos "hacia adelante". En cada salto entrenaremos y simularemos dos modelos:
- uno con el uso de una DNN preentrenada, es decir, realizaremos el ajuste preciso en un nuevo rango en cada salto;
- el segundo, terminando de entrenar la DNN.opt obtenida tras la optimización en la etapa de ajuste preciso en un nuevo rango temporal.
#---prepare----
evalq({
step <- 1:10
dt <- PrepareData(Data, Open, High, Low, Close, Volume)
DTforv <- foreach(i = step, .packages = "dplyr" ) %do% {
SplitData(dt, 4000, 1000, 350, 10, start = i*100) %>%
CappingData(., impute = T, fill = T, dither = F, pre.outl = pre.outl)%>%
NormData(., preproc = preproc) -> DTn
foreach(i = 1:4) %do% {
DTn[[i]] %>% dplyr::select(-c(v.rstl, v.pcci))
} -> DTn
list(pretrain = DTn[[1]],
train = DTn[[2]],
val = DTn[[3]],
test = DTn[[4]]) -> DTn
list(
pretrain = list(
x = DTn$pretrain %>% dplyr::select(-c(Data, Class)) %>% as.data.frame(),
y = DTn$pretrain$Class %>% as.data.frame()
),
train = list(
x = DTn$train %>% dplyr::select(-c(Data, Class)) %>% as.data.frame(),
y = DTn$train$Class %>% as.data.frame()
),
test = list(
x = DTn$val %>% dplyr::select(-c(Data, Class)) %>% as.data.frame(),
y = DTn$val$Class %>% as.data.frame()
),
test1 = list(
x = DTn$test %>% dplyr::select(-c(Data, Class)) %>% as.data.frame(),
y = DTn$test$Class %>% as.vector()
)
)
}
}, env)
Realizamos la primera parte del forward test usando la DNN preentrenada y los hiperparámetros óptimos obtenidos según la variable de entrenamiento SRBM + upperLayer + BP.
#----#---SRBM + upperLayer + BP---- evalq({ #--BestParams-------------------------- best.par <- OPT_Res3$Best_Par %>% unname # n1, n2, fact1, fact2, dr1, dr2, Lr.rbm , Lr.top, Lr.fine n1 = best.par[1]; n2 = best.par[2] fact1 = best.par[3]; fact2 = best.par[4] dr1 = best.par[5]; dr2 = best.par[6] Lr.rbm = best.par[7] Lr.top = best.par[8] Lr.fine = best.par[9] Ln <- c(0, 2*n1, 2*n2, 0) foreach(i = step, .packages = "darch" ) %do% { DTforv[[i]] -> X if(i==1) Res3$Dnn -> Dnn #----train/test------- fineTuneBP(Ln, fact1, fact2, dr1, dr2, Dnn, Lr.fine) -> Dnn.opt predict(Dnn.opt, newdata = X$test$x %>% tail(100) , type = "class") -> Ypred yTest <- X$test$y[ ,1] %>% tail(100) #numIncorrect <- sum(Ypred != yTest) #Score <- 1 - round(numIncorrect/nrow(xTest), 2) Evaluate(actual = yTest, predicted = Ypred)$Metrics[ ,2:5] %>% round(3) } -> Score3_dnn }, env)
Segunda etapa del forward test usando el Dnn.opt, obtenido en la optimización:
evalq({
foreach(i = step, .packages = "darch" ) %do% {
DTforv[[i]] -> X
if(i==1) {Res3$Dnn.opt -> Dnn}
#----train/test-------
fineTuneBP(Ln, fact1, fact2, dr1, dr2, Dnn, Lr.fine) -> Dnn.opt
predict(Dnn.opt, newdata = X$test$x %>% tail(100) , type = "class") -> Ypred
yTest <- X$test$y[ ,1] %>% tail(100)
#numIncorrect <- sum(Ypred != yTest)
#Score <- 1 - round(numIncorrect/nrow(xTest), 2)
Evaluate(actual = yTest, predicted = Ypred)$Metrics[ ,2:5] %>%
round(3)
} -> Score3_dnnOpt
}, env)
Comparamos los resultados de la simulación y los reunimos en un recuadro:
env$Score3_dnn env$Score3_dnnOpt
| iter | Score3_dnn | Score3_dnnOpt |
|---|---|---|
| Accuracy Precision Recall F1 | Accuracy Precision Recall F1 | |
| 1 | -1 0.76 0.737 0.667 0.7 1 0.76 0.774 0.828 0.8 |
-1 0.77 0.732 0.714 0.723 1 0.77 0.797 0.810 0.803 |
| 2 | -1 0.79 0.88 0.746 0.807 1 0.79 0.70 0.854 0.769 |
-1 0.78 0.836 0.78 0.807 1 0.78 0.711 0.78 0.744 |
| 3 | -1 0.69 0.807 0.697 0.748 1 0.69 0.535 0.676 0.597 |
-1 0.67 0.824 0.636 0.718 1 0.67 0.510 0.735 0.602 |
| 4 | -1 0.71 0.738 0.633 0.681 1 0.71 0.690 0.784 0.734 |
-1 0.68 0.681 0.653 0.667 1 0.68 0.679 0.706 0.692 |
| 5 | -1 0.56 0.595 0.481 0.532 1 0.56 0.534 0.646 0.585 |
-1 0.55 0.578 0.500 0.536 1 0.55 0.527 0.604 0.563 |
| 6 | -1 0.61 0.515 0.829 0.636 1 0.61 0.794 0.458 0.581 |
-1 0.66 0.564 0.756 0.646 1 0.66 0.778 0.593 0.673 |
| 7 | -1 0.67 0.55 0.595 0.571 1 0.67 0.75 0.714 0.732 |
-1 0.73 0.679 0.514 0.585 1 0.73 0.750 0.857 0.800 |
| 8 | -1 0.65 0.889 0.623 0.733 1 0.65 0.370 0.739 0.493 |
-1 0.68 0.869 0.688 0.768 1 0.68 0.385 0.652 0.484 |
| 9 | -1 0.55 0.818 0.562 0.667 1 0.55 0.222 0.500 0.308 |
-1 0.54 0.815 0.55 0.657 1 0.54 0.217 0.50 0.303 |
| 10 | -1 0.71 0.786 0.797 0.791 1 0.71 0.533 0.516 0.525 |
-1 0.71 0.786 0.797 0.791 1 0.71 0.533 0.516 0.525 |
Podemos ver en el recuadro que los dos primeros saltos dan buenos resultados. La calidad en los 2 primeros saltos de las dos variantes es prácticamente igual, y luego cae. Por lo tanto, podemos decir que después de la optimización y la simulación, la DNN garantizará la calidad de la clasificación en el nivel del conjunto de prueba durante al menos las próximas 200-250 barras.
Existen muchas otras combinaciones en cuanto al entrenamiento del modelo con forward tests, sobre las que hablamos en el anterior artículo, y también multitud de hiperparámetros que tenemos que regular.
Conclusión
- El paquete darch v.0.12 proporciona acceso a una enorme lista de hiperparámetros de la DNN, ofreciendo grandes posibilidades de ajuste preciso y profundo.
- El uso del enfoque bayesiano para la optimización de los hiperparámetros de la DNN ofrece un amplio surtido de modelos con buena calidad, que se pueden usar para crear nuestros propios conjuntos.
- La optimización de los hiperparámetros de la DNN con el método bayesiano aumenta la calidad de clasificación en un 7-10%.
- Para obtener el mejor resultado, es necesario realizar varias optimizaciones (10 — 20) eligiendo sucesivamente el mejor resultado.
- El proceso de optimización se puede continuar por etapas, presentando como valores iniciales los parámetros obtenidos en las pasadas preliminares.
- El uso de las DNN obtenidas al optimizar los hiperparámetros posibilita el aumento de la calidad de clasificación en el forward test a nivel de prueba en un segmento con una longitud igual al conjunto de prueba.
Para la posterior mejora tiene sentido incluir en la lista de parámetros a optimizar la función de entrenamiento rpropagation en sus 4 variantes, la normalización de pesos de las neuronas en las capas ocultas normalizeWeights(TRUE, FALSE) y el límite superior de esta normalización normalizeWeightsBound. Podemos experimentar también con otros parámetros, que, a nuestro juicio, pueden influir en la calidad de la clasificación del modelo. Una de las principales ventajas del paquete darch es precisamente que ofrece acceso a todos los parámetros de la neurored. Podemos determinar de forma experimental cómo influye cada parámetro en la calidad de la clasificación.
A pesar del considerable gasto de tiempo, el uso de la optimziación bayesiana es muy oportuno.
Otra posibilidad de mejorar la calidad de la clasificación es el uso de un conjunto de neuroredes. Analizaremos esta opción para el refuerzo de las diferentes variantes en la siguiente parte del artículo.
Anexos
En GitHub/PartV encontrará:
1 FUN_Optim.R — funciones necesarias para realizar todos los cálculos descritos en este artículo.
2. RUN_Optim.R — cálculos realizados en este artículo.
3. SessionInfo. txt — paquetes usados en los cálculos.
Traducción del ruso hecha por MetaQuotes Ltd.
Artículo original: https://www.mql5.com/ru/articles/4225
Advertencia: todos los derechos de estos materiales pertenecen a MetaQuotes Ltd. Queda totalmente prohibido el copiado total o parcial.
Este artículo ha sido escrito por un usuario del sitio web y refleja su punto de vista personal. MetaQuotes Ltd. no se responsabiliza de la exactitud de la información ofrecida, ni de las posibles consecuencias del uso de las soluciones, estrategias o recomendaciones descritas.
 ZUP - zigzag universal con patrones Pesavento. Búsqueda de patrones
ZUP - zigzag universal con patrones Pesavento. Búsqueda de patrones
 LifeHack para tráders: cocinamos ForEach usando #define
LifeHack para tráders: cocinamos ForEach usando #define
 Visualizando la optimización de una estrategia comercial en MetaTrader 5
Visualizando la optimización de una estrategia comercial en MetaTrader 5
 Construcción automática de las líneas de apoyo y resistencia
Construcción automática de las líneas de apoyo y resistencia
- Aplicaciones de trading gratuitas
- 8 000+ señales para copiar
- Noticias económicas para analizar los mercados financieros
Usted acepta la política del sitio web y las condiciones de uso
Otro retrasado aquí
https://github.com/yanyachen/rBayesianOptimization/blob/master/R/Utility_Max.R
También puse maxit = 1 en lugar de 100.
A través de ... no se puede pasar, puede simplemente cargar su función Utility_Max en R y utilizar la versión corregida.
Lo he comprobado en la optimización del conjunto de redes neuronales del artículo PartVI. Ni maxit ni control tiene un efecto visible en el tiempo de computación. La mayor influencia es el número de neuronas en la capa oculta. Lo dejé así
OPT_Res <- BayesianOptimization(fitnes, bounds = bonds, init_grid_dt = NULL, init_points = 20, n_iter = 20, acq = "ucb", kappa = 2.576, eps = 0.0, verbose = TRUE, maxit = 100, control = c(100, 50, 8)) elapsed = 14.42 Round = 1 numFeature = 9.0000 r = 7.0000 nh = 36.0000 fact = 9.0000 Value = 0.7530 elapsed = 42.94 Round = 2 numFeature = 4.0000 r = 8.0000 nh = 46.0000 fact = 6.0000 Value = 0.7450 elapsed = 9.50 Round = 3 numFeature = 11.0000 r = 5.0000 nh = 19.0000 fact = 5.0000 Value = 0.7580 elapsed = 14.17 Round = 4 numFeature = 10.0000 r = 4.0000 nh = 35.0000 fact = 4.0000 Value = 0.7480 elapsed = 12.36 Round = 5 numFeature = 8.0000 r = 4.0000 nh = 23.0000 fact = 6.0000 Value = 0.7450 elapsed = 25.61 Round = 6 numFeature = 12.0000 r = 8.0000 nh = 44.0000 fact = 7.0000 Value = 0.7490 elapsed = 8.03 Round = 7 numFeature = 12.0000 r = 9.0000 nh = 9.0000 fact = 2.0000 Value = 0.7470 elapsed = 14.24 Round = 8 numFeature = 8.0000 r = 4.0000 nh = 45.0000 fact = 2.0000 Value = 0.7620 elapsed = 9.05 Round = 9 numFeature = 7.0000 r = 8.0000 nh = 20.0000 fact = 10.0000 Value = 0.7390 elapsed = 17.53 Round = 10 numFeature = 12.0000 r = 9.0000 nh = 20.0000 fact = 6.0000 Value = 0.7410 elapsed = 4.77 Round = 11 numFeature = 9.0000 r = 2.0000 nh = 7.0000 fact = 2.0000 Value = 0.7570 elapsed = 8.87 Round = 12 numFeature = 6.0000 r = 1.0000 nh = 40.0000 fact = 8.0000 Value = 0.7730 elapsed = 14.16 Round = 13 numFeature = 8.0000 r = 6.0000 nh = 41.0000 fact = 10.0000 Value = 0.7390 elapsed = 21.61 Round = 14 numFeature = 9.0000 r = 6.0000 nh = 47.0000 fact = 7.0000 Value = 0.7620 elapsed = 5.14 Round = 15 numFeature = 13.0000 r = 3.0000 nh = 3.0000 fact = 5.0000 Value = 0.7260 elapsed = 5.66 Round = 16 numFeature = 6.0000 r = 9.0000 nh = 1.0000 fact = 9.0000 Value = 0.7090 elapsed = 7.26 Round = 17 numFeature = 9.0000 r = 2.0000 nh = 25.0000 fact = 1.0000 Value = 0.7550 elapsed = 32.09 Round = 18 numFeature = 11.0000 r = 7.0000 nh = 38.0000 fact = 6.0000 Value = 0.7600 elapsed = 17.18 Round = 19 numFeature = 5.0000 r = 3.0000 nh = 46.0000 fact = 6.0000 Value = 0.7500 elapsed = 11.08 Round = 20 numFeature = 6.0000 r = 4.0000 nh = 20.0000 fact = 6.0000 Value = 0.7590 elapsed = 4.47 Round = 21 numFeature = 6.0000 r = 2.0000 nh = 4.0000 fact = 2.0000 Value = 0.7390 elapsed = 5.27 Round = 22 numFeature = 6.0000 r = 2.0000 nh = 21.0000 fact = 10.0000 Value = 0.7520 elapsed = 7.96 Round = 23 numFeature = 7.0000 r = 1.0000 nh = 41.0000 fact = 7.0000 Value = 0.7730 elapsed = 12.31 Round = 24 numFeature = 7.0000 r = 3.0000 nh = 41.0000 fact = 3.0000 Value = 0.7730 elapsed = 7.64 Round = 25 numFeature = 8.0000 r = 4.0000 nh = 16.0000 fact = 7.0000 Value = 0.7420 elapsed = 6.24 Round = 26 numFeature = 13.0000 r = 5.0000 nh = 6.0000 fact = 1.0000 Value = 0.7600 elapsed = 8.41 Round = 27 numFeature = 11.0000 r = 8.0000 nh = 8.0000 fact = 7.0000 Value = 0.7420 elapsed = 8.48 Round = 28 numFeature = 6.0000 r = 7.0000 nh = 15.0000 fact = 2.0000 Value = 0.7580 elapsed = 10.11 Round = 29 numFeature = 12.0000 r = 6.0000 nh = 17.0000 fact = 4.0000 Value = 0.7310 elapsed = 6.03 Round = 30 numFeature = 8.0000 r = 3.0000 nh = 12.0000 fact = 1.0000 Value = 0.7540 elapsed = 8.58 Round = 31 numFeature = 13.0000 r = 5.0000 nh = 18.0000 fact = 2.0000 Value = 0.7300 elapsed = 6.78 Round = 32 numFeature = 13.0000 r = 2.0000 nh = 15.0000 fact = 8.0000 Value = 0.7320 elapsed = 9.54 Round = 33 numFeature = 10.0000 r = 3.0000 nh = 37.0000 fact = 9.0000 Value = 0.7420 elapsed = 8.19 Round = 34 numFeature = 6.0000 r = 1.0000 nh = 42.0000 fact = 3.0000 Value = 0.7630 elapsed = 12.34 Round = 35 numFeature = 7.0000 r = 2.0000 nh = 43.0000 fact = 8.0000 Value = 0.7570 elapsed = 20.47 Round = 36 numFeature = 7.0000 r = 8.0000 nh = 39.0000 fact = 2.0000 Value = 0.7670 elapsed = 11.51 Round = 37 numFeature = 5.0000 r = 9.0000 nh = 18.0000 fact = 3.0000 Value = 0.7540 elapsed = 32.71 Round = 38 numFeature = 7.0000 r = 7.0000 nh = 40.0000 fact = 6.0000 Value = 0.7540 elapsed = 28.33 Round = 39 numFeature = 7.0000 r = 9.0000 nh = 38.0000 fact = 5.0000 Value = 0.7550 elapsed = 22.87 Round = 40 numFeature = 12.0000 r = 6.0000 nh = 48.0000 fact = 3.0000 Value = 0.7580 Best Parameters Found: Round = 12 numFeature = 6.0000 r = 1.0000 nh = 40.0000 fact = 8.0000 Value = 0.7730 maxit = 100, control = c(100, 50, 8))Mejor 10
Valor - F1 promedio. No es un mal rendimiento.
Para acelerar los cálculos, tenemos que reescribir algunas funciones del paquete. Lo primero es sustituir todas las funciones ncol(), nrow(), que son muchas, por dim()[1], dim()[2]. Se ejecutan decenas de veces más rápido. Y probablemente, ya que sólo hay operaciones matriciales, usar la GPU (paquete gpuR). No voy a ser capaz de hacerlo yo mismo, ¿puedo sugerir al desarrollador?
Suerte
Lo he comprobado en la optimización del conjunto de redes neuronales del documento PartVI. Ni maxit ni control tienen un tiempo de ejecución visible. La mayor influencia es el número de neuronas en la capa oculta. Lo dejé así
Mejor 10
Valor - F1 medio. No es una mala actuación.
Para acelerar los cálculos, tenemos que reescribir algunas funciones del paquete. Lo primero es sustituir todas las funciones ncol(), nrow() por dim()[1], dim()[2]. Se ejecutan decenas de veces más rápido. Y probablemente, ya que sólo hay operaciones matriciales, usar la GPU (paquete gpuR). No podré hacerlo yo mismo, ¿puedo sugerírselo al desarrollador?
Suerte
Sólo optimizar algunos parámetros, he optimizado 20 piezas, y cuando los puntos conocidos se convierten en 20-40 piezas, a continuación, el cálculo de sólo GPfit tomó decenas de minutos, en tales condiciones, verá la aceleración.
Y el número de neuronas afecta sólo el tiempo de cálculo de la propia NS.
Sólo optimizar algunos parámetros, he optimizado 20 piezas, y cuando los puntos conocidos se convierten en 20-40 piezas, a continuación, el cálculo de sólo GPfit tomó decenas de minutos, en tales condiciones se verá la aceleración.
Y el número de neuronas afecta sólo el tiempo de cálculo de la propia NS.
Supongo que sí.
Nuevo artículo Deep Neural Networks (Part V). Bayesian optimization of DNN hyperparameters ha sido publicado:
Autor: Vladimir Perervenko