
Глубокие нейросети (Часть VII). Ансамбль нейросетей: stacking
Содержание
- Введение
- 1. Подготовка входных данных для обучаемого объединителя
- 2. Базовые модели сравнения
- 3. Библиотеки keras/tensorflow. Общее описание и установка
- 4. Объединитель выходов bagging ансамбля — нейросеть
- 5. Анализ результатов экспериментов
- Заключение
- Приложение
Введение
Модели базового уровня ансамбля (индивидуальные классификаторы) обучаются на полном наборе. Затем метамодель обучается на выходах ансамбля, полученных при предикте тестового набора. Выходы базовых классификаторов ансамбля при этом становятся входными данными для нового обучаемого классификатора, который сам представляет собой объединитель. Такой подход называется «сложным обобщением» или "обобщением через обучение", чаще просто stacking.
Одна из основных проблем этого объединителя — построение учебного набора для метаклассификатора.
Поэкспериментируем с построением и тестированием stacking-ансамблей. В них будем использовать ансамбль нейросетевых классификаторов ELM с оптимальными гиперпараметрами, полученными ранее. В первом эксперименте используем выходы обрезанного ансамбля а во втором — все выходы ансамбля. Объединителями в обоих вариантах станут полносвязные нейросети, но с различной структурой. В дальнейших экспериментах проверим, как влияют на качество классификации нейросети мультимодальность и многозадачность.
Чтобы оценить качество предсказания в этих вариантах, используем базовые модели сравнения.
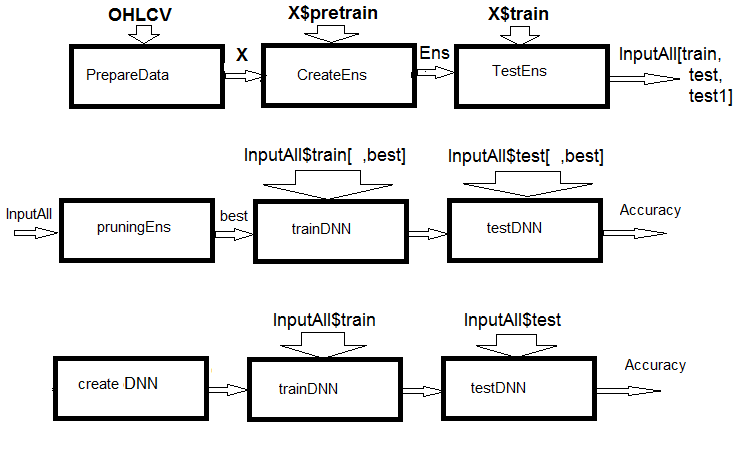
Рис.1. Структурная схема расчетов
Как видно из рисунка, эксперимент состоит из трех частей.
- Готовим входные данные для ансамбля, обучаем ансамбль ELM и получаем предсказания на train/test/test1 наборах. Эти наборы и будут входами InputAll для обучаемых объединителей.
- Обрежем ансамбль: выберем лучшие предсказания ELM по информационной важности. Протестируем базовые модели сравнения для получения метрик-ориентиров. Обучим и протестируем DNN на этих данных, вычислим метрики модели и сравним с метриками базовой модели.
- Создадим мультимодальную и многозадачную нейросети, обучим их и протестируем на наборах InputAll. Вычислим метрики полученных моделей и сравним с метриками базовой модели.
1. Готовим входные данные для обучаемого объединителя
Для проведения экспериментов будем использовать версию R 3.4.4. В ней есть несколько новых пакетов, которые мы до сих пор не использовали.
Запускаем RStudio. Загружаем из GitHub/Part_I файл Cotir.RData с котировками из терминала и файлы Importar.R, Libary.R, FunPrepareData_VII.R, FUN_Stacking.R с функциями подготовки данных из GitHub/Part_VII. Обратите внимание: последовательность загрузки файлов важна! Я немного доработал функции, чтобы ускорить расчеты и улучшить читабельность скриптов. Добавлен также ряд предикторов, необходимых для экспериментов.
Будем использовать те же котировки и разбивать их на такие же выборки, как и в предыдущих статьях этой серии. Напишем скрипт подготовки исходных данных. Не буду снова останавливаться на подробностях расчета — они описаны ранее. Сделанные изменения связаны с применением пакета dplyr и импортом пакетов и функций в рабочее окружение. Очень полезный пакет dplyr, упрощающий манипулирование данными, при отладке иногда преподносит сюрпризы, отнимающие много часов поиска ошибки.
Поясню, о чем идет речь. При загрузке библиотеки dplyr в консоли мы получаем такие предупреждения:
> library(dplyr) Attaching package: ‘dplyr’ The following objects are masked from ‘package:stats’: filter, lag The following objects are masked from ‘package:base’: intersect, setdiff, setequal, union
Налицо конфликт имен функций. Чтобы снять проблему, приходилось явно указывать, из какого пакета использовать конкретную функцию. Например, dplyr::filter(), dplyr::lag. Вторая проблема: часто нам нужна всего одна-две функции из пакета, но загрузить его нужно целиком. А ведь некоторые пакеты (например, caret) очень тяжелые и тянут за собой зависимые пакеты, которые нам не нужны. В этом смысле стиль импорта функций и пакетов в Python логичнее. Например:
from theano import function, config, shared, tensor import numpy as np import time
В первой строке импортируется ряд функций из пакета theano, во второй строке — пакет numpy под псевдонимом np и в третьей — пакет time. Для получения такой же возможности в R есть пакет importar, который имеет всего две функции import() и import_fun(). Первая дает возможность импортировать пакеты, вторая — функции. Правда, первую пришлось переименовать в import_pack(), чтобы она не пересекалась с функцией reticulate::import().
Кроме того, добавлены новые переменные, которые понадобятся для проведения экспериментов. Сформируем два набора данных — data1 и data2. Упорядочим в них предикторы по информационной важности.
Ниже приведен скрипт подготовки исходных данных для экспериментов. Он находится в файле Prepare.R.
#--0--Library------------- # source(file = "importar.R") # source(file = "Library.R") # source(file = "FunPrepareData_VII.R") # source(file = "FUN_Stacking.R") #--1-prepare---- evalq({ # объединяем котировки OHLCV, Med, Typ, W в датафрейм # вычисляем предикторы и целевую dt <- PrepareData(Data, Open, High, Low, Close, Volume) # разделяем исходный набор на pretrain/train/val/test DT <- SplitData(dt$feature, 4000, 1000, 500, 250, start = 1) # определяем параметры выбросов pre.outl <- PreOutlier(DT$pretrain) # импутируем выбросы во всех наборах DTcap <- CappingData(DT, impute = T, fill = T, dither = F, pre.outl = pre.outl) # устанавливаем метод нормирования предикторов meth <- "spatialSign" #"expoTrans" "range" "spatialSign", # определяем параметры нормирования preproc <- PreNorm(DTcap$pretrain, meth = meth, rang = c(-0.95, 0.95)) # нормируем предикторы во всех наборах DTcap.n <- NormData(DTcap, preproc = preproc) }, env)
В блоке 0 (Library) загружаем необходимые библиотеки и функции. Четыре файла со скриптами нужно загружать в указанной последовательности. В блоке 1 (prepare) создаем предикторы и нормализуем их, убрав выбросы. Метод нормализации можно изменять.
Теперь сформируем два набора данных — data1 и data2. В первом наборе в качестве предикторов будут цифровые фильтры и их первые разности, а в качестве целевой — знак изменения первой разности ЗигЗага. Во втором наборе предикторами будут первые разности котировок High/Low/Close и разности котировок CO/HO/LO/HL, а целевой — первая разность Зигзага. Скрипт приведен ниже и находится в файле Prepare.R.
#--2-Data X------------- evalq({ foreach(i = 1:length(DTcap)) %do% { DTcap.n[[i]] ->.; dp$select(., Data, ftlm, stlm, rbci, pcci, fars, v.fatl, v.satl, v.rftl, v.rstl,v.ftlm, v.stlm, v.rbci, v.pcci, Class)} -> data1 X1 <- vector(mode = "list", 4) foreach(i = 1:length(X1)) %do% { data1[[i]] %>% dp$select(-c(Data, Class)) %>% as.data.frame() -> x data1[[i]]$Class %>% as.numeric() %>% subtract(1) -> y list(x = x, y = y)} -> X1 list(pretrain = X1[[1]] , train = X1[[2]] , test = X1[[3]] , test1 = X1[[4]] ) -> X1 }, env) #----------------- evalq({ foreach(i = 1:length(DTcap.n)) %do% { DTcap.n[[i]] ->.; dp$select(., Data, CO, HO, LO, HL, dC, dH, dL)} -> data2 X2 <- vector(mode = "list", 4) foreach(i = 1:length(X2)) %do% { data2[[i]] %>% dp$select(-Data) %>% as.data.frame() -> x DT[[i]]$dz -> y list(x = x, y = y)} -> X2 list(pretrain = X2[[1]] , train = X2[[2]] , test = X2[[3]] , test1 = X2[[4]] ) -> X2 }, env)
Упорядочим предикторы в обоих наборах по уменьшению их информационной важности. Посмотрим, как они ранжированы. Скрипт приведен ниже, находится в файле Prepare.R.
#---3--bestF----------------------------------- #require(clusterSim) evalq({ orderF(x = X1$pretrain$x %>% as.matrix(), type = "metric", s = 1, 4, distance = NULL, # "d1" - Manhattan, "d2" - Euclidean, #"d3" - Chebychev (max), "d4" - squared Euclidean, #"d5" - GDM1, "d6" - Canberra, "d7" - Bray-Curtis method = "kmeans" ,#"kmeans" (default) , "single", #"ward.D", "ward.D2", "complete", "average", "mcquitty", #"median", "centroid", "pam" Index = "cRAND") %$% stopri[ ,1] -> orderX1 }, env) colnames(env$X1$pretrain$x)[env$orderX1] [1] "v.fatl" "v.rbci" "v.ftlm" "fars" "v.satl" "stlm" [7] "rbci" "ftlm" "v.stlm" "v.rftl" "pcci" "v.rstl" [13] "v.pcci evalq({ orderF(x = X2$pretrain$x %>% as.matrix(), type = "metric", s = 1, 4, distance = NULL, # "d1" - Manhattan, "d2" - Euclidean, #"d3" - Chebychev (max), "d4" - squared Euclidean, #"d5" - GDM1, "d6" - Canberra, "d7" - Bray-Curtis method = "kmeans" ,#"kmeans" (default) , "single", #"ward.D", "ward.D2", "complete", "average", "mcquitty", #"median", "centroid", "pam" Index = "cRAND") %$% stopri[ ,1] -> orderX2 }, env) colnames(env$X2$pretrain$x)[env$orderX2] [1] "dC" "CO" "HO" "LO" "dH" "dL" "HL"
Интересен порядок котировочных предикторов.
Чтобы подготовить входные данные для объединителей, нам нужно:
- создать ансамбль ELM и обучим его на обучающем наборе X1$pretrain;
- сделать предикт набора X1$train с помощью обученного ансамбля. Это будет обучающий набор InputTrain;
- сделать предикт набора X1$test с помощью обученного ансамбля. Это будет тестовый набор InputTest;
- сделать предикт набора X1$test1 с помощью обученного ансамбля. Это будет тестовый набор InputTest1.
Определим переменные и константы, напишем функции createEns() и GetInputData() — она будет возвращать значение всеx выходов ансамбля. Значения параметров функции createEns() мы уже получили после оптимизации ансамбля. У вас могут быть другие значения. Скрипт, приведенный ниже, находится в файле FUN_Stacking().
#----Library------------- import_fun(rminer, holdout, holdout) #source(file = "FunPrepareData_VII.R") #----Input------------- evalq({ #type of activation function. Fact <- c("sig", #: sigmoid "sin", #: sine "radbas", #: radial basis "hardlim", #: hard-limit "hardlims", #: symmetric hard-limit "satlins", #: satlins "tansig", #: tan-sigmoid "tribas", #: triangular basis "poslin", #: positive linear "purelin") #: linear n <- 500 #---createENS---------------------- createEns <- function(numFeature = 8L, r = 7L, nh = 5L, fact = 7L, order, X){ # определяем индексы лучших предикторов bestF <<- order %>% head(numFeature) # выбираем лучшие предикторы для обучающего набора Xtrain <- X$pretrain$x[ , bestF] #setMKLthreads(1) k <- 1 rng <- RNGseq(n, 12345) #---creste Ensemble--- Ens <<- foreach(i = 1:n, .packages = "elmNN") %do% { rngtools::setRNG(rng[[k]]) idx <- rminer::holdout(Ytrain, ratio = r/10, mode = "random")$tr k <- k + 1 elmtrain(x = Xtrain[idx, ], y = Ytrain[idx], nhid = nh, actfun = Fact[fact]) } return(Ens) } #---GetInputData -FUN----------- GetInputData <- function(Ens, X){ #---predict-InputTrain-- Xtest <- X$train$x[ , bestF] foreach(i = 1:n, .packages = "elmNN", .combine = "cbind") %do% { predict(Ens[[i]], newdata = Xtest) } -> predEns #[ ,n] #---predict--InputTest---- Xtest1 <- X$test$x[ , bestF] foreach(i = 1:n, .packages = "elmNN", .combine = "cbind") %do% { predict(Ens[[i]], newdata = Xtest1) } -> InputTest #[ ,n] #---predict--InputTest1---- Xtest2 <- X$test1$x[ , bestF] foreach(i = 1:n, .packages = "elmNN", .combine = "cbind") %do% { predict(Ens[[i]], newdata = Xtest2) } -> InputTest1 #[ ,n] #---res------------------------- return(list(InputTrain = predEns, InputTest = InputTest, InputTest1 = InputTest1)) } }, env)
Создаем ансамбль и вычисляем входы для обучаемых объединителей:
#---4--createEns---------------- evalq({ Ytrain <- X1$pretrain$y Ytest <- X1$train$y Ytest1 <- X1$test$y Ytest2 <- X1$test1$y Ens <- vector(mode = "list", n) createEns(order = orderX1, X = X1) -> Ens GetInputData(Ens, X1) -> res }, env)
Структура результата:
> env$res %>% str() List of 3 $ InputTrain: num [1:1001, 1:500] 0.811 0.882 0.924 0.817 0.782 ... $ InputTest : num [1:501, 1:500] 0.5 0.383 0.366 0.488 0.359 ... $ InputTest1: num [1:251, 1:500] 0.32 0.246 0.471 0.563 0.451 ...
2. Базовые модели сравнения
Мы будем создавать два обучаемых объединителя. Один заменит усреднение выходов лучших нейросетей ансамбля, а второй заменит обрезку и усреднение. Поэтому нам понадобятся показатели качества классификации для обоих вариантов.
Ансамбль нейросетей
Для первого варианта базовой моделью сравнения будет ансамбль ELM с оптимальными параметрами.
Поскольку во втором варианте 500 входных данных, мы будем использовать пакет varbvs. В нем есть быстрые алгоритмы для подбора байесовских моделей выбора переменных и вычисления коэффициентов Байеса, где результат моделируется с использованием линейной или логистической регрессии. Алгоритмы основаны на вариационных приближениях, описанных в статье "Масштабируемый вариационный вывод для выбора байесовских переменных в регрессии и его точность в исследованиях генетических ассоциаций". Это ПО было применено к большим наборам данных с более чем миллионом переменных и тысячами образцов.
Для первого варианта напишем дополнительные функции getBest(), testAver(), testVot() и вычислим метрики. Функции находятся в файле FUN_Stacking.R.
evalq({
getBest <- function(Ens, x, y, nb){
n <- length(Ens)
foreach(i = 1:n, .packages = "elmNN", .combine = "cbind") %do% {
predict(Ens[[i]], newdata = x)} -> y.pr
foreach(i = 1:n, .combine = "c") %do% {
median(y.pr[ ,i])} ->> th
foreach(i = 1:n, .combine = "c") %do% {
ifelse(y.pr[ ,i] > th[i], 1, 0) -> Ypred
Evaluate(actual = y, predicted = Ypred)$Metrics$F1 %>%
mean()
} -> Score
Score %>% order(decreasing = TRUE) %>% head(2*nb + 1) -> best
y.pr[ ,best] %>%
apply(1, sum) %>%
divide_by(length(best)) %>%
median() -> med
return(list(Score = Score, bestNN = best, med = med))
}
testAver <- function(Ens, x, y, best, med){
n <- length(Ens)
foreach(i = 1:n, .packages = "elmNN", .combine = "cbind") %:%
when(i %in% best) %do% {
predict(Ens[[i]], newdata = x)} %>%
apply(1, sum) %>% divide_by(length(best)) -> ensPred
ifelse(ensPred > med, 1, 0) -> clAver
Evaluate(actual = y, predicted = clAver)$Metrics[ ,2:5] %>%
round(3) -> Score
return(list(Score = Score, Ypred = ensPred, clAver = clAver))
}
testVot <- function(Ens, x, y, best){
n <- length(Ens)
foreach(i = 1:n, .packages = "elmNN", .combine = "cbind") %:%
when(i %in% best) %do% {
predict(Ens[[i]], newdata = x)} %>%
apply(2, function(x) ifelse(x > th[i], 1, -1)) %>%
apply(1, function(x) sum(x)) -> vot
ifelse(vot > 0, 1, 0) -> ClVot
Evaluate(actual = y, predicted = ClVot)$Metrics[ ,2:5] %>%
round(3) -> Score
return(list(Score = Score, Ypred = ClVot))
}
}, env) Эти функции мы уже рассматривали, поэтому я не буду останавливаться на их описании. А вот на их выходах стоит заострить внимание.
- Функция getBest() возвращает метрики (Score), индексы лучших индивидуальных классификаторов ансамбля (bestNN), медиану усредненного выхода ансамбля (med), которая будет использована при тестировании модели. В окружение сбрасывается вектор медиан th[500] всех выходов ансамбля.
- Функция testAver() возвращает метрики (Score), усредненный непрерывный предикт ансамбля (Ypred) и номинальный предикт ансамбля (clAver).
- Функция testVot() возвращает метрики (Score) и номинальный предикт ансамбля (Ypred).
Протестируем созданный ансамбль на двух тестовых наборах с применением усреднения и голосованием большинством и посмотрим метрики.
#--2---test---- evalq({ Ytrain <- X1$pretrain$y Ytest <- X1$train$y Ytest1 <- X1$test$y Ytest2 <- X1$test1$y Ens <- vector(mode = "list", n) Ens <- createEns(order = orderX1, X = X1) #---3------ resBest <- getBest(Ens, x = X1$train$x[ , bestF], y = Ytest, nb = 3) #---4--averaging--- ScoreAver <- testAver(Ens, x = X1$test$x[ , bestF], y = Ytest1, best = resBest$bestNN, med = resBest$med) ScoreAver1 <- testAver(Ens, x = X1$test1$x[ , bestF], y = Ytest2, best = resBest$bestNN, med = resBest$med) #---5--voting---- ScoreVot <- testVot(Ens, x = X1$test$x[ , bestF], y = Ytest1, best = resBest$bestNN) ScoreVot1 <- testVot(Ens, x = X1$test1$x[ , bestF], y = Ytest2, best = resBest$bestNN) }, env) > env$ScoreAver$Score Accuracy Precision Recall F1 0 0.75 0.708 0.778 0.741 1 0.75 0.794 0.727 0.759 > env$ScoreAver1$Score Accuracy Precision Recall F1 0 0.753 0.750 0.826 0.786 1 0.753 0.758 0.664 0.708 > env$ScoreVot$Score Accuracy Precision Recall F1 0 0.752 0.702 0.800 0.748 1 0.752 0.808 0.712 0.757 > env$ScoreVot1$Score Accuracy Precision Recall F1 0 0.741 0.739 0.819 0.777 1 0.741 0.745 0.646 0.692
Хорошие показатели на обоих тестовых наборах. Ниже, при анализе результатов, мы разложим ошибку классификации на bias/variance/noise и оценим вклад каждой составляющей в общую ошибку.
Для второго варианта (500 входов) скрипт обучения модели приведен ниже. Находится в файле varb.R.
library(varbvs) evalq({ vr <- varbvs(X = res$InputTrain, Z = NULL, y = Ytest, family = "binomial", optimize.eta = TRUE, logodds = seq(-6,-2, 0.25), nr = 250, initialize.params = TRUE, maxiter = 1e5, verbose = FALSE) summary(vr, cred.int = 0.95, nv = 7, nr = 1e5) %>% print() }, env) Summary of fitted Bayesian variable selection model: family: binomial num. hyperparameter settings: 17 samples: 1001 iid variable selection prior: yes variables: 500 fit prior var. of coefs (sa): yes covariates: 1 fit approx. factors (eta): yes maximum log-likelihood lower bound: -579.4602 Hyperparameters: estimate Pr>0.95 candidate values sa 8.09 [7.63,8.54] NA--NA logodds -2.26 [-2.75,-2.00] (-6.00)--(-2.00) Selected variables by probability cutoff: >0.10 >0.25 >0.50 >0.75 >0.90 >0.95 3 3 3 3 3 3 Top 7 variables by inclusion probability: index variable prob PVE coef* Pr(coef.>0.95) X18 18 X18 1.00000 NA 4.529 [+3.861,+5.195] X5 5 X5 1.00000 NA 1.955 [+1.543,+2.370] X255 255 X255 1.00000 NA 2.097 [+1.537,+2.660] X109 109 X109 0.00948 NA -1.033 [-2.008,-0.057] X404 404 X404 0.00467 NA -0.665 [-1.350,+0.024] X275 275 X275 0.00312 NA -0.726 [-1.735,+0.286] X343 343 X343 0.00299 NA -0.604 [-1.353,+0.149] *See help(varbvs) about interpreting coefficients in logistic regression.
Используя полученную модель, вычислим предикт на двух тестовых наборах и рассчитаем метрики.
env$vr$pip %>% order() %>% tail(7) -> bestNN_vr evalq({ predict(vr, res$InputTest) -> pr.vr1 Evaluate(actual = Ytest1, predicted = pr.vr1)$Metrics[ ,2:5] %>% round(3) -> metr.test confus(table(Ytest1, pr.vr1)) -> cm1 predict(vr, res$InputTest1) -> pr.vr2 Evaluate(actual = Ytest2, predicted = pr.vr2)$Metrics[ ,2:5] %>% round(3) -> metr.test1 confus(table(Ytest2, pr.vr2)) -> cm2 }, env) > env$metr.test Accuracy Precision Recall F1 0 0.78 0.750 0.783 0.766 1 0.78 0.808 0.779 0.793 > env$metr.test1 Accuracy Precision Recall F1 0 0.729 0.765 0.732 0.748 1 0.729 0.689 0.726 0.707
Получился отличный результат, лучше, чем метрики ансамбля.
Все данные, необходимые для продолжения экспериментов, готовы.
Поскольку в дальнейшем мы предполагаем использовать библиотеки keras / tensorflow, ниже коротко рассмотрим их.
3. Библиотеки keras/nensorflow. Общее описание и установка
Бурно развивающаяся область изучения глубоких нейросетей пополнилась рядом библиотек с открытым кодом. В их числе — TensorFlow(Google), CNTK(Microsoft), Apache MXNet и многие другие. Благодаря тому, что все эти и другие крупнейшие разработчики ПО входят в Консорциум R, для всех этих библиотек предоставляется АПИ к R.
Все вышеперечисленные библиотеки низкоуровневые. Для начинающих они сложны в изучении и использовании. Учитывая это, команда Rstudio разработала пакет keras для R.
Keras — это высокоуровневое API нейронных сетей. Пакет разработан с упором на возможность быстрого прототипирования и экспериментальной проверки работоспособности модели. Вот ключевые особенности Keras:
- Позволяет одинаково работать на CPU или на графическом процессоре.
- Дружественный API, который позволяет легко прототипировать модели глубокого обучения.
- Встроенная поддержка сверточных сетей (для компьютерного зрения), рекурентных сетей (для обработки последовательности) и любых их комбинаций.
- Поддерживает произвольные сетевые архитектуры: модели с несколькими входами или несколькими выходами, совместное использование слоев, совместное использование моделей и т. д. Это означает, что Keras подходит для построения, по существу, любой модели глубокого обучения, от сети памяти до нейронной машины Тьюринга.
- Он способен работать поверх нескольких back-end, включая TensorFlow, CNTK или Theano.
Keras — это API, предназначенный для людей, а не для машин. Пакет снижает когнитивную нагрузку: предлагает последовательные и простые API, минимизирует количество действий пользователя и обеспечивает эффективную обратную связь по ошибкам пользователя. Все это делает Keras легким в изучении и простым в использовании. Но это не связано уменьшением гибкости: поскольку Keras интегрируется с низкоуровневыми языками глубокого обучения (в частности, TensorFlow), он позволяет реализовать все, что вы могли бы создать на базовом языке.
Можно разработать модель Keras с использованием нескольких модулей глубокого обучения. Любая модель Keras, которая использует только встроенные слои, может переноситься без изменений по всем этим бэкэндам: вы можете обучить модель с одним бэкэндом и загрузить ее в другой (например, для развертывания). Доступные бэкэнды включают:
- TensorFlow backend (from Google)
- CNTK backend (from Microsoft)
- Theano backend
Можно обучить модель Keras на нескольких различных аппаратных платформах, не только CPU:
- NVIDIA GPUs
- Google TPUs, via the TensorFlow backend and Google Cloud
- OpenCL-enabled GPUs, such as those from AMD, via the PlaidML Keras backend
Инсталляция keras и tensorflow backend
Keras и TensorFlow можно настроить для работы на CPU или графических процессорах. Версия CPU намного проще в установке и настройке, поэтому это лучший выбор для начала работы с пакетом. Вот руководство по версиям CPU и GPU с веб-сайта TensorFlow:
- TensorFlow только с CPU. Мы рекомендуем сначала установить эту версию, особенно если в вашей системе нет графического процессора NVIDIA®.
- TensorFlow с поддержкой GPU. Обычно программы TensorFlow работают на GPU быстрее, чем на процессоре. Поэтому, если ваша система имеет графический процессор NVIDIA®, отвечающий всем предварительным требованиям, и вам необходимо запустить высокопроизводительные приложения, вы должны в конечном итоге установить эту версию.
Единственный поддерживаемый метод установки в Windows — conda. Это означает, что перед установкой Keras вы должны установить Anaconda 3.x(Phyton 3.5.x/3.6.x) для Windows. Я установил Anaconda3(Python3.6).
Сначала устанавливаем keras пакет из CRAN:
install.packages("keras") Интерфейс Keras R по умолчанию использует TensorFlow. Чтобы установить и основную библиотеку Keras, и бэкэнд TensorFlow, используйте функцию install_keras ():
# default installation library(keras) install_keras()
Так Keras и TensorFlow будут установлены на основе CPU. Если вам нужна индивидуальная установка — например, с графическим процессором NVIDIA, изучите документацию. Для установки TensorFlow определенной версии или использующую GPU нужно выполнить:
# install with GPU version of TensorFlow # (NOTE: only do this if you have an NVIDIA GPU + CUDA!) install_keras(tensorflow = "gpu") # install a specific version of TensorFlow install_keras(tensorflow = "1.5") install_keras(tensorflow = "1.5-gpu")
Более подробно см. здесь.
Для проведения экспериментов c TensorFlow предназначен поддерживающий пакет tfruns. Это набор инструментов для управления обучением и экспериментами TensorFlow из R.
- Отслеживайте гиперпараметры, метрики, выходные данные и исходный код каждого тренировочного цикла.
- Сравнивайте гиперпараметры и метрики между прогонами, чтобы найти наиболее эффективную модель.
- Автоматически создавайте отчеты для визуализации отдельных тренировочных прогонов или сравнений между прогонами.
- Никаких изменений в исходном коде не требуется (данные запуска автоматически фиксируются для всех моделей Keras и tfestimators).
Наилучшее качество визуализации процесса и результатов обучения DNN предоставляет TensorBoard.
Знатокам глубокого обучения дается возможность работать напрямую с низкоуровневой библиотекой TensorFlow с помощью пакета tensorflow.
Все эти пакеты базируются на основном — reticulate, который представляет собой R-интерфейс к Python-модулям, функциям и классам. При вызове в Python типы данных R автоматически преобразуются в их эквивалентные типы Python. Возвращаемые с Python значения преобразуются обратно в типы R.
Все эти пакеты отлично документированы, снабжены множеством примеров, постоянно развиваются. Благодаря этому мы можем использовать в экспертах и индикаторах терминала наиболее продвинутые модели глубокого обучения (DNN, RNN, CNN, LSTM, VAE и др), обучения с подкреплением (RL) и множество других наработок Python. Единственное ограничение — знания и опыт разработчика.
Отмечу еще два интересных пакета: kerasR и kerasformula. По первому есть тесты, подтверждающие скорость работы, более высокую, чем у оригинальной "tensorflow-1.5". Вторая предлагает упрощенный вариант задания модели с помощью формулы.
Цель этой статьи — только привести примеры для простого старта в новой области. В наши задачи не входит охватить все многообразие возможностей и получить высокие показатели качества модели.
Перед началом экспериментов нужно проверить, установлен ли Python и взаимодействует ли с ним R.
> library(reticulate)
> py_config()
python: K:\Anaconda3\envs\r-tensorflow\python.exe
libpython: K:/Anaconda3/envs/r-tensorflow/python36.dll
pythonhome: K:\ANACON~1\envs\R-TENS~1
version: 3.6.5 | packaged by conda-forge | (default, Apr 6 2018, 16:13:55)
[MSC v.1900 64 bit (AMD64)]
Architecture: 64bit
numpy: K:\ANACON~1\envs\R-TENS~1\lib\site-packages\numpy
numpy_version: 1.14.2
tensorflow: K:\ANACON~1\envs\R-TENS~1\lib\site-packages\tensorflow
python versions found:
K:\Anaconda3\envs\r-tensorflow\python.exe
K:\ANACON~1\python.exe
K:\Anaconda3\python.exe Посмотрим, какая версия tensorflow используется:
> library(tensorflow) > tf_config() TensorFlow v1.5.1 (K:\ANACON~1\envs\R-TENS~1\lib\site-packages\tensorflow) Python v3.6 (K:\Anaconda3\envs\r-tensorflow\python.exe)
Все готово для продолжения экспериментов.
4. Объединитель выходов bagging ансамбля — нейросеть
Проведем два эксперимента. В первом вместо усреднения лучших выходов ансамбля применим функцию softmax. Во втором заменим обрезание и усреднение нейросетью, подав на ее вход все 500 выходов ансамбля. Структурная схема экспериментов приведена на рисунке ниже.
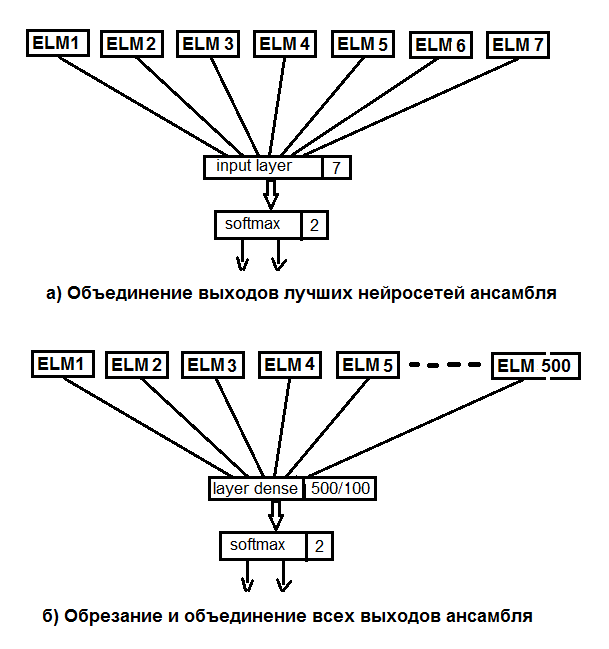
Рис.2. Замена усреднения выходов ансамбля нейросетью
Основная структура данных Keras — это модель, способ организации слоев. Простейший тип — последовательная модель, (Sequential model), представляющая собой линейную совокупность слоев.
Сначала создадим простейшую последовательную модель, а потом начнем добавлять слои с помощью оператора pipe (%>%). Для первого эксперимента создадим нейросеть, состоящую только из входного и выходного слоя. На вход будем подавать выходы ансамбля после обрезки, полученные на тестовом наборе. Для валидации будем использовать 20% обучающего набора. Создание, обучение и тестирование нейросетей в этом пакете чрезвычайно просто.
Задаем постоянные параметры модели, определяем обучающий и тестовые наборы для DNN.
#===========Keras=========================================== library(keras) num_classes <- 2L batch_size <- 32L epochs <- 300L #--------- bestNN <- env$resBest$bestNN x_train <- env$res$InputTrain[ ,bestNN] y_train <- env$Ytest %>% to_categorical() x_test <- env$res$InputTest[ ,bestNN] y_test <- env$Ytest1 %>% to_categorical() x_test1 <- env$res$InputTest1[ ,bestNN] y_test1 <- env$Ytest2 %>% to_categorical()
Создаем модель. Код скрипта представлен ниже. Определяем и компилируем модель со структурой NN(7, 2) — 7 нейронов на входе и 2 на выходе. Оптимизатор — функция "rmsprop ", функция потерь — 'binary'_crossentropy' , метрики по результатам тестирования — "accuracy".
##----model--keras------------------------- # define model model <- keras_model_sequential() # add layers and compile model %>% layer_dense(units = num_classes, input_shape = dim(x_train)[2]) %>% layer_activation(activation = 'softmax') %>% compile( loss = 'binary_crossentropy', optimizer = optimizer_rmsprop(), metrics = 'accuracy' )
Обучаем и тестируем модель (скрипт представлен ниже). Сохраняем историю обучения. Дополнительно указываем:
- не нужно выводить в терминал результат каждой итерации;
- не нужно показывать в Viever/Rstudio графики в реальном времени;
- нужно перемешивать входные данные после каждой эпохи обучения.
Для валидации используем 20% от обучающего набора.
## Training & Evaluation --------------------------- # Fit model to data model %>% fit( x_train, y_train, batch_size = batch_size, epochs = epochs, verbose = 0, view_metrics = FALSE, shuffle = TRUE, validation_split = 0.2) -> history # Output metrics score <- model %>% evaluate(x_test, y_test, verbose = 0) cat('Test loss:', score[[1]] %>% round(3), '\n') Test loss: 0.518 cat('Test accuracy:', score[[2]] %>% round(3), '\n') Test accuracy: 0.754 #--------------- score1 <- model %>% evaluate(x_test1, y_test1, verbose = 0) cat('Test loss:', score1[[1]] %>% round(3), '\n') Test loss: 0.55 cat('Test accuracy:', score1[[2]] %>% round(3), '\n') Test accuracy: 0.737
Количественные результаты неплохие, они практические равны результатам усреднения ансамбля. Посмотрим на историю обучения и тестирования этой модели:
#--plot------------------------
plot(history) 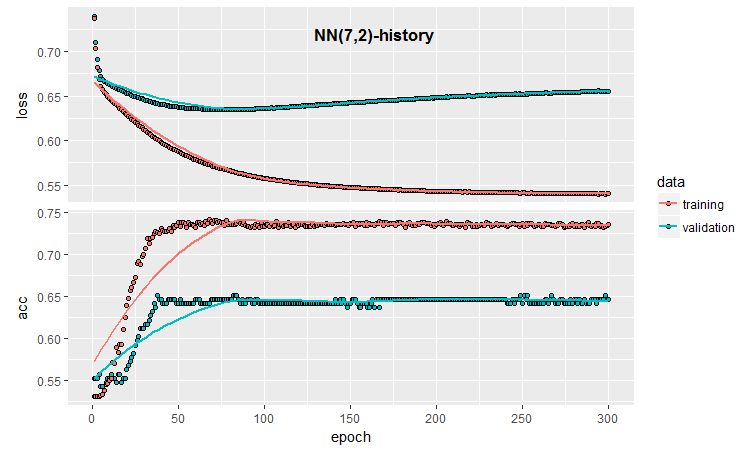
Рис.3. История обучения модели (7, 2)
Из графика видно, что приблизительно после 30 эпох модель явно переобучена, а после 50 эпох Аccuracy выходит на плато.
Еще раз хочу напомнить, что процесс инициализации нейросети случаен. Т.е. при каждом новом создании, обучении и тестировании модели мы будем получать различные результаты.
Даже в этой минимальной конфигурации нейросети у нас много возможностей влиять на качество классификации и переобучение. Бегло перечислю некоторые: ранняя остановка обучения ( earlystopping ); способ инициализации нейронов; регуляризация активационной функции и т.д. Из них проверим только раннюю остановку обучения, добавление шума во входные данные, регуляризацию активационной функции и выведем более подробную графическую картину результатов обучения. Для этого используем предоставленную keras возможность обратного выхода во время обучения (callback).
callback_early_stopping(monitor = "val_loss", min_delta = 0, patience = 0, verbose = 0, mode = c("auto", "min", "max")) callback_tensorboard(log_dir = NULL, histogram_freq = 0, batch_size = 32, write_graph = TRUE, write_grads = FALSE, write_images = FALSE, embeddings_freq = 0, embeddings_layer_names = NULL, embeddings_metadata = NULL)
Определим функции обратного вызова.
early_stopping <- callback_early_stopping(monitor = "val_acc", min_delta = 1e-5, patience = 20, verbose = 0, mode = "auto") log_dir <- paste0(getwd(),"/run_1") tensboard <- callback_tensorboard(log_dir = log_dir, histogram_freq = 1, batch_size = 32, write_graph = TRUE, write_grads = TRUE, write_images = FALSE)
В первой мы указали, что отслеживать нужно значение accuracy. Если ее значение становится меньше min_delta в течение patiente эпох, то обучение следует прекратить. Во второй задали путь к директории, где нужно сохранить результаты обучения для последующего воспроизведения, и указали, что именно сохранять. Напишем полный скрипт с применением этих функций и посмотрим на результат.
##=====Variant earlystopping================================= #--prepare data-------------------------- library(reticulate) library(keras) py_set_seed(12345) num_classes <- 2L batch_size <- 32L learning_rate <- 0.005 epochs <- 100L #--------- bestNN <- env$resBest$bestNN x_train <- env$res$InputTrain[ ,bestNN] y_train <- env$Ytest %>% to_categorical() x_test <- env$res$InputTest[ ,bestNN] y_test <- env$Ytest1 %>% to_categorical() x_test1 <- env$res$InputTest1[ ,bestNN] y_test1 <- env$Ytest2 %>% to_categorical() ##----model--keras------------------------- # define model model <- keras_model_sequential() # add layers and compile model %>% layer_gaussian_noise(stddev = 0.05, input_shape = dim(x_train)[2], name = "GN") %>% layer_dense(units = num_classes, name = "dense1") %>% layer_activation_softmax(name = "soft") %>% layer_activity_regularization(l2 = 1.0, name = "reg") %>% #l1 = 0.01, compile( loss = 'binary_crossentropy', optimizer = optimizer_rmsprop(lr = learning_rate, decay = 0.01), metrics = 'accuracy' ) ## Training & Evaluation --------------------------- # Fit model to data model %>% fit( x_train, y_train, batch_size = batch_size, epochs = epochs, verbose = 0, view_metrics = TRUE , shuffle = TRUE, validation_split = 0.2, callbacks = list(early_stopping, tensboard)) -> history
Метрики на двух тестовых наборах и история обучения с ранним выходом.
# Output metrics > score <- model %>% evaluate(x_test, y_test, verbose = 0) > cat('Test loss:', score[[1]] %>% round(3), '\n') Test loss: 0.539 > cat('Test accuracy:', score[[2]] %>% round(3), '\n') Test accuracy: 0.756 > #--------------- > score1 <- model %>% evaluate(x_test1, y_test1, verbose = 0) > cat('Test loss:', score1[[1]] %>% round(3), '\n') Test loss: 0.571 > cat('Test accuracy:', score1[[2]] %>% round(3), '\n') Test accuracy: 0.713
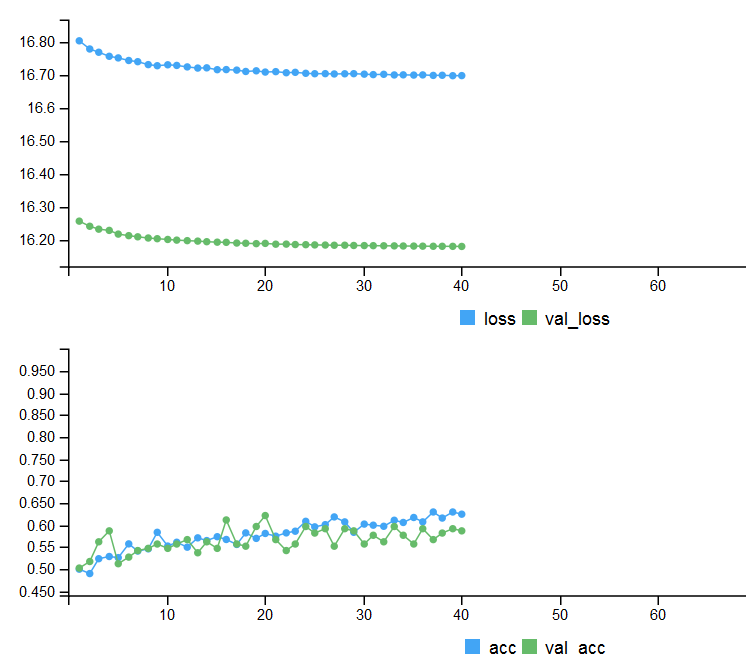
Рис. 4. История обучения модели с ранней остановкой
Для вывода подробной графической информации о процессе обучения нейросети используем возможности tensorboard:
> tensorboard(log_dir = log_dir) TensorBoard 1.7.0 at http://127.0.0.1:7451 (Press CTRL+C to quit) Started TensorBoard at http://127.0.0.1:7451
В браузере откроется страница, где можно рассмотреть все внутренные детали нейросети. Вот примеры скриншотов:
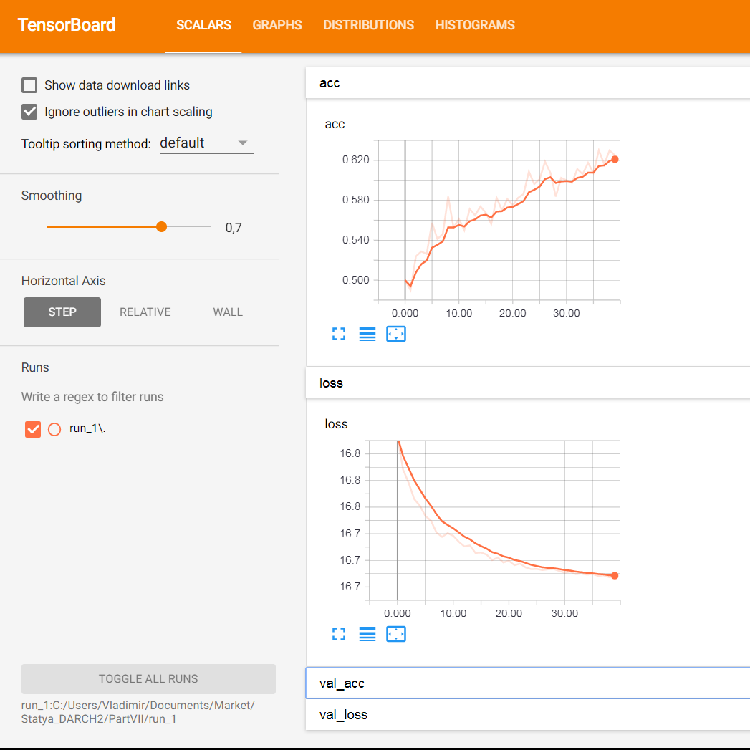
Рис. 5. Графики потерь и точности при обучении на обучающем наборе, валидационные данные на val_acc и val_loss
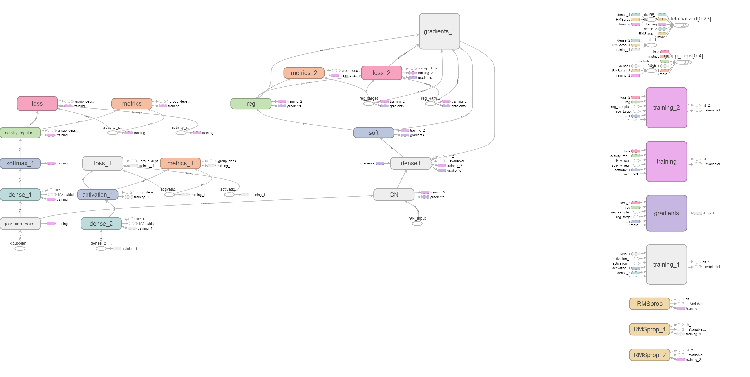
Рис. 6. Вычислительный граф нейросети
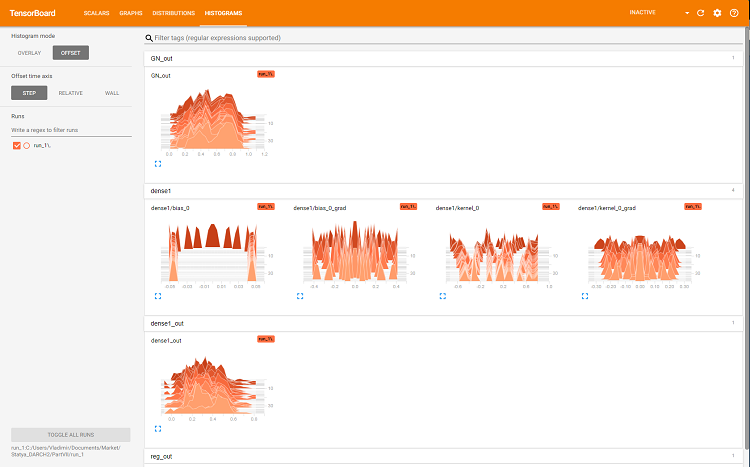
Рис. 7. Гистограммы слоя dense
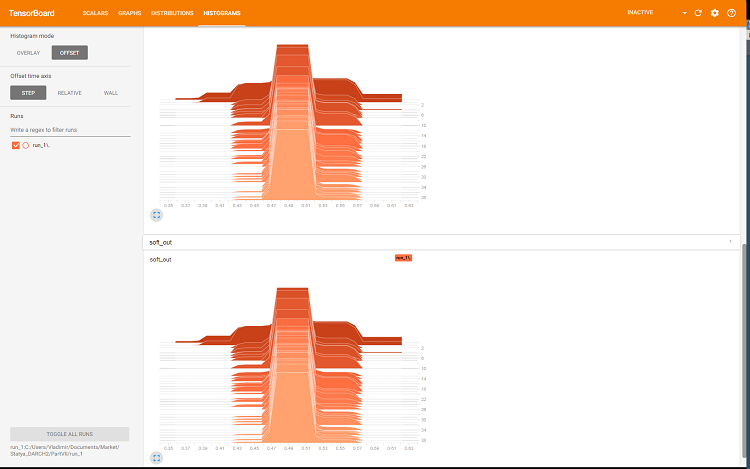
Рис. 8. Гистограмма выходов sotmax и regularization
Эти графики — мощный инструмент для отладки параметров нейросети, но их подробный разбор выходит за рамки этой статьи.
При каждом новом запуске tensorboard нужно изменять путь сохранения log_dir или удалять ранее использованный.
Посмотрим, как изменится качество классификации с теми же параметрами, но для валидации используем тестовый набор. Скрипт приведен ниже и находится в файле:
library(reticulate) library(keras) py_set_seed(12345) num_classes <- 2L batch_size <- 32L learning_rate <- 0.005 epochs <- 100L #--------- bestNN <- env$resBest$bestNN x_train <- env$res$InputTrain[ ,bestNN] y_train <- env$Ytest %>% to_categorical() x_test <- env$res$InputTest[ ,bestNN] y_test <- env$Ytest1 %>% to_categorical() x_test1 <- env$res$InputTest1[ ,bestNN] y_test1 <- env$Ytest2 %>% to_categorical() #---------------------------------------- early_stopping <- callback_early_stopping(monitor = "val_acc", min_delta = 1e-5, patience = 20, verbose = 0, mode = "auto") log_dir <- paste0(getwd(),"/run_2") tensboard <- callback_tensorboard(log_dir = log_dir, histogram_freq = 1, batch_size = 32, write_graph = TRUE, write_grads = TRUE, write_images = FALSE) ##----model--keras------------------------- # define model model <- keras_model_sequential() # add layers and compile model %>% layer_gaussian_noise(stddev = 0.05, input_shape = dim(x_train)[2], name = "GN") %>% layer_dense(units = num_classes, name = "dense1") %>% layer_activation_softmax(name = "soft") %>% layer_activity_regularization(l2 = 1.0, name = "reg") %>% #l1 = 0.01, compile( loss = 'binary_crossentropy', optimizer = optimizer_rmsprop(lr = learning_rate, decay = 0.01), metrics = 'accuracy' ) ## Training & Evaluation --------------------------- # Fit model to data model %>% fit( x_train, y_train, batch_size = batch_size, epochs = epochs, verbose = 0, view_metrics = TRUE , shuffle = TRUE, validation_data = list(x_test, y_test), callbacks = list(early_stopping, tensboard)) -> history
Посмотрим метрики на втором тестовом наборе:
#--model--test1------------------------------------------------- predict(model, x_test1) -> Ypr.test1 Ypr.test1 %>% max.col()-1 -> y_pr_test1 #Ypr.test1 %>% apply(1, function(x) which.max(x)) %>% subtract(1) -> y_pr_test1 evalq(res_mod_test1 <- Eval(Ytest2, y_pr_test1), env) > env$res_mod_test1 $metrics Accuracy Precision Recall F1 0 0.713 0.704 0.826 0.760 1 0.713 0.730 0.575 0.644 $confMatr Confusion Matrix and Statistics predicted actual 0 1 0 114 24 1 48 65 Accuracy : 0.7131 95% CI : (0.6529, 0.7683) No Information Rate : 0.6454 P-Value [Acc > NIR] : 0.013728 Kappa : 0.4092 Mcnemar's Test P-Value : 0.006717 Sensitivity : 0.7037 Specificity : 0.7303 Pos Pred Value : 0.8261 Neg Pred Value : 0.5752 Prevalence : 0.6454 Detection Rate : 0.4542 Detection Prevalence : 0.5498 Balanced Accuracy : 0.7170 'Positive' Class : 0
Они практически не отличаются от первого варианта. Можем визуально сравнить два варианта с помощью tensorboard.
#-----plot------------------ tensorboard(log_dir = c(paste0(getwd(),"/run_1"), paste0(getwd(),"/run_2")))
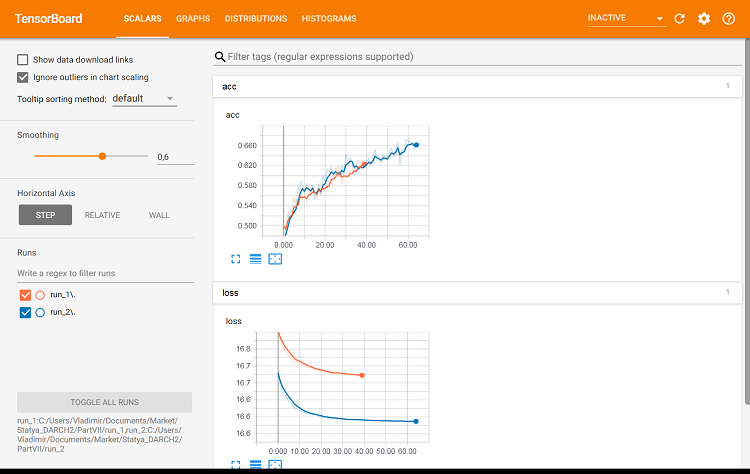
Рис. 9. Метрики на обучающем наборе
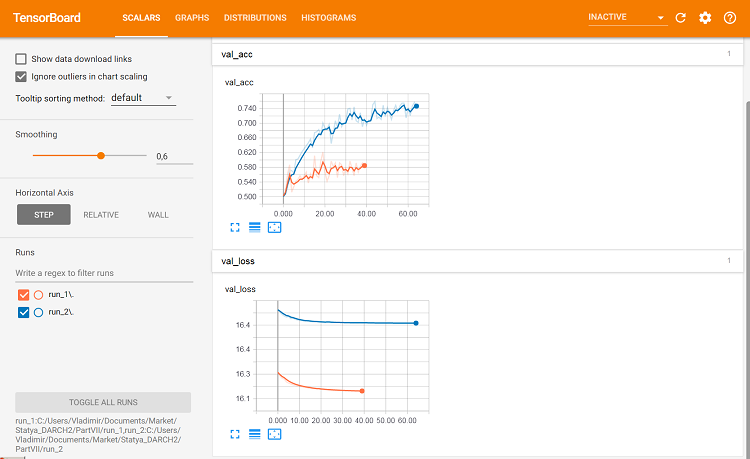
Рис. 10. Метрики на валидационном наборе
А вот здесь уже видны различия.
Проведем последний эксперимент. На вход многослойной нейросети подадим все 500 выходов ансамбля. Таким образом нейросеть выполнит обрезку и объединение одновременно. Скрипт приведен ниже и находится в файле modelDNN_500.R.
library(reticulate) library(keras) py_set_seed(12345) num_classes <- 2L batch_size <- 32L learning_rate <- 0.0001 epochs <- 100L #--------- x_train <- env$res$InputTrain y_train <- env$Ytest %>% to_categorical() x_test <- env$res$InputTest y_test <- env$Ytest1 %>% to_categorical() x_test1 <- env$res$InputTest1 y_test1 <- env$Ytest2 %>% to_categorical() #---------------------------------------- early_stopping <- callback_early_stopping(monitor = "val_acc", min_delta = 1e-5, patience = 20, verbose = 0, mode = "auto")
Мы загрузили библиотеки, константы, определили наборы для обучения и тестирования, а также функцию ранней остановки.
##----modelDNN--keras------------------------- # define model modDNN <- keras_model_sequential() # add layers and compile modDNN %>% layer_gaussian_noise(stddev = 0.001, input_shape = dim(x_train)[2], name = "GN") %>% layer_batch_normalization() %>% layer_dense(units = 100, activation = "elu", name = "dense1") %>% layer_dropout(rate = 0.5, name = "dp1") %>% layer_batch_normalization() %>% layer_dense(units = 50, activation = "elu", name = "dense2") %>% layer_batch_normalization() %>% layer_dropout(rate = 0.5, name = "dp2") %>% layer_dense(units = 10, activation = "elu", name = "dense3") %>% layer_batch_normalization() %>% layer_dropout(rate = 0.2, name = "dp3") %>% layer_dense(units = num_classes, activation = "softmax", name = "soft") %>% compile( loss = 'binary_crossentropy', optimizer = optimizer_rmsprop(lr = learning_rate, decay = 0.0001), metrics = 'accuracy' )
Итак, мы определили нейросеть, указав последовательность и параметры слоев. Также мы указали компилятору, какую функцию потерь, оптимизатор и метрику использовать при обучении модели. Теперь обучим модель:
## Training & Evaluation --------------------------- # Fit model to data modDNN %>% fit( x_train, y_train, batch_size = batch_size, epochs = epochs, verbose = 0, view_metrics = TRUE , shuffle = TRUE, validation_split = 0.2, #validation_data = list(x_test, y_test), callbacks = list(early_stopping)) -> history
При обучении метрики выведем и перемешаем входные данные, для валидации будем использовать 20% обучающего набора и применим ранний останов. Протестируем модель на тестовом наборе:
#--model--test------------------------- predict(modDNN, x_test) -> Ypr.test Ypr.test %>% apply(1, function(x) which.max(x)) %>% subtract(1) -> y_pr_test evalq(res_mod_test <- Eval(Ytest1, y_pr_test), env)
Смотрим результат в цифрах на уровне усреднения ансамбля:
> env$res_mod_test $metrics Accuracy Precision Recall F1 0 0.752 0.702 0.800 0.748 1 0.752 0.808 0.712 0.757 $confMatr Confusion Matrix and Statistics predicted actual 0 1 0 184 46 1 78 193 Accuracy : 0.7525 95% CI : (0.7123, 0.7897) No Information Rate : 0.523 P-Value [Acc > NIR] : < 2.2e-16 Kappa : 0.5068 Mcnemar's Test P-Value : 0.005371 Sensitivity : 0.7023 Specificity : 0.8075 Pos Pred Value : 0.8000 Neg Pred Value : 0.7122 Prevalence : 0.5230 Detection Rate : 0.3673 Detection Prevalence : 0.4591 Balanced Accuracy : 0.7549 'Positive' Class : 0
Построим историю обучения:
plot(history)
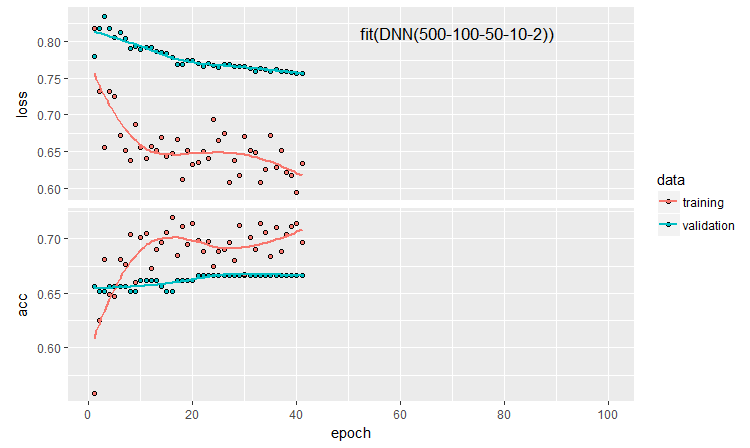
Рис. 11. История обучения нейросети DNN500
Чтобы улучшить качество классификации, мы можем модифицировать множество гиперпараметров: способ инициализации нейронов, регуляризацию активации нейронов и их весов и т.д.. Результаты, полученные почти с интуитивными параметрами, обнадеживают качеством, но огорчают потолком. Без оптимизации получить Accuracy больше 0.82 не получилось. Вывод: нужно опимизировать гиперпараметры нейросети. В предыдущих частях мы экспериментировали с байесовской оптимизацией. Думаю, она применима и здесь, но это отдельная и непростая тема.
Последовательный способ определения модели дает возможность построить и настроить модели различной сложности и глубины. Но с помощью функционального АПИ keras можно создавать более сложные структуры нейросетей: например, со множеством входов и выходов. Об этом мы поговорим в следующей статье.
5. Анализ результатов экспериментов
Итак, у нас есть результаты обучения и тестирования пяти моделей:
- ансамбль с усреднением (EnsAver);
- ансамбль с голосованием большинством (EnsVot);
- модель логистической регрессии varb;
- нейросеть DNN(7,2);
- нейросеть DNN500.
Соберем в одну таблицу показатели качества всех этих моделей, разложим ошибку классификации на составляющие и оценим их вклад в общую ошибку. Используем функцию randomUniformForest::biasVarCov() (Bias-Variance-Covariance Decomposition). Подробней об этой функции смотрите в описании пакета. Код декомпозиции ошибки классификации ансамблей EnsAver и EnsVot приведен ниже. Для остальных моделей скрипты аналогичны.
#---bias--test------------------------------- import_fun(randomUniformForest, biasVarCov, BiasVar) evalq({ target = Ytest1 biasAver <- BiasVar(predictions = ScoreAver$clAver, target = target, regression = FALSE, idx = 1:length(target)) biasVot <- BiasVar(predictions = ScoreVot$ClVot, target = target, regression = FALSE, idx = 1:length(target)) }, env) ----------------------------- Noise: 0.2488224 Squared bias: 0.002107561 Variance of estimator: 0.250475 Covariance of estimator and target: 0.1257046 Assuming binary classification with classes {0,1}, where '0' is the majority class. Misclassification rate = P(Y = 1)P(Y = 0) + {P(Y = 1) - P(Y_hat = 1)}^2 + P(Y_hat = 0)P(Y_hat = 1) - 2*Cov(Y, Y_hat) Misclassification rate = P(Y = 1) + P(Y_hat = 1) - 2*E(Y*Y_hat) = 0.2499958 --------------------- Noise: 0.2488224 Squared bias: 0.004079665 Variance of estimator: 0.2499721 Covariance of estimator and target: 0.1274411 Assuming binary classification with classes {0,1}, where '0' is the majority class. Misclassification rate = P(Y = 1)P(Y = 0) + {P(Y = 1) - P(Y_hat = 1)}^2 + P(Y_hat = 0)P(Y_hat = 1) - 2*Cov(Y, Y_hat) Misclassification rate = P(Y = 1) + P(Y_hat = 1) - 2*E(Y*Y_hat) = 0.2479918
Компактный вывод:
> env$biasAver $predError [1] 0.2499958 $squaredBias [1] 0.002107561 $predictionsVar [1] 0.250475 $predictionsTargetCov [1] 0.1257046
| 95%CI Acc | Precision | Recall | F1 | PredErr | sqBias | predVar | predTargCov | |
|---|---|---|---|---|---|---|---|---|
| EnsAver | 0.7102, 0.7880 (0.7500) | 0.708 0.794 | 0.778 0.727 | 0.741 0.759 | 0.2499 | 0.0021 | 0.2505 | 0.1257 |
| EnsVot | 0.7123, 0.7897 (0.7525) | 0.702 0.808 | 0.800 0.712 | 0.748 0.757 | 0.248 | 0.0041 | 0.25 | 0.1274 |
| varb | 0.7416, 0.8159 (0.7804) | 0.790 0.808 | 0.783 0.779 | 0.766 0.793 | 0.2199 | 0.000398 | 0.25 | 0.13964 |
| DNN(7, 2) | 0.7165, 0.7935 (0.7565) | 0.765 0.751 | 0.678 0.823 | 0.719 0.785 | 0.2460 | 0.000195 | 0.2498 | 0.1264 |
| DNN500 | 0.7123, 0.7897 (0.7525) | 0.702 0.808 | 0.800 0.712 | 0.748 0.757 | 0.2779 | 0.01294 | 0.2452 | 0.1145 |
Что мы видим в сводной таблице:
- Лучший показатель Accuracy среди базовых моделей — у varb, объединяющей 500 выходов ансамбля. Среди обучаемых объединителей по этому показателю лучшая модель — DNN(7,2), объединяющая 7 лучших выходов ансамбля.
- Наименьшая ошибка теста для тестового образца (PredErr) — у varb и DNN(7,2).
- Квадрат смещения (sqBias ) у тех же двух моделей на порядок лучше остальных.
- Дисперсия ошибки (PredVar) у всех моделей практически одинакова. Это выглядит странно: ансамбль должен был дать снижение дисперсии, а мы получили низкое смещение.
- Наилучшая ковариация между оценкой и ответом (predictionTargetCov ) — у varb. Как самостоятельная величина она ни о чем не говорит, используется только для сравнения моделей.
- У модели DNN500 все показатели наихудшие. Вывод: усложнение моделей для простых задач не приводит к улучшению результатов.
Заключение
Ансамбль нейросетевых ELM классификаторов с усреднением или простым голосованием показывает неплохое качество классификации при очень высокой скорости вычисления. Улучшить качество можно, оптимизировав порог перевода выходов из непрерывного в номинальный и откалибровав выходы перед усреднением. Заметного уменьшения дисперсии ошибки не обнаружено.
Замена усреднения выходов ансамбля функцией softmax простой нейросети на порядок уменьшает смещение без заметного уменьшения дисперсии. Применение более сложных моделей нейросети для замены обрезки и усреднения не показали хороших результатов.
Модель логистической регрессии, полученная с помошью байесовских процессов выбора предикторов (пакет varbvs) показывает очень хорошие результаты. Можно использовать лучшие выходы, определенные этим пакетом для нейросети.
24% шума, которые выглядят неустранимыми начиная с препроцессинга, еще раз подсказывают, что нужно на каком-то этапе переразметить шумовые примеры в отдельный класс.
Нужно использовать возможности keras и работать с последовательностями (таймсериями), которыми и являются наши данные. Это может улучшить показатели качества классиификации.
Приложение
В GitHub/PartVII находятся следующие файлы:
- Importar.R — функции импорта пакетов.
- Library.R — необходимые библиотеки.
- FunPrepareData_VII.R — функции подготовки исходных данных.
- FunStacking.R — функции создания и тестирования ансамбля.
- Prepare.R — функции и скрипты подготовки исходных данных для обучаемых объединителей.
- Varb.R — скрипты базовой модели varb.
- model_DNN7_2.R — скрипты нейросети DNN(7-2).
- model_DNN_500.R — скрипты нейросети DNN500.
- SessionInfo_VII.txt — перечень пакетов, использованных в скриптах статьи.
Предупреждение: все права на данные материалы принадлежат MetaQuotes Ltd. Полная или частичная перепечатка запрещена.
Данная статья написана пользователем сайта и отражает его личную точку зрения. Компания MetaQuotes Ltd не несет ответственности за достоверность представленной информации, а также за возможные последствия использования описанных решений, стратегий или рекомендаций.
 Тестирование паттернов валютных пар: Использование и перспективы для реальной торговли. Часть IV
Тестирование паттернов валютных пар: Использование и перспективы для реальной торговли. Часть IV
 Визуализация результатов оптимизации по выбранному критерию
Визуализация результатов оптимизации по выбранному критерию
- Бесплатные приложения для трейдинга
- 8 000+ сигналов для копирования
- Экономические новости для анализа финансовых рынков
Вы принимаете политику сайта и условия использования
Vladimir Perervenko
Нейросети будем строить с помощью пакета keras/TensorFlow из Python
А чем не устраивает в R
keras: R Interface to 'Keras'
kerasR: R Interface to the Keras Deep Learning Library
Приветствую СС.
Так его и использую. keras for R. KerasR пробовал, но в связи с тем, что бекенд tensorflow очень быстро развивается( уже версия 1.8) , надежнее все таки использовать пакет, разрабатываемый и поддерживаемый коллективом Rstudio.
Удачи
У меня в ближайших планах обучение с подкреплением в тандеме с нейросетями.
Самое вкусное, интересно будет почитать ваше видение применения RL в задачах трейдинга, и реализации DQN, DDPG, A3C, TRPO
Обсуждение и вопросы по коду можно сделать в ветке
Удачи
Обсуждение и вопросы по коду можно сделать в ветке
Удачи
быстрая интеграция с python для глубокого обучения и mt5
https://github.com/TheSnowGuru/PyTrader-python-mt5-trading-api-connector