
种群优化算法:微人工免疫系统(Micro-AIS)
内容
1. 概述
免疫系统是一种了不起的机制,在保护我们的身体免受外部威胁方面发挥着重要作用。就像一个无形的盾牌,它可以对抗细菌、病毒和真菌,保持我们的身体健康。但是,如果我们能运用这种强大的机制来解决复杂的优化和学习问题呢?这正是人工免疫系统(AIS)优化方法中采用的方式。
人体免疫系统是一个由细胞、组织和器官组成的复杂系统,可保护身体免受感染、疾病和其它外部影响。它的工作原理是识别和破坏外来物质和微生物,如细菌、病毒、真菌、等等。
AIS 算法针对这些过程运用抗原(输入)、抗体(溶液)和杀伤细胞(优化过程)的概念进行建模,从而以最优方式解决问题。抗原代表需要优化的输入。抗体代表了这个问题的潜在解。杀手细胞是寻找优化问题的最佳解的优化过程。
人工免疫系统(AIS)优化方法于 1990 年代提出。针对这种方法的早期研究可以追溯到 1980 年代中期,Farmer、Packard、Perelson(1986)、以及 Bersini 和 Varela(1990)都为其做出了重大贡献。
从那时起,AIS 方法不断发展,并成为科学界一直研究的主题。已经提出了该方法的许多变体和修订版本,以及它在各种优化和学习问题中的应用。身体免疫系统在抵御感染和肿瘤等外部影响方面也起着重要作用。它有能力识别和检测异常,并攻击恶意个体,同时维护区分和存储有关它们的信息以备将来使用。
Micro-AIS(微免疫算法)是免疫系统(AIS)算法的修改版,旨在解决优化问题。它与传统 AIS 的不同之处在于,它使用更简单的免疫系统模型和更简单的免疫信息处理操作。
Micro-AIS 的基本思路是模拟免疫细胞试剂群的创造和进化。个体在解搜索空间中移动,彼此之间不交互,也不交换有关解的信息。同时,个体可以学习和适应不断变化的任务条件。
传统的 AIS 运用复杂的免疫系统模型,其中包括多种类型的细胞和分子,例如 B 细胞、T 细胞、抗体、细胞因子、等等。Micro-AIS 采用一个更简单的模型,该模型仅包含抗体。此外,Micro-AIS 采用更简单的免疫信息处理操作,例如突变和选择。
与 AIS 相比,Micro-AIM 的优势之一是其简单性和易于实现。然而,在某些情况下,更复杂的免疫系统模型也许能更有效地解决复杂问题。
总体而言,Micro-AIS 和传统 AIS 之间的选择取决于特定的应用环境。如果优化问题比较简单,需要快速解决,那么 Micro-AIS 也许是一个不错的选择。如果问题更复杂,并且需要更准确的解,那么传统的 AIS 也许是更合适的选择。
2. 算法
Micro-AIS 使用“亲和度”的概念来判定适用性,其是抗体和抗原之间相似性的度量。亲和度衡量抗体和抗原之间的相似程度。亲和度越高,抗体和抗原越相似。在 Micro-AIS 中,亲和度用于选择最佳抗体,并通过突变和交叉产生新抗体。具有较高亲和度的抗体更有可能被选中用于产生新的抗体。
亲和度可以定义为对象的特征向量,和分类器的权重向量之间的距离。距离越小,向量越相似,亲和度越高。通常,亲和度可以定义为两个浮点数之间距离的函数。可以根据 Micro-AIS 算法的具体应用和要求选择距离函数。例如,对于优化问题,距离通常定义为欧几里得距离、曼哈顿距离、或其它类型的距离。
然而,使用 Micro-AIS 的实验表明,在这种搜索策略中使用亲和度并不是最有效的方式,可直接用适应度函数值取代。
最初的 Micro-AIS 使用突变到基因的概率应用。适应度越大,突变的可能性就越低。由于效率低下,这种方式也不得不放弃,这很容易验证 — 只需添加几行代码即可。
Micro-AIS 伪代码:
- 创建抗体克隆群体,并在搜索空间中随机分布它们。抗体及其克隆代表了优化问题的潜在解。克隆是通过随机生成基因型创造得来,基因型决定了优化问题的参数值。
- 定义适应度,这是解的品质度量。适应度值可以通过估测每种抗体的目标函数来计算。
- 对于每种抗体,根据递减进度规则创建相应数量的克隆体:第一种抗体(就适应度而言)产生的克隆多于第二种,第二种抗体多于第三种,依此类推。因此,克隆的数量不是对应于适应度,而是对应于严格定义的进度规则。适应度更强的抗体比之适应度较差的抗体产生更多的克隆,且比例始终相同。
- 将突变应用于克隆基因。对于每个克隆,都会发生基因型突变,这令我们能够创建新的解,并探索优化问题的参数空间。
- 判定克隆的适应度。
- 在计算突变和适应度之后,将克隆群体添加到亲本抗体群体当中。
- 按适应度降序对群体(抗体 + 克隆)进行排序,以便随后在下一次迭代中选择创建新克隆群体的最佳解,从而实现后代克隆和亲本抗体之间的竞争。
- 从步骤 2 开始重复,直到满足停止准则。停止准则可以提前定义,例如达到某个适应度值、或达到最大迭代次数。
克隆中基因的突变是由方程遵照具有均匀分布的随机数生成:
X' = X + dist * rnd * k * mutation
其中:
- X' - 克隆基因的新值(坐标)
- X - 亲本抗体基因值
- dist - 亲本基因增量
- rnd - 随机数在 [-1.0;1.0] 范围内均匀分布
- k - 根据当前局面均匀递减比率
- mutation - 突变率,事实上 - 刻度增量因子(外部参数)
“k” 比率的计算方法如下:
k = (epochs - epochsCNT) / epochs
其中:
- epochs - 局数的限值
- epochsCNT - 局数(迭代)计数器
如果 “rnd” 大于 0,则 “dist” 增量大小是取从优化参数的最大值到 “X” 的距离;如果不是 0,则取从 “X” 到优化参数的最小值的距离。
因此,突变允许我们随机更改解的参数值,从而确保探索问题空间。递减系数 “k” 令我们能够降低以后迭代中参数发生太突然变化的可能性,从而提高算法到最优解的收敛性,并优化找到的坐标。
我们编写一个充当抗体的结构,S_Agent。该结构仅包含两个字段:
- c:个体坐标数组
- f:适应度指数
Init 方法初始化结构体的字段,更改 “c” 数组的大小,并将初始值 “-DBL_MAX” 分配给 “f” 字段。
//—————————————————————————————————————————————————————————————————————————————— struct S_Agent { void Init (int coords) { ArrayResize (c, coords); f = -DBL_MAX; } double c []; //coordinates double f; //fitness }; //——————————————————————————————————————————————————————————————————————————————
声明 C_AO_Micro_AIS 微免疫系统算法类,其定义各种字段和方法。
类字段:
- cB:最佳坐标数组。
- fB:最佳坐标的适应度指数。
- a:S_Agent 类型的个体数组。
- rangeMax:最大搜索范围值的数组。
- rangeMin:最小搜索范围值的数组。
- rangeStep: 搜索步骤数组。
“coords”、“popSize”、“minClonesNumber”、“cloneStep”、“mutation” 和 “epochs” 接受算法外部参数。
类方法:
- Init:按给定的值数初始化类的字段。
- Moving:移动个体。
- Revision:执行修订。
该类还定义了 “SeInDiSp”、“RNDfromCI” 和 “Sorting” 私密方法,分别用于常规化、随机数生成、和排序。
//—————————————————————————————————————————————————————————————————————————————— class C_AO_Micro_AIS { //---------------------------------------------------------------------------- public: double cB []; //best coordinates public: double fB; //FF of the best coordinates public: S_Agent a []; //agent public: double rangeMax []; //maximum search range public: double rangeMin []; //manimum search range public: double rangeStep []; //step search public: void Init (const int coordsP, //coordinates number const int popSizeP, //population size const int minClonesNumberP, //minimum number of clones const int cloneStepP, //clone step const double mutationP, //mutation const int epochP); //total epochs public: void Moving (); public: void Revision (); //---------------------------------------------------------------------------- private: int coords; //coordinates number private: int popSize; //population size private: int minClonesNumber; //minimum number of clones private: int cloneStep; //clone step private: double mutation; //mutation private: int epochs; //total epochs private: int epochsCNT; //epoch counter private: int parentsNumb; //number of parents private: bool revision; private: S_Agent parents []; //parents private: int ind []; private: double val []; private: S_Agent pTemp []; private: int cCnt []; //clone counters for each antibody private: double SeInDiSp (double In, double InMin, double InMax, double Step); private: double RNDfromCI (double min, double max); private: void Sorting (S_Agent &p [], int size); }; //——————————————————————————————————————————————————————————————————————————————
若要初始化类对象,需实现 Init 方法,以便采用指定数值初始化类字段。
在该方法开始时,调用 MathSrand 函数初始化随机数生成器,并调用 GetMicrosecondCount 函数重置其状态。
然后将 “-DBL_MAX” 和 “false” 分别分配给 “fB” 和 “revision” 变量。我们还采用方法的输入值初始化私密字段。
接下来,计算 “cCnt” 数组的值,为循环中的每种抗体存储克隆数。我们应用进度方程,其中进度的第一项是 “a1”,进度的差值是 “d”,进度的所有项之和是 “Sn”。进度值存储在 “cCnt” 数组之中。
然后,该方法判定 “parentsNumb” 变量的值,当作 “cCnt” 数组的大小。
接下来,更改数组大小:“ind”、“val”、“pTemp”、“a”、“parents”、“rangeMax”、“rangeMin”、“rangeStep” 和 “cB”。数组大小根据 “popSize” 和 “parentsNumb” 值进行设置。
接下来,在循环中,数组 “a” 的元素调用 S_Agent 类的 Init 方法进行初始化,并且 “parents” 数组的元素也调用 Init 方法进行初始化。
在方法结束时,“rangeMax”、“rangeMin”、“rangeStep” 和 “cB” 数组的大小会根据 “coords” 值而变化。
因此,Init 方法初始化 C_AO_Micro_AIS 类的字段,并计算 “cCnt” 数组的进度值。
//—————————————————————————————————————————————————————————————————————————————— void C_AO_Micro_AIS::Init (const int coordsP, //coordinates number const int popSizeP, //population size const int minClonesNumberP, //minimum number of clones const int cloneStepP, //clone step const double mutationP, //mutation const int epochP) //total epochs { MathSrand ((int)GetMicrosecondCount ()); // reset of the generator fB = -DBL_MAX; revision = false; coords = coordsP; popSize = popSizeP; minClonesNumber = minClonesNumberP; cloneStep = cloneStepP; mutation = mutationP; epochs = epochP; epochsCNT = 1; //---------------------------------------------------------------------------- int Sn = popSize; //sum int a1 = minClonesNumber; //first member of progression int d = cloneStep; //progression difference int an = 0; //n th member of progression, int Ssum = 0; ArrayResize (cCnt, 1); for (int n = 1;; n++) { an = a1 + (n - 1) * d; Ssum = n * (a1 + an) / 2; if (Ssum == Sn) { ArrayResize (cCnt, n); cCnt [n - 1] = an; break; } else { if (Ssum < Sn) { ArrayResize (cCnt, n); cCnt [n - 1] = an; } else { if (n == 1) { ArrayResize (cCnt, n); cCnt [n - 1] = Sn; break; } else { n--; an = a1 + (n - 1) * d; int diff = Sn - ((n) * (a1 + an) / 2); int index = ArraySize (cCnt) - 1; while (true) { if (index < 0) index = ArraySize (cCnt) - 1; cCnt [index]++; index--; diff--; if (diff <= 0) break; } break; } } } } parentsNumb = ArraySize (cCnt); ArrayReverse (cCnt, 0, WHOLE_ARRAY); ArrayResize (ind, popSize + parentsNumb); ArrayResize (val, popSize + parentsNumb); ArrayResize (pTemp, popSize + parentsNumb); ArrayResize (a, popSize); for (int i = 0; i < popSize; i++) a [i].Init (coords); ArrayResize (parents, popSize + parentsNumb); for (int i = 0; i < popSize + parentsNumb; i++) parents [i].Init (coords); ArrayResize (rangeMax, coords); ArrayResize (rangeMin, coords); ArrayResize (rangeStep, coords); ArrayResize (cB, coords); } //——————————————————————————————————————————————————————————————————————————————
调用 Moving 类方法,我们实现了在搜索空间中移动抗体。
在方法的开头,检查 “revision” 变量值。如果是 “false”,则抗体克隆要按照生成的均匀分布坐标被放置在搜索空间当中。
在群体中生成抗体克隆后,“revision” 变量设置为 “true”,方法退出。
如果 “revision” 变量的值不是 “false”,则执行下一个代码模块。
随后是一个嵌套的 “for” 循环,其迭代覆盖 “parentsNumb” 亲本个体的数量。在该循环中,将发生以下情况:
- 嵌套的 “for” 循环迭代覆盖给定 “cCnt[i]” 亲本抗体的克隆数量。
- 在该循环中,嵌套的 “for” 循环迭代覆盖所有个体 “c” 坐标。
- “a[indx].c[c]” 坐标值设置为等于 “parents[i].c[c]” 坐标的值。
随后执行以下代码模块:
- “k” 变量的值计算为 “epochs” 和 “epochsCNT” 之间的差值除以 “epochs”。
- “rnd” 随机数在 [-1.0;1.0] 范围内产生。
- 如果 “rnd” 大于 0.0,则 “dist” 变量值计算为 “rangeMax[c]” 和 “a[indx].c[c]” 之间的差值。否则,“dist” 等于 “a[indx].c[c]” 和 “rangeMin[c]” 之间的差值。
- “a[indx].c[c]” 按照方程式 “a[indx].c[c] + dist * rnd * k * mutation” 重新计算。此处 “mutation” 是突变比率。
- “a[indx].c[c]” 传递至 SeInDiSp 函数,该函数以“rangeStep[c]” 为步长,在从 “rangeMin[c]” 到 “rangeMax[c]” 的范围内对其进行常规化。
//—————————————————————————————————————————————————————————————————————————————— void C_AO_Micro_AIS::Moving () { //---------------------------------------------------------------------------- if (!revision) { for (int i = 0; i < popSize; i++) { for (int c = 0; c < coords; c++) { a [i].c [c] = RNDfromCI (rangeMin [c], rangeMax [c]); a [i].c [c] = SeInDiSp (a [i].c [c], rangeMin [c], rangeMax [c], rangeStep [c]); } } revision = true; return; } //---------------------------------------------------------------------------- int indx = 0; double min = DBL_MAX; double max = -DBL_MAX; double dist = 0.0; int cnt = 0; double rnd = 0.0; for (int i = 0; i < parentsNumb; i++) { for (int cl = 0; cl < cCnt [i]; cl++) { for (int c = 0; c < coords; c++) { a [indx].c [c] = parents [i].c [c]; //---------------------------------------------------------------------- double k = ((double)epochs - (double)epochsCNT) / (double)epochs; rnd = RNDfromCI (-1.0, 1.0); if (rnd > 0.0) dist = (rangeMax [c] - a [indx].c [c]); else dist = (a [indx].c [c] - rangeMin [c]); a [indx].c [c] = a [indx].c [c] + dist * rnd * k * mutation; a [indx].c [c] = SeInDiSp (a [indx].c [c], rangeMin [c], rangeMax [c], rangeStep [c]); } indx++; } } } //——————————————————————————————————————————————————————————————————————————————
最后,我们实现 Revision 方法。该方法对 Micro-AIS 算法中个体群体的当前状态执行审计。
第一个代码模块(由注释分隔),检查克隆群体的适应度值,并更新最佳全局解。
然后,在一个循环中,将克隆复制到载体末端的亲本抗原群体之中。
此后,采用 “parents” 和 “parentsNumb + popSize” 参数调用 Sorting 函数。该函数按适应度分数降序对 “parents” 数组进行排序。
在方法结束时,“epochsCNT” 变量递增,它负责计算算法中的局数。
因此,Revision 方法修改抗体(个体)群体的当前状态,找到具有最佳适应度指数值的个体,并更新亲本个体数组。
//—————————————————————————————————————————————————————————————————————————————— void C_AO_Micro_AIS::Revision () { //---------------------------------------------------------------------------- int indx = -1; for (int i = 0; i < popSize; i++) { if (a [i].f > fB) indx = i; } if (indx != -1) { fB = a [indx].f; ArrayCopy (cB, a [indx].c, 0, 0, WHOLE_ARRAY); } //---------------------------------------------------------------------------- for (int i = parentsNumb; i < parentsNumb + popSize; i++) { parents [i] = a [i - parentsNumb]; } Sorting (parents, parentsNumb + popSize); epochsCNT++; } //——————————————————————————————————————————————————————————————————————————————
3. 测试结果
测试台上 Micro-AIS 算法的打印输出:
C_AO_Micro_AIS:50:1:2:0.3
=============================
5 Hilly's; Func runs: 10000; result: 0.7954680903046107
25 Hilly's; Func runs: 10000; result: 0.5192246492565626
500 Hilly's; Func runs: 10000; result: 0.30860655744850657
=============================
5 Forest's; Func runs: 10000; result: 0.7295587642801589
25 Forest's; Func runs: 10000; result: 0.36878621216829993
500 Forest's; Func runs: 10000; result: 0.09398090798741626
=============================
5 Megacity's; Func runs: 10000; result: 0.37666666666666665
25 Megacity's; Func runs: 10000; result: 0.15866666666666668
500 Megacity's; Func runs: 10000; result: 0.028016666666666672
=============================
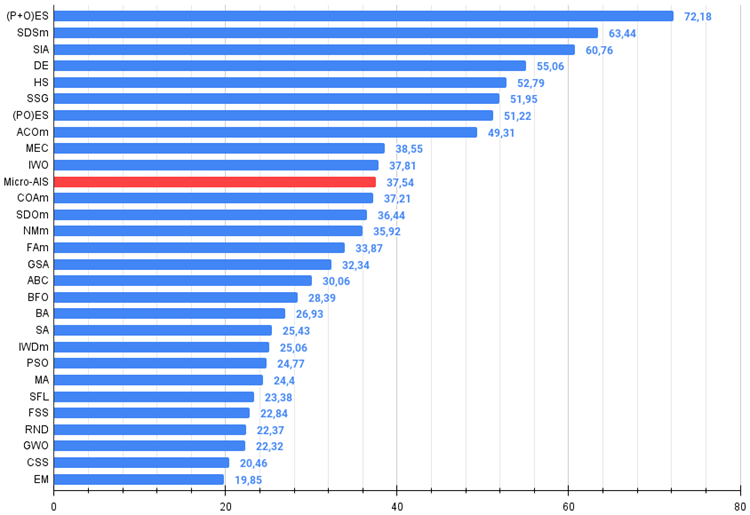
All score: 3.37898 (37.54%)
从上一篇文章开始,我已转向了测试结果的绝对值。故此,现在很容易在表格中导航,并比较不同算法的结果。37.54% 不是一个杰出的结果,但它仍然处于表格上半部分的位置。
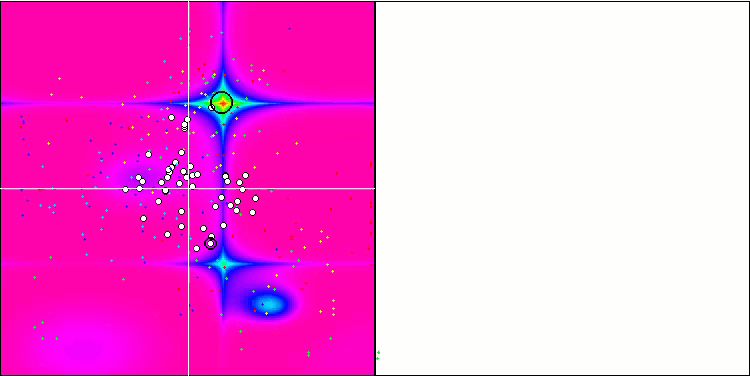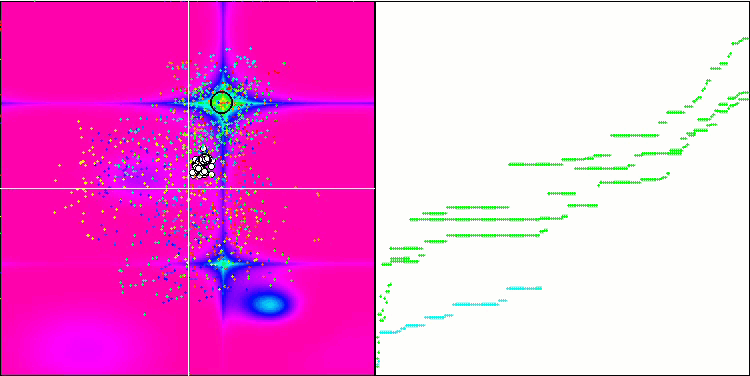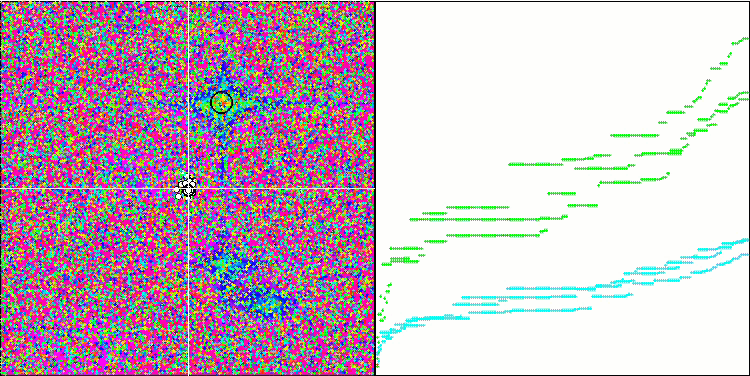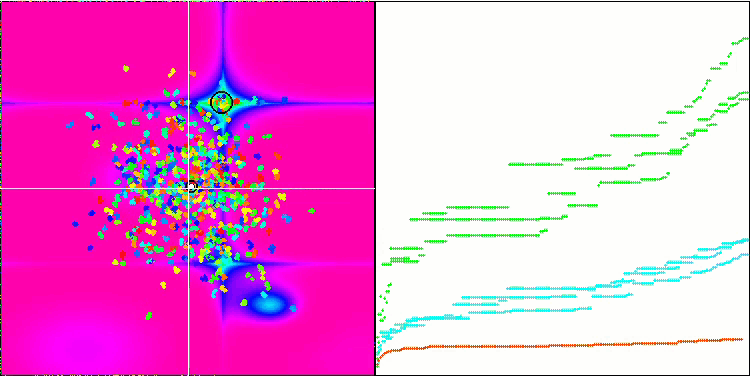
优化过程的可视化表明,该算法坚持不懈地探索显著的局部极值,从而达成更好的解。这导致个体集中在空间的不同区域。收敛图显示了异常行为。典型情况,在所研究的算法中,在迭代的前半部分,收敛性急剧增加,然后收敛率逐渐降低。不过,在该算法中,收敛图是 S-形的。仅在前 10-20% 的迭代中观察到收敛性急剧增加,然后收敛速度降低,但在接近优化结束时,可以再次看到收敛性显著加速。
收敛图的这种行为可以根据线性定律缩小突变期间增量范围的策略来解释。不过,由于算法关于测试函数表面的信息“积累”不均匀,因此根据线性定律不会发生收敛。缩小突变范围只有在接近优化结束时才会开始发挥更明显的作用。用非线性定律的其它变体代替缩小突变范围的线性定律并没有导致收敛性的改善,但也许研究人员在选择收窄范围定律的其它变体时有建设性想象的空间。
收敛图还有留有很多遐想余地,不过,它的出现给出了希望,即搜索策略有进一步改进的潜力。
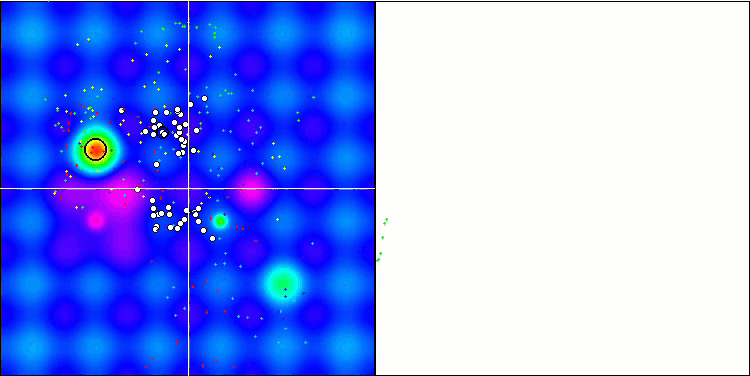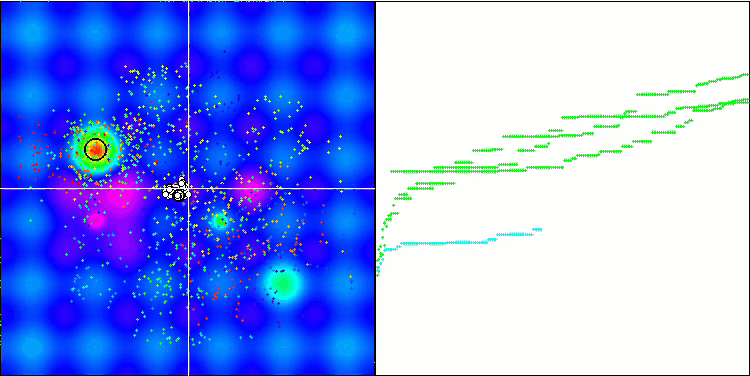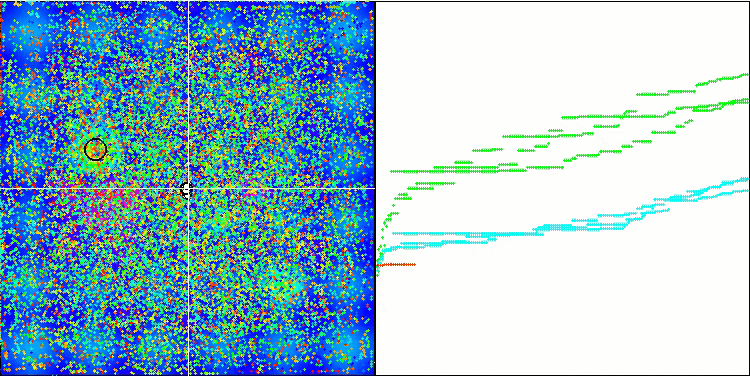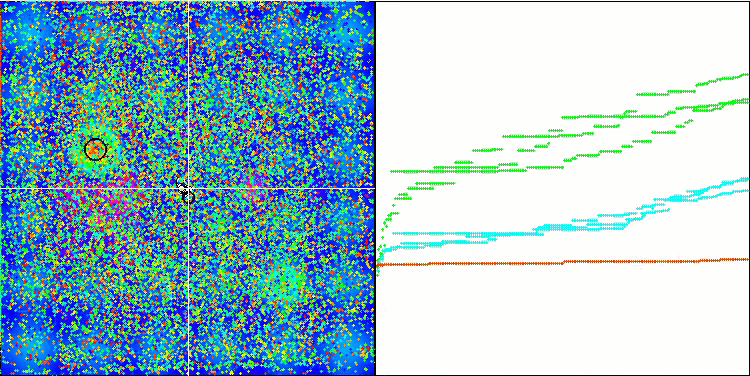
Micro-AIS 依据 Hilly 测试函数
Micro-AIS 依据 Forest 测试函数
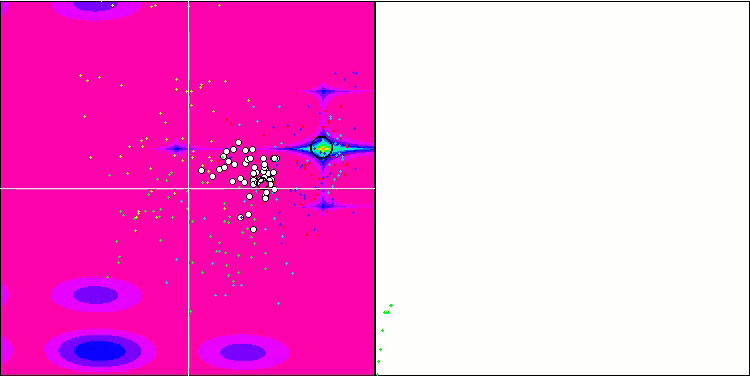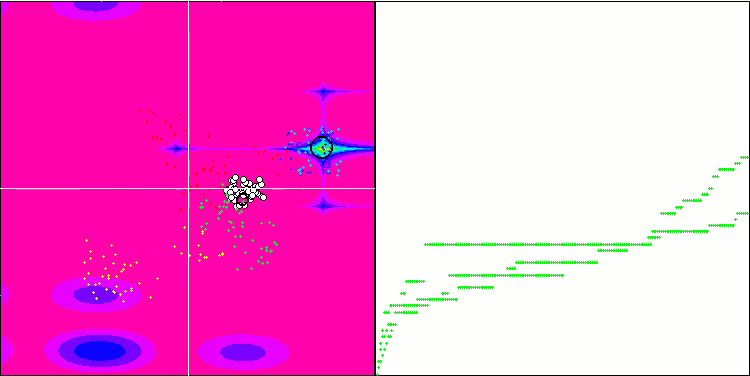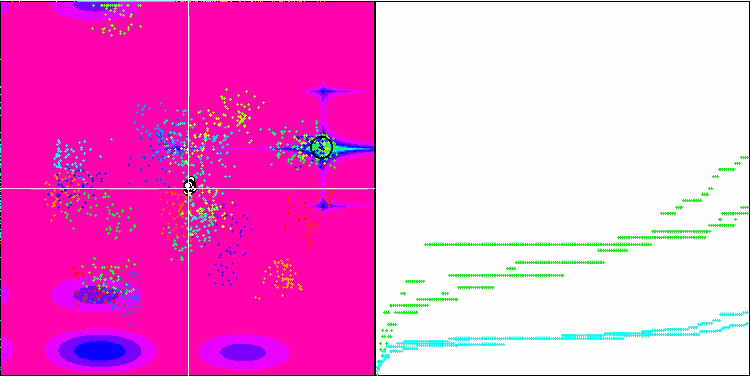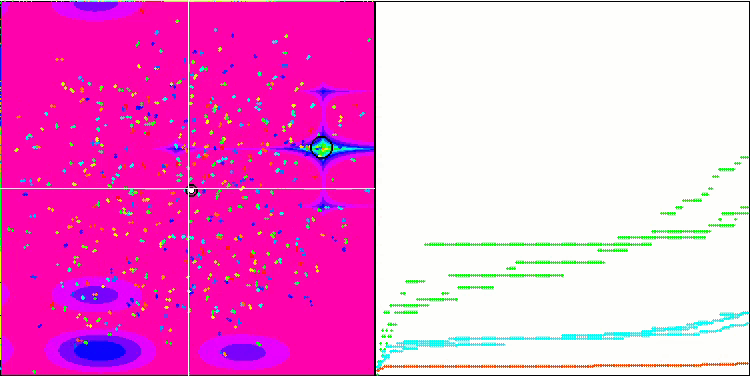
Micro-AIS 依据 Megacity 测试函数
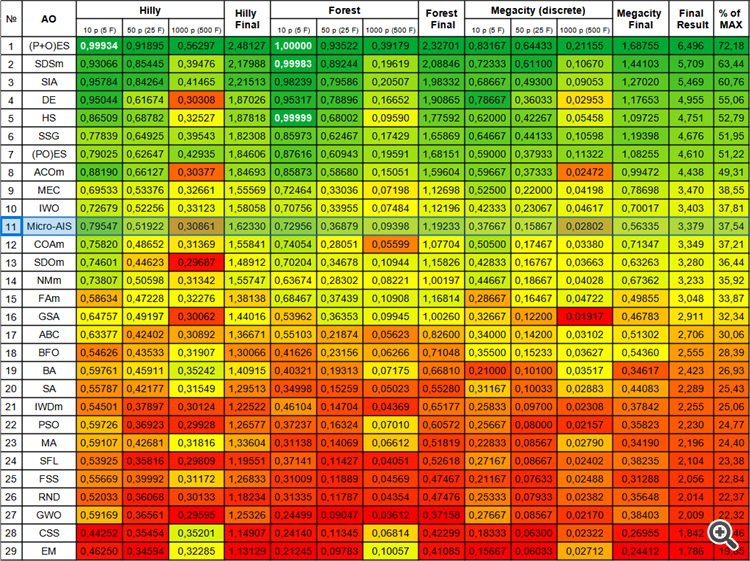
Micro-AIS 算法排在评级表的第 11 行,占据了应有的位置,领先于布谷鸟优化算法、人工蜂群和模拟退火等知名和流行的算法。这表明该算法在解决复杂优化问题方面的效率。
然而,根据颜色表,我们可以注意到具有大量变量的函数的性能下降,这表明该算法的扩展能力较弱。这可能是由于 Micro-AIS 使用更简单的免疫系统模型,以及更简单的免疫信息处理操作,这可能会限制其在高维空间中找到最佳解的能力。
然而,这并不意味着 Micro-AIS 不能用于解决具有大量变量的问题。也许,可以通过修改算法,或将其与其它优化方法结合运用来提高其性能。
| # | AO | 说明 | Hilly | Hilly 最终 | Forest | Forest 最终 | Megacity (离散) | Megacity 最终 | 最终结果 | 最大 % | ||||||
| 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | ||||||||
| 1 | (P+O)ES | (P+O) 进化策略 | 0.99934 | 0.91895 | 0.56297 | 2.48127 | 1.00000 | 0.93522 | 0.39179 | 2.32701 | 0.83167 | 0.64433 | 0.21155 | 1.68755 | 6.496 | 72.18 |
| 2 | SDSm | 随机扩散搜索 M | 0.93066 | 0.85445 | 0.39476 | 2.17988 | 0.99983 | 0.89244 | 0.19619 | 2.08846 | 0.72333 | 0.61100 | 0.10670 | 1.44103 | 5.709 | 63.44 |
| 3 | SIA | 模拟各向同性退火 | 0.95784 | 0.84264 | 0.41465 | 2.21513 | 0.98239 | 0.79586 | 0.20507 | 1.98332 | 0.68667 | 0.49300 | 0.09053 | 1.27020 | 5.469 | 60.76 |
| 4 | DE | 差分进化 | 0.95044 | 0.61674 | 0.30308 | 1.87026 | 0.95317 | 0.78896 | 0.16652 | 1.90865 | 0.78667 | 0.36033 | 0.02953 | 1.17653 | 4.955 | 55.06 |
| 5 | HS | 谐声搜索 | 0.86509 | 0.68782 | 0.32527 | 1.87818 | 0.99999 | 0.68002 | 0.09590 | 1.77592 | 0.62000 | 0.42267 | 0.05458 | 1.09725 | 4.751 | 52.79 |
| 6 | SSG | 树苗播种和生长 | 0.77839 | 0.64925 | 0.39543 | 1.82308 | 0.85973 | 0.62467 | 0.17429 | 1.65869 | 0.64667 | 0.44133 | 0.10598 | 1.19398 | 4.676 | 51.95 |
| 7 | (PO)ES | (PO) 进化策略 | 0.79025 | 0.62647 | 0.42935 | 1.84606 | 0.87616 | 0.60943 | 0.19591 | 1.68151 | 0.59000 | 0.37933 | 0.11322 | 1.08255 | 4.610 | 51.22 |
| 8 | ACOm | 蚁群优化 M | 0.88190 | 0.66127 | 0.30377 | 1.84693 | 0.85873 | 0.58680 | 0.15051 | 1.59604 | 0.59667 | 0.37333 | 0.02472 | 0.99472 | 4.438 | 49.31 |
| 9 | MEC | 心智进化计算 | 0.69533 | 0.53376 | 0.32661 | 1.55569 | 0.72464 | 0.33036 | 0.07198 | 1.12698 | 0.52500 | 0.22000 | 0.04198 | 0.78698 | 3.470 | 38.55 |
| 10 | IWO | 入侵性杂草优化 | 0.72679 | 0.52256 | 0.33123 | 1.58058 | 0.70756 | 0.33955 | 0.07484 | 1.12196 | 0.42333 | 0.23067 | 0.04617 | 0.70017 | 3.403 | 37.81 |
| 11 | Micro-AIS | 微人工免疫系统 | 0.79547 | 0.51922 | 0.30861 | 1.62330 | 0.72956 | 0.36879 | 0.09398 | 1.19233 | 0.37667 | 0.15867 | 0.02802 | 0.56335 | 3.379 | 37.54 |
| 12 | COAm | 布谷鸟优化算法 M | 0.75820 | 0.48652 | 0.31369 | 1.55841 | 0.74054 | 0.28051 | 0.05599 | 1.07704 | 0.50500 | 0.17467 | 0.03380 | 0.71347 | 3.349 | 37.21 |
| 13 | SDOm | 螺旋动力学优化 M | 0.74601 | 0.44623 | 0.29687 | 1.48912 | 0.70204 | 0.34678 | 0.10944 | 1.15826 | 0.42833 | 0.16767 | 0.03663 | 0.63263 | 3.280 | 36.44 |
| 14 | NMm | Nelder-Mead 方法 M | 0.73807 | 0.50598 | 0.31342 | 1.55747 | 0.63674 | 0.28302 | 0.08221 | 1.00197 | 0.44667 | 0.18667 | 0.04028 | 0.67362 | 3.233 | 35.92 |
| 15 | FAm | 萤火虫算法 M | 0.58634 | 0.47228 | 0.32276 | 1.38138 | 0.68467 | 0.37439 | 0.10908 | 1.16814 | 0.28667 | 0.16467 | 0.04722 | 0.49855 | 3.048 | 33.87 |
| 16 | GSA | 重力搜索算法 | 0.64757 | 0.49197 | 0.30062 | 1.44016 | 0.53962 | 0.36353 | 0.09945 | 1.00260 | 0.32667 | 0.12200 | 0.01917 | 0.46783 | 2.911 | 32.34 |
| 17 | ABC | 人工蜂群 | 0.63377 | 0.42402 | 0.30892 | 1.36671 | 0.55103 | 0.21874 | 0.05623 | 0.82600 | 0.34000 | 0.14200 | 0.03102 | 0.51302 | 2.706 | 30.06 |
| 18 | BFO | 细菌觅食优化 | 0.54626 | 0.43533 | 0.31907 | 1.30066 | 0.41626 | 0.23156 | 0.06266 | 0.71048 | 0.35500 | 0.15233 | 0.03627 | 0.54360 | 2.555 | 28.39 |
| 19 | BA | 蝙蝠算法 | 0.59761 | 0.45911 | 0.35242 | 1.40915 | 0.40321 | 0.19313 | 0.07175 | 0.66810 | 0.21000 | 0.10100 | 0.03517 | 0.34617 | 2.423 | 26.93 |
| 20 | SA | 模拟退火 | 0.55787 | 0.42177 | 0.31549 | 1.29513 | 0.34998 | 0.15259 | 0.05023 | 0.55280 | 0.31167 | 0.10033 | 0.02883 | 0.44083 | 2.289 | 25.43 |
| 21 | IWDm | 智能水滴 M | 0.54501 | 0.37897 | 0.30124 | 1.22522 | 0.46104 | 0.14704 | 0.04369 | 0.65177 | 0.25833 | 0.09700 | 0.02308 | 0.37842 | 2.255 | 25.06 |
| 22 | PSO | 粒子群优化 | 0.59726 | 0.36923 | 0.29928 | 1.26577 | 0.37237 | 0.16324 | 0.07010 | 0.60572 | 0.25667 | 0.08000 | 0.02157 | 0.35823 | 2.230 | 24.77 |
| 23 | MA | 猴子算法 | 0.59107 | 0.42681 | 0.31816 | 1.33604 | 0.31138 | 0.14069 | 0.06612 | 0.51819 | 0.22833 | 0.08567 | 0.02790 | 0.34190 | 2.196 | 24.40 |
| 24 | SFL | 蛙跳 | 0.53925 | 0.35816 | 0.29809 | 1.19551 | 0.37141 | 0.11427 | 0.04051 | 0.52618 | 0.27167 | 0.08667 | 0.02402 | 0.38235 | 2.104 | 23.38 |
| 25 | FSS | 鱼群搜索 | 0.55669 | 0.39992 | 0.31172 | 1.26833 | 0.31009 | 0.11889 | 0.04569 | 0.47467 | 0.21167 | 0.07633 | 0.02488 | 0.31288 | 2.056 | 22.84 |
| 26 | RND | 随机 | 0.52033 | 0.36068 | 0.30133 | 1.18234 | 0.31335 | 0.11787 | 0.04354 | 0.47476 | 0.25333 | 0.07933 | 0.02382 | 0.35648 | 2.014 | 22.37 |
| 27 | GWO | 灰狼优化器 | 0.59169 | 0.36561 | 0.29595 | 1.25326 | 0.24499 | 0.09047 | 0.03612 | 0.37158 | 0.27667 | 0.08567 | 0.02170 | 0.38403 | 2.009 | 22.32 |
| 28 | CSS | 收费系统搜索 | 0.44252 | 0.35454 | 0.35201 | 1.14907 | 0.24140 | 0.11345 | 0.06814 | 0.42299 | 0.18333 | 0.06300 | 0.02322 | 0.26955 | 1.842 | 20.46 |
| 29 | EM | 类电磁算法 | 0.46250 | 0.34594 | 0.32285 | 1.13129 | 0.21245 | 0.09783 | 0.10057 | 0.41085 | 0.15667 | 0.06033 | 0.02712 | 0.24412 | 1.786 | 19.85 |
总结
微人工免疫系统(Micro-AIS)优化方法是一种有趣且有前途的方法,可以根据免疫系统功能原理解决优化问题。它允许使用免疫系统的强大机制来解决复杂的优化和学习问题。
Micro-AIS 的优点包括少量的外部参数和简单的算法实现。这令它在实际运用于任务中非常有吸引力。
不过,Micro-AIS 算法也有缺点。它容易卡在局部极值,收敛性低。甚至,算法在具有大量变量的函数上性能下降,这表明其扩展能力较弱。
然而,Micro-AIS 是一种很有前途的优化方法,可以通过修改算法、或与其它优化方法相结合来改进。总体而言,使用微人工免疫系统的优化方法是对优化领域的重要贡献,可以成为解决机器学习、人工智能、生物信息学等各个领域复杂问题的实用工具。
该算法给人的印象是一种 “模板”,可以使用各种方法构建,并扩展搜索能力。这得益于简单的架构,这为试验这种非常有趣的算法留下了非常大的空间。
图例 1. 算法的颜色渐变是依据相关测试
图例 2. 算法测试结果的直方图(标尺从 0 到 100,越多越好,
其中 100 是最大可能的理论结果(存档提供了一个用于计算评级表格的脚本)。
Micro-AIS 算法的优缺点:
- 少量外部参数。
- 简单的算法实现。
- 容易卡住。
- 低收敛性。
本文附有一个存档,其中包含前几篇文章中讲述的算法代码的当前更新版本。文章作者不对规范算法讲述的绝对准确性负责。它们当中进行了多处修改,从而提升搜索能力。文章中表述的结论和论断是基于实验的结果。
本文由MetaQuotes Ltd译自俄文
原文地址: https://www.mql5.com/ru/articles/13951
注意: MetaQuotes Ltd.将保留所有关于这些材料的权利。全部或部分复制或者转载这些材料将被禁止。
本文由网站的一位用户撰写,反映了他们的个人观点。MetaQuotes Ltd 不对所提供信息的准确性负责,也不对因使用所述解决方案、策略或建议而产生的任何后果负责。
 数据科学和机器学习(第 18 部分):掌握市场复杂性博弈,截断型 SVD 对比 NMF
数据科学和机器学习(第 18 部分):掌握市场复杂性博弈,截断型 SVD 对比 NMF
 开发多币种 EA 交易(第 3 部分):架构修改
开发多币种 EA 交易(第 3 部分):架构修改
 种群优化算法:细菌觅食优化 — 遗传算法(BFO-GA)
种群优化算法:细菌觅食优化 — 遗传算法(BFO-GA)

显然,你遇到过这种情况。如果你在该示例中保留六个刻度线,它就会起作用。因此,您现在可以调整本系列文章的算法,只需使用更简单的示例即可。
这是不现实的简化。据我所知,运行次数是有限制的,而不是参数数量。
我从测试拟合函数的设置中提取参数,是为了在比较算法时,能或多或少了解优化空间的实际大小。;-).
这是不现实的简化。据我所知,运行次数是有限制的,而不是参数数量。
我从测试拟合函数的设置中提取参数,是为了在比较算法时,或多或少了解优化空间的实际大小。;-).
老实说,我不明白为什么 MQ 会从字面上理解这个步骤。这是最小离散化的程度。它与优化算法的关系几乎为零。
坦率地说,我不明白 MQ 为什么要从字面上理解 "步骤"。它是最小离散度的级别。它与优化算法几乎毫无关系。
在特征的真实表示中,是的,而且不是几乎,而是零关系。
对于二元 GA 来说,这方面的情况要复杂一些,有一些细微差别。
我之前说过,不能将文章中的算法与标准 GA 进行正面比较,这是不对的。标准 GA 是一种复杂的算法,它考虑到了许多细微差别,这些细微差别可以在大多数用户的个人电脑上工作:工作速度、新解决方案的独特性、节省内存。
据我所知,自定义优化只在一个内核的终端图上完成,而我在测试器中讨论的是多线程优化(对于我在文章 中描述的粒子群算法,对于大多数其他算法,通过类比也应该是可行的,因为通常有一个将任务划分为代理组的原则)。但是,测试器只支持最原始的示例(我给出了上面的测试),这就把这个想法扼杀在了萌芽状态。
分享链接。这个?
分享链接。这个?
是的。