Discusión sobre el artículo "Tercera generación de neuroredes: "Neuroredes profundas"" - página 2
Está perdiendo oportunidades comerciales:
- Aplicaciones de trading gratuitas
- 8 000+ señales para copiar
- Noticias económicas para analizar los mercados financieros
Registro
Entrada
Usted acepta la política del sitio web y las condiciones de uso
Si no tiene cuenta de usuario, regístrese
No es una pregunta para mí. ¿Eso es todo lo que tienes que decir sobre el artículo?
¿Qué pasa con el artículo? Es una típica reescritura. Es lo mismo en otras fuentes, sólo que con palabras ligeramente diferentes. Incluso las fotos son las mismas. No vi nada nuevo, es decir, de autor.
Quería probar los ejemplos, pero es un fastidio. La sección es para MQL5, pero los ejemplos son para MQL4.
vlad1949
Querido Vlad
Mirado a través de los archivos, usted tiene bastante viejo R documentación. Sería bueno cambiar a las copias adjuntas.
vlad1949
Querido Vlad
¿Por qué no se ejecuta en el probador?
Tengo todo funciona sin problemas. Pero el esquema es sin indicador: el Asesor Experto se comunica directamente con R.
Jeffrey Hinton, inventor de las redes profundas: "Las redes profundas sólo son aplicables a datos en los que la relación señal-ruido es grande. Las series financieras son tan ruidosas que las redes profundas no son aplicables. Lo hemos intentado y no ha habido suerte".
Escuche sus conferencias en YouTube.
Jeffrey Hinton, inventor de las redes profundas: "Las redes profundas sólo son aplicables a datos en los que la relación señal-ruido es grande. Las series financieras son tan ruidosas que las redes profundas no son aplicables. Lo hemos intentado y no ha habido suerte".
Escucha sus conferencias en youtube.
Teniendo en cuenta su post en el hilo paralelo.
El ruido se entiende de forma diferente en tareas de clasificación que en radioingeniería. Un predictor se considera ruidoso si está débilmente relacionado (tiene débil poder predictivo) para la variable objetivo. Un significado completamente distinto. Hay que buscar predictores que tengan poder predictivo para diferentes clases de la variable objetivo.
Yo entiendo el ruido de forma parecida. Las series financieras dependen de un gran número de predictores, la mayoría de los cuales nos son desconocidos y que introducen ese "ruido" en las series. Utilizando únicamente los predictores disponibles públicamente, somos incapaces de predecir la variable objetivo, independientemente de las redes o métodos que utilicemos.
vlad1949
Querido Vlad
¿Por qué no se ejecuta en el probador?
Tengo todo funciona sin problemas. Cierto el esquema sin indicador: el asesor se comunica directamente con R.
llllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllll
Buenas tardes SanSanych.
Pues la idea principal es hacer multidivisa con varios indicadores.
De lo contrario, por supuesto, usted puede embalar todo en un Asesor Experto.
Pero si la formación, pruebas y optimización se llevará a cabo sobre la marcha, sin interrumpir el comercio, entonces la variante con un Asesor Experto será un poco más difícil de implementar.
Buena suerte
PS. ¿Cuál es el resultado de las pruebas?
Saludos SanSanych.
Aquí tienes algunos ejemplos para determinar el número óptimo de clusters que encontré en algún foro anglosajón. No he podido utilizarlos todos con mis datos. Muy interesante el paquete 11 "clusterSim".
--------------------------------------------------------------------------------------
En el próximo post cálculos con mis datos
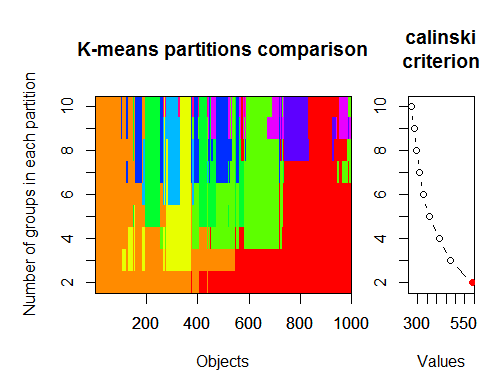
El número óptimo de conglomerados puede determinarse mediante varios paquetes y utilizando más de 30 criterios de optimalidad. Según mis observaciones, el criterio más utilizado es el de Calinsky.
Tomemos los datos brutos de nuestro conjunto del indicador dt . Contiene 17 predictores, el objetivo y y el cuerpo de la vela z.
En las últimas versiones de los paquetes "magrittr" y "dplyr " hay muchas nuevas características agradables, una de ellas es 'pipe' - %>%. Es muy conveniente cuando no necesitas guardar resultados intermedios. Preparemos los datos iniciales para el clustering. Tomemos la matriz inicial dt, seleccionemos las últimas 1000 filas de ella, y luego seleccionemos 17 columnas de nuestras variables de ellas. Obtenemos una notación más clara, nada más.
1.
2. Criterio de Calinsky: Otro enfoque para diagnosticar cuántos conglomerados se ajustan a los datos. En este caso
probamos de 1 a 10 grupos.
3. Determinar el modelo y el número de grupos óptimos según el criterio de información bayesiano para la maximización de expectativas, inicializado por la agrupación jerárquica para modelos de mezcla gaussiana parametrizados.Modelos de mezcla gaussiana