Diskussion zum Artikel "Neuronale Netzwerke der dritten Generation: Tiefe Netzwerke" - Seite 2
Sie verpassen Handelsmöglichkeiten:
- Freie Handelsapplikationen
- Über 8.000 Signale zum Kopieren
- Wirtschaftsnachrichten für die Lage an den Finanzmärkte
Registrierung
Einloggen
Sie stimmen der Website-Richtlinie und den Nutzungsbedingungen zu.
Wenn Sie kein Benutzerkonto haben, registrieren Sie sich
Das ist keine Frage für mich. Ist das alles, was Sie über den Artikel zu sagen haben?
Was ist mit dem Artikel? Es ist eine typische Umschreibung. Es ist das Gleiche in anderen Quellen, nur in etwas anderen Worten. Sogar die Bilder sind die gleichen. Ich habe nichts Neues, d. h. nichts Autorisches, gefunden.
Ich wollte die Beispiele ausprobieren, aber das ist schade. Der Abschnitt ist für MQL5, aber die Beispiele sind für MQL4.
vlad1949
Lieber Vlad!
Ich habe in den Archiven nachgesehen, du hast eine ziemlich alte R-Dokumentation. Es wäre gut, auf die beigefügten Kopien zu wechseln.
vlad1949
Lieber Vlad!
Warum hast du im Tester nicht funktioniert?
Ich habe alles funktioniert ohne Probleme. Aber das System ist ohne einen Indikator: der Expert Advisor kommuniziert direkt mit R.
Jeffrey Hinton, Erfinder der tiefen Netze: "Tiefe Netze sind nur auf Daten anwendbar, bei denen das Signal-Rausch-Verhältnis groß ist. Finanzreihen sind so verrauscht, dass tiefe Netze nicht anwendbar sind. Wir haben es versucht und hatten kein Glück."
Hören Sie sich seine Vorträge auf YouTube an.
Jeffrey Hinton, Erfinder der tiefen Netze: "Tiefe Netze sind nur auf Daten anwendbar, bei denen das Signal-Rausch-Verhältnis groß ist. Finanzreihen sind so verrauscht, dass tiefe Netze nicht anwendbar sind. Wir haben es versucht und hatten kein Glück."
Hören Sie sich seine Vorträge auf youtube an.
In Anbetracht Ihres Beitrags im parallelen Thread.
Rauschen wird bei Klassifizierungsaufgaben anders verstanden als in der Funktechnik. Ein Prädiktor gilt als verrauscht, wenn er nur schwach mit der Zielvariablen zusammenhängt (eine schwache Vorhersagekraft hat). Eine völlig andere Bedeutung. Man sollte nach Prädiktoren suchen, die eine Vorhersagekraft für verschiedene Klassen der Zielvariablen haben.
Ich habe ein ähnliches Verständnis von Rauschen. Finanzreihen hängen von einer großen Anzahl von Prädiktoren ab, von denen die meisten uns unbekannt sind und die dieses "Rauschen" in die Reihen einbringen. Wenn wir nur die öffentlich verfügbaren Prädiktoren verwenden, sind wir nicht in der Lage, die Zielvariable vorherzusagen, egal welche Netze oder Methoden wir verwenden.
vlad1949
Lieber Vlad!
Warum hast du im Tester nicht funktioniert?
Ich habe alles funktioniert ohne Probleme. Richtig das Schema ohne Indikator: der Advisor kommuniziert direkt mit R.
llllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllll
Guten Tag SanSanych.
Also die Hauptidee ist es, Multicurrency mit mehreren Indikatoren zu machen.
Ansonsten kann man natürlich alles in einen Expert Advisor packen.
Aber wenn das Training, Testen und Optimieren on the fly erfolgen soll, ohne den Handel zu unterbrechen, dann wird die Variante mit einem Expert Advisor etwas schwieriger zu realisieren sein.
Viel Erfolg!
PS. Was ist das Ergebnis der Tests?
Grüße SanSanych.
Hier sind einige Beispiele zur Bestimmung der optimalen Anzahl von Clustern, die ich in einem englischsprachigen Forum gefunden habe. Ich konnte nicht alle davon mit meinen Daten verwenden. Sehr interessant 11 Paket "clusterSim".
--------------------------------------------------------------------------------------
Im nächsten Beitrag Berechnungen mit meinen Daten
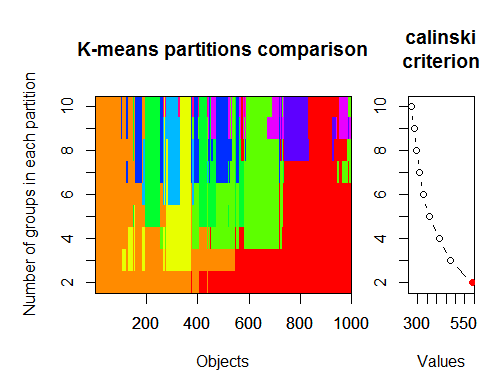
Die optimale Anzahl von Clustern kann mit mehreren Paketen und unter Verwendung von mehr als 30 Optimalitätskriterien bestimmt werden. Nach meinen Beobachtungen istdas am häufigsten verwendete Kriterium das Calinsky-Kriterium.
Nehmen wir die Rohdaten aus unserem Satz des Indikators dt . Er enthält 17 Prädiktoren, das Ziel y und den Candlestick-Körper z.
In den neuesten Versionen der Pakete "magrittr" und "dplyr" gibt es viele neue nette Funktionen, eine davon ist "pipe" - %>%. Sie ist sehr praktisch, wenn Sie Zwischenergebnisse nicht speichern müssen. Bereiten wir die Ausgangsdaten für das Clustering vor. Nehmen Sie die Ausgangsmatrix dt, wählen Sie die letzten 1000 Zeilen aus und wählen Sie dann 17 Spalten unserer Variablen aus. Wir erhalten eine klarere Notation, mehr nicht.
1.
2. Calinsky-Kriterium: Ein weiterer Ansatz, um festzustellen, wie viele Cluster zu den Daten passen. In diesem Fall
versuchen wir 1 bis 10 Gruppen.
3. das optimale Modell und die Anzahl der Cluster nach dem Bayes'schen Informationskriterium für die Erwartungsmaximierung bestimmen, initialisiert durch hierarchisches Clustering für parametrisierte Gaußsche Mischmodelle.Gaußsche Mischungsmodelle