Discussione sull’articolo "Reti neurali di terza generazione: Reti profonde" - pagina 2
Ti stai perdendo delle opportunità di trading:
- App di trading gratuite
- Oltre 8.000 segnali per il copy trading
- Notizie economiche per esplorare i mercati finanziari
Registrazione
Accedi
Accetti la politica del sito e le condizioni d’uso
Se non hai un account, registrati
Non è una domanda per me. È tutto quello che hai da dire sull'articolo?
Cosa c'è da dire sull'articolo? È una tipica riscrittura. È la stessa cosa in altre fonti, solo con parole leggermente diverse. Anche le immagini sono le stesse. Non ho visto nulla di nuovo, cioè di autoriale.
Volevo provare gli esempi, ma è un peccato. La sezione è per MQL5, ma gli esempi sono per MQL4.
vlad1949
Caro Vlad!
Ho guardato negli archivi, hai una documentazione R piuttosto vecchia. Sarebbe opportuno passare alle copie allegate.
vlad1949
Caro Vlad!
Perché non è riuscito a funzionare nel tester?
Ho tutto funziona senza problemi. Ma lo schema è senza indicatore: l'Expert Advisor comunica direttamente con R.
Jeffrey Hinton, inventore delle reti profonde: "Le reti profonde sono applicabili solo a dati in cui il rapporto segnale/rumore è elevato. Le serie finanziarie sono così rumorose che le reti profonde non sono applicabili. Ci abbiamo provato e non abbiamo avuto fortuna".
Ascoltate le sue conferenze su YouTube.
Jeffrey Hinton, inventore delle reti profonde: "Le reti profonde sono applicabili solo a dati in cui il rapporto segnale/rumore è elevato. Le serie finanziarie sono così rumorose che le reti profonde non sono applicabili. Ci abbiamo provato e non abbiamo avuto fortuna".
Ascoltate le sue lezioni su youtube.
Considerando il suo post nel thread parallelo.
Il rumore è inteso in modo diverso nei compiti di classificazione rispetto all'ingegneria radiofonica. Un predittore è considerato rumoroso se è debolmente correlato (ha un debole potere predittivo) alla variabile target. Un significato completamente diverso. Si dovrebbero cercare predittori che abbiano potere predittivo per diverse classi della variabile target.
Ho una concezione simile del rumore. Le serie finanziarie dipendono da un gran numero di predittori, la maggior parte dei quali è sconosciuta e introduce il "rumore" nelle serie. Utilizzando solo i predittori pubblicamente disponibili, non siamo in grado di prevedere la variabile target, indipendentemente dalle reti o dai metodi utilizzati.
vlad1949
Caro Vlad!
Perché non è riuscito a funzionare nel tester?
Ho tutto funziona senza problemi. Vero lo schema senza indicatore: il consulente comunica direttamente con R.
llllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllll
Buon pomeriggio SanSanych.
Quindi l'idea principale è quella di fare multicurrency con diversi indicatori.
Altrimenti, ovviamente, è possibile racchiudere tutto in un Expert Advisor.
Ma se la formazione, i test e l'ottimizzazione saranno effettuati al volo, senza interrompere il trading, allora la variante con un Expert Advisor sarà un po' più difficile da implementare.
Buona fortuna
PS. Qual è il risultato dei test?
Saluti SanSanych.
Ecco alcuni esempi di determinazione del numero ottimale di cluster che ho trovato su alcuni forum di lingua inglese. Non sono riuscito a utilizzarli tutti con i miei dati. Molto interessante il pacchetto 11 "clusterSim".
--------------------------------------------------------------------------------------
Nel prossimo post i calcoli con i miei dati
Il numero ottimale di cluster può essere determinato da diversi pacchetti e utilizzando più di 30 criteri di ottimizzazione. Secondo le mie osservazioni, il criterio più utilizzato è il criterio di Calinsky.
Prendiamo i dati grezzi del nostro set dall'indicatore dt . Esso contiene 17 predittori, il target y e il corpo della candela z.
Nelle ultime versioni dei pacchetti "magrittr" e "dplyr" ci sono molte nuove funzionalità, una delle quali è la "pipe" - %>%. È molto comodo quando non è necessario salvare i risultati intermedi. Prepariamo i dati iniziali per il clustering. Prendiamo la matrice iniziale dt, selezioniamo le ultime 1000 righe e poi selezioniamo le 17 colonne delle nostre variabili. Otteniamo una notazione più chiara, niente di più.
1.
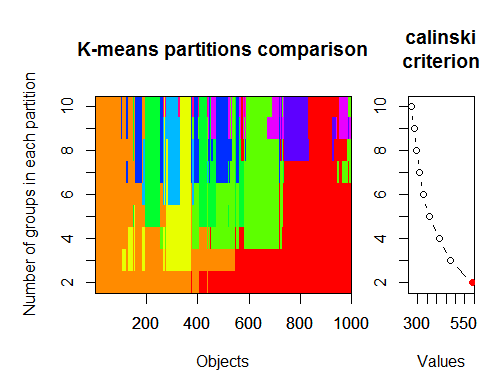
2. Criterio di Calinsky: un altro approccio per diagnosticare quanti cluster sono adatti ai dati. In questo caso
proviamo da 1 a 10 gruppi.
3. Determinare il modello ottimale e il numero di cluster in base al Criterio di informazione bayesiano per la massimizzazione dell'aspettativa, inizializzato dal clustering gerarchico per i modelli a miscela gaussiana parametrizzati.Modelli a miscela gaussiana