Discussion de l'article "Réseaux de neurones de troisième génération : Réseaux profonds" - page 2
Vous manquez des opportunités de trading :
- Applications de trading gratuites
- Plus de 8 000 signaux à copier
- Actualités économiques pour explorer les marchés financiers
Inscription
Se connecter
Vous acceptez la politique du site Web et les conditions d'utilisation
Si vous n'avez pas de compte, veuillez vous inscrire
Ce n'est pas une question pour moi. C'est tout ce que vous avez à dire sur l'article ?
Qu'est-ce qu'il y a à dire sur l'article ? C'est une réécriture typique. C'est la même chose dans d'autres sources, mais avec des mots légèrement différents. Même les images sont les mêmes. Je n'ai rien vu de nouveau, c'est-à-dire d'auteur.
Je voulais essayer les exemples, mais c'est dommage. La section est pour MQL5, mais les exemples sont pour MQL4.
vlad1949
Cher Vlad !
J'ai regardé dans les archives, vous avez une documentation R assez ancienne. Il serait bon de passer aux copies ci-jointes.
vlad1949
Cher Vlad !
Pourquoi n'avez-vous pas réussi à faire fonctionner le testeur ?
J'ai tout fonctionne sans problème. Mais le schéma est sans indicateur : l'Expert Advisor communique directement avec R.
Jeffrey Hinton, inventeur des réseaux profonds : "Les réseaux profonds ne sont applicables qu'aux données dont le rapport signal/bruit est élevé. Les séries financières sont tellement bruyantes que les réseaux profonds ne sont pas applicables. Nous avons essayé, mais sans succès."
Écoutez ses conférences sur YouTube.
Jeffrey Hinton, inventeur des réseaux profonds : "Les réseaux profonds ne sont applicables qu'aux données dont le rapport signal/bruit est élevé. Les séries financières sont tellement bruyantes que les réseaux profonds ne sont pas applicables. Nous avons essayé, mais sans succès."
Écoutez ses conférences sur YouTube.
Compte tenu de votre message dans le fil de discussion parallèle.
Le bruit est compris différemment dans les tâches de classification et dans l'ingénierie radio. Un prédicteur est considéré comme bruyant s'il est faiblement lié (a un faible pouvoir prédictif) à la variable cible. Une signification complètement différente. Il faut rechercher des prédicteurs qui ont un pouvoir prédictif pour différentes classes de la variable cible.
J'ai une compréhension similaire du bruit. Les séries financières dépendent d'un grand nombre de prédicteurs, dont la plupart nous sont inconnus et qui introduisent ce "bruit" dans les séries. En utilisant uniquement les prédicteurs accessibles au public, nous sommes incapables de prédire la variable cible, quels que soient les réseaux ou les méthodes que nous utilisons.
vlad1949
Cher Vlad !
Pourquoi n'avez-vous pas réussi à faire fonctionner le testeur ?
J'ai tout fonctionne sans problème. Vrai le schéma sans indicateur : le conseiller communique directement avec R.
llllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllllll
Bonjour SanSanych.
L'idée principale est donc de faire de la multidevise avec plusieurs indicateurs.
Sinon, bien sûr, vous pouvez tout mettre dans un Expert Advisor.
Mais si la formation, les tests et l'optimisation doivent être mis en œuvre à la volée, sans interrompre le trading, alors la variante avec un Expert Advisor sera un peu plus difficile à mettre en œuvre.
Bonne chance !
PS. Quel est le résultat des tests ?
Bonjour SanSanych.
Voici quelques exemples de détermination du nombre optimal de clusters que j'ai trouvés sur un forum anglophone. Je n'ai pas pu tous les utiliser avec mes données. Le paquet 11 "clusterSim" est très intéressant.
--------------------------------------------------------------------------------------
Dans le prochain post les calculs avec mes données
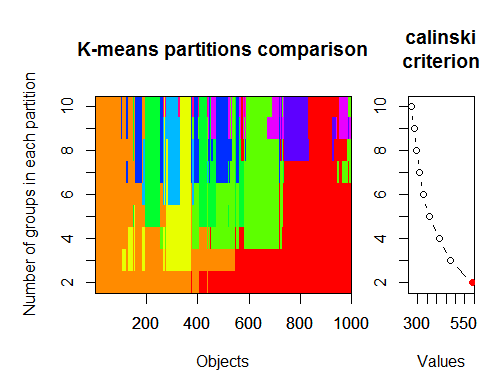
Le nombre optimal de clusters peut être déterminé par plusieurs packages et en utilisant plus de 30 critères d'optimalité. D'après mes observations, le critère le plus utilisé est lecritère de Calinsky.
Prenons les données brutes de notre ensemble à partir de l'indicateur dt . Il contient 17 prédicteurs, la cible y et le corps de chandelier z.
Les dernières versions des paquets "magrittr" et "dplyr" contiennent de nombreuses nouvelles fonctionnalités, dont l'une est le "pipe" - %>%. Elle est très pratique lorsque vous n'avez pas besoin de sauvegarder les résultats intermédiaires. Préparons les données initiales pour le clustering. Prenons la matrice initiale dt, sélectionnons-en les 1000 dernières lignes, puis sélectionnons-y les 17 colonnes de nos variables. Nous obtenons une notation plus claire, rien de plus.
1.
2. Critère de Calinsky : une autre approche pour diagnostiquer le nombre de grappes qui conviennent aux données. Dans ce cas
nous essayons de 1 à 10 groupes.
3) Déterminer le modèle optimal et le nombre de grappes selon le critère d'information bayésien pour la maximisation de l'espérance, initialisé par le regroupement hiérarchique pour les modèles de mélange gaussien paramétrés.Modèles de mélange gaussien