
Redes neurais de maneira fácil (Parte 15): Agrupamento de dados via MQL5
Conteúdo
- Introdução
- 1. Princípios de construção de modelos
- 2. Criando um programa OpenCL
- 3. Trabalho preparatório do lado do programa principal
- 4. Construindo uma classe que gera o algoritmo k-médias
- 5. Teste
- Fim do artigo
- Referências
- Programas utilizados no artigo
Introdução
No artigo anterior, nos familiarizamos com o método de agrupamento k-médias e analisamos sua implementação usando a linguagem Python. Mas muitas vezes o uso da integração impõe certas restrições e custos adicionais, especialmente se falamos do estado atual de integração, que não permite o uso de dados de programas embutidos, como indicadores ou manipulação de eventos de terminal. E se um grande número de indicadores clássicos for implementado em várias bibliotecas, o algoritmo de indicadores personalizados precisará ser repetido em seu script. E o que fazer se não se tiver o código fonte do indicador e um entendimento de seu algoritmo? Ou você planeja usar os resultados de agrupamento em outros programas MQL5. Nesses casos, a implementação do método de agrupamento via ferramentas MQL5 nos ajudará.
1. Princípios de construção de modelos
Vejamos novamente o algoritmo do método de agrupamento k-médias:
- Determinamos k pontos aleatórios a partir da amostra de treinamento como centros de agrupamento.
- Preparamos um loop de operações:
- Determinamos a distância de cada ponto a cada centro;
- Com ajuda do centro mais próximo, determinamos se o ponto pertence ao agrupamento;
- Com a média aritmética, determinamos um novo centro para cada agrupamento.
- Repetimos as operações no loop até que os centros do agrupamento “parem”.
E antes de escrever o código do método, vamos falar um pouco sobre os principais pontos de nossa implementação.
As principais operações do algoritmo visto acima são realizadas dentro do loop. No início do corpo do loop, precisamos encontrar a distância de cada elemento da amostra de treinamento até o centro de cada agrupamento. Como se pode ver, esta operação para cada elemento da amostra de treinamento é absolutamente independente de outros elementos. Portanto, podemos usar a tecnologia OpenCL para aplicar computação paralela. Além disso, as operações também são independentes na hora de calcular a distância aos centros dos diferentes agrupamentos. Isso significa que podemos paralelizar operações em um espaço de tarefas bidimensional.
O próximo passo é determinar se um elemento da sequência pertence a um determinado agrupamento. Ao realizar esta operação, também é monitorada a independência dos cálculos para cada elemento da sequência. E aqui, relativamente aos elementos individuais do conjunto de treinamento, podemos usar a tecnologia OpenCL para aplicar computação paralela.
E na conclusão das operações no corpo do loop, definimos novos centros de agrupamentos. Para fazer isso, precisamos passar por todos os elementos da amostra de treinamento e calcular os valores da média aritmética no que diz respeito a cada elemento do vetor de descrição do estado do sistema e de cada agrupamento. É importante ressaltar que apenas os elementos pertencentes a este agrupamento são levados em consideração na hora de calcular o centro do agrupamento. Os demais elementos são ignorados. Assim, os valores de cada elemento envolve apenas um. E aqui também podemos usar a tecnologia de computação paralela em espaço bidimensional. Em um eixo teremos elementos do vetor de descrição do estado do sistema, e no outro eixo, os agrupamentos analisados.
Depois de agrupar os dados para avaliar o desempenho do nosso modelo, temos que calcular a função de perda. Para fazer isso, como mencionado acima, precisamos calcular o desvio médio aritmético do estado do sistema em relação ao centro do agrupamento correspondente. É claro que não podemos dividir explicitamente em fluxos o cálculo da média aritmética. Em vez disso, podemos dividir esta tarefa em 2 subtarefas. Primeiro calculamos a distância aos respectivos centros. E podemos facilmente paralelizar essa tarefa no que toca a um único estado do sistema. E só então calculamos a média aritmética do vetor distância resultante.
2. Criando um programa OpenCL
Bem, assim, temos quatro subtarefas separadas para aplicar computação paralela. Graças aos artigos anteriores desta série, ficamos sabendo que para aplicar a computação paralela via tecnologia OpenCL, precisamos criar um programa separado para carregar e executar operações do lado do OpenCL context. Criaremos núcleos executáveis do programa em um arquivo separado "unsupervised.cl" respeitando a ordem das tarefas descritas acima.
Começaremos este serviço escrevendo um kernel KmeansCulcDistance, no qual prepararemos operações para calcular distâncias dos estados do sistema aos centros atuais de todos os agrupamentos. Como mencionado acima, iniciaremos a execução deste kernel em um espaço de tarefas bidimensional. Em uma dimensão, teremos estados separados do sistema a partir da amostra de treinamento, e já na segunda, os agrupamentos do nosso modelo.
Nos parâmetros de entrada do kernel, indicamos ponteiros para 3 buffers de dados e o tamanho do vetor que descreve um estado do sistema analisado. Dois dos buffers especificados conterão os dados iniciais. Esta será a própria amostra de treinamento e a matriz de vetores centros de agrupamentos. O terceiro buffer de dados será o tensor de resultado.
No corpo do kernel, primeiro obtemos os identificadores do fluxo de trabalho atual em ambas as dimensões e o número total de agrupamentos segundo o número de threads em execução na 2ª dimensão. Precisamos desses dados para determinar o deslocamento em relação dos elementos desejados em todos os tensores mencionados acima. Imediatamente, determinaremos os deslocamentos nos tensores dos dados iniciais e inicializaremos a variável em zero para calcular a distância até o centro do agrupamento.
Em seguida, geramos um loop com o número de iterações igual ao tamanho do vetor de descrição de um estado do sistema em estudo. No corpo deste loop, somamos as distâncias quadradas entre os valores dos elementos correspondentes dos vetores de estado do sistema e o centro do agrupamento.
Após a conclusão de todas as iterações do loop, basta salvar o valor recebido no elemento correspondente do buffer de resultados. Lembre que, do ponto de vista matemático, para determinar a distância entre dois pontos no espaço, precisamos extrair a raiz quadrada do valor resultante. Mas, neste caso, não estamos interessados na distância exata entre os dois pontos. Só precisamos definir distâncias menores. Portanto, para economizar recursos, não extrair a raiz quadrada.
__kernel void KmeansCulcDistance(__global double *data, __global double *means, __global double *distance, int vector_size ) { int m = get_global_id(0); int k = get_global_id(1); int total_k = get_global_size(1); double sum = 0.0; int shift_m = m * vector_size; int shift_k = k * vector_size; for(int i = 0; i < vector_size; i++) sum += pow(data[shift_m + i] - means[shift_k + i], 2); distance[m * total_k + k] = sum; }
Já demos começo, escrevemos o código para o primeiro kernel e estamos seguindo em frente para trabalhar no próximo subprocesso. Segundo o algoritmo do nosso método, temos que determinar a qual dos agrupamentos cada estado dos apresentados na amostra de treinamento pertence. Para isso, precisamos determinar qual dos centros do agrupamento está mais próximo do estado analisado. Já calculamos as distâncias no kernel anterior. Agora precisamos determinar apenas o número com o valor mínimo. Obviamente, realizaremos todas as operações relativamente a um único estado do sistema.
Para gerar este processo, vamos criar o kernel KmeansClustering. Assim como o kernel anterior, nos parâmetros ele receberá ponteiros para 3 buffers de dados e o número total de agrupamentos. Por mais estranho que pareça, mas de 3 buffers, apenas um distance levará os dados iniciais, enquanto os outros dois conterão os resultados das operações. No buffer clusters colocamos o índice do agrupamento ao qual pertence o estado analisado do sistema.
Nos terceiro buffer flags botamos o sinalizador de alteração do agrupamento em comparação com o estado anterior. A análise desses sinalizadores nos dirá o ponto de interrupção do processo de treinamento do modelo. A lógica por trás desse processo é bastante simples. Se nenhum estado do sistema mudar a associação com um agrupamento, então, como consequência, os centros dos agrupamentos também não mudarão. Isso significa que dar continuação a operações cíclicas não faz sentido. Este é o ponto de parada para o treinamento do modelo.
Mas vamos voltar ao nosso algoritmo do kernel. Vamos iniciá-lo em um espaço de tarefas unidimensional no que diz respeito aos estados do sistema analisados. Portanto, no corpo do kernel, determinaremos o número do estado analisado e o deslocamento correspondente nos buffers de dados. Devo dizer que ambos os buffers de resultados contêm um valor para cada estado. Por essa razão, o deslocamento nos buffers especificados será igual ao ID do fluxo. Isso significa que nos resta determinar o deslocamento apenas no buffer de dados iniciais que contém as distâncias calculadas aos centros do agrupamento.
Aqui vamos preparar 2 variáveis privadas. Uma value, onde registraremos a distância até o centro. E na segunda result, onde colocaremos número do agrupamento. No estágio inicial, armazenaremos os valores do agrupamento com o identificador "0" nelas.
E só então geramos um loop sobre as distâncias aos centros de todos os agrupamentos. Como já armazenamos os valores do agrupamento com índice "0" em variáveis, iniciamos o loop a partir do próximo agrupamento.
No corpo do loop, verificamos a distância até o próximo centro. E se for maior ou igual ao armazenado anteriormente na variável, passamos a verificar o próximo agrupamento.
Ao encontrar um centro mais próximo, reescrevemos o valor de nossas variáveis privadas. Nelas, salvaremos a distância menor e o número do agrupamento correspondente.
Após a conclusão de todas as iterações do nosso loop, o identificador do agrupamento mais próximo do estado analisado será armazenado na variável result. É aqui que nos referimos ao estado atual. Mas antes de salvar o valor recebido no elemento correspondente do buffer de resultados, verificamos se o número do agrupamento mudou em relação à iteração anterior e salvamos o resultado da comparação no buffer de sinalizadores.
__kernel void KmeansClustering(__global double *distance, __global double *clusters, __global double *flags, int total_k ) { int i = get_global_id(0); int shift = i * total_k; double value = distance[shift]; int result = 0; for(int k = 1; k < total_k; k++) { if(value <= distance[shift + k]) continue; value = distance[shift + k]; result = k; } flags[i] = (double)(clusters[i] != (double)result); clusters[i] = (double)result; }
E ao final do algoritmo de agrupamento, precisamos atualizar os valores dos vetores centrais de todos os agrupamentos que são coletados na matriz means. Para realizar esta tarefa, vamos criar outro kernel, o KmeansUpdating. Como os kernels discutidos acima, aquele considerado nos parâmetros receberá ponteiros para 3 buffers de dados e uma constante. Dois buffers contêm os dados iniciais e o outro buffer é de resultados. Como mencionado acima, iniciaremos este kernel para que execute tarefas em um espaço bidimensional. Mas ao contrário do kernel KmeansCulcDistance na primeira dimensão do espaço de tarefas, vamos iterar sobre os elementos do vetor de descrição de estado do sistema, e na constantetotal_m especificamos o número de elementos na amostra de treinamento.
No corpo do kernel, primeiro definiremos os IDs de thread em ambas as dimensões. Como antes, vamos usá-los para determinar elementos analisados e deslocamentos em buffers de dados. Imediatamente, determinaremos o comprimento do vetor de descrição de um estado do sistema, que é igual ao número total de threads em execução na primeira dimensão. Além disso, inicializamos 2 variáveis privadas nas quais somaremos os valores dos elementos correspondentes da descrição do estado do sistema e seu número.
Realizaremos diretamente as operações de soma no seguinte loop gerado, cujo número de iterações será igual ao número de elementos na amostra de treinamento. Lembre-se de que somaremos apenas os elementos que pertencem ao agrupamento analisado. Para fazer isso, no corpo do loop, primeiro verificamos a qual agrupamento o elemento atual pertence. E se não corresponder ao analisado, passamos para o próximo elemento.
Quando o elemento passa na nossa verificação de se agrupamento analisado corresponde, adicionamos o valor do elemento respectivo do vetor de descrição do estado do sistema e aumentamos o contador em "1".
Depois de sair do loop, basta dividir a soma acumulada pelo número de elementos somados. Mas aqui devemos lembrar sobre a possibilidade de receber um erro crítico, nomeadamente o de divisão por zero. Claro, dada a organização do algoritmo, isso é improvável. No entanto, para manter a confiabilidade do nosso programa, adicionaremos essa verificação. E preste atenção, se não houver elementos pertencentes ao agrupamento analisado, não redefinimos seu valor, mas o deixamos igual.
__kernel void KmeansUpdating(__global double *data, __global double *clusters, __global double *means, int total_m ) { int i = get_global_id(0); int vector_size = get_global_size(0); int k = get_global_id(1); double sum = 0; int count = 0; for(int m = 0; m < total_m; m++) { if(clusters[m] != k) continue; sum += data[m * vector_size + i]; count++; } if(count > 0) means[k * vector_size + i] = sum / count; }
Nesta fase, já criamos 3 kernels para implementar o algoritmo de agrupamento de dados k-médias. Mas antes de passar para a criação de objetos do programa principal, temos que criar outro kernel para calcular a função de perda.
Determinaremos o valor da função de perda em 2 etapas. Primeiro, identificamos o desvio de cada elemento individual da amostra de treinamento em relação ao centro do agrupamento correspondente. E então calculamos o desvio médio aritmético para toda a amostra. Podemos dividir as operações do primeiro estágio em threads e realizar cálculos paralelos usando ferramentas OpenCL. Para implementar esta funcionalidade, vamos criar o kernel KmeansLoss, que receberá ponteiros para 4 buffers e uma constante em seus parâmetros. Três buffers levarão os dados iniciais e um buffer para gravar os resultados.
Vamos lançar o kernel em um espaço de tarefas unidimensional com o número de threads igual ao número de elementos na amostra de treinamento. No corpo do kernel, primeiro determinaremos o número do padrão analisado a partir da amostra de treinamento. Em seguida, determinamos a qual agrupamento ele pertence. Desta vez não vamos recalcular as distâncias aos centros de todos os agrupamentos. Em vez disso, simplesmente extraímos o valor correspondente do buffer de clusters com ajuda do número do elemento. Foi nesse buffer que salvamos o número do agrupamento no kernel KmeansClustering discutido acima.
Agora podemos determinar o deslocamento para o início dos vetores que precisamos nos tensores da amostra de treinamento e na matriz de centros de agrupamento.
Em seguida, basta calcular a distância entre os dois vetores. Para isso, inicializamos uma variável privada para acumular a soma dos desvios e preparamos um loop percorrendo todos os elementos do vetor de descrição de um estado do sistema analisado. No corpo deste loop, somaremos os desvios quadrados dos elementos correspondentes dos vetores.
Após a conclusão de todas as iterações do loop, transferiremos o valor acumulado para o elemento correspondente do buffer de resultado loss.
__kernel void KmeansLoss(__global double *data, __global double *clusters, __global double *means, __global double *loss, int vector_size ) { int m = get_global_id(0); int c = clusters[m]; int shift_c = c * vector_size; int shift_m = m * vector_size; double sum = 0; for(int i = 0; i < vector_size; i++) sum += pow(data[shift_m + i] - means[shift_c + i], 2); loss[m] = sum; }
Bem, aqui consideramos os algoritmos para construir todos os processos do lado do OpenCL context. Agora podemos passar para a organização de processos no lado do programa principal.
3. Trabalho preparatório do lado do programa principal
Temos que criar uma nova classe CKmeans do lado do programa principal. Vamos salvar o código desta classe no arquivo "kmeans.mqh". Mas antes de prosseguir diretamente para a tarefa a ser realizada na nova classe, faremos um pequeno trabalho preparatório. Em primeiro lugar, para transferir dados para o OpenCL context, usaremos o objeto de classe CBufferDouble já conhecido por nós nesta série de artigos. Em vez de reescrever o código da classe especificada, vamos simplesmente incluir a biblioteca criada anteriormente.
#include "..\NeuroNet_DNG\NeuroNet.mqh"
Em seguida, carregaremos o código do programa OpenCL criado acima como um recurso.
#resource "unsupervised.cl" as string cl_unsupervised
Em seguida, passamos para a criação de constantes nomeadas. Neste artigo, precisaremos de várias dessas constantes. E aqui deve ser dito que precisamos cuidar da singularidade das constantes que criamos a fim de poder compartilhá-las com uma biblioteca previamente criada.
Primeiro, precisamos de uma constante para identificar a nova classe.
#define defUnsupervisedKmeans 0x7901
Em segundo lugar, precisaremos de constantes para identificar os kernels e seus parâmetros. Kernels são identificados através de numeração contínua no que diz respeito a um programa OpenCL. Ao mesmo tempo, os parâmetros são numerados dentro de um único kernel. Para melhorar a legibilidade do código, decidi agrupar as constantes de acordo com os kernels aos quais pertencem.
#define def_k_kmeans_distance 0 #define def_k_kmd_data 0 #define def_k_kmd_means 1 #define def_k_kmd_distance 2 #define def_k_kmd_vector_size 3
#define def_k_kmeans_clustering 1 #define def_k_kmc_distance 0 #define def_k_kmc_clusters 1 #define def_k_kmc_flags 2 #define def_k_kmc_total_k 3
#define def_k_kmeans_updates 2 #define def_k_kmu_data 0 #define def_k_kmu_clusters 1 #define def_k_kmu_means 2 #define def_k_kmu_total_m 3
#define def_k_kmeans_loss 3 #define def_k_kml_data 0 #define def_k_kml_clusters 1 #define def_k_kml_means 2 #define def_k_kml_loss 3 #define def_k_kml_vector_size 4
Depois de criar constantes nomeadas, vamos para a próxima etapa do trabalho preparatório. Quando falamos de implementação computação multithread em modelos de aprendizado supervisionado, inicializamos o objeto para trabalhar com o OpenCL context no construtor da classe dispatcher de rede neural. Dentro da estrutura deste artigo, planejamos usar a classe de agrupamento CKmeans sem usar nenhum outro modelo. E parece que podemos mover a função de inicialização da instância do objeto COpenCLMy dentro de nossa nova classe CKmeans. No entanto, não excluo o uso de agrupamento como parte de outros modelos mais complexos. Mas isso está além do escopo deste artigo, e retornaremos a essa questão em artigos ulteriores desta série. No entanto, devemos considerar essa possibilidade. Por isso é que decidi criar uma função separada para inicializar uma instância da classe de objeto COpenCLMy.
Vejamos o algoritmo da função OpenCLCreate. Ele é construído de tal forma que recebe o teste do programa OpenCL como parâmetros e retorna um ponteiro para uma instância de um objeto inicializado. No corpo da função, primeiro criaremos uma nova instância da classe COpenCLMy. E verificamos imediatamente o resultado da operação de criação de novo objeto.
COpenCLMy *OpenCLCreate(string programm) { COpenCL *result = new COpenCLMy(); if(CheckPointer(result) == POINTER_INVALID) return NULL;
Em seguida, vamos chamar o método de inicialização do novo objeto, passando para ele uma variável string com o texto do programa nos parâmetros OpenCL. E novamente verificamos o resultado da operação. Se de repente recebermos um erro ao realizar esta operação, excluímos o objeto criado acima e saímos do método, retornando um ponteiro vazio.
if(!result.Initialize(programm, true)) { delete result; return NULL; }
Após a inicialização bem-sucedida do programa, procedemos à criação de kernels no OpenCL context. Primeiro, especificaremos o número de kernels a serem criados e, em seguida, criaremos todos os kernels descritos acima, um por um. Ao mesmo tempo, não se esqueça de controlar o processo, verificando o resultado de cada operação.
O código abaixo mostra um exemplo de inicialização de apenas um kernel. O resto é inicializado da mesma maneira. O código completo de todos os métodos e funções pode ser encontrado no anexo.
if(!result.SetKernelsCount(4)) { delete result; return NULL; } //--- if(!result.KernelCreate(def_k_kmeans_distance, "KmeansCulcDistance")) { delete result; return NULL; } //--- ........... //--- return result; }
Depois de criar todos os kernels com sucesso, saímos do método retornando um ponteiro para a instância do objeto criado.
Desta forma, concluímos o trabalho preparatório e passamos diretamente ao trabalho na nova classe de agrupamento de dados.
4. Construindo uma classe que gera o algoritmo k-médias
Iniciando o trabalho em uma nova classe de agrupamento de dados CKmeans vamos discutir o seu conteúdo. Que funcionalidade deve ter? E quais métodos e variáveis precisaremos para gerar essa funcionalidade. Vamos declarar todas as variáveis no bloco protectec.
Antes de tudo, precisaremos de variáveis para armazenar os hiperparâmetros do modelo: o número de agrupamentos criados (m_iClusters) e o tamanho do vetor de descrição de um estado individual do sistema (m_iVectorSize).
Após avaliar a qualidade do modelo treinado, calculamos a função de perda, cujo valor é armazenado na variável m_dLoss.
Além disso, para entender o estado do modelo (treinado ou não), precisamos do sinalizador m_bTrained.
Parece-me que esta lista de variáveis será suficiente para criar a funcionalidade necessária. Em seguida, passamos a declarar os objetos usados. Não vamos demorar muito aqui e vamos declarar uma instância de classe para trabalhar com o OpenCL context (c_OpenCL). E também precisamos de buffers de dados para armazenar informações e trocá-las com o OpenCL context. Faremos com que seus nomes sejam iguais aos usados acima ao desenvolver o programa OpenCL:
- c_aDistance;
- c_aMeans;
- c_aClasters;
- c_aFlags;
- c_aLoss.
Depois de declarar as variáveis, passamos a trabalhar nos métodos de classe. Aqui não esconderemos nada e tornaremos públicos todos os métodos.
E começaremos, é claro, com o construtor e o destruidor da classe. No primeiro, vamos criar instâncias dos objetos usados e definir valores iniciais para as variáveis.
void CKmeans::CKmeans(void) : m_iClusters(2), m_iVectorSize(1), m_dLoss(-1), m_bTrained(false) { c_aMeans = new CBufferDouble(); if(CheckPointer(c_aMeans) != POINTER_INVALID) c_aMeans.BufferInit(m_iClusters * m_iVectorSize, 0); c_OpenCL = NULL; }
E no destruidor de classes, limpamos a memória e excluímos todos os objetos criados na classe.
void CKmeans::~CKmeans(void) { if(CheckPointer(c_aMeans) == POINTER_DYNAMIC) delete c_aMeans; if(CheckPointer(c_aDistance) == POINTER_DYNAMIC) delete c_aDistance; if(CheckPointer(c_aClasters) == POINTER_DYNAMIC) delete c_aClasters; if(CheckPointer(c_aFlags) == POINTER_DYNAMIC) delete c_aFlags; if(CheckPointer(c_aLoss) == POINTER_DYNAMIC) delete c_aLoss; }
Em seguida, criaremos um método de inicialização para nossa classe, nos parâmetros para os quais passaremos um ponteiro para o objeto de trabalho com o OpenCL context e os hiperparâmetros do modelo. No corpo do método, primeiro criamos um pequeno bloco de controles no qual verificamos os dados recebidos nos parâmetros.
Depois disso, salvamos os hiperparâmetros obtidos nas variáveis correspondentes e inicializamos o buffer da matriz de vetores de agrupamentos médios com valores zero. Ao mesmo tempo, não nos esquecemos de verificar o resultado das operações de inicialização do buffer.
bool CKmeans::Init(COpenCLMy *context, int clusters, int vector_size) { if(CheckPointer(context) == POINTER_INVALID || clusters < 2 || vector_size < 1) return false; //--- c_OpenCL = context; m_iClusters = clusters; m_iVectorSize = vector_size; if(CheckPointer(c_aMeans) == POINTER_INVALID) { c_aMeans = new CBufferDouble(); if(CheckPointer(c_aMeans) == POINTER_INVALID) return false; } c_aMeans.BufferFree(); if(!c_aMeans.BufferInit(m_iClusters * m_iVectorSize, 0)) return false; m_bTrained = false; m_dLoss = -1; //--- return true; }
Após a inicialização, temos que treinar o modelo. Criamos essa funcionalidade no método Study. Nos parâmetros do método, passaremos a amostra de treinamento e o sinalizador de inicialização da matriz de centros de agrupamento. O objetivo deste sinalizador é permitir que a inicialização de uma matriz seja desativada ao continuar a treinar um modelo parcial ou totalmente pré-treinado carregado desde um arquivo.
No corpo do método, criamos um bloco de controles. Primeiro, verificamos se os ponteiros de objeto recebidos nos parâmetros da amostra de treinamento e no OpenCL context são válidos.
Em seguida, verificamos a presença de dados na amostra de treinamento e se seu número é um múltiplo do tamanho do vetor de descrição de um único estado do sistema especificado durante a inicialização.
Também adicionaremos uma verificação de que o número de elementos na amostra de treinamento é pelo menos 10 vezes maior que o número de agrupamentos.
bool CKmeans::Study(CBufferDouble *data, bool init_means = true) { if(CheckPointer(data) == POINTER_INVALID || CheckPointer(c_OpenCL) == POINTER_INVALID) return false; //--- int total = data.Total(); if(total <= 0 || m_iClusters < 2 || (total % m_iVectorSize) != 0) return false; //--- int rows = total / m_iVectorSize; if(rows <= (10 * m_iClusters)) return false;
O próximo passo é inicializar a matriz de centros de agrupamento. Obviamente, antes de inicializar a matriz, verificaremos o estado do sinalizador de inicialização que recebemos nos parâmetros do método.
Vamos inicializar a matriz com vetores selecionados aleatoriamente a partir da amostra de treinamento. E aqui precisamos criar um algoritmo que exclua a inicialização de vários agrupamentos com o mesmo estado do sistema. Para fazer isso, criaremos uma matriz de sinalizadores com o número de elementos igual ao número de estados do sistema na amostra de treinamento. No estágio inicial, inicializamos essa matriz com valores false. Em seguida, geramos um loop com o número de iterações igual ao número de agrupamentos em nosso modelo. No corpo do loop, geraremos aleatoriamente um número dentro do tamanho da amostra de treinamento e verificaremos o sinalizador em relação ao índice resultante. Se o estado do sistema fornecido já tiver inicializado qualquer agrupamento, diminuiremos os estados do contador de iteração e passaremos para a próxima iteração do loop.
Se o elemento selecionado ainda não participou da inicialização dos agrupamentos, determinamos o deslocamento na amostra de treinamento para o início do estado dado do sistema na amostra de treinamento e na matriz de vetores centrais. Em seguida, elaboramos um loop aninhado para copiar dados. E antes de passar para a próxima iteração do loop, vamos alterar o sinalizador com o índice trabalhado.
bool flags[]; if(ArrayResize(flags, rows) <= 0 || !ArrayInitialize(flags, false)) return false; //--- for(int i = 0; (i < m_iClusters && init_means); i++) { Comment(StringFormat("Cluster initialization %d of %d", i, m_iClusters)); int row = (int)((double)MathRand() * MathRand() / MathPow(32767, 2) * (rows - 1)); if(flags[row]) { i--; continue; } int start = row * m_iVectorSize; int start_c = i * m_iVectorSize; for(int c = 0; c < m_iVectorSize; c++) { if(!c_aMeans.Update(start_c + c, data.At(start + c))) return false; } flags[row] = true; }
Após inicializar a matriz de centros, passamos a verificar a validade dos ponteiros e, se necessário, criar novas instâncias de objetos buffer para registrar a matriz de distância (c_aDistance), o vetor de identificação do agrupamento para cada estado do sistema (c_aClasters) e o vetor de sinalizadores de alteração de agrupamento para estados de sistema individuais (c_aFlags). Ao mesmo tempo, não se esqueça de controlar a execução das operações.
if(CheckPointer(c_aDistance) == POINTER_INVALID) { c_aDistance = new CBufferDouble(); if(CheckPointer(c_aDistance) == POINTER_INVALID) return false; } c_aDistance.BufferFree(); if(!c_aDistance.BufferInit(rows * m_iClusters, 0)) return false;
if(CheckPointer(c_aClasters) == POINTER_INVALID) { c_aClasters = new CBufferDouble(); if(CheckPointer(c_aClasters) == POINTER_INVALID) return false; } c_aClasters.BufferFree(); if(!c_aClasters.BufferInit(rows, 0)) return false;
if(CheckPointer(c_aFlags) == POINTER_INVALID) { c_aFlags = new CBufferDouble(); if(CheckPointer(c_aFlags) == POINTER_INVALID) return false; } c_aFlags.BufferFree(); if(!c_aFlags.BufferInit(rows, 0)) return false;
Por fim, criaremos buffers no OpenCL context.
if(!data.BufferCreate(c_OpenCL) || !c_aMeans.BufferCreate(c_OpenCL) || !c_aDistance.BufferCreate(c_OpenCL) || !c_aClasters.BufferCreate(c_OpenCL) || !c_aFlags.BufferCreate(c_OpenCL)) return false;
Neste ponto, concluímos a etapa de trabalho preparatório e procedemos à organização das operações cíclicas diretamente no processo de treinamento do modelo. Lembre-se das principais fases do algoritmo considerado:
- Determinar as distâncias entre cada elemento da amostra de treinamento e cada centro do agrupamento;
- Distribuir os estados do sistema por agrupamentos (por distância mínima);
- Atualizar os centros de agrupamento.
Veja estes passos do algoritmo. Para cada etapa acima, já criamos kernels no programa OpenCL. Portanto, resta-nos gerar uma chamada cíclica dos kernels correspondentes.
Elaboramos um loop de treinamento e no corpo do loop seremos os primeiros a chamar o kernel para cálculo de distâncias aos centros dos agrupamentos. Já carregamos todos os buffers necessários na memória do OpenCL context. Portanto, vamos direto para a especificação dos parâmetros do kernel. Aqui indicamos ponteiros para os buffers de dados usados e o tamanho do vetor que descreve um estado do sistema. Observe que para especificar um parâmetro em particular, usamos um par de constantes - "identificador de kernel/identificador de parâmetro"
int count = 0; do { if(!c_OpenCL.SetArgumentBuffer(def_k_kmeans_distance, def_k_kmd_data, data.GetIndex())) return false; if(!c_OpenCL.SetArgumentBuffer(def_k_kmeans_distance, def_k_kmd_means, c_aMeans.GetIndex())) return false; if(!c_OpenCL.SetArgumentBuffer(def_k_kmeans_distance, def_k_kmd_distance, c_aDistance.GetIndex())) return false; if(!c_OpenCL.SetArgument(def_k_kmeans_distance, def_k_kmd_vector_size, m_iVectorSize)) return false;
Em seguida, precisamos especificar a dimensão do espaço de tarefas e o deslocamento em cada um deles. Planejamos iniciar este kernel em um espaço de tarefas bidimensional. Vamos criar 2 matrizes estáticas com o número de elementos igual ao espaço da tarefa:
- global_work_size - para especificar a dimensão do espaço de tarefas;
- global_work_offset - para especificar o deslocamento em cada dimensão.
Nelas, indicaremos o deslocamento zero em ambas as dimensões. O tamanho da primeira dimensão será igual ao número de estados individuais do sistema na amostra de treinamento. E indicamos o tamanho da segunda dimensão igual ao número de agrupamentos em nosso modelo.
uint global_work_offset[2] = {0, 0}; uint global_work_size[2]; global_work_size[0] = rows; global_work_size[1] = m_iClusters;
Depois disso, basta executar o kernel e ler os resultados das operações.
if(!c_OpenCL.Execute(def_k_kmeans_distance, 2, global_work_offset, global_work_size)) return false; if(!c_aDistance.BufferRead()) return false;
De maneira semelhante, chamamos o segundo kernel, que determina se os estados do sistema pertencem a agrupamentos específicos. Observe que este kernel será inciado em um espaço de tarefa unidimensional. Portanto, precisaremos de outras matrizes para indicar o tamanho e o deslocamento.
if(!c_OpenCL.SetArgumentBuffer(def_k_kmeans_clustering, def_k_kmc_flags, c_aFlags.GetIndex())) return false; if(!c_OpenCL.SetArgumentBuffer(def_k_kmeans_clustering, def_k_kmc_clusters, c_aClasters.GetIndex())) return false; if(!c_OpenCL.SetArgumentBuffer(def_k_kmeans_clustering, def_k_kmc_distance, c_aDistance.GetIndex())) return false; if(!c_OpenCL.SetArgument(def_k_kmeans_clustering, def_k_kmc_total_k, m_iClusters)) return false; uint global_work_offset1[1] = {0}; uint global_work_size1[1]; global_work_size1[0] = rows; if(!c_OpenCL.Execute(def_k_kmeans_clustering, 1, global_work_offset1, global_work_size1)) return false; if(!c_aFlags.BufferRead()) return false;
Observe que, depois de enfileirar o kernel, apenas lemos os dados do buffer de sinalizadores. Agora temos dados suficientes para determinar o fim do treinamento do modelo. E o carregamento de dados de índices de agrupamentos intermediários não é significativo, mas implica em custos adicionais. É por isso que desistimos dele nesta fase.
Após a distribuição dos elementos da amostra de treinamento por agrupamentos, verificamos se houve redistribuição dos elementos por agrupamentos. Para fazer isso, verificamos o valor máximo do buffer de dados do sinalizador. Como você se lembra, no código do correspondente do kernel preenchemos o buffer de sinalizadores com o resultado booleano da comparação dos IDs de agrupamento da iteração anterior e do novo que está sendo atribuído. Se igual, escrevemos no buffer "0". Se o agrupamento mudou, gravamos "1". Não nos importa quantos elementos mudaram o agrupamento. Basta-nos saber a sua presença. Por isso, verificamos o valor máximo. E se for igual a "0", ou seja, nenhum dos elementos alterou o agrupamento, consideramos o treinamento do modelo concluído. Lemos o buffer de identificação do agrupamento de cada elemento da sequência e saímos do loop.
m_bTrained = (c_aFlags.Maximum() == 0); if(m_bTrained) { if(!c_aClasters.BufferRead()) return false; break; }
Caso o processo de aprendizado ainda não tenha sido concluído, procede-se à organização da chamada do 3º kernel - atualização dos vetores centrais dos agrupamentos. Vamos rodar este kernel, assim como o primeiro, em um espaço de tarefas bidimensional. Logo, usaremos as matrizes criadas quando o primeiro kernel foi chamado. Vamos alterar apenas o tamanho da primeira dimensão.
if(!c_OpenCL.SetArgumentBuffer(def_k_kmeans_updates, def_k_kmu_data, data.GetIndex())) return false; if(!c_OpenCL.SetArgumentBuffer(def_k_kmeans_updates, def_k_kmu_means, c_aMeans.GetIndex())) return false; if(!c_OpenCL.SetArgumentBuffer(def_k_kmeans_updates, def_k_kmu_clusters, c_aClasters.GetIndex())) return false; if(!c_OpenCL.SetArgument(def_k_kmeans_updates, def_k_kmu_total_m, rows)) return false; global_work_size[0] = m_iVectorSize; if(!c_OpenCL.Execute(def_k_kmeans_updates, 2, global_work_offset, global_work_size)) return false; if(!c_aMeans.BufferRead()) return false; count++; Comment(StringFormat("Study iterations %d", count)); } while(!m_bTrained && !IsStopped());
Após executar o kernel para controle visual do processo de aprendizado, exibiremos o número de iterações de treinamento concluídas no campo de comentários do gráfico e passaremos para a próxima iteração do loop.
Observe que durante todo o treinamento do modelo, não limpamos a memória do OpenCL context e não copiamos novamente os dados para ele. Lembramos que essas operações também exigem recursos. E para aumentar a eficiência do uso de recursos e reduzir o tempo geral de treinamento do modelo, eliminamos esses custos. Mas essa abordagem só é possível se a memória de contexto for suficiente para armazenar todos os dados. Caso contrário, precisaremos reconsiderar o uso da memória de context, liberando dados antigos e carregando novos dados antes de executar cada kernel.
No entanto, após a conclusão do processo de aprendizado, antes de sair do método, limpamos a memória de context e removemos alguns dos buffers dela.
data.BufferFree(); c_aDistance.BufferFree(); c_aFlags.BufferFree(); //--- return true; }
Deve-se dizer que o treinamento do modelo não é um fim em si mesmo. Treinamos o modelo para aproveitar seus resultados e aplicá-los a novos dados. Para realizar esta funcionalidade, criaremos o método Clustering. Na verdade, seu algoritmo é uma versão um tanto truncada do método de aprendizado discutido acima, no qual o loop de aprendizado e o terceiro kernel são excluídos. E apenas os 2 primeiros kernels são chamados uma vez. Você pode se familiarizar com seu código no anexo.
O próximo método que veremos é o método para calcular o valor da função de perda GetLoss. É importante ressaltar aqui que, para economizar recursos ao treinar o modelo, não calculamos os valores da função de perda. Portanto, nos parâmetros, o método recebe um ponteiro para a amostra de dados, para a qual o erro será calculado. Mas se antes no início do método criávamos um bloco de controles, agora chamamos o método de agrupamento. E, claro, não nos esquecemos de conferir o resultado da execução do método.
double CKmeans::GetLoss(CBufferDouble *data) { if(!Clustering(data)) return -1;
Essa abordagem nos permite resolver 2 tarefas ao mesmo tempo com uma ação. Primeiro, este é o agrupamento da nova amostra em si. Afinal, para calcular os desvios, precisamos entender a quais agrupamentos os elementos da amostra pertencem.
Em segundo lugar, o método Clustering já contém todos os controles necessários, então não precisamos repeti-los.
Em seguida, contamos o número de estados do sistema na amostra e inicializamos o buffer para determinar os desvios parciais.
int total = data.Total(); int rows = total / m_iVectorSize; //--- if(CheckPointer(c_aLoss) == POINTER_INVALID) { c_aLoss = new CBufferDouble(); if(CheckPointer(c_aLoss) == POINTER_INVALID) return -1; } if(!c_aLoss.BufferInit(rows, 0)) return -1;
E vamos transferir os dados iniciais para a memória de context. Observe que não passamos buffers de médias e IDs de agrupamento para a memória de context. A razão é que eles já estão na memória do OpenCL context. Não os removemos depois de fazer o agrupamento de dados e, neste estágio, podemos economizar alguns recursos.
if(!data.BufferCreate(c_OpenCL) || !c_aLoss.BufferCreate(c_OpenCL)) return -1;
Em seguida, chamaremos o correspondente kernel. O procedimento para chamar o kernel é completamente idêntico aos exemplos discutidos acima. Então não vamos nos debruçar sobre isso. O código completo de todos os métodos e funções pode ser encontrado no anexo.
Mas no kernel, calculamos o desvio para cada estado individual. Agora temos que determinar o desvio médio. Para isso, geramos um loop no qual simplesmente somamos todos os valores do buffer. E então dividimos pelo número total de elementos na amostra analisada.
m_dLoss = 0; for(int i = 0; i < rows; i++) m_dLoss += c_aLoss.At(i); m_dLoss /= rows;
No final do método, limpamos a memória de context e retornamos o valor resultante.
data.BufferFree();
c_aLoss.BufferFree();
return m_dLoss;
}
Nesta fase, criamos todas as funcionalidades necessárias para o treinamento do modelo e posterior agrupamento de dados. Mas sabemos que o treinamento de um modelo é bastante demorado e não será repetido antes de cada «uso industrial» do modelo ser iniciado. Portanto, devemos organizar o processo de salvar o modelo em um arquivo e restaurá-lo de volta ao seu pleno funcionamento a partir do arquivo. Esta funcionalidade é executada nos métodos Save e Load respectivamente. Como parte de nossa série de artigos, já criamos métodos semelhantes mais de uma vez, porque, eles estão presentes em todas as classes. Você pode encontrar seu código no anexo, e ficarei feliz em responder a todas as suas perguntas no fórum no tópico dedicado a este artigo.
Como resultado, a estrutura final de nossa classe terá a seguinte forma. E o código completo de todos os métodos e classes pode ser encontrado no anexo.
class CKmeans : public CObject { protected: int m_iClusters; int m_iVectorSize; double m_dLoss; bool m_bTrained; COpenCLMy *c_OpenCL; //--- CBufferDouble *c_aDistance; CBufferDouble *c_aMeans; CBufferDouble *c_aClasters; CBufferDouble *c_aFlags; CBufferDouble *c_aLoss; public: CKmeans(void); ~CKmeans(void); //--- bool SetOpenCL(COpenCLMy *context); bool Init(COpenCLMy *context, int clusters, int vector_size); bool Study(CBufferDouble *data, bool init_means = true); bool Clustering(CBufferDouble *data); double GetLoss(CBufferDouble *data); //--- virtual bool Save(const int file_handle); virtual bool Load(const int file_handle); //--- virtual int Type(void) { return defUnsupervisedKmeans; } };
5. Teste
E assim caminhamos para a culminação do processo. Criamos uma nova classe de agrupamento de dados e gostaríamos de avaliar seu valor prático. Vamos treinar o modelo. Para isso, criaremos o EA "kmeans.mq5". O código do EA está no anexo.
Os parâmetros externos do EA serão totalmente transferidos a partir daqueles usados anteriormente, só que aumentaremos o período de estudo para 15 anos. Afinal, o destaque do aprendizado não supervisionado está justamente na possibilidade de utilizar um grande conjunto de dados não marcados. Não exibi nos parâmetros o número de agrupamentos do modelo, pois gerei o processo de aprendizado em um loop com uma gama bastante ampla de agrupamentos. Para encontrar o número ideal de agrupamentos, passamos por várias opções que variam de 50 a 1000 agrupamentos. Mais precisamente, usamos um incremento de 50 agrupamentos. Lembre que essas são as configurações de agrupamento que usamos no artigo anterior ao testar o script de Python. Também pegamos os parâmetros de teste de experimentos anteriores:
- Instrumento: EURUSD;
- Período gráfico: H1;
Como resultado do treinamento, obtivemos um gráfico das dependências da função de perda em relação ao número de agrupamentos, mostrado abaixo.
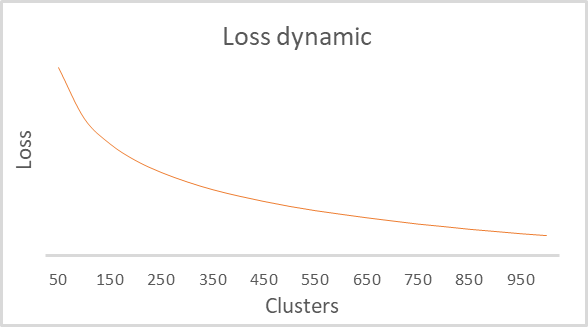
Como se pode ver no gráfico, a quebra acabou sendo bastante estendida na faixa de 100 a 500 agrupamentos. Com isso, é preciso dizer que foram analisados 92 estados do sistema. E a forma do gráfico em si é completamente idêntica àquela construída pelo script de Python no artigo anterior. Isto confirma indiretamente a precisão da classe que construímos.
Fim do artigo
Neste artigo, criamos uma nova classe CKmeans para implementar um dos métodos de agrupamento k-médias mais comuns. E até conseguimos treinar o modelo com um número diferente de agrupamentos. Com base nos resultados dos testes, podemos concluir que o modelo é capaz de identificar cerca de 500 padrões. Um resultado semelhante foi obtido por testes semelhantes em Python. Isso quer dizer que replicamos corretamente o algoritmo do método. No próximo artigo, discutiremos possíveis métodos de uso prático de resultados de agrupamento.
Referências
- Redes neurais de maneira fácil
- Redes neurais de maneira fácil (Parte 2): treinamento e teste da rede
- Redes neurais de maneira fácil (Parte 3): redes convolucionais
- Redes neurais de maneira fácil (Parte 4): redes recorrentes
- Redes neurais de maneira fácil (Parte 5): cálculos em paralelo com OpenCL
- Redes neurais de maneira fácil (Parte 6): experimentos com a taxa de aprendizado da rede neural
- Redes neurais de maneira fácil (Parte 7): métodos de otimização adaptativos
- Redes neurais de maneira fácil (Parte 8): mecanismos de atenção
- Redes neurais de maneira fácil (Parte 9): documentação do trabalho
- Redes neurais de maneira fácil (Parte 10): atenção multi-cabeça
- Redes neurais de maneira fácil (Parte 11): uma visão sobre GPT
- Redes neurais de maneira fácil (Parte 12): dropout
- Redes neurais de maneira fácil (Parte 13): normalização em lote
- Redes neurais de maneira fácil (Parte 14): agrupamento de dados
Programas utilizados no artigo
| # | Nome | Tipo | Canal Equidistante |
|---|---|---|---|
| 1 | kmeans.mq5 | Expert Advisor | EA para treinamento de modelos |
| 2 | kmeans.mqh | Biblioteca de classe | Biblioteca para gerar o método k-médias |
| 3 | unsupervised.cl | Biblioteca | Biblioteca de código de programa OpenCL para gerar o método k-médias |
| 4 | NeuroNet.mqh | Biblioteca de classe | Biblioteca de classes para criar uma rede neural |
| 5 | NeuroNet.cl | Biblioteca | Biblioteca de código de programa OpenCL |
Traduzido do russo pela MetaQuotes Ltd.
Artigo original: https://www.mql5.com/ru/articles/10947
 Como desenvolver um sistema de negociação baseado no indicador OBV
Como desenvolver um sistema de negociação baseado no indicador OBV
 Ciência de Dados e Aprendizado de Máquina (Parte 03): Regressões Matriciais
Ciência de Dados e Aprendizado de Máquina (Parte 03): Regressões Matriciais
 Redes neurais de maneira fácil (Parte 16): Uso prático do agrupamento
Redes neurais de maneira fácil (Parte 16): Uso prático do agrupamento
 Como desenvolver um sistema de negociação baseado no indicador SAR Parabólico
Como desenvolver um sistema de negociação baseado no indicador SAR Parabólico
- Aplicativos de negociação gratuitos
- 8 000+ sinais para cópia
- Notícias econômicas para análise dos mercados financeiros
Você concorda com a política do site e com os termos de uso