Популяционные алгоритмы оптимизации: Гибридный алгоритм оптимизации бактериального поиска с генетическим алгоритмом (Bacterial Foraging Optimization - Genetic Algorithm, BFO-GA)
Inhalt
1. Einführung
2. Der Algorithmus
3. Testergebnisse
1. Einführung
Der hybride Optimierungsalgorithmus BFO-GA kombiniert zwei verschiedene Optimierungsalgorithmen: den Foraging-Optimierungsalgorithmus (BFO) und den genetischen Algorithmus (GA). Dieser hybride Algorithmus wurde entwickelt, um die Effizienz der Optimierung zu verbessern und einige der Unzulänglichkeiten der einzelnen Algorithmen zu überwinden.
BFO (Bacterial Foraging Optimization, Optimierung gemäß bakterieller Nahrungssuche) ist ein Optimierungsalgorithmus, der durch das Futtersuchverhalten von Bakterien inspiriert wurde. Sie wurde im Jahr 2002 von Rahul K. Kujur vorgeschlagen. BFO modelliert die bakterielle Bewegung mit drei Hauptmechanismen: Übergänge, Diffusion und Positionsaktualisierung. Jedes Bakterium im Algorithmus stellt eine Lösung des Optimierungsproblems dar, und die Nahrung entspricht der optimalen Lösung. Die Bakterien bewegen sich durch den Suchraum, um die beste Nahrung zu finden.
Der genetische Algorithmus (GA) ist ein Optimierungsalgorithmus, der sich an den Prinzipien der natürlichen Selektion und der Genetik orientiert. Sie wurde in den 1970er Jahren von John Holland entwickelt. GA arbeitet mit einer Population von Individuen, die Lösungen für ein Optimierungsproblem darstellen. Die Individuen durchlaufen die Operationen der Kreuzung (Kombination genetischer Informationen) und der Mutation (zufällige Veränderungen der genetischen Informationen), um neue Generationen zu schaffen. Nach mehreren Generationen strebt GA danach, die optimale Lösung zu finden.
Der hybride BFO-GA-Algorithmus kombiniert die Vorteile beider Algorithmen. BFO hat gute lokale Such- und schnelle Konvergenzfähigkeiten, kann aber bei der globalen Suche Schwierigkeiten haben. Andererseits hat GA eine gute Fähigkeit zur globalen Suche, kann aber langsam konvergieren und neigt dazu, in lokalen Optima stecken zu bleiben. Der hybride BFO-GA-Algorithmus versucht, diese Einschränkungen zu überwinden, indem er BFO für die globale Suche und GA für die Verfeinerung lokaler Optima verwendet.
Der Hybridalgorithmus BFO-GA (Bacterial Foraging Optimization - Genetic Algorithm) wurde 2007 von D. N. Kim, A. Abraham und J. H. Cho. Dieser Algorithmus kombiniert Optimierungsprinzipien, die auf dem Verhalten von schwärmenden Bakterien basieren, mit genetischen Operatoren wie Selektion, Crossover und Mutation.
BFO-GA verwendet den bakteriellen Algorithmus als Grundlage, erweitert ihn aber um genetische Auswahl-, Crossover- und Mutationsoperatoren. Dieser Algorithmus verwendet Bakterienschwärme, um die optimale Lösung zu finden.
BFO-GA verwendet einen auf der Roulette-Methode basierenden Algorithmus als Auswahloperator. Bei dieser Methode werden die Elternbakterien mit Wahrscheinlichkeiten proportional zu ihrer Fitness ausgewählt. So ist es wahrscheinlicher, dass fittere Bakterien für die Kreuzung ausgewählt werden.
Die Kreuzung im BFO-GA-Algorithmus wird mit dem arithmetischen Crossover-Operator durchgeführt. Dabei werden die genetischen Informationen der beiden Elternbakterien kombiniert, sodass neue Nachkommen mit neuen Genkombinationen entstehen.
Die Mutation in BFO-GA wird mit dem nicht-uniformen Mihaljevic-Mutator durchgeführt. Dieser Mutator verändert die genetische Information in Bakterien, indem er zufällige Änderungen vornimmt. Ungleichmäßigkeit der Mutation bedeutet, dass die Mutationswahrscheinlichkeit in Abhängigkeit von verschiedenen Faktoren variieren kann.
Diese modifizierenden Operatoren werden nach Abschluss der Chemotaxis- und Reproduktionsverfahren des bakteriellen Algorithmus vor den Eliminierungs- und Dispersionsverfahren dieses Algorithmus durchgeführt. Mit anderen Worten: Nachdem sich die Bakterien auf die optimale Lösung zubewegt und Nachkommen produziert haben, werden genetische Operatoren eingesetzt, um die Lösungen zu diversifizieren und zu verbessern.
2. Der Algorithmus
Der BFO-GA-Algorithmus nutzt die Modellierung des Verhaltens von schwärmenden Bakterien, um die optimale Lösung für ein Optimierungsproblem zu finden. Es simuliert die Bewegung von Bakterien im Parameterraum, wobei jedes Bakterium eine mögliche Lösung für ein Problem darstellt. Bakterien bewegen sich durch den Parameterraum, indem sie zwei Hauptbewegungsarten ausführen: Bewegung in Richtung eines Nährstoffgradienten und Bewegung in eine zufällige Richtung.
Der BFO-GA-Algorithmus umfasst die folgenden Schritte:
- Initialisierung: Schaffung einer Anfangspopulation von Bakterien, von denen jede eine potenzielle Lösung für ein Problem darstellt. Jedes Bakterium hat seine eigenen Parameter, die während des Optimierungsprozesses geändert werden können.
- Schätzung des Nährstoffgradienten: Die Berechnung des Werts der Fitnessfunktion für jedes Bakterium in einer Population und der anschließende Vergleich der beiden letzten Werte, wobei der Unterschied zwischen den Werten die Qualität der von jedem Bakterium präsentierten Lösung bestimmt.
- Bewegung in Richtung des Gefälles: Jedes Bakterium bewegt sich in Richtung des Nahrungsgradienten, wodurch es sich mit einem konstanten Vektor der Positionsveränderung zu optimaleren Lösungen hinbewegen kann.
- Bewegung in eine zufällige Richtung: Einige Bakterien bewegen sich auch nach dem Zufallsprinzip, um zu vermeiden, dass sie in lokalen Optima stecken bleiben. Diese Bewegung basiert auf einer zufälligen Änderung der Parameter des Bakteriums im Falle einer vorherigen erfolglosen Bewegung.
- Genetische Operatoren: Anwendung der genetischen Operatoren - Selektion, Kreuzung und Mutation - auf eine Bakterienpopulation. Dadurch können wir neue Parameterkombinationen schaffen und die Qualität der Lösungen verbessern.
- Wiederholung der Schritte 2-5: Wiederholung der Schritte der Bewegung in der Gradientenrichtung, der Bewegung in der Zufallsrichtung und der Anwendung genetischer Operatoren, bis ein Stoppkriterium erreicht ist, z. B. das Erreichen der maximalen Anzahl von Iterationen oder das Erreichen eines bestimmten Fitnessfunktionswertes.
Theoretisch sollte der hybride BFO-GA-Algorithmus eine Reihe von Vorteilen gegenüber BFO haben, die erwähnenswert sind:
- Erkundung und Verwertung: BFO-GA ist in der Lage, den Parameterraum durch die zufällige Bewegung der Bakterien zu erkunden und durch die Bewegung in Richtung des Nahrungsgradienten auszunutzen. Dadurch kann der Algorithmus optimale Lösungen finden, indem er lokale Optima vermeidet und zu einem globalen Optimum konvergiert.
- Anpassungsfähigkeit: BFO-GA hat die Fähigkeit, sich an veränderte Optimierungsbedingungen anzupassen. Während des Betriebs des Algorithmus können sich die Parameter der Bakterien ändern, wodurch sie sich an neue Bedingungen anpassen und optimalere Lösungen finden können.
- Eine Parametereinstellung ist nicht erforderlich: Wie jeder Optimierungsalgorithmus verfügt auch BFO-GA über eine Reihe von Parametern, die angepasst werden können, um bessere Ergebnisse zu erzielen. Dazu gehören Parameter in Bezug auf die Größe der Bakterienpopulation, Bewegungsschritte in Richtung des Gradienten und zufällige Bewegungen, Wahrscheinlichkeiten der genetischen Operatoren und andere. Die Änderung der Parameter hat keinen signifikanten Einfluss auf das Ergebnis.

Abbildung 1. Vererbung der elterlichen „Gene“ durch die Tochterbakterien
Kommen wir nun von den theoretischen Grundlagen zur praktischen Umsetzung.
Zunächst habe ich die Auswahl nach der Roulette-Methode aufgegeben (die Methode wurde im Algorithmus der künstlichen Bienenkolonie verwendet). Die Versuche ergaben bessere Ergebnisse bei der BFO-GA mit der Verwendung von einfachen Zufallsstichproben aus einer elterlichen Population.
Zweitens beschloss ich, den uneinheitlichen Mihaljevic-Mutator durch eine Potenzgesetz-Mutation zu ersetzen (in einigen Quellen wird er als eine Art Potenzgesetz-Mutator bezeichnet). Es handelt sich dabei um die Erzeugung einer Zufallszahl mit einer Potenzgesetzverteilung, die in dem Artikel über Smart Cephalopod erwähnt wird.
Drittens wird der arithmetische Crossover-Operator durch eine einfache zufällige Vererbung von Genen verschiedener Elternteile ersetzt, die nach dem Zufallsprinzip gemäß einer Gleichverteilungsregel ausgewählt werden.
Um ein Bakterium zu beschreiben, schreiben wir die Struktur S_Bacteria, die die folgenden Felder enthält:
- c: Array von Bakterienkoordinaten stellt die aktuellen Koordinaten der Bakterien im Parameterraum dar. Die Größe des Arrays „c“ hängt von der Anzahl der Variablen im Optimierungsproblem ab.
- cLast: Array der vorherigen Koordinaten des Bakteriums, um die vorherige Position des Bakteriums im Parameterraum zu speichern, aus dem die neue Position berechnet wird.
- v: Array von Bakterienvektoren, die die aktuelle Bewegungsrichtung der Bakterien im Parameterraum darstellen. Die Größe des Arrays „v“ hängt von der Anzahl der Variablen im Optimierungsproblem ab.
- f: Der aktuelle Wert der Gesundheit des Bakteriums (Fitness) bestimmt die Qualität der aktuellen Position des Bakteriums im Parameterraum. Je größer der „f“-Wert, desto besser.
- fLast: Früherer Gesundheitswert des Bakteriums, der zur Ermittlung der früheren Qualität der Position des Bakteriums dient und bei der Bestimmung des Nährstoffgradienten verwendet wird.
- lifeCNT: Der Lebenszähler der Bakterien zeigt die Anzahl der Iterationen an, die das Bakterium an seiner aktuellen Position verbracht hat. Das Feld wird verwendet, um die Anzahl der Schritte zu begrenzen, die Bakterien in einer Richtung machen können.
//—————————————————————————————————————————————————————————————————————————————— struct S_Bacteria { double c []; //coordinates double cLast []; //coordinates double v []; //vector double f; //current health double fLast; //previous health double lifeCNT; //life counter }; //——————————————————————————————————————————————————————————————————————————————
Wir deklarieren die Klasse C_AO_BFO_GA, die den BFO-GA-Algorithmus implementiert. Schauen wir uns die einzelnen Felder und Klassenmethoden an:
Felder der Klasse:
- b: Ist ein Array von Bakterien. Jedes Element des Arrays ist ein Objekt vom Typ „S_Bacteria“, das ein Bakterium im Algorithmus darstellt.
- rangeMax: Array der maximalen Suchbereichswerte für jede Variable im Optimierungsproblem.
- rangeMin: Array der minimalen Suchbereichswerte für jede Variable.
- rangeStep: Array von Suchschritten für jede Variable.
- cB: Feld der besten vom Algorithmus gefundenen Koordinaten.
- fB: Wert der Fitnessfunktion für die besten Koordinaten.
Methoden der Klasse:
- Init: Initialisiert die Parameter des BFO-GA-Algorithmus, wie „paramsP“ (Anzahl der optimierten Parameter), „populationSizeP“ (Populationsgröße), „lambda“ (Bewegungslänge der Bakterien), „reproductionP“ (Reproduktionswahrscheinlichkeit), „lifeCounterP“ (Lebenszähler) - wenn die Anzahl der Leben erreicht ist, machen die Bakterien einen Salto, und „powerMutP“ (Mutationskraft).
- Swimming: Ermöglicht die Bewegung von Bakterien im Parameterraum.
- Evaluation: Überarbeitung der Population und Aktualisierung der Gesamtlösung.
Private Methoden der Klasse:
- NewVector: Erzeugt einen neuen Vektor für den angegebenen Bakterienparameter „paramInd“.
- PowerDistribution: Wendet eine Leistungsverteilung auf den In-Eingang mit den angegebenen Parametern „outMin“, „outMax“ und „power“ an.
- Sorting: Sortiert die Bakterien in einer Population nach ihren Fitnessfunktionswerten in absteigender Reihenfolge.
- SeInDiSp: Normalisiert die Eingabe-Variablen im Intervall [InMin, InMax] mit „Step“.
- RNDfromCI: Erzeugt eine Zufallszahl in einem bestimmten Intervall [min, max].
- Scale: Skaliert die Eingabe-Variable vom Intervall [InMIN, InMAX] zum Intervall [OutMIN, OutMAX].
//—————————————————————————————————————————————————————————————————————————————— class C_AO_BFO_GA { //---------------------------------------------------------------------------- public: S_Bacteria b []; //bacteria public: double rangeMax []; //maximum search range public: double rangeMin []; //manimum search range public: double rangeStep []; //step search public: double cB []; //best coordinates public: double fB; //FF of the best coordinates public: void Init (const int paramsP, //number of opt. parameters const int populationSizeP, //population size const double lambdaP, //lambda const double reproductionP, //probability of reproduction const int lifeCounterP, //life counter const double powerMutP); //mutation power public: void Swimming (); public: void Evaluation (); //---------------------------------------------------------------------------- private: double NewVector (int paramInd); private: S_Bacteria bT []; //bacteria private: double v []; private: int ind []; private: double val []; private: int populationSize; //population size private: int parameters; //number of optimized parameters private: double lambda; //lambda private: double reproduction; //probability of reproduction private: int lifeCounter; //life counter private: double powerMut; //mutation power private: bool evaluation; private: double PowerDistribution (const double In, const double outMin, const double outMax, const double power); private: void Sorting (); private: double SeInDiSp (double In, double InMin, double InMax, double Step); private: double RNDfromCI (double min, double max); private: double Scale (double In, double InMIN, double InMAX, double OutMIN, double OutMAX, bool revers); }; //——————————————————————————————————————————————————————————————————————————————
Die Init-Methode wird zur Initialisierung der Klasse verwendet. Die Methode initialisiert die Parameter des BFO-GA-Algorithmus.
Eingaben zur Methode:
- paramsP: Anzahl der optimierten Parameter.
- populationSizeP: Größe der Population.
- lambdaP: Der Parameter lambda, der die Entfernung bestimmt, die die Bakterien bei jedem Schritt zurücklegen.
- reproductionP: Reproduktionswahrscheinlichkeit.
- lifeCounterP: Lebenszähler.
- powerMutP: Mutationsleistung.
Innerhalb der Methode werden die Felder der Klasse C_AO_BFO_GA mit Anfangswerten initialisiert und die Arraygrößen festgelegt.
//—————————————————————————————————————————————————————————————————————————————— void C_AO_BFO_GA::Init (const int paramsP, //number of opt. parameters const int populationSizeP, //population size const double lambdaP, //lambda const double reproductionP, //probability of reproduction const int lifeCounterP, //life counter const double powerMutP) //mutation power { fB = -DBL_MAX; evaluation = false; parameters = paramsP; populationSize = populationSizeP; lambda = lambdaP; reproduction = reproductionP; lifeCounter = lifeCounterP; powerMut = powerMutP; ArrayResize (rangeMax, parameters); ArrayResize (rangeMin, parameters); ArrayResize (rangeStep, parameters); ArrayResize (v, parameters); ArrayResize (ind, populationSize); ArrayResize (val, populationSize); ArrayResize (b, populationSize); ArrayResize (bT, populationSize); for (int i = 0; i < populationSize; i++) { ArrayResize (b [i].c, parameters); ArrayResize (b [i].cLast, parameters); ArrayResize (b [i].v, parameters); b [i].f = -DBL_MAX; b [i].fLast = -DBL_MAX; ArrayResize (bT [i].c, parameters); ArrayResize (bT [i].cLast, parameters); ArrayResize (bT [i].v, parameters); } ArrayResize (cB, parameters); } //——————————————————————————————————————————————————————————————————————————————
Wir werden die Methode Swimming schreiben, um bakterielle Chemotaxis, Replikation, Selektion, Merkmalsvererbung und Mutation durchzuführen:
void Swimming ()
{
} Bei der ersten Iteration ist das Flag „evaluation“ gleich „false“. Wir verteilen die Bakterien zufällig mit einer Gleichverteilung über den gesamten Suchraum. In diesem Stadium sind der aktuelle Gesundheitszustand (Fitness) und der vorherige unbekannt. Weisen wir den DBL_MAX-Wert den entsprechenden Variablen zu. Außerdem fügen wir einen zufälligen Bewegungsvektor zu den neu geschaffenen Bakterien hinzu.
Wir überprüfen die Bakterienkoordinaten auf Überschreitung der Grenzen und führen den Schritt der Änderung der optimierten Parameter durch. Der Bewegungsvektor jedes Bakteriums ermöglicht es ihnen, gleichmäßig vorwärts zu schwimmen.
if (!evaluation) { fB = -DBL_MAX; for (int s = 0; s < populationSize; s++) { for (int k = 0; k < parameters; k++) { b [s].c [k] = RNDfromCI (rangeMin [k], rangeMax [k]); b [s].c [k] = SeInDiSp (b [s].c [k], rangeMin [k], rangeMax [k], rangeStep [k]); v [k] = rangeMax [k] - rangeMin [k]; b [s].v [k] = NewVector (k); b [s].f = -DBL_MAX; b [s].fLast = -DBL_MAX; b [s].lifeCNT = 0; } } evaluation = true; return; }
Die Wahrscheinlichkeit der Vermehrung (Replikation) von Bakterien wird erst bei der zweiten und den folgenden Iterationen überprüft. Wenn die Fortpflanzungswahrscheinlichkeit erfüllt ist, geschieht Folgendes:
- „bacterium original“: Die beste Hälfte der sortierten Bakterienpopulation setzt ihre Bewegung in dieselbe Richtung fort. Zu diesem Zweck fügen wir den Koordinaten der vorherigen Position einen für jedes Bakterium individuellen Bewegungsvektor hinzu.
- „bacterium clone“: Die zweite Hälfte der Population besteht aus Tochterbakterien, die aus den „Genen“ (Koordinaten) verschiedener Elternbakterien gewonnen werden. Auf diese Weise erben sie Eigenschaften und erfüllen die kombinatorischen Fähigkeiten des Algorithmus (siehe Abbildung 1 oben).
- Geklonte Bakterien, die Gene von verschiedenen Eltern geerbt haben, erben auch eine Komponente des Bewegungsvektors der Eltern, und das geerbte Gen unterliegt einer Mutation gemäß einer Potenzgesetzverteilung.
Die geklonten Bakterien erben also die Eigenschaften der verschiedenen Eltern. In diesem Fall verändern sich die Gene mit hoher Wahrscheinlichkeit in ihrer Umgebung. Die Wahrscheinlichkeit nimmt mit zunehmender Entfernung von den Werten des Elterngens ab. Auf diese Weise können wir die erfolgreichen Eigenschaften der Eltern kombinieren, denn nur die besten Mitglieder der Population können ihre Gene an ihre Erben weitergeben. Außerdem bewirken die vererbten Komponenten des Bewegungsvektors, dass sich die Tochterbakterien in der nächsten Iteration entsprechend den besten Komponenten der elterlichen Vektoren bewegen.
Im Idealfall sollte dies der Bewegung eines Fischschwarms ähneln, aber diese Analogie lässt sich aufgrund des Einflusses vieler Zufallsfaktoren auf die Bewegung von Bakterien nicht immer bestätigen. Zu diesen Faktoren gehören die Fitnesslandschaft, die Ersetzung durch glücklichere Mitglieder der Population und die unvermeidliche Änderung des Bewegungsvektors aufgrund der Begrenzung der Anzahl der Leben.
Der Lebenszähler der Bakterien in der besseren Hälfte der Kolonie erhöht sich um eins, während er bei geklonten Bakterien zurückgesetzt wird.
double r = RNDfromCI (0.0, 1.0); if (r < reproduction) { int st = populationSize / 2; //bacterium original-------------------------------------------------------- for (int s = 0; s < st; s++) { for (int k = 0; k < parameters; k++) { b [s].c [k] = b [s].cLast [k] + b [s].v [k]; b [s].c [k] = SeInDiSp (b [s].c [k], rangeMin [k], rangeMax [k], rangeStep [k]); } b [s].lifeCNT++; } //bacterium clone----------------------------------------------------------- for (int s = st; s < populationSize; s++) { for (int k = 0; k < parameters; k++) { int i = (int)RNDfromCI (0, st); if (i >= st) i = st - 1; b [s].c [k] = b [i].cLast [k]; b [s].c [k] = PowerDistribution (b [s].c [k], rangeMin [k], rangeMax [k], powerMut); b [s].c [k] = SeInDiSp (b [s].c [k], rangeMin [k], rangeMax [k], rangeStep [k]); } b [s].lifeCNT = 0; } }
Wenn die Reproduktionswahrscheinlichkeit nicht erfüllt ist, wird die Schleife für die gesamte Population wiederholt:
for (int s = 0; s < populationSize; s++)
Zunächst wird geprüft, ob die Lebensdauer der Bakterien erreicht ist. Wenn die Bakterien die Grenze ihrer Lebensdauer erreicht haben, schlagen sie Purzelbäume und beginnen, sich in eine neue Richtung zu bewegen, indem sie ihren Bewegungsvektor ändern. Der Alterszähler wird zurückgesetzt.
Nachdem geprüft wurde, ob die Bakterien die Lebensgrenze erreicht haben, mutieren diejenigen, die sie erreicht haben, und bei der nächsten Iteration beginnen sie, sich in eine neue Richtung zu bewegen, indem sie ihren Bewegungsvektor ändern. Der Alterszähler wird zurückgesetzt.
if (b [s].lifeCNT >= lifeCounter) { for (int k = 0; k < parameters; k++) { b [s].v [k] = NewVector (k); b [s].c [k] = PowerDistribution (b [s].cLast [k], rangeMin [k], rangeMax [k], powerMut); b [s].c [k] = SeInDiSp (b [s].c [k], rangeMin [k], rangeMax [k], rangeStep [k]); } b [s].lifeCNT = 0; }
Wenn das Bakterium seine Lebensgrenze noch nicht erreicht hat, prüfen wir, ob eine positive Chemotaxis vorliegt, d. h. ob sich das Bakterium in Richtung eines zunehmenden Nahrungsgradienten bewegt. Die Bewegung des Bakteriums gilt als korrekt, wenn die Fitnesswerte in den beiden vorherigen Iterationen gleich sind. Warum genau gleich? In der nächsten Phase, der Evaluierungsmethode, prüfen wir, ob sich die Gesundheit der Bakterien positiv verändert hat. Wenn die Änderungen positiv sind, wird der aktuelle Zustand dem vorherigen Zustand zugeordnet. Wenn die Gesundheitszustände in den letzten beiden Iterationen gleich sind, deutet dies darauf hin, dass die Richtung der Bewegung der Bakterien auf der Suche nach mehr Nahrung richtig ist. Das Bakterium kann sich also nur in Richtung mehr Nahrung bewegen. Andernfalls beginnen die Bakterien an Ort und Stelle zu taumeln. Dies ist jedoch kein Problem, da das festsitzende Bakterium früher oder später entweder zu einem neuen Zustand mutiert oder die Gene von glücklicheren Eltern und deren Bewegungsvektor erbt.
Der Lebenszähler von Bakterien, die sich in die richtige Richtung bewegen, steigt allmählich an. Das bedeutet, dass selbst erfolgreiche Bakterien früher oder später Mutationen durchlaufen, wie im vorigen Codeblock erwähnt.
if (b [s].f == b [s].fLast) { for (int k = 0; k < parameters; k++) { b [s].c [k] = b [s].cLast [k] + b [s].v [k]; b [s].c [k] = SeInDiSp (b [s].c [k], rangeMin [k], rangeMax [k], rangeStep [k]); } b [s].lifeCNT++; }
Wird bei einem Bakterium keine positive Chemotaxis beobachtet, so ändert das Bakterium seinen Bewegungsvektor und macht einen Purzelbaum. Auch der Lebenszähler solcher Bakterien tickt weiter. Es kam die Idee auf, den Wert des Lebenszählers solcher erfolglosen Bakterien intensiver zu erhöhen, obwohl ich diese Idee nicht getestet habe.
for (int k = 0; k < parameters; k++) { b [s].v [k] = NewVector (k); b [s].c [k] = b [s].cLast [k] + b [s].v [k]; b [s].c [k] = SeInDiSp (b [s].c [k], rangeMin [k], rangeMax [k], rangeStep [k]); } b [s].lifeCNT++;
Um einen Purzelbaum (Änderung des Bewegungsvektors) durchzuführen, verwenden Sie die Methode NewVector, die für den optimierten Parameter mit dem Index „paramInd“ ausgeführt wird:
- eine Zufallszahl „r“ wird mit Hilfe der Funktion „RNDfromCI“ mit einer Gleichverteilung im Intervall [-1,0, 1,0] erzeugt.
- wird ein neuer Wert der Vektorkomponente berechnet, indem der zulässige Wertebereich „v“ des optimierten Parameters mit dem Verhältnis „lambda“ und der Zufallszahl „r“ multipliziert wird.
//—————————————————————————————————————————————————————————————————————————————— double C_AO_BFO_GA::NewVector (int paramInd) { double r = RNDfromCI (-1.0, 1.0); return lambda * v [paramInd] * r; } //——————————————————————————————————————————————————————————————————————————————Die Bewertungsmethode wird verwendet, um den Wettbewerb zwischen den Bakterien zu regeln und die globale Lösung zu aktualisieren.
Zu Beginn der Methode wird jedes Bakterium in der Population auf positive Chemotaxis getestet: Wenn der aktuelle Wert der Fitnessfunktion „b[s].f“ größer ist als der vorherige Wert „b[s].fLast“, werden die vorherige Fitness und die Koordinaten (von denen aus sich die Bakterien bewegen werden) aktualisiert.
Die Population wird dann mit der Sortiermethode in absteigender Reihenfolge der Fitnessfunktionswerte anhand des fLast-Wertes der Bakterien sortiert.
Danach wird die globale Lösung aktualisiert.
//—————————————————————————————————————————————————————————————————————————————— void C_AO_BFO_GA::Evaluation () { for (int s = 0; s < populationSize; s++) { if (b [s].f > b [s].fLast) { b [s].fLast = b [s].f; ArrayCopy (b [s].cLast, b [s].c, 0, 0, WHOLE_ARRAY); } } Sorting (); if (b [0].fLast > fB) { fB = b [0].fLast; ArrayCopy (cB, b [0].cLast, 0, 0, WHOLE_ARRAY); } } //——————————————————————————————————————————————————————————————————————————————
3. Testergebnisse
Testergebnisse von BFO-GA:
C_AO_BFO_GA:50;0.01;0.8;50;10.0
=============================
5 Hilly's; Func runs: 10000; result: 0.8914957961234459
25 Hilly's; Func runs: 10000; result: 0.5511088047221568
500 Hilly's; Func runs: 10000; result: 0.31529201642392934
=============================
5 Forest's; Func runs: 10000; result: 0.9698155977381523
25 Forest's; Func runs: 10000; result: 0.39612322805995287
500 Forest's; Func runs: 10000; result: 0.06304955092899359
=============================
5 Megacity's; Func runs: 10000; result: 0.7266666666666667
25 Megacity's; Func runs: 10000; result: 0.275
500 Megacity's; Func runs: 10000; result: 0.035250000000000004
=============================
All score: 4.22380 (46.93%)
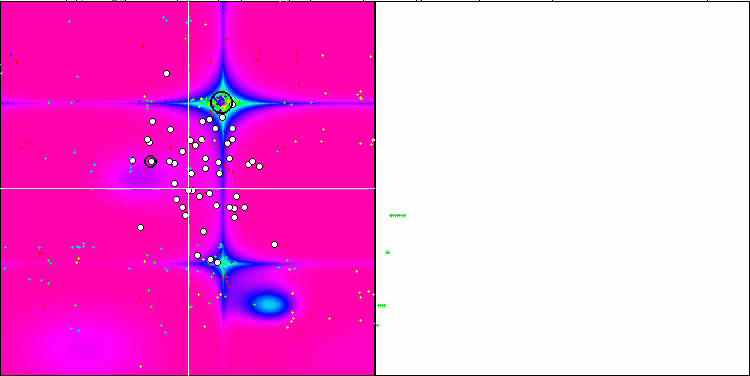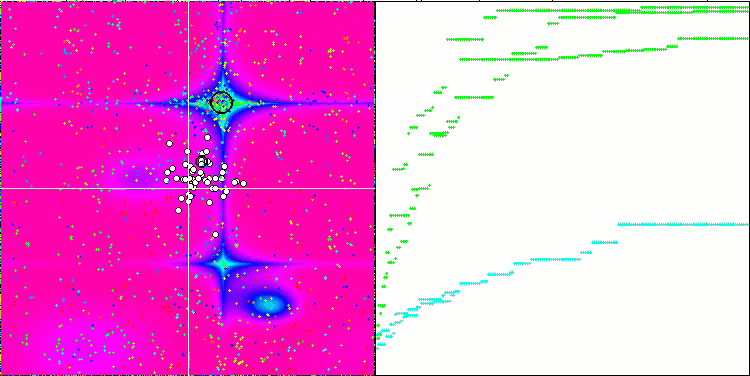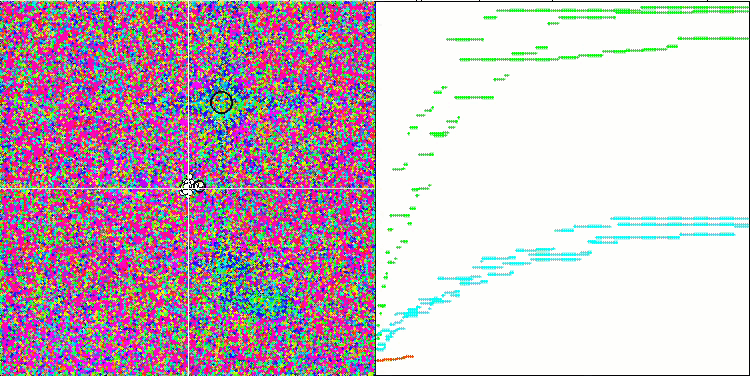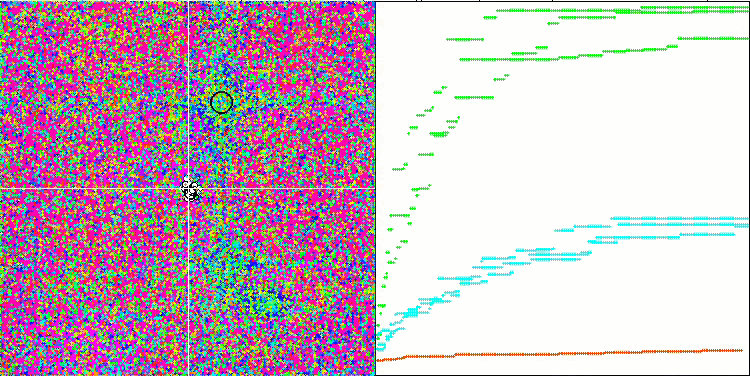
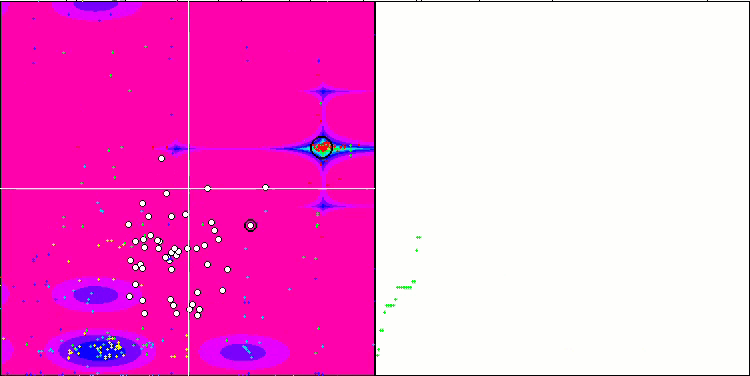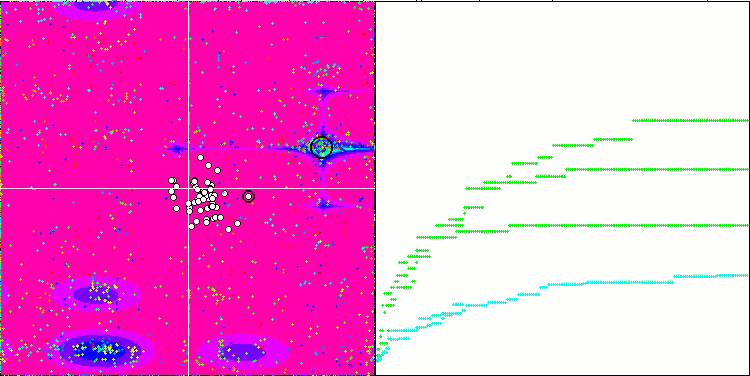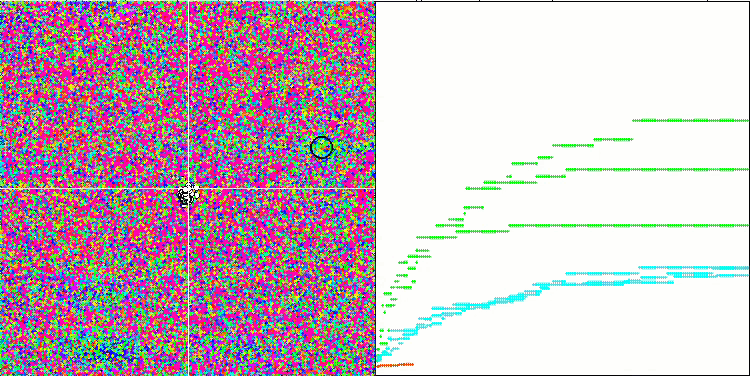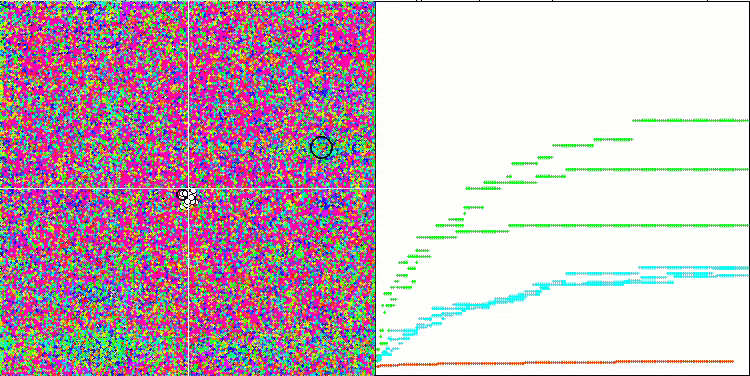
Eine visuelle Analyse des Tests zeigt, dass der BFO-GA-Algorithmus den Bereich des globalen Extremums schnell erkennt, während er der Untersuchung lokaler Extrema weniger Aufmerksamkeit schenkt, was dazu führen könnte, dass er stecken bleibt, wenn sich das globale Extremum als fehlerhaft erweist. Im Allgemeinen konvergiert der Algorithmus recht zuverlässig, wenn auch etwas langsam. Es wird ein gewisses „Schwärmen“ beobachtet, was ein gutes Zeichen ist, aber es gibt keine vollständige Kommunikation zwischen den Bakterien, und sie verhalten sich als unabhängige Populationseinheiten.
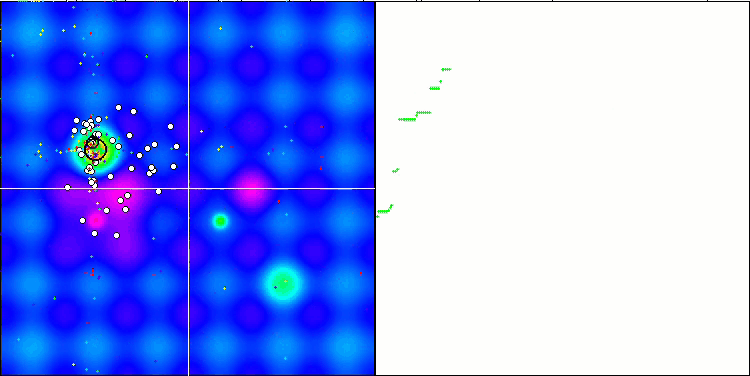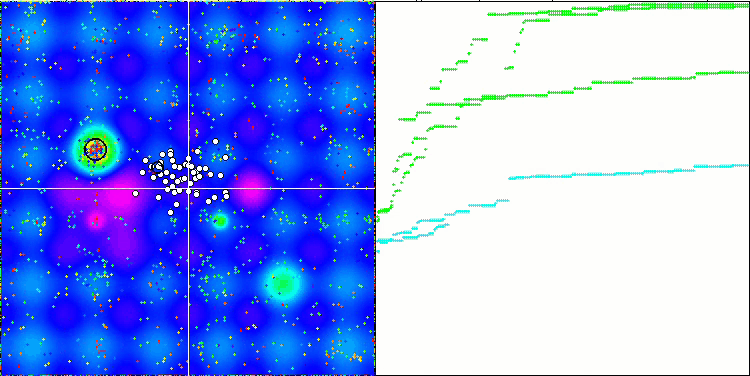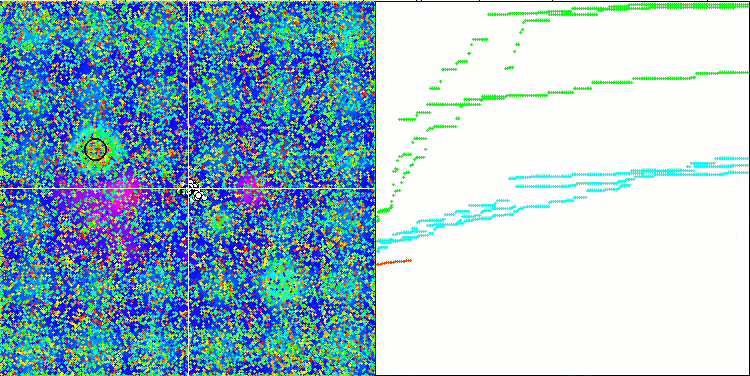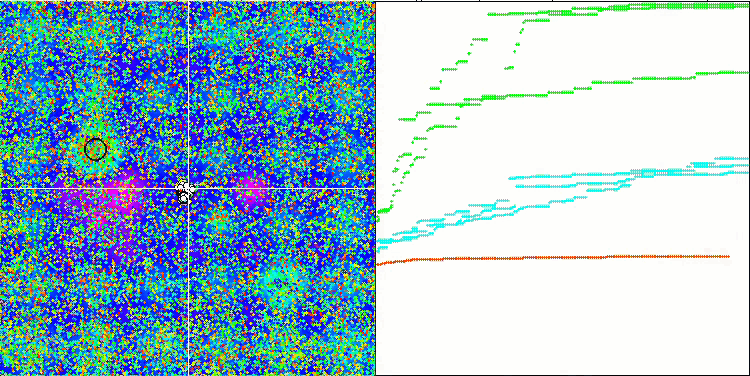
BFO-GA mit der Testfunktion Hilly
BFO-GA mit der Testfunktion Forest
BFO-GA mit der Testfunktion Megacity
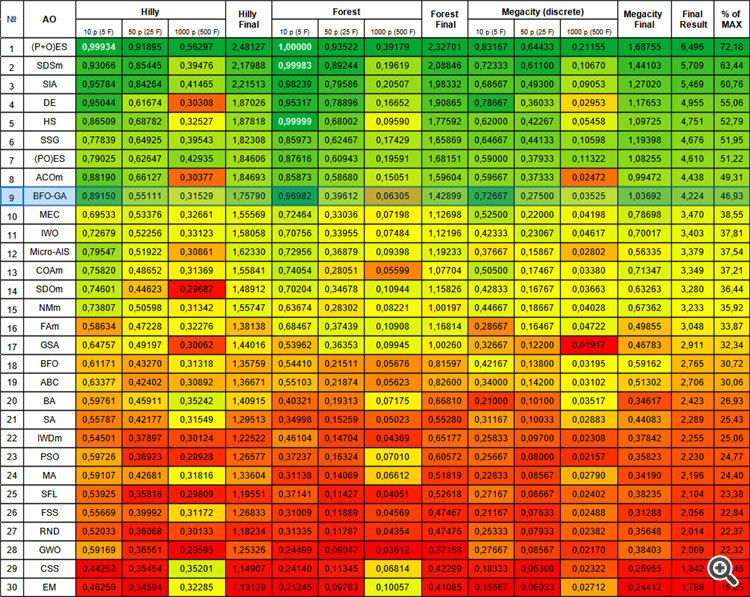
In der Gesamtbewertungstabelle nimmt der BFO-GA-Algorithmus den 9. Platz ein, was ein recht gutes Ergebnis ist. Dies zeigt, dass die Veränderungen, die beim Hinzufügen von Operatoren des genetischen Algorithmus zum ursprünglichen BFO-Algorithmus vorgenommen wurden, nicht umsonst waren und zu angemessenen Ergebnissen führten. Dieser Algorithmus zeigte überwiegend die besten Ergebnisse bei Testfunktionen mit einer kleinen Anzahl von Variablen und übertraf damit die meisten anderen Algorithmen.
| # | AO | Beschreibung | Hilly | Hilly final | Forest | Forest final | Megacity (discrete) | Megacity final | Final result | % of MAX | ||||||
| 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | ||||||||
| 1 | (P+O)ES | (P+O) Entwicklungsstrategien | 0.99934 | 0.91895 | 0.56297 | 2.48127 | 1.00000 | 0.93522 | 0.39179 | 2.32701 | 0.83167 | 0.64433 | 0.21155 | 1.68755 | 6.496 | 72.18 |
| 2 | SDSm | stochastische Diffusionssuche M | 0.93066 | 0.85445 | 0.39476 | 2.17988 | 0.99983 | 0.89244 | 0.19619 | 2.08846 | 0.72333 | 0.61100 | 0.10670 | 1.44103 | 5.709 | 63.44 |
| 3 | SIA | Simuliertes isotropes Abkühlen | 0.95784 | 0.84264 | 0.41465 | 2.21513 | 0.98239 | 0.79586 | 0.20507 | 1.98332 | 0.68667 | 0.49300 | 0.09053 | 1.27020 | 5.469 | 60.76 |
| 4 | DE | differentielle Evolution | 0.95044 | 0.61674 | 0.30308 | 1.87026 | 0.95317 | 0.78896 | 0.16652 | 1.90865 | 0.78667 | 0.36033 | 0.02953 | 1.17653 | 4.955 | 55.06 |
| 5 | HS | Harmoniesuche | 0.86509 | 0.68782 | 0.32527 | 1.87818 | 0.99999 | 0.68002 | 0.09590 | 1.77592 | 0.62000 | 0.42267 | 0.05458 | 1.09725 | 4.751 | 52.79 |
| 6 | SSG | Setzen, Säen und Wachsen | 0.77839 | 0.64925 | 0.39543 | 1.82308 | 0.85973 | 0.62467 | 0.17429 | 1.65869 | 0.64667 | 0.44133 | 0.10598 | 1.19398 | 4.676 | 51.95 |
| 7 | (PO)ES | (PO) Entwicklungsstrategien | 0.79025 | 0.62647 | 0.42935 | 1.84606 | 0.87616 | 0.60943 | 0.19591 | 1.68151 | 0.59000 | 0.37933 | 0.11322 | 1.08255 | 4.610 | 51.22 |
| 8 | ACOm | Ameisen-Kolonie-Optimierung M | 0.88190 | 0.66127 | 0.30377 | 1.84693 | 0.85873 | 0.58680 | 0.15051 | 1.59604 | 0.59667 | 0.37333 | 0.02472 | 0.99472 | 4.438 | 49.31 |
| 9 | BFO-GA | Optimierung der bakteriellen Futtersuche - ga | 0.89150 | 0.55111 | 0.31529 | 1.75790 | 0.96982 | 0.39612 | 0.06305 | 1.42899 | 0.72667 | 0.27500 | 0.03525 | 1.03692 | 4.224 | 46.93 |
| 10 | MEC | Evolutionäre Berechnung des Geistes | 0.69533 | 0.53376 | 0.32661 | 1.55569 | 0.72464 | 0.33036 | 0.07198 | 1.12698 | 0.52500 | 0.22000 | 0.04198 | 0.78698 | 3.470 | 38.55 |
| 11 | IWO | Optimierung mit invasiven Unkräutern | 0.72679 | 0.52256 | 0.33123 | 1.58058 | 0.70756 | 0.33955 | 0.07484 | 1.12196 | 0.42333 | 0.23067 | 0.04617 | 0.70017 | 3.403 | 37.81 |
| 12 | Micro-AIS | Künstliches Mikro-Immunsystem | 0.79547 | 0.51922 | 0.30861 | 1.62330 | 0.72956 | 0.36879 | 0.09398 | 1.19233 | 0.37667 | 0.15867 | 0.02802 | 0.56335 | 3.379 | 37.54 |
| 13 | COAm | Kuckuck-Optimierungsalgorithmus M | 0.75820 | 0.48652 | 0.31369 | 1.55841 | 0.74054 | 0.28051 | 0.05599 | 1.07704 | 0.50500 | 0.17467 | 0.03380 | 0.71347 | 3.349 | 37.21 |
| 14 | SDOm | Optimierung der Spiraldynamik M | 0.74601 | 0.44623 | 0.29687 | 1.48912 | 0.70204 | 0.34678 | 0.10944 | 1.15826 | 0.42833 | 0.16767 | 0.03663 | 0.63263 | 3.280 | 36.44 |
| 15 | NMm | Nelder-Mead-Verfahren M | 0.73807 | 0.50598 | 0.31342 | 1.55747 | 0.63674 | 0.28302 | 0.08221 | 1.00197 | 0.44667 | 0.18667 | 0.04028 | 0.67362 | 3.233 | 35.92 |
| 16 | FAm | Firefly-Algorithmus M | 0.58634 | 0.47228 | 0.32276 | 1.38138 | 0.68467 | 0.37439 | 0.10908 | 1.16814 | 0.28667 | 0.16467 | 0.04722 | 0.49855 | 3.048 | 33.87 |
| 17 | GSA | Algorithmus für die Schwerkraftsuche | 0.64757 | 0.49197 | 0.30062 | 1.44016 | 0.53962 | 0.36353 | 0.09945 | 1.00260 | 0.32667 | 0.12200 | 0.01917 | 0.46783 | 2.911 | 32.34 |
| 18 | BFO | Optimierung der bakteriellen Futtersuche | 0.61171 | 0.43270 | 0.31318 | 1.35759 | 0.54410 | 0.21511 | 0.05676 | 0.81597 | 0.42167 | 0.13800 | 0.03195 | 0.59162 | 2.765 | 30.72 |
| 19 | ABC | Künstliches Bienenvolk (Artificial Bee Colony, ABC) | 0.63377 | 0.42402 | 0.30892 | 1.36671 | 0.55103 | 0.21874 | 0.05623 | 0.82600 | 0.34000 | 0.14200 | 0.03102 | 0.51302 | 2.706 | 30.06 |
| 20 | BA | Fledermaus-Algorithmus | 0.59761 | 0.45911 | 0.35242 | 1.40915 | 0.40321 | 0.19313 | 0.07175 | 0.66810 | 0.21000 | 0.10100 | 0.03517 | 0.34617 | 2.423 | 26.93 |
| 21 | SA | simuliertes Abkühlen | 0.55787 | 0.42177 | 0.31549 | 1.29513 | 0.34998 | 0.15259 | 0.05023 | 0.55280 | 0.31167 | 0.10033 | 0.02883 | 0.44083 | 2.289 | 25.43 |
| 22 | IWDm | intelligente Wassertropfen M | 0.54501 | 0.37897 | 0.30124 | 1.22522 | 0.46104 | 0.14704 | 0.04369 | 0.65177 | 0.25833 | 0.09700 | 0.02308 | 0.37842 | 2.255 | 25.06 |
| 23 | PSO | Partikelschwarmoptimierung | 0.59726 | 0.36923 | 0.29928 | 1.26577 | 0.37237 | 0.16324 | 0.07010 | 0.60572 | 0.25667 | 0.08000 | 0.02157 | 0.35823 | 2.230 | 24.77 |
| 24 | MA | Affen-Algorithmus | 0.59107 | 0.42681 | 0.31816 | 1.33604 | 0.31138 | 0.14069 | 0.06612 | 0.51819 | 0.22833 | 0.08567 | 0.02790 | 0.34190 | 2.196 | 24.40 |
| 25 | SFL | schlurfender Froschsprung | 0.53925 | 0.35816 | 0.29809 | 1.19551 | 0.37141 | 0.11427 | 0.04051 | 0.52618 | 0.27167 | 0.08667 | 0.02402 | 0.38235 | 2.104 | 23.38 |
| 26 | FSS | Fischschulsuche | 0.55669 | 0.39992 | 0.31172 | 1.26833 | 0.31009 | 0.11889 | 0.04569 | 0.47467 | 0.21167 | 0.07633 | 0.02488 | 0.31288 | 2.056 | 22.84 |
| 27 | RND | zufällig | 0.52033 | 0.36068 | 0.30133 | 1.18234 | 0.31335 | 0.11787 | 0.04354 | 0.47476 | 0.25333 | 0.07933 | 0.02382 | 0.35648 | 2.014 | 22.37 |
| 28 | GWO | Grauer-Wolf-Optimierung | 0.59169 | 0.36561 | 0.29595 | 1.25326 | 0.24499 | 0.09047 | 0.03612 | 0.37158 | 0.27667 | 0.08567 | 0.02170 | 0.38403 | 2.009 | 22.32 |
| 29 | CSS | Suche geladener Systeme | 0.44252 | 0.35454 | 0.35201 | 1.14907 | 0.24140 | 0.11345 | 0.06814 | 0.42299 | 0.18333 | 0.06300 | 0.02322 | 0.26955 | 1.842 | 20.46 |
| 30 | EM | elektromagnetismusähnlicher Algorithmus | 0.46250 | 0.34594 | 0.32285 | 1.13129 | 0.21245 | 0.09783 | 0.10057 | 0.41085 | 0.15667 | 0.06033 | 0.02712 | 0.24412 | 1.786 | 19.85 |
Zusammenfassung
Wir sehen deutliche Verbesserungen der BFO-GA-Ergebnisse im Vergleich zum ursprünglichen BFO-Algorithmus, was auf die Verwendung der Operatoren des genetischen Algorithmus zurückzuführen ist: Auswahl, Mutation und Vererbung. BFO verfügte bisher nicht über Mechanismen zum Verlassen von lokalen Extremen, und dieses Problem ist nun nicht mehr vorhanden. Der Algorithmus bietet enorme Möglichkeiten zum Experimentieren, indem er die Reihenfolge der Komponenten der bakteriellen Suchstrategie und der GA-Operatoren kombiniert. Viele von Ihnen habe noch nicht ausprobiert.
Im BFO-Algorithmus ist mir früher ein Fehler bei der Berechnung der Anzahl der Bakterienleben unterlaufen, der nun korrigiert wurde. BFO verbesserte seine Ergebnisse leicht und rückte eine Zeile über ABC auf. Ich möchte anmerken, dass es beim Schreiben von Optimierungsalgorithmen schwierig ist, Fehler im Code und in der Logik der Suchstrategie zu erkennen. Selbst wenn Fehler auftreten, setzt der Algorithmus die Suche fast immer fort. Fehler beeinträchtigen nicht immer die Ergebnisse. Manchmal verbessern sich die Ergebnisse erheblich und der „Fehler“ wird Teil der Strategie. Bei Optimierungsalgorithmen sollte man immer bedenken, dass große Änderungen in der Logik nicht immer zu signifikanten Verbesserungen der Ergebnisse führen, während kleine Änderungen manchmal zu dramatischen Verbesserungen führen können. Die korrigierte BFO-Version finden Sie im Archiv.
Bild 2. Farbabstufung der Algorithmen gemäß den einschlägigen Tests
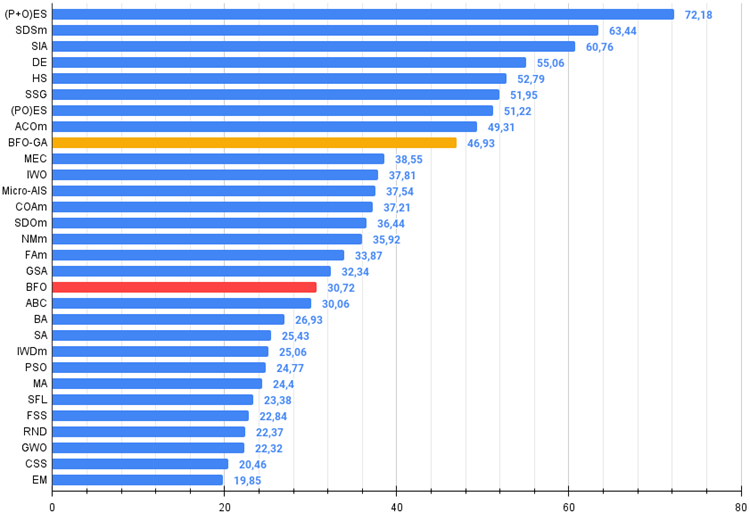
Abb. 3. Das Histogramm der Algorithmus-Testergebnisse (auf einer Skala von 0 bis 100, größer ist besser,
wobei 100 das höchstmögliche, theoretische Ergebnis ist das Archiv enthält ein Skript zur Berechnung der Bewertungstabelle)
Vor- und Nachteile der BFO-GA:
- Algorithmus mit hoher Arbeitsgeschwindigkeit.
- Einfache Umsetzung.
- Gute Konvergenz bei Funktionen mit einer kleinen Anzahl von Parametern.
- Geringe Konvergenz bei Aufgaben mit einem großen Suchraum.
Dem Artikel ist ein Archiv mit aktualisierten Versionen der in früheren Artikeln beschriebenen Algorithmuscodes beigefügt. Der Autor des Artikels übernimmt keine Verantwortung für die absolute Richtigkeit der Beschreibung der kanonischen Algorithmen. An vielen von ihnen wurden Änderungen vorgenommen, um die Suchmöglichkeiten zu verbessern. Die in den Artikeln dargelegten Schlussfolgerungen und Urteile beruhen auf den Ergebnissen der Versuche.
Übersetzt aus dem Russischen von MetaQuotes Ltd.
Originalartikel: https://www.mql5.com/ru/articles/14011
 The Disagreement Problem: Diving Deeper into The Complexity Explainability in AI
The Disagreement Problem: Diving Deeper into The Complexity Explainability in AI
 Neural networks made easy (Part 68): Offline Preference-guided Policy Optimization
Neural networks made easy (Part 68): Offline Preference-guided Policy Optimization
 Advanced Variables and Data Types in MQL5
Advanced Variables and Data Types in MQL5
 Data label for time series mining (Part 6):Apply and Test in EA Using ONNX
Data label for time series mining (Part 6):Apply and Test in EA Using ONNX
- Freie Handelsapplikationen
- Über 8.000 Signale zum Kopieren
- Wirtschaftsnachrichten für die Lage an den Finanzmärkte
Sie stimmen der Website-Richtlinie und den Nutzungsbedingungen zu.