
Neuroredes profundas (Parte IV). Creación, entrenamiento y simulación de un modelo de neurored
Índice
- Introducción
- 1 Breve descripción de las posibilidades del paquete
- 1.1. Funciones de inicialización de las neuronas
- 1.2. Funciones de activación de las neuronas
- 1.3. Métodos de entrenamiento
- 1.4. Métodos de regularización y estabilización
- 1.5. Métodos y parámetros de entrenamiento de un RBM
- 1.6. Métodos y parámetros de entrenamiento de una DNN
- 2. Comprobando la calidad del funcionamiento de una DNN dependiendo de los parámetros utilizados
- 2.1. Experimentos
- 2.1.1. Datos de entrada (preparación)
- 2.1.2. Modelo básico de comparación
- 2.1.3. Estructura de una DNN
- 2.1.4. Variantes de entrenamiento
- 2.2. Análisis de resultados
- Conclusión
- Anexos
Introducción
Principales vertientes de investigación y aplicación
En la actualidad, en la investigación y aplicación de redes neuronales profundas se han formado dos tendencias principales. Estas se diferencian en la aproximación a la inicialización de los pesos de las neuronas en las capas ocultas.
Enfoque № 1. Las redes neuronales son extremadamente sensibles al método de inicialización de las neuronas en las capas ocultas, especialmente al aumentar el número de capas ocultas (más de 3). El primero en intentar resolver este problema fue el profesor G.Hynton. La esencia de su propuesta era la siguiente: iniciar un peso de neuronas en las capas ocultas con los pesos obtenidos en el entrenamiento no supervisado de redes autoasociativas, compuestas a partir de una RBM (máquina de Boltzmann restringida) o un AE (autoencoder). Estas Stacked RBM (SRBM) y Stacked AE (SAE) se entrenan con una gran matriz de datos no marcados. El objetivo de este entrenamiento es detectar las estructuras (presentaciones, formas) ocultas y las dependencias en los datos. La inicialización de neuronas con pesos MLP, obtenidos durante el pre-entrenamiento, ubica el MLP en el espacio de soluciones más cercano al óptimo. Esto da la posibilidad de reducir el número de datos marcados y épocas de entrenamiento en el caso de realizar un ajuste preciso (entrenamiento) adecuado del MLP. Para muchas zonas de aplicación práctica (especialmente al procesar "grandes datos") nos encontramos ante ventajas de enorme importancia.
Enfoque № 2: Otro grupo de científicos, bajo el mando de Yoshua Benjio, ha desarrollado métodos específicos de inicialización de neuronas ocultas, funciones especiales de activación, métodos de estabilización y entrenamiento. Los éxitos en este plano se relacionan con el desarrollo vertiginoso de las neuroredes convolucionales profundas y las neuroredes recurrentes (DCNN, RNN). Estas neuroredes han mostrado una efectividad máxima en el reconocimiento de imágenes, el análisis y la clasificación de textos y la traducción del habla viva de un idioma a otro. Las ideas y métodos desarrollados para estas neuroredes han comenzado a cambiar con éxito también para la MLP.
Hoy en día, ambas vertientes se usan de forma activa en la práctica. Sin embargo, las neuroredes con pre-entrenamiento necesitan menos ejemplos para el entrenamiento y menos recursos computacionales, con resultados prácticamente idénticos. Esta es una ventaja muy importante. Nosotros nos inclinamos por el uso de neuroredes profundas con pre-entrenamiento. En nuestra opinión, el entrenamiento no supervisado es el futuro.
Paquetes en el lenguaje R que permiten desarrollar y usar DNN en la práctica
Para crear y usar DNN con diferente nivel de complejidad y diferentes posibilidades en el lenguaje R se ha desarrollado una serie de paquetes.
Paquetes que permiten crear, entrenar y probar DNN con pre-entrenamiento:
- deepnet — es un paquete sencillo, no está saturado con multitud de parámetros y ajustes. Permite crear tanto neuroredes con pre-entrenamiento SAE, como SRBM. En el artículo anterior hemos analizado la implementación práctica de expertos usando este paquete. El uso de RBM para el pre-entrenamiento da resultados menos estables. El paquete se ha diseñado para tener un primer contacto con el tema y estudiar las peculiaridades del funcionamiento de estas neuroredes. Si su uso se enfoca de forma adecuada, podrá utilizarse en los expertos. RcppDL — versión de este paquete (un poco recortada) en С++.
- darch
v.0.12 — paquete complejo, flexible, con multitud de parámetros, modos y ajustes (los valores por defecto recomendados
ya vienen establecidos). Permite construir y ajustar una neurored de cualquier complejidad y configuración. Para el
pre-entrenamiento se usa SRBM. El paquete se ha pensado para los usuarios preparados. Más abajo hablamos con detalle de sus
posibilidades.
Paquetes que permiten crear, entrenar y probar DNN sin pre-entrenamiento:
- H2O — paquete para procesar "grandes datos" (>1M líneas y >1K columnas). La neurored utilizada en él dispone de un desarrollado sistema de regularización. Las posibilidades de nuestra esfera de aplicación son excesivas, pero esto no nos impide utilizarlo.
- mxnetpermite
crear, no solo MLP, sino también redes recurrentes complejas, convulucionales y LSTM. El paquete dispone de API en varios
lenguajes, incluidos R y Python. La ideología se diferencia mucho de las enumeradas más arriba. Esto es así porque, en esencia, los
desarrolladores han escrito los paquetes para Python. El paquete mxnet para R ha sido simplificado y tiene una funcionalidad
ligeramente menor en comparación con el paquete para Python. Pero este hecho no menoscaba sus ventajas.
En el entorno de Python, el tema de la redes recurrentes y profundas ha sido ampliamente desarrollado. Existen muchos paquetes interesantes para construir tales tipos de neuroredes, que en R simplemente no existen. Aquí tenemos los paquetes R que permiten ejecutar los programas/módulos descritos para Python:
- PythonInR y reticulate son dos paquetes que dan la posibilidad de ejecutar cualquier código de Python en R. Para ello, deberá tener instalado Python 2/3 en su computadora.
- kerasr— es una interfaz R para la popular biblioteca de aprendizaje profundo keras.
- tensorflow — paquete que proporciona acceso a TensorFow API completo en el entorno R.
No hace mucho, la compañía Microsoft colgó la biblioteca cntk v.2.1(Computatinal Network Toolkit) GitHub, y ahora puede ser utilizada como backend para Keras, con el que R "se lleva bien". Sería desable ponerlo a prueba en nuestras tareas.
Yándex tampoco se queda atrás: en open source ha colgado su propia biblioteca CatBoost. Con su ayuda se puede entrenar de forma efectiva un modelo con datos heterogéneos, incluidos los datos que difícilmente se pueden imaginar en forma de números (por ejemplo, tipos de nube o categorías de mercancías). El código fuente, la documentación, las referencias y las herramientas necesarias ya han sido publicadas en GitHub bajo la licencia Apache 2.0. A pesar de que no se trata de una neurored, sino de árboles mejorados, será necesario realizar una prueba del algoritmo, tanto más que con él se ha implementado una API de R.
1 Breve descripción de las posibilidades del paquete
El paquete darch ver. 0.12.0 ofrece un amplio surtido de funciones que permiten no solo crear y entrenar un modelo, sino también montarlo - literalmente - bloque a bloque y construirlo según sus preferencias. En comparación con la anterior versión del paquete (0.10.0), que ya analizamos en el artículo anterior, han tenido lugar cambios sustanciales. Se han añadido nuevas funciones de activación, inicialización y estabilización, pero, lo que es más importante, todo se ha hecho en una función darch(), que simultáneamente también es un constructor. Se da soporte al uso de mapas gráficos. Después de entrenar el modelo, la función retorna un objeto de clase DArch. La estructura del objeto se muestra en la fig.1. Las funciones predict() y darchTest() retornan el pronóstico según los nuevos datos o la métrica de la clasificación.

Fig. 1. Estructura del objeto DArch
Todos los parámetros tienen valores por defecto que, normalmente, no son óptimos. Toda esta montaña de parámetros se puede unir en tres grupos: comunes, para RBM y para NN. Más abajo analizaremos algunos de ellos con más detalle.
| Funciones | Tipos |
|---|---|
| Funciones de inicialización |
|
| Función de activación |
|
| Función de aprendizaje |
|
| Nivel de aprendizaje |
|
| Función de estabilización |
|
| Momentum |
|
| Condiciones de parada |
|
Una red profunda consta de un número n de RBM (n = layers -1), conectadas en una red autoasociativa (SRBM), y propiamente de la neurored MLP con un número layers de capas. El pre-entrenamiento capa por capa de RBM se realiza con datos no marcados. El entrenamiento preciso de la neurored se realiza de forma supervisada y con datos marcados.
La división de estos estados de entrenamiento con la ayuda de los parámetros nos da la posibilidad de usar datos de diferente volumen (¡pero no con diferente estructura!) u obtener varios modelos diferentes, entrenados con precisión sobre la base de un pre-entrenamiento. Además, si los datos para el pre-entrenamiento y el ajuste de precisión son iguales, será posible realizar el entrenamiento de una vez, sin dividirlo en dos estadios. Podemos saltarnos el pre-entrenamiento (rbm.numEpochs = 0; darch.numEpochs = 10)) y utilizar una red multicapa o, al contrario, entrenar solo RBM (rbm.numEpochs = 10; darch.numEpochs = 0). Además, tendremos acceso a todos los parámetros internos.
La neurored simplificada se podrá terminar de entrenar en lo sucesivo un número indefinido de veces con los nuevos datos. Son muy pocos los modelos capaces de ofrecer esta posibilidad. En la fig.2 se muestra el esquema estructural de una neurored profunda inicializada por máquinas restringidas de Bolzmann (DNRBM).
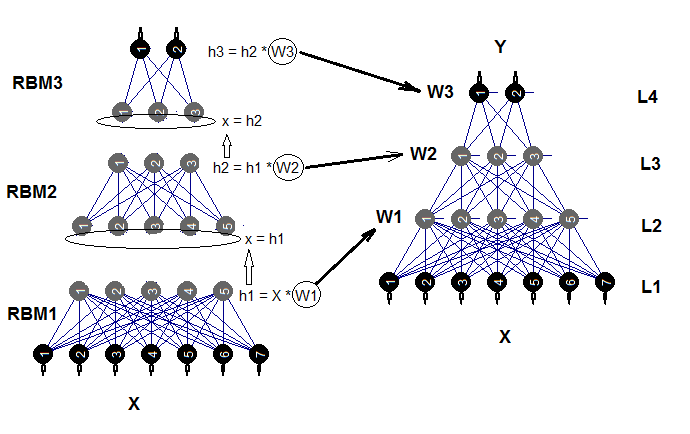
Fig.2. Esquema estructural de DNSRBM
1.1. Funciones de inicialización de las neuronas
En el paquete se usan dos funciones principales de inicialización de neuronas.
- generateWeightsUniform() usa la función runif(n, min, max) y se implementa de la forma que sigue:
> generateWeightsUniform function (numUnits1, numUnits2, weights.min = getParameter(".weights.min", -0.1, ...), weights.max = getParameter(".weights.max", 0.1, ...), ...) { matrix(runif(numUnits1 * numUnits2, weights.min, weights.max), nrow = numUnits1, ncol = numUnits2) } <environment: namespace:darch>
Aquí, numUnits1 es el número de neuronas en la capa anterior, y numUnits2, el número de neuronas en la capa actual.
- generateWeightsNormal() usa la función rnorm(n, mean, sd) y se implementa en el paquete así:
> generateWeightsNormal function (numUnits1, numUnits2, weights.mean = getParameter(".weights.mean", 0, ...), weights.sd = getParameter(".weights.sd", 0.01, ...), ...) { matrix(rnorm(numUnits1 * numUnits2, weights.mean, weights.sd), nrow = numUnits1, ncol = numUnits2) } <environment: namespace:darch>
Las otras cuatro funciones usan estas dos, pero definen min, max, mean y sd con fórmulas específicas. Podrá familiarizarse con ellas llamando en el terminal el nombre de la función sin parántesis.
1.2. Funciones de activación de las neuronas
Junto con las funciones de activación estándar, el paquete ofrece un amplio conjunto de nuevas funciones. Aquí tenemos algunas de ellas:
x <- seq(-5, 5, 0.1) par(mfrow = c(2,3)) plot(x, y = 1/(1 + exp(-x)), t = "l", main = "sigmoid") abline(v = 0, col = 2) plot(x, y = tanh(x), t = "l", main = "tanh") abline(v = 0, h = 0, col = 2) plot(x, y = log(1 + exp(x)), t = "l", main = "softplus"); abline(v = 0, col = 2) plot(x, y = ifelse(x > 0, x ,exp(x) - 1), t = "l", main = "ELU") abline(h = 0, v = 0, col = 2) plot(x, y = ifelse(x > 0, x , 0), t = "l", main = "ReLU") abline(h = 0, v = 0, col = 2) par(mfrow = c(1,1))
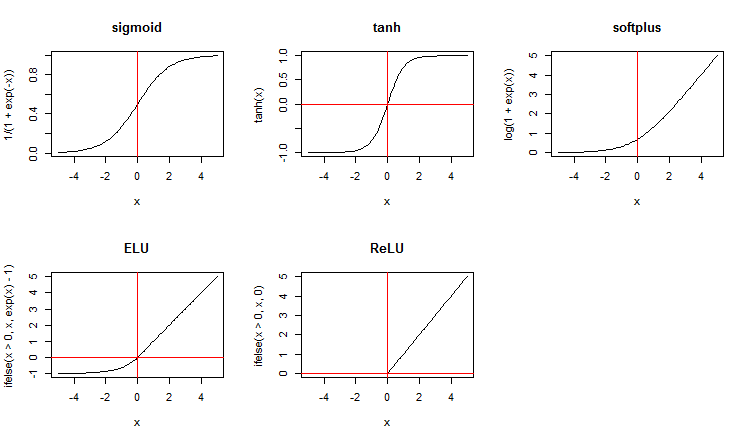
Fig.3. Funciones de activación de las neuronas
Vamos a analizar por separado la función de activación maxout. La función proviene de las redes convolucionales. La capa oculta de la neurored se divide en módulos del tamaño (poolSize). El número de neuronas en la capa oculta debe ser divisible por el tamaño de agregación (pool). Para el entrenamiento, se elige una neurona con una activación máxima del pool y luego se transmite a la entrada. La función de activación de las neuronas en el pool se indica por separado. Dicho de manera más simple, se trata de una capa doble (convolución + maxpooling) con posibilidades de salto de filtración limitadas. De acuerdo con las publicaciones, ofrece buenos resultados formando pareja con dropout. En la fig. 4. se muestra de forma esquemática una capa oculta con 8 neuronas y dos tamaños de pool.
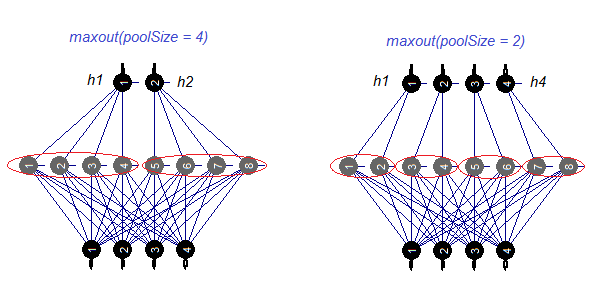
Fig.4. Función de activación maxout
1.3. Métodos de entrenamiento
Por desgracia, en el paquete se muestran solo dos métodos de entrenamiento: backpropagation y rprop , la versión básica y la mejorada con actualización de pesos al darse la propagación inversa y sin ella. Se ha previsto la posibilidad de entrenar con la ayuda del multiplicador bp.learnRateScale.
1.4. Métodos de regularización y estabilización
- dropout es un método de eliminación (regularización de pesos) de parte de las neuronas de la capa oculta en el entrenamiento. Las neuronas se regularizan en orden aleatorio con respecto al número de neuronas que deben ser eliminadas, y que se indica con el parámetro darch.dropout. El nivel de eliminación en cada capa oculta puede ser diferente. La máscara de eliminación se puede generar para cada mini-muestra (batch) o para cada época.
- dropconnect se relaciona con la desconexión entre parte de las neuronas de la capa actual y las neuronas de la capa anterior. Las conexiones se cortan en orden aleatorio con respecto al número de conexiones que deben ser cortadas, se indica con el mismo parámetro darch.dropout (normalmente, no superior a 0.5). Según ciertas publicaciones, muestra un resultado mejor que dropout.
- dither
es un método de prevención de re-entrenamiento por medio de la reducción (dithering) de los datos de entrada.
- weightDecay — el peso de cada neurona se multiplicará por (1 — weightDecay) antes de la acutalización.
- normalizeWeights es una manera de normalizar el vector de entrada de los pesos de las neuronas con una posible limitación desde arriba (L2 norm)
Los primeros tres métodos se aplican solo por separado.
1.5. Métodos y parámetros de entrenamiento de un RBM
Hay dos métodos de entrenamiento de SRBM: el entrenamiento de RBM uno a uno en rbm.numEpochs o el entrenamiento por turnos de cada RBM una época cada vez. La posibilidad de elegir uno de estos métodos se regula con la ayuda del parámetro rbm.consecutive: TRUE o default indicará el primer método, FALSE, el segundo. En la fig.5 se muestra el esquema de entrenamiento en las dos variantes. Con la ayuda del parámetro rbm.lastLayer podemos indicar en qué capa de SRBM hay que establecer el re-entrenamiento. Con 0 — entrenamos todas las capas, con (-1) — no entrenamos la capa superior. Esto tiene sentido, ya que la capa superior de una neurored se debe entrenar por separado y de forma más prolongada. El resto de los parámetros son comprensibles y no necesitan de aclaraciones adicionales.
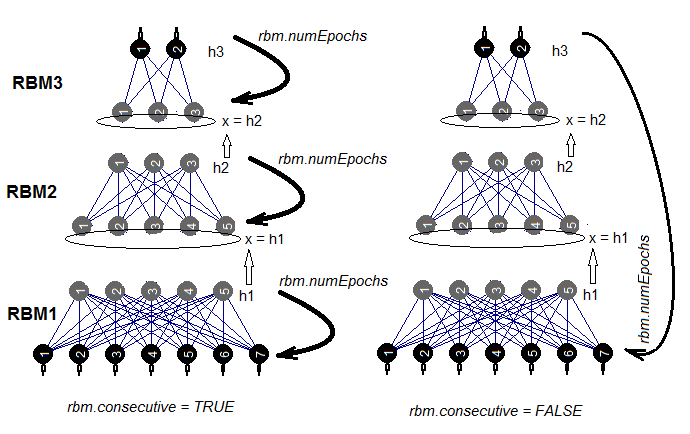
Fig.5. Dos métodos de entrenamiento de SRBM
1.6. Métodos y parámetros de entrenamiento de una DNN
Podemos entrenar DNN de dos formas: con pre-entrenamiento y sin él. Estos dos métodos se diferencian de forma cardinal el uno del otro en cuanto a los parámetros usados. Así, para entrenar con pre-entranemiento, no tiene sentido alguno usar los métodos específicos de inicialización y regularización. Lo que es más, su uso puede empeorar el resultado. El asunto es que después del pre-entrenamiento, los pesos de las neuronas de las capas ocultas se ubican en una zona cercana a los valores óptimos, y solo necesitarán un pequeño ajuste sutil. Y al contrario, para conseguir el mismo resultado, al entrenar sin pre-entrenamiento, se deberán usar todos los métodos disponibles de inicialización y regularización. Normalmente, el entrenamiento según este método ocupa más tiempo.
Bien, a nosotros nos interesa el entrenamiento con pre-entrenamiento. Normalmente, se divide en 2 etapas.
- Entrenamiento de SRBM con un gran volumen de datos no marcados. Los parámetros del pre-entrenamiento los indicaremos aparte. Como
resultado, obtendremos una neurored inicializada con los pesos de SRBM. A continuación, entrenamos la capa de la neurored con los
datos no marcados con nuestros propios parámetros de entrenamiento. Como resultado, obtenemos una neurored con una capa
superior entrenada y pesos iniciados en las capas inferiores. La guardamos como un objeto independiente para su uso
posterior.
- En la segunda etapa de ajuste preciso, usamos solo unos pocos ejemplos marcados, un nivel bajo de entrenamiento y un pequeño número de épocas de entrenamiento para todas las capas de la neurored, puliendo sutilmente la red. La red entrenada está lista.
La posibilidad de dividir las etapas del pre-entrenamiento, el entrenamiento preciso y los posteriores entrenamientos finales otorga una flexibilidad increíble en la construcción de algoritmos de entrenamiento no solo de una DNN, sino también, lo que es más importante, del entrenamiento de los comités de DNN. En la fig.6 se presentan unas cuantas variantes de entrenamiento de DNN y los componentes de DNN.
- Variante а: durante el ajuste preciso, guardamos DNN cada n épocas. De esta forma, obtenemos una serie de DNN con
diferente grado de entrenamiento. Podemos utilizar posteriormente cada una de esas redes por separado o como parte del comité. La
desventaja de esta opción es que todos las DNN se entrenan con los mismos datos, ya que los parámetros de entrenamiento son iguales
para todos ellos.
- Variante b: ajustamos con precisión una DNN de forma paralela con diferentes conjuntos de datos (ventana móvil, ventana de crecimiento, etcétera) y diferentes parámetros. Obtenemos un comité de DNN, que nos dará predicciones menos correlacionadas que la variante а.
- Variante c: ajustamos con precisión una DNN secuencialmente con diferentes conjuntos de datos y diferentes parámetros, guardando los modelos intermedios. En esto consiste precisamente el entrenamiento adicional: se puede realizar constantemente, mientras vayan llegando suficientes porciones de datos nuevos.
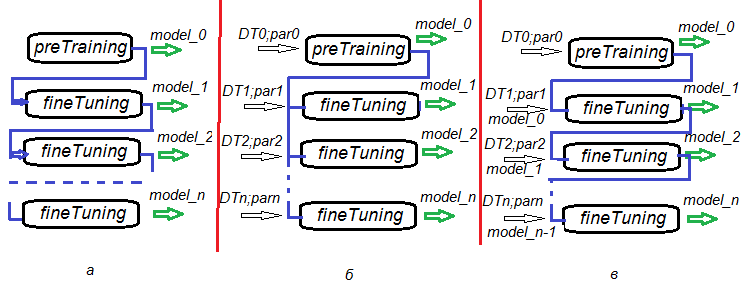
Fig.6. Variantes de entrenemiento de una DNN
2. Comprobación de la calidad del funcionamiento de una DNN, dependiendo de la calidad de los parámetros aplicados.
2.1. Experimentos
2.1.1. Datos de entrada (preparación)
Usaremos los datos y funciones de las anteriores partes del artículo (1, 2, 3). En ellas analizamos con bastante detalle diferentes variantes de pre-entrenamiento de datos. Por eso, aquí solo destacaremos brevemente las etapas de pre-entrenamiento que vamos a ejecutar. Los datos originales son OHLCV, los mismos que antes. Los datos de entrada son filtros digitales, los de salida, ZigZag. Podemos usar las funciones y la imagen del espacio de trabajo de Cotir.RData.
Las etapas de preparación de los datos que se ejecutarán están incluidas en funciones aparte:
- PrepareData() — creamos el conjunto inicial de datos dataSet, lo limpiamos de NA;
- SplitData() — dividimos el conjunto inicial dataSet en los subconjuntos pretrain, train, val, test;
- CappingData() — definimos e imputamos los datos atípicos en todos los subconjuntos.
Para ahorrar espacio en el artículo, no vamos a mostrar la lista de estas funciones. Se puede descargar de GitHub; además, ya han sido analizadas con detalle en las anteriores partes. Los resultados de ejecución los veremos más abajo. Asimismo, no analizaremos todos los métodos de transformación durante el pre-procesamiento. Muchos de ellos ya son ampliamente conocidos y utilizados. Nos detendremos en el menos conocido y más raramente usado: la discretización (con y sin supervisión). En la segunda parte del artículo se analizaron dos paquetes de discretización con supervisión (discretization y smbinning). En ellos se usan diferentes algoritmos para la discretización.
Vamos a analizar diferentes métodos de división de las variables continuas en rangos, y también veremos cómo usar estas variables discretizadas en los modelos.
¿Qué es el binning?
El binning es un término usado al modelar puntos (scoring modeling). Asimismo, en el aprendizaje de máquinas se conoce como Discretization al proceso de transformación de una variable continua en un número final de intervalos (rangos). Esto ayudará a comprender mejor su distribución y conexión con la variable objeto binaria. Los rangos (bins) creados en el proceso pueden convertirse en atributos de la característica predictora para usar modelos.
¿Por qué el binning?
A pesar del recelo que provoca el binning, este también tiene ventajas indiscutibles.
- Nos permite usar los datos ausentes (NA) y otros cálculos especiales (por ejemplo, divisiones por cero) para incluirlos en el modelo.
- Controla y mitiga el impacto de los datos atípicos en el modelo.
- Resuelva el problema de la presencia de diferentes escalas entre los predictores, haciendo comparables los coeficientes de peso en el modelo final.
Discretización sin supervisión
Unsupervised Discretization divide una función continua en rangos sin tener en cuenta cualquier otra información. Se trata de un apartado con dos opciones: intervalos de igual longitud e intervalos con igual frecuencia.
| Opción |
Objetivo |
Ejemplo |
Desventaja |
|---|---|---|---|
| Intervalos de igual longitud |
Comprensión de la distribución de la variable |
Histograma clásico, cuyos búnkeres tienen una misma longitud y pueden ser calculados con el uso de varias normas (Sturges, Rice,
etcétera) |
El número de entradas en el búnker puede ser demasiado pequeño para realizar el cálculo correctamente |
| Intervalos de igual frecuencia |
Analizar la interrelación con la vavirable binaria objetivo con la ayuda de índices tales como bad rate |
Quartilies o Percentiles | Los puntos de eliminación elegidos no pueden maximizar la diferencia entre los rangos al compararse con la variable
objetivo |
Discretización supervisada
La discretización supervisada divide una variable continua en rangos (bins) que son representados en la variable continua. La idea básica consiste en encontrar los puntos de división que maximizan la diferencia entre grupos.
Hoy, con ayuda de diferentes algoritmos, como por ejemplo ChiMerge o Recursive Partitioning, los analistas pueden encontrar rápidamente los puntos óptimos en segundos y valorar la interrelación con la variable objetivo, usando índices como el peso de los datos reales (Weight of Evidence, WoE) y el valor de la información (Information Value, IV).
El peso de los datos reales o WoE puede usarse como herramienta para transformar los predictores en la etapa de pre-procesado para los algoritmos de entrenamiento con supervisión. Al discretizar los predictores, podemos sustituirlos por nuevas variables nominales o bien por los valores de sus datos reales. La segunda variante es interesante porque permite apartarse de la transformación de las variables (factores) nominales en dummy, lo que proporciona, según algunos datos, un incremento positivo en forma de clasificación.
Los datos reales y el valor de la información (abreviado, IV) desempeñan dos papeles distintos al analizar los datos:
- El peso de los datos reales describe la interrelación entre la variable predictiva y la variable binaria objetivo.
- El valor de la información (IV) mide la fuerza de estas relaciones.
Vamos a analizar qué es precisamente el peso de los datos reales y el IV, en imágenes y fórmulas. Vamos a recordar el gráfico de la variable v.fatl, dividido en 10 segmentos de frecuencia equivalente en la segunda parte del artículo.
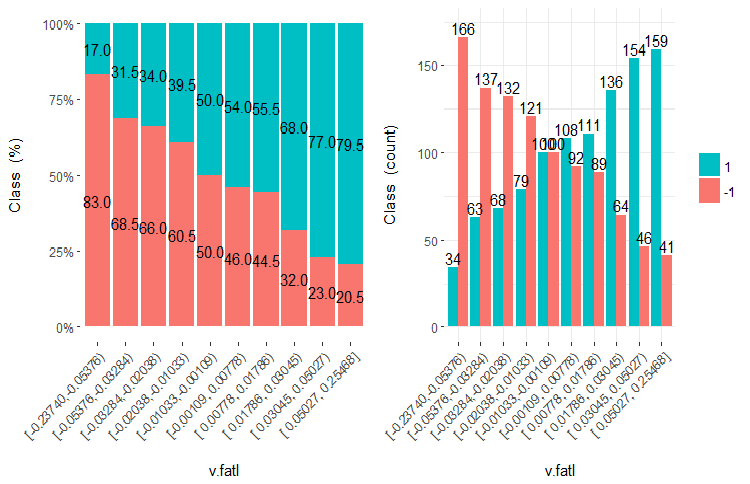
Fig.7. Variable v.fatl, dividida en 10 segmentos de la misma frecuencia
Capacidad predictiva de los datos (WOE)
Como podemos ver, en cada rango hay ejemplos que entran tanto en la clase "1", como en la clase "-1". La capacidad predictiva de los datos (WOE) WoEi se calcula según la fórmula
WoEi = ln(Gi/Bi)*100
donde:
Gi — es la frecuencia relativa de ejemplos "buenos" (en nuestro caso, "buenos" = "1") en cada rango de la variable;
Bi — es la frecuencia relativa de ejemplos "malos" (en nuestro caso, "malos" = "-1") en cada rango de la variable.
Si WoEi = 1, quiere decir que tenemos aproximadamente la misma cantidad de ejemplos "buenos" y "malos" en este rango, entonces la capacidad predictiva de este rango es igual a 0. Si hay más ejemplos "buenos", entonces WOE >0, y al contrario.
Valor informativo (IV)
Es la medida más extendida para definir la significación de las variables y la medición de la diferencia en la distribución de "buenos" y "malos" ejemplos. El valor de la información se define con la fórmula:
IV = ∑ (Gi – Bi) ln (Gi/Bi)
El valor de la información de una variables es igual a la suma de todos los rangos de la variable. En general, los valores de ese coeficiente pueden ser interpretados de la siguiente forma:
- menor a 0,02 — variable estadísticamente insignificante;
- 0,02 – 0,1 — variable estadísticamente poco significante;
- 0,1 – 0,3 — variable estadísticamente significante;
- 0,3 y superior — variable estadísticamente muy significante.
A continuación, usando diferentes algoritmos y criterios de optimización, los rangos se unen/separan para obtener la máxima diferencia de este criterio entre rangos. Por ejemplo, el paquete smbinning usa Recursive Partitioning para categorizar las variables numéricas y IV para definir los puntos de distribución óptimos, mientras que el paquete discretization resuelve esta tarea con la ayuda de ChiMerge y MDL. Asimismo, debemos recordar que los puntos de división son obtenidos con el conjunto de entranemiento, y los conjuntos de validación y de prueba, con su uso.
Hay varios paquetes que permiten de una u otra manera discretizar las variables numéricas. Son discretization, smbinning, Information, InformationValue y woebinning. Teniendo en cuenta el hecho de que debemos no solo discretizar el conjunto de entrenamiento, sino luego utilizar esta información para dividir los conjuntos de validación y pruebas, y también que es muy deseable controlar visualmente los resultados, he elegido el paquete woebinning.
El paquete divide de forma automática las variables numéricas y los factores y los liga a la variable objetivo binaria. Se contemplan dos enfoques:
- la implementación de la clasificación fina y bruta une secuencialmente clases y niveles granulados;
- el enfoque, semejante a un árbol, segmenta los rangos originales usando la división binaria.
Ambos procedimientos combinan rangos separados basados en valores análogos de WOE y se detienen sobre la base de los criterios basados en IV. El paquete se puede utilizar con una sola variable o con un frame de datos completo. Proporciona herramientas flexibles para explorar diversas soluciones de binning y para desplegarlas con nuevos datos.
Procedamos a los cálculos. En nuestro espacio de trabajo ya se han cargado las cotizaciones del terminal (o una imagen del entorno de trabajo de Cotir.RData de GitHub). Secuencia de cálculos y resultados:
- PrepareData() — creamos un conjunto inicial de datos dt[7906, 14], lo limpiamos de NA. El conjunto de datos incluye la etiqueta temporal Data, parámetros de entrada (12) y la variable objetivo Class (factor con dos niveles "-1" y "+1").
- SplitData() — dividimos el conjunto inicial de datos dt[] en los subconjuntos pretrain, train, val, test con una proporción 2000/1000/500/500, uniéndolos en un frame de datos DT[4, 4000, 14].
- CappingData() — definimos e importamos los valores atípicos en todos los subcojuntos, obtenemos el conjunto DTcap[4, 4000, 14]. Aunque la discretización es tolerante con los valores atípicos, los imputamos todos. También se puede experimentar sin este paso. Recordemos que los parámetros de los valores atípicos (pre.outl) se definen en el subconjunto pretrain. Procesamos las muestras train/val/test usando estos parámetros.
- NormData() — normalizamos el conjunto usando el método spatialSing del paquete caret. Al igual que sucede con la imputación de valores atípicos, los parámetros de normalización (preproc) se definen con el subconjunto pretrain. Las muestras train/val/test se procesan usando estos parámetros. Obtenemos DTcap.n[4, 4000, 14].
- DiscretizeData() — definimos los parámetros de discretización (preCut), la calidad de las variables y sus rangos para WOE y IV.
evalq({
dt <- PrepareData(Data, Open, High, Low, Close, Volume)
DT <- SplitData(dt, 2000, 1000, 500,500)
pre.outl <- PreOutlier(DT$pretrain)
DTcap <- CappingData(DT, impute = T, fill = T, dither = F,
pre.outl = pre.outl)
preproc <- PreNorm(DTcap, meth = meth)
DTcap.n <- NormData(DTcap, preproc = preproc)
preCut <- PreDiscret(DTcap.n)
}, env)
Vamos a reunir los datos de la discretización de todas las variables en un recuadro y a echarles un vistazo:
evalq(tabulate.binning <- woe.binning.table(preCut), env) > env$tabulate.binning $`WOE Table for v.fatl` Final.Bin Total.Count Total.Distr. 0.Count 1.Count 0.Distr. 1.Distr. 1.Rate WOE IV 1 <= -0.3904381926 154 7.7% 130 24 13.2% 2.4% 15.6% 171.3 0.185 2 <= -0.03713814085 769 38.5% 498 271 50.4% 26.8% 35.2% 63.2 0.149 3 <= 0.1130198981 308 15.4% 141 167 14.3% 16.5% 54.2% -14.5 0.003 4 <= Inf 769 38.5% 219 550 22.2% 54.3% 71.5% -89.7 0.289 6 Total 2000 100.0% 988 1012 100.0% 100.0% 50.6% NA 0.626 $`WOE Table for ftlm` Final.Bin Total.Count Total.Distr. 0.Count 1.Count 0.Distr. 1.Distr. 1.Rate WOE IV 1 <= -0.2344708291 462 23.1% 333 129 33.7% 12.7% 27.9% 97.2 0.204 2 <= -0.01368798447 461 23.1% 268 193 27.1% 19.1% 41.9% 35.2 0.028 3 <= 0.1789073635 461 23.1% 210 251 21.3% 24.8% 54.4% -15.4 0.005 4 <= Inf 616 30.8% 177 439 17.9% 43.4% 71.3% -88.4 0.225 6 Total 2000 100.0% 988 1012 100.0% 100.0% 50.6% NA 0.463 $`WOE Table for rbci` Final.Bin Total.Count Total.Distr. 0.Count 1.Count 0.Distr. 1.Distr. 1.Rate WOE IV 1 <= -0.1718377948 616 30.8% 421 195 42.6% 19.3% 31.7% 79.4 0.185 2 <= -0.09060410462 153 7.6% 86 67 8.7% 6.6% 43.8% 27.4 0.006 3 <= 0.3208178176 923 46.2% 391 532 39.6% 52.6% 57.6% -28.4 0.037 4 <= Inf 308 15.4% 90 218 9.1% 21.5% 70.8% -86.1 0.107 6 Total 2000 100.0% 988 1012 100.0% 100.0% 50.6% NA 0.335 $`WOE Table for v.rbci` Final.Bin Total.Count Total.Distr. 0.Count 1.Count 0.Distr. 1.Distr. 1.Rate WOE IV 1 <= -0.1837437563 616 30.8% 406 210 41.1% 20.8% 34.1% 68.3 0.139 2 <= 0.03581374495 461 23.1% 253 208 25.6% 20.6% 45.1% 22.0 0.011 3 <= 0.2503922644 461 23.1% 194 267 19.6% 26.4% 57.9% -29.5 0.020 4 <= Inf 462 23.1% 135 327 13.7% 32.3% 70.8% -86.1 0.161 6 Total 2000 100.0% 988 1012 100.0% 100.0% 50.6% NA 0.331 $`WOE Table for v.satl` Final.Bin Total.Count Total.Distr. 0.Count 1.Count 0.Distr. 1.Distr. 1.Rate WOE IV 1 <= -0.01840058612 923 46.2% 585 338 59.2% 33.4% 36.6% 57.3 0.148 2 <= 0.3247097195 769 38.5% 316 453 32.0% 44.8% 58.9% -33.6 0.043 3 <= 0.4003869443 154 7.7% 32 122 3.2% 12.1% 79.2% -131.4 0.116 4 <= Inf 154 7.7% 55 99 5.6% 9.8% 64.3% -56.4 0.024 6 Total 2000 100.0% 988 1012 100.0% 100.0% 50.6% NA 0.330 $`WOE Table for v.stlm` Final.Bin Total.Count Total.Distr. 0.Count 1.Count 0.Distr. 1.Distr. 1.Rate WOE IV 1 <= -0.4030051922 154 7.7% 118 36 11.9% 3.6% 23.4% 121.1 0.102 2 <= -0.1867821117 462 23.1% 282 180 28.5% 17.8% 39.0% 47.3 0.051 3 <= 0.1141896118 615 30.8% 301 314 30.5% 31.0% 51.1% -1.8 0.000 4 <= Inf 769 38.5% 287 482 29.0% 47.6% 62.7% -49.4 0.092 6 Total 2000 100.0% 988 1012 100.0% 100.0% 50.6% NA 0.244 $`WOE Table for pcci` Final.Bin Total.Count Total.Distr. 0.Count 1.Count 0.Distr. 1.Distr. 1.Rate WOE IV 1 <= -0.1738420887 616 30.8% 397 219 40.2% 21.6% 35.6% 61.9 0.115 2 <= -0.03163945242 307 15.3% 165 142 16.7% 14.0% 46.3% 17.4 0.005 3 <= 0.2553612644 615 30.8% 270 345 27.3% 34.1% 56.1% -22.1 0.015 4 <= Inf 462 23.1% 156 306 15.8% 30.2% 66.2% -65.0 0.094 6 Total 2000 100.0% 988 1012 100.0% 100.0% 50.6% NA 0.228 $`WOE Table for v.ftlm` Final.Bin Total.Count Total.Distr. 0.Count 1.Count 0.Distr. 1.Distr. 1.Rate WOE IV 1 <= -0.03697698898 923 46.2% 555 368 56.2% 36.4% 39.9% 43.5 0.086 2 <= 0.2437475615 615 30.8% 279 336 28.2% 33.2% 54.6% -16.2 0.008 3 <= Inf 462 23.1% 154 308 15.6% 30.4% 66.7% -66.9 0.099 5 Total 2000 100.0% 988 1012 100.0% 100.0% 50.6% NA 0.194 $`WOE Table for v.rftl` Final.Bin Total.Count Total.Distr. 0.Count 1.Count 0.Distr. 1.Distr. 1.Rate WOE IV 1 <= -0.1578370554 616 30.8% 372 244 37.7% 24.1% 39.6% 44.6 0.060 2 <= 0.1880959621 768 38.4% 384 384 38.9% 37.9% 50.0% 2.4 0.000 3 <= 0.3289762494 308 15.4% 129 179 13.1% 17.7% 58.1% -30.4 0.014 4 <= Inf 308 15.4% 103 205 10.4% 20.3% 66.6% -66.4 0.065 6 Total 2000 100.0% 988 1012 100.0% 100.0% 50.6% NA 0.140 $`WOE Table for stlm` Final.Bin Total.Count Total.Distr. 0.Count 1.Count 0.Distr. 1.Distr. 1.Rate WOE IV 1 <= -0.4586732186 154 7.7% 60 94 6.1% 9.3% 61.0% -42.5 0.014 2 <= -0.1688696056 462 23.1% 266 196 26.9% 19.4% 42.4% 32.9 0.025 3 <= 0.2631157075 922 46.1% 440 482 44.5% 47.6% 52.3% -6.7 0.002 4 <= 0.3592235072 154 7.7% 97 57 9.8% 5.6% 37.0% 55.6 0.023 5 <= 0.4846279843 154 7.7% 81 73 8.2% 7.2% 47.4% 12.8 0.001 6 <= Inf 154 7.7% 44 110 4.5% 10.9% 71.4% -89.2 0.057 8 Total 2000 100.0% 988 1012 100.0% 100.0% 50.6% NA 0.122 $`WOE Table for v.rstl` Final.Bin Total.Count Total.Distr. 0.Count 1.Count 0.Distr. 1.Distr. 1.Rate WOE IV 1 <= -0.4541701981 154 7.7% 94 60 9.5% 5.9% 39.0% 47.3 0.017 2 <= -0.3526306487 154 7.7% 62 92 6.3% 9.1% 59.7% -37.1 0.010 3 <= -0.2496412214 154 7.7% 53 101 5.4% 10.0% 65.6% -62.1 0.029 4 <= -0.08554320418 307 15.3% 142 165 14.4% 16.3% 53.7% -12.6 0.002 5 <= 0.360854678 923 46.2% 491 432 49.7% 42.7% 46.8% 15.2 0.011 6 <= Inf 308 15.4% 146 162 14.8% 16.0% 52.6% -8.0 0.001 8 Total 2000 100.0% 988 1012 100.0% 100.0% 50.6% NA 0.070 $`WOE Table for v.pcci` Final.Bin Total.Count Total.Distr. 0.Count 1.Count 0.Distr. 1.Distr. 1.Rate WOE IV 1 <= -0.4410911486 154 7.7% 92 62 9.3% 6.1% 40.3% 41.9 0.013 2 <= -0.03637567714 769 38.5% 400 369 40.5% 36.5% 48.0% 10.5 0.004 3 <= 0.1801156117 461 23.1% 206 255 20.9% 25.2% 55.3% -18.9 0.008 4 <= 0.2480148615 154 7.7% 84 70 8.5% 6.9% 45.5% 20.6 0.003 5 <= 0.3348752487 154 7.7% 67 87 6.8% 8.6% 56.5% -23.7 0.004 6 <= 0.4397404288 154 7.7% 76 78 7.7% 7.7% 50.6% -0.2 0.000 7 <= Inf 154 7.7% 63 91 6.4% 9.0% 59.1% -34.4 0.009 9 Total 2000 100.0% 988 1012 100.0% 100.0% 50.6% NA 0.042
En el recuadro para cada variable se muestran los resultados:
- Final.Bin — límites de los rangos;
- Total.Count — número total de ejemplos en el rango;
- Total.Distr — número relativo de ejemplos;
- 0.Count — número de ejemplos relacionados con la clase "0";
- 1.Count — número de ejemplos relacionados con la clase "1";
- 0.Distr — número relativo de ejemplos relacionados con la clase "0";
- 1.Distr — número relativo de ejemplos relacionados con la clase "1";
- 1.Rate — porcentaje del número de ejemplos de la clase "1" con respecto al número de ejemplos de la clase "0";
- WOE — capacidad predictiva de los rangos;
- IV — significación estadística de los rangos (¡acumulativa!).
Gráficamente, resultará más obvio. Mostramos los gráficos WOE de todas las variables de forma creciente de los IV basados en este recuadro:
> evalq(woe.binning.plot(preCut), env)
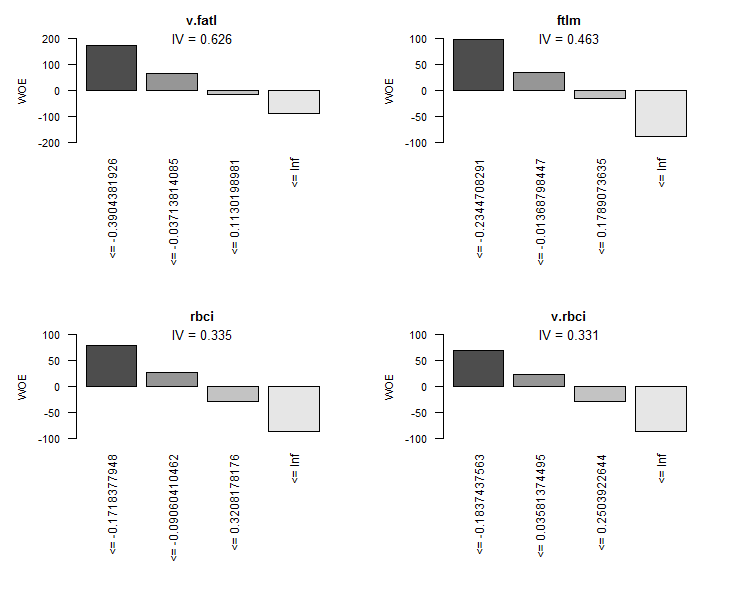
Fig.8. WOE de las 4 mejores variables
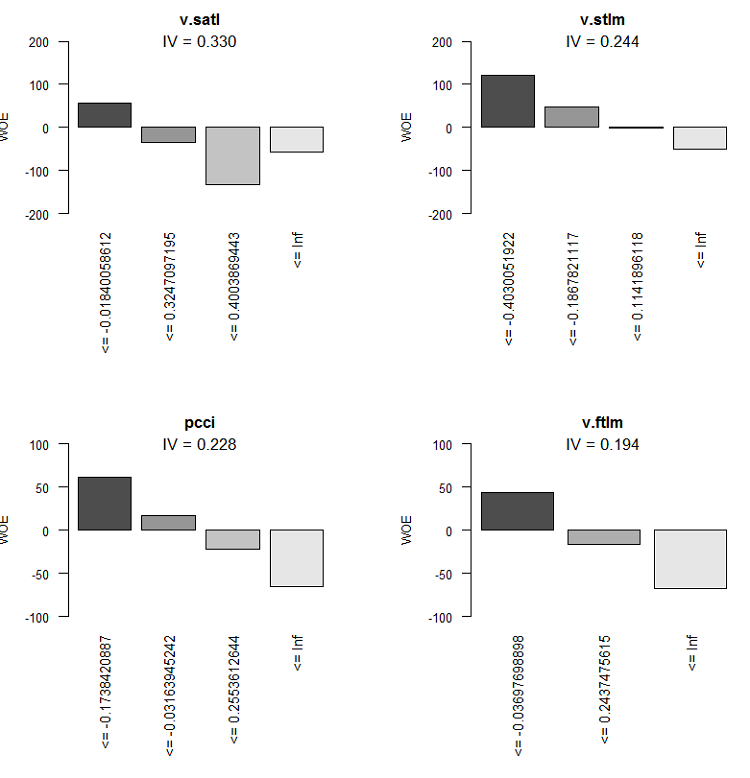
Fig.9. WOE de las variables 5-8
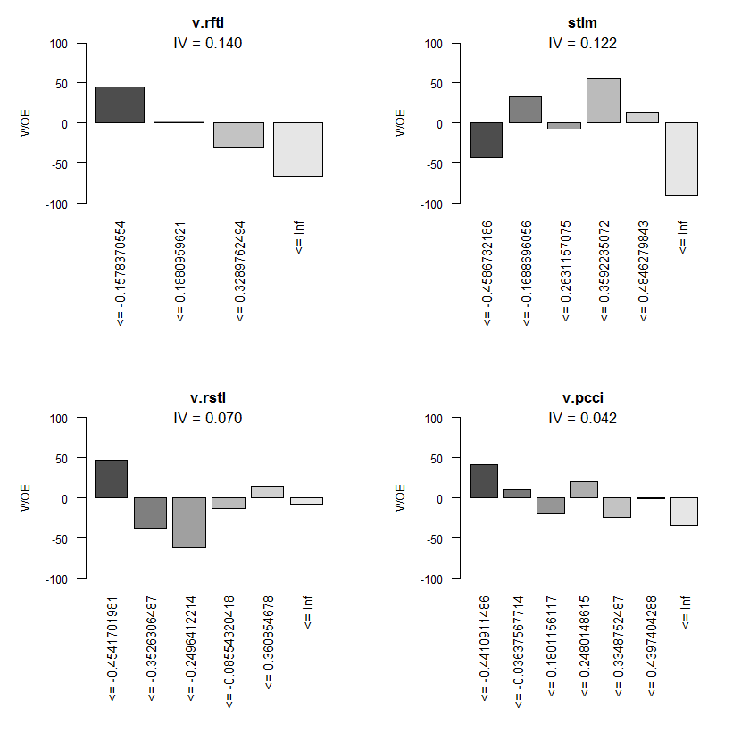
Fig.10. WOE de las variables de entrada 9-12
Gráfico del rango total de las variables según el valor de la información de su IV.
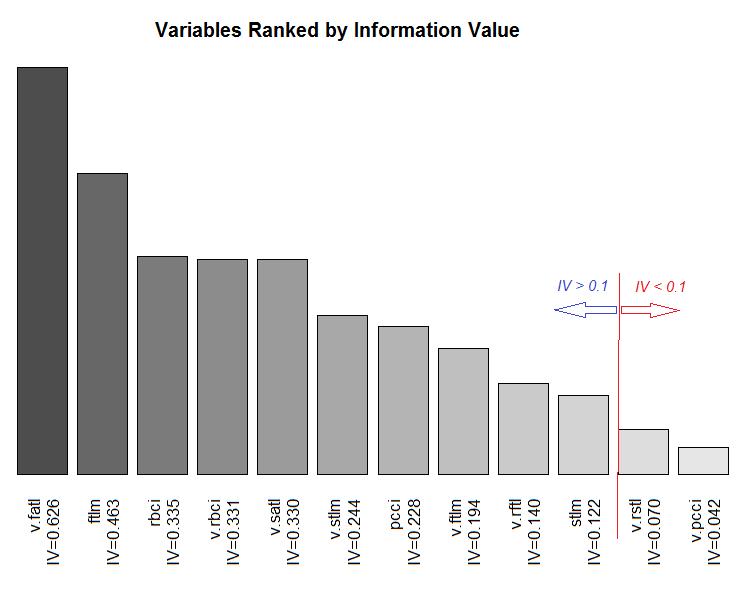
Fig.11. Intervalo de las variables en función del valor de la información de su IV
Las dos variables poco significativas v.rstl y v.pcci, que tienen IV < 0.1, podemos no utilizarlas en lo sucesivo. A partir de los
gráficos, vemos que de las 10 variables significativas, solo dos - v.satl y STLM - tienen una relación no lineal con la variable
objetivo, las otras están asociadas a ella de forma lineal.
Para realizar experimentos adicionales, tendremos que crear tres grupos:
- DTbin — conjunto de datos en el que los predictores numéricos continuos son transformados en factores con un número de niveles igual al número de rangos en los que están divididos;
- DTdum — conjunto de datos en el que los predictores-factores del conjunto DTbin han sido convertidos en variables binarias-dummy;
- DTwoe — conjunto de datos en el que los predictores-factores han sido convertidos en variables numéricas, reemplazando sus niveles con los valores WOE de estos niveles.
El primer conjunto DTbin los necesitamos para entrenar y obtener las métricas del modelo básico. Los conjuntos segundo y tercero serán utilizados para el entrenamiento de DNN y para comparar la eficacia de estos dos métodos de transformación.
En el paquete woebinning existe la función woe.binning.deploy(), que nos ayudará a resolver de forma relativamente fácil este problema. A la función se deberá transmitir:
- el frame de datos con los predictores y la variable objeto, además, la variable debe tener el valor 0 o 1;
- los parámetros de discretización obtenidos en la etapa anterior (preCut);
- los nombres de las variables que debemos categorizar. Si tenemos que categorizar todas las variables, solo indicaremos el nombre del frame de datos;
- indicar con qué IV mínimo no se categorizan las variables;
- especificar qué variables adicionales (excepto las categorizadas) queremos obtener. Existen dos variantes: "woe" y "dum".
La función devuelve el frame de datos que contiene las variables originales, las variables categorizadas y las variables adicionales (si es que se han encargado). Los nombres de las variables creadas nuevamente son generados mediante la adición del prefijo o sufijo correspondiente al nombre de la variable original. Por lo tanto, los prefijos de todas las variables adicionales son "dum" o "woe", y a las variables categorizadas se les añade el sufijo "binned". Escribimos la función DiscretizeData(), que, usando woe.binning.deploy(), transformará el conjunto de datos original.
DiscretizeData <- function(X, preCut, var){ require(foreach) require(woeBinning) DTd <- list() foreach(i = 1:length(X)) %do% { X[[i]] %>% select(-Data) %>% targ.int() %>% woe.binning.deploy(preCut, min.iv.total = 0.1, add.woe.or.dum.var = var) -> res return(res) } -> DTd list(pretrain = DTd[[1]] , train = DTd[[2]] , val = DTd[[3]] , test = DTd[[4]] ) -> DTd return(DTd) }
Parámetros de entrada de la función: datos originales (list X) con los espacios pretrain/train/val/test, parámetros de discretización preCut, tipo de variable adicional (string var).
La función elimina de cada variable el espacio "Data" y cambia el factor-variable "Class" por la variable numérica "Cl". Después de esto, suministra a la entrada de la función woe.binning.deploy(), en cuyos parámetros de entrada indicaremos el IV = 0.1 mínimo para incluir las variables en el conjunto de salida. En la salida, obtendremos una lista con los mismos espacios pretrain/train/val/test, pero en cada uno de ellos, a las variables originales se les añadirán las categorizadas y, si así lo especificamos, las adicionales. Calculamos los conjuntos necesarios y les añadiremos los datos "brutos" del conjunto DTcap.n.
evalq({
require(dplyr)
require(foreach)
DTbin = DiscretizeData(DTcap.n, preCut = preCut, var = "")
DTwoe = DiscretizeData(DTcap.n, preCut = preCut, var = "woe")
DTdum = DiscretizeData(DTcap.n, preCut = preCut, var = "dum")
X.woe <- list()
X.bin <- list()
X.dum <- list()
foreach(i = 1:length(DTcap.n)) %do% {
DTbin[[i]] %>% select(contains("binned")) -> X.bin[[i]]
DTdum[[i]] %>% select(starts_with("dum")) -> X.dum[[i]]
DTwoe[[i]] %>% select(starts_with("woe")) %>%
divide_by(100) -> X.woe[[i]]
return(list(bin = X.bin[[i]], woe = X.woe[[i]],
dum = X.dum[[i]], raw = DTcap.n[[i]]))
} -> DTcut
list(pretrain = DTcut[[1]],
train = DTcut[[2]],
val = DTcut[[3]],
test = DTcut[[4]] ) -> DTcut
rm(DTwoe, DTdum, X.woe, X.bin, X.dum)
},
env)
Puesto que hemos obtenido el WOE en tanto por ciento, dividiéndolo entre 100 obtendremos los valores de las variables que se podrán suministrar a las entradas de la neurored sin realizar una normalización adicional. Vamos a ver la estructura del espacio obtenido, por ejemplo, DTcut$val.
> env$DTcut$val %>% str() List of 4 $ bin:'data.frame': 501 obs. of 10 variables: ..$ v.fatl.binned: Factor w/ 5 levels "(-Inf,-0.3904381926]",..: 1 1 3 2 4 3 4 4 4 4 ... ..$ ftlm.binned : Factor w/ 5 levels "(-Inf,-0.2344708291]",..: 2 1 1 1 2 2 3 4 4 4 ... ..$ rbci.binned : Factor w/ 5 levels "(-Inf,-0.1718377948]",..: 2 1 2 1 2 3 3 3 4 4 ... ..$ v.rbci.binned: Factor w/ 5 levels "(-Inf,-0.1837437563]",..: 1 1 3 2 4 3 4 4 4 4 ... ..$ v.satl.binned: Factor w/ 5 levels "(-Inf,-0.01840058612]",..: 1 1 1 1 1 1 1 1 1 2 ... ..$ v.stlm.binned: Factor w/ 5 levels "(-Inf,-0.4030051922]",..: 2 2 3 2 3 2 3 3 4 4 ... ..$ pcci.binned : Factor w/ 5 levels "(-Inf,-0.1738420887]",..: 1 1 4 2 4 2 4 2 2 3 ... ..$ v.ftlm.binned: Factor w/ 4 levels "(-Inf,-0.03697698898]",..: 1 1 3 2 3 2 3 3 2 2 ... ..$ v.rftl.binned: Factor w/ 5 levels "(-Inf,-0.1578370554]",..: 2 1 1 1 1 1 1 2 2 2 ... ..$ stlm.binned : Factor w/ 7 levels "(-Inf,-0.4586732186]",..: 2 2 2 2 1 1 1 1 1 2 ... $ woe:'data.frame': 501 obs. of 10 variables: ..$ woe.v.fatl.binned: num [1:501] 1.713 1.713 -0.145 0.632 -0.897 ... ..$ woe.ftlm.binned : num [1:501] 0.352 0.972 0.972 0.972 0.352 ... ..$ woe.rbci.binned : num [1:501] 0.274 0.794 0.274 0.794 0.274 ... ..$ woe.v.rbci.binned: num [1:501] 0.683 0.683 -0.295 0.22 -0.861 ... ..$ woe.v.satl.binned: num [1:501] 0.573 0.573 0.573 0.573 0.573 ... ..$ woe.v.stlm.binned: num [1:501] 0.473 0.473 -0.0183 0.473 -0.0183 ... ..$ woe.pcci.binned : num [1:501] 0.619 0.619 -0.65 0.174 -0.65 ... ..$ woe.v.ftlm.binned: num [1:501] 0.435 0.435 -0.669 -0.162 -0.669 ... ..$ woe.v.rftl.binned: num [1:501] 0.024 0.446 0.446 0.446 0.446 ... ..$ woe.stlm.binned : num [1:501] 0.329 0.329 0.329 0.329 -0.425 ... $ dum:'data.frame': 501 obs. of 41 variables: ..$ dum.v.fatl.-Inf.-0.3904381926.binned : num [1:501] 0 0 0 0 0 0 0 0 0 0 ... ..$ dum.v.fatl.-0.03713814085.0.1130198981.binned : num [1:501] 0 0 0 0 0 0 0 0 0 0 ... ..$ dum.v.fatl.-0.3904381926.-0.03713814085.binned: num [1:501] 0 0 0 0 0 0 0 0 0 0 ... ..$ dum.v.fatl.0.1130198981.Inf.binned : num [1:501] 0 0 0 0 0 0 0 0 0 0 ... ..$ dum.ftlm.-0.2344708291.-0.01368798447.binned : num [1:501] 0 0 0 0 0 0 0 0 0 0 ... ..$ dum.ftlm.-Inf.-0.2344708291.binned : num [1:501] 0 0 0 0 0 0 0 0 0 0 ... ..$ dum.ftlm.-0.01368798447.0.1789073635.binned : num [1:501] 0 0 0 0 0 0 0 0 0 0 ... ..$ dum.ftlm.0.1789073635.Inf.binned : num [1:501] 0 0 0 0 0 0 0 0 0 0 ... ....................................................................................... ..$ dum.stlm.-Inf.-0.4586732186.binned : num [1:501] 0 0 0 0 0 0 0 0 0 0 ... ..$ dum.stlm.-0.1688696056.0.2631157075.binned : num [1:501] 0 0 0 0 0 0 0 0 0 0 ... ..$ dum.stlm.0.2631157075.0.3592235072.binned : num [1:501] 0 0 0 0 0 0 0 0 0 0 ... ..$ dum.stlm.0.3592235072.0.4846279843.binned : num [1:501] 0 0 0 0 0 0 0 0 0 0 ... ..$ dum.stlm.0.4846279843.Inf.binned : num [1:501] 0 0 0 0 0 0 0 0 0 0 ... $ raw:'data.frame': 501 obs. of 14 variables: ..$ Data : POSIXct[1:501], format: "2017-02-23 15:30:00" "2017-02-23 15:45:00" ... ..$ ftlm : num [1:501] -0.223 -0.374 -0.262 -0.31 -0.201 ... ..$ stlm : num [1:501] -0.189 -0.257 -0.271 -0.389 -0.473 ... ..$ rbci : num [1:501] -0.0945 -0.1925 -0.1348 -0.1801 -0.1192 ... ..$ pcci : num [1:501] -0.5714 -0.2602 0.4459 -0.0478 0.2596 ... ..$ v.fatl: num [1:501] -0.426 -0.3977 0.0936 -0.1512 0.1178 ... ..$ v.satl: num [1:501] -0.35 -0.392 -0.177 -0.356 -0.316 ... ..$ v.rftl: num [1:501] -0.0547 -0.2065 -0.3253 -0.4185 -0.4589 ... ..$ v.rstl: num [1:501] 0.0153 -0.0273 -0.0636 -0.1281 -0.15 ... ..$ v.ftlm: num [1:501] -0.321 -0.217 0.253 0.101 0.345 ... ..$ v.stlm: num [1:501] -0.288 -0.3 -0.109 -0.219 -0.176 ... ..$ v.rbci: num [1:501] -0.2923 -0.2403 0.1909 0.0116 0.2868 ... ..$ v.pcci: num [1:501] -0.0298 0.3738 0.6153 -0.5643 0.2742 ... ..$ Class : Factor w/ 2 levels "-1","1": 1 1 1 1 2 2 2 2 2 1 ...
Como podemos ver, en el espacio bin hay 10 factores-variables con diferente cantidad de niveles y el sufijo "binned". El espacio woe contiene 10 variables, cuyos niveles de factor han sido cambiados a WOE (tienen el prefijo "woe"). En el espacio dum hay 41 variables numéricas dummy con el valor (0, 1), que han sido obtenidas a partir de las variables de factor con la codificación uno para uno (tienen el prefijo "dum"). En el espacio raw, hay 14 variables: son Data — timestamp, Class — variable factor-objetivo y 12 predictores numéricos.
Tenemos toda la información necesaria para los experimentos adicionales. En este momento, en el entorno env deben encontrase ya los objetos de la lista a continuación. Guardamos el área de trabajo con estos objetos en el archivo PartIV.RData.
> ls(env) [1] "Close" "Data" "dt" "DT" "DTbin" "DTcap" "DTcap.n" "DTcut" "High" [10] "i" "Low" "Open" "pre.outl" "preCut" "preproc" "Volume"
2.1.2. Modelo básico de comparación
Como modelo base usaremos el modelo OneR, sencillo, fiable y fácil de interpretar, e implementado en el paquete homónimo OneR. Los detalles sobre el algoritmo se pueden encontrar en la descripción del paquete. El modelo solo funciona con los datos de intervalo, pero el paquete tiene funciones auxiliares que pueden discretizar las variables numéricas de diferentes maneras. Como ya hemos transformado los predictores en factores, no los necesitaremos.
Ahora vamos a explicar el cálculo mostrado más abajo. Creamos los conjuntos train/val/test extrayendo de DTcut los espacios correspondientes y añadiéndoles la variable objetivo Class. Entrenamos el modelo con el conjunto train.
> evalq({
+ require(OneR)
+ require(dplyr)
+ require(magrittr)
+ train <- cbind(DTcut$train$bin, Class = DTcut$train$raw$Class) %>% as.data.frame()
+ val <- cbind(DTcut$val$bin, Class = DTcut$val$raw$Class) %>% as.data.frame()
+ test <- cbind(DTcut$test$bin, Class = DTcut$test$raw$Class) %>% as.data.frame()
+ model <- train, formula = NULL, ties.method = "chisq", #c("first","chisq"
+ verbose = TRUE) #FALSE, TRUE
+ }, env)
Loading required package: OneR
Attribute Accuracy
1 * v.satl.binned 63.14%
2 v.fatl.binned 62.64%
3 ftlm.binned 62.54%
4 pcci.binned 61.44%
5 v.rftl.binned 59.74%
6 v.rbci.binned 58.94%
7 rbci.binned 58.64%
8 stlm.binned 58.04%
9 v.stlm.binned 57.54%
10 v.ftlm.binned 56.14%
---
Chosen attribute due to accuracy
and ties method (if applicable): '*'
Warning message:
In train, formula = NULL, ties.method = "chisq", verbose = TRUE) :
data contains unused factor levels
El modelo ha elegido la variable v.satl.binned
con una precisión básica de 63.14% como base para
la construcción de las normas. Veamos la información general del modelo:
> summary(env$model) Call: train, formula = NULL, ties.method = "chisq", verbose = FALSE) Rules: If v.satl.binned = (-Inf,-0.01840058612] then Class = -1 If v.satl.binned = (-0.01840058612,0.3247097195] then Class = 1 If v.satl.binned = (0.3247097195,0.4003869443] then Class = 1 If v.satl.binned = (0.4003869443, Inf] then Class = 1 Accuracy: 632 of 1001 instances classified correctly (63.14%) Contingency table: v.satl.binned Class (-Inf,-0.01840058612] (-0.01840058612,0.3247097195] (0.3247097195,0.4003869443] (0.4003869443, Inf] Sum -1 * 325 161 28 37 551 1 143 * 229 * 35 * 43 450 Sum 468 390 63 80 1001 --- Maximum in each column: '*' Pearson's Chi-squared test: X-squared = 74.429, df = 3, p-value = 4.803e-16
Representación gráfica del resultado del entrenamiento:
plot(env$model)

Fig.12. Distribución de las categorías de la variable v.satl.binned por clases en el modelo
La precisición de la predicción en el entrenamiento no es muy alta. Veamos qué precisión muestra el modelo en el conjunto de validación:
> evalq(res.val <- eval_model(predict(model, val %>% as.data.frame()), val$Class), + env) Confusion matrix (absolute): Real Prediction -1 1 Sum -1 106 87 193 1 100 208 308 Sum 206 295 501 Confusion matrix (relative): Real Prediction -1 1 Sum -1 0.21 0.17 0.39 1 0.20 0.42 0.61 Sum 0.41 0.59 1.00 Accuracy: 0.6267 (314/501) Error rate: 0.3733 (187/501) Error rate reduction (vs. base rate): 0.0922 (p-value = 0.04597)
y en el conjunto de prueba:
> evalq(res.test <- eval_model(predict(model, test %>% as.data.frame()), test$Class), + env) Confusion matrix (absolute): Real Prediction -1 1 Sum -1 130 102 232 1 76 193 269 Sum 206 295 501 Confusion matrix (relative): Real Prediction -1 1 Sum -1 0.26 0.20 0.46 1 0.15 0.39 0.54 Sum 0.41 0.59 1.00 Accuracy: 0.6447 (323/501) Error rate: 0.3553 (178/501) Error rate reduction (vs. base rate): 0.1359 (p-value = 0.005976)
Los resultados no han sido nada esperanzadores. Error rate reduction muestra cómo ha aumentado la precisión con respecto al volumen básico (0.5). Un valor "p" bajo (< 0.05) indica que el modelo verdaderamente es capaz de dar pronósticos mejores que el nivel básico. La precisión del conjunto de prueba es de 0.6447 (323/501), mayor que la precisión del conjunto de validación. Esta cifra será un punto de referencia para la comparación con los resultados de nuestras predicciones de los modelos futuros.
2.1.3. Estructura de una DNN
Para el entrenamiento y las pruebas utilizaremos tres conjuntos de datos:
- DTcut$$raw — 12 variables de entrada (los valores atípicos han sido imputados y normalizados).
- DTcut$$dum — 41 variables binarias.
- DTcut$$woe — 10 variables numéricas.
Con todos los conjuntos usamos la variable objetivo Class = factor con dos niveles. Estructura de la neurored:
- DNNraw - layers = c(12, 16, 8(2), 2), funciones de activación c(tanh, maxout(lin), softmax)
- DNNwoe - layers = c(10, 16, 8(2), 2), funciones de activación c(tanh, maxout(lin), softmax)
- DNNdum - layers = c(41, 50, 8(2), 2), funciones de activación c(ReLU, maxout(ReLU), softmax)
La siguiente figura muestra la estructura de la neurored DNNwoe. La red neuronal consta de una capa de entrada, una capa de salida y dos capas ocultas. Las otras dos (DNNdum, DNNraw) tienen una estructura similar, pero difieren en el número de neuronas de las capas y en las funciones de activación.
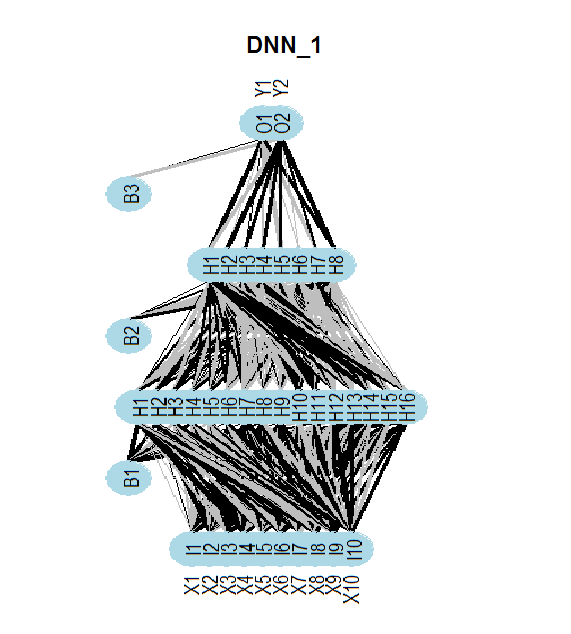
Fig.13. Estructura de la neurored DNNwoe
2.1.4. Variantes de entrenamiento
Con pre-entrenamiento
El entrenamiento se lleva a cabo en dos etapas:
- pre-entrenamiento con el conjunto SRBM /pretrain y entrenamiento posterior solo de la capa superior de DNN, con el conjunto de validación train y los parámetros par_0;
- el entrenamiento preciso de toda la red con los conjuntos train/val y los parámetros par_1.
Podemos guardar los modelos intermedios del entrenamiento preciso, pero también podemos omitir el guardado. El mejor modelo según las conclusiones del entrenamiento final ha sido guardado. Los parámetros de estas dos etapas deben incluir:
- par_0 — parámetros generales de la neurored, parámetros de entrenamiento de RBM y parámetros de la capa superior de DNN;
- par_1 — parámetros de entrenamiento de todas las capas de DNN.
Todos los parámetros de DArch tienen los valores por defecto. Pero si necesitamos ciertos ajustes en una cierta etapa de la formación, podemos solicitarlos con una lista, y se anulará la configuración predeterminada. Después de la primera etapa de entrenamiento, obtenemos la estructura de DArch con los parámetros y los resultados del entrenamiento (error de entrenamiento, pruebas, etc.), así como la red neuronal, iniciada por los pesos SRBM entrenados. Para llevar a cabo la segunda etapa del entrenamiento, tenemos que usar la estructura de DArch, obtenida en la primera etapa, y la lista de parámetros para esta fase de entrenamiento. Y, por supuesto, necesitaremos los conjuntos de entrenamiento y validación.
Ahora echamos un vistazo a los parámetros necesarios para la primera etapa de entrenamiento (pre-entrenamiento de SRBM y entrenamiento de la capa superior de la neurored) y ejecutamos esta:
##=====CODE I etap=========================== evalq({ require(darch) require(dplyr) require(magrittr) Ln <- c(0, 16, 8, 0)# // el número de neuronas de entrada y salida se define de forma automática a partir del conjunto de datos nEp_0 <- 25 #------------------ par_0 <- list( layers = Ln, # // sacamos este parámetro fuera de la lista (para que las manipulaciones sean más sencillas) seed = 54321,# // si queremos obtener datos idénticos al realizar la inicialización logLevel = 5, # // qué nivel de muestra de información necesitamos # params RBM======================== rbm.consecutive = F, # each RBM is trained one epoch at a time rbm.numEpochs = nEp_0, rbm.batchSize = 50, rbm.allData = TRUE, rbm.lastLayer = -1, # // no entrenar la capa superior de SRBM rbm.learnRate = 0.3, rbm.unitFunction = "tanhUnitRbm", # params NN ======================== darch.batchSize = 50, darch.numEpochs = nEp_0,# // sacamos este parámetro fuera de la lista para que las manipulaciones sean más sencillas darch.trainLayers = c(F,F,T), #обучать //solo la capa superior darch.unitFunction = c("tanhUnit","maxoutUnit", "softmaxUnit"), bp.learnRate = 0.5, bp.learnRateScale = 1, darch.weightDecay = 0.0002, darch.dither = F, darch.dropout = c(0.1,0.2,0.1), darch.fineTuneFunction = backpropagation, #rpropagation normalizeWeights = T, normalizeWeightsBound = 1, darch.weightUpdateFunction = c("weightDecayWeightUpdate", "maxoutWeightUpdate", "weightDecayWeightUpdate"), darch.dropout. T, darch.maxout.poolSize = 2, darch.maxout.unitFunction = "linearUnit") #--------------------------- DNN_default <- darch(darch = NULL, paramsList = par_0, x = DTcut$pretrain$woe %>% as.data.frame(), y = DTcut$pretrain$raw$Class %>% as.data.frame(), xValid = DTcut$train$woe %>% as.data.frame(), yValid = DTcut$train$raw$Class %>% as.data.frame() ) }, env)Resultado tras finalizar la primera etapa de entrenamiento:
........................... INFO [2017-09-11 14:12:19] Classification error on Train set (best model): 31.95% (639/2000) INFO [2017-09-11 14:12:19] Train set (best model) Cross Entropy error: 1.233 INFO [2017-09-11 14:12:19] Classification error on Validation set (best model): 35.86% (359/1001) INFO [2017-09-11 14:12:19] Validation set (best model) Cross Entropy error: 1.306 INFO [2017-09-11 14:12:19] Best model was found after epoch 3 INFO [2017-09-11 14:12:19] Final 0.632 validation Cross Entropy error: 1.279 INFO [2017-09-11 14:12:19] Final 0.632 validation classification error: 34.42% INFO [2017-09-11 14:12:19] Fine-tuning finished after 5.975 secs
Segunda etapa de entrenamiento de toda la neurored:
##=====CODE II etap=========================== evalq({ require(darch) require(dplyr) require(magrittr) nEp_1 <- 100 bp.learnRate <- 1 par_1 <- list( layers = Ln, seed = 54321, logLevel = 5, rbm.numEpochs = 0,# ¡No entrenamos SRBM! darch.batchSize = 50, darch.numEpochs = nEp_1, darch.trainLayers = c(T,T,T), #TRUE, darch.unitFunction = c("tanhUnit","maxoutUnit", "softmaxUnit"), bp.learnRate = bp.learnRate, bp.learnRateScale = 1, darch.weightDecay = 0.0002, darch.dither = F, darch.dropout = c(0.1,0.2,0.1), darch.fineTuneFunction = backpropagation, #rpropagation backpropagation normalizeWeights = T, normalizeWeightsBound = 1, darch.weightUpdateFunction = c("weightDecayWeightUpdate", "maxoutWeightUpdate", "weightDecayWeightUpdate"), darch.dropout. T, darch.maxout.poolSize = 2, darch.maxout.unitFunction = exponentialLinearUnit, darch.elu.alpha = 2) #------------------------------ DNN_1 <- darch( darch = DNN_default, paramsList = par_1, x = DTcut$train$woe %>% as.data.frame(), y = DTcut$train$raw$Class %>% as.data.frame(), xValid = DTcut$val$woe %>% as.data.frame(), yValid = DTcut$val$raw$Class %>% as.data.frame() ) }, env)
Resultado de la segunda etapa de entrenamiento:
........................... INFO [2017-09-11 15:48:37] Finished epoch 100 of 100 after 0.279 secs (3666 patterns/sec) INFO [2017-09-11 15:48:37] Classification error on Train set (best model): 31.97% (320/1001) INFO [2017-09-11 15:48:37] Train set (best model) Cross Entropy error: 1.225 INFO [2017-09-11 15:48:37] Classification error on Validation set (best model): 31.14% (156/501) INFO [2017-09-11 15:48:37] Validation set (best model) Cross Entropy error: 1.190 INFO [2017-09-11 15:48:37] Best model was found after epoch 96 INFO [2017-09-11 15:48:37] Final 0.632 validation Cross Entropy error: 1.203 INFO [2017-09-11 15:48:37] Final 0.632 validation classification error: 31.44% INFO [2017-09-11 15:48:37] Fine-tuning finished after 37.22 secs
Gráfico de variación del error de predicción en el proceso de entrenamiento de la segunda etapa:
plot(env$DNN_1, y = "raw")
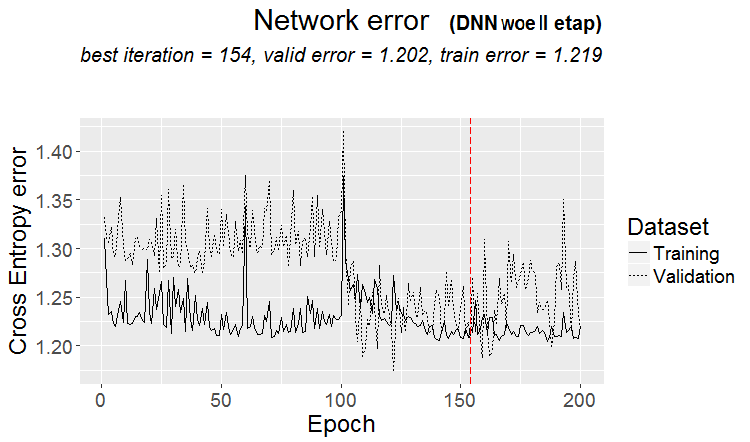
Fig.14. Variación del error de clasificación en la segunda etapa de entrenamiento
Vamos a echar un vistazo al error de clasificación del modelo final en el conjunto de prueba:
#-----------
evalq({
xValid = DTcut$test$woe %>% as.data.frame()
yValid = DTcut$test$raw$Class %>% as.vector()
Ypredict <- predict(DNN_1, newdata = xValid, type = "class")
numIncorrect <- sum(Ypredict != yValid)
cat(paste0("Incorrect classifications on all examples: ", numIncorrect, " (",
round(numIncorrect/nrow(xValid)*100, 2), "%)\n"))
caret::confusionMatrix(yValid, Ypredict)
}, env)
Incorrect classifications on all examples: 166 (33.13%)
Confusion Matrix and Statistics
Referencia
Prediction -1 1
-1 129 77
1 89 206
Accuracy : 0.6687
95% CI : (0.6255, 0.7098)
No Information Rate : 0.5649
P-Value [Acc > NIR] : 1.307e-06
Kappa : 0.3217
Mcnemar's Test P-Value : 0.3932
Sensitivity : 0.5917
Specificity : 0.7279
Pos Pred Value : 0.6262
Neg Pred Value : 0.6983
Prevalence : 0.4351
Detection Rate : 0.2575
Detection Prevalence : 0.4112
Balanced Accuracy : 0.6598
'Positive' Class : -1
#----------------------------------------
La precisión en este conjunto de datos (woe) con estos parámetros - ni mucho menos óptimos - es un poco superior a la del modelo básico. Existe un potencial significativo para aumentar la precisión mediante la optimización de los hiperparámetros DNN. Al repetir los cálculos, los datos obtenidos podrían no coincidir con los mostrados en este artículo.
Para los cálculos sucesivos con otros conjuntos de datos, reuniremos nuestros scripts de una forma compacta. Escribimos la función de entrenamiento para el conjunto woe:
#------------------- DNN.train.woe <- function(param, X){ require(darch) require(magrittr) darch( darch = NULL, paramsList = param[[1]], x = X[[1]]$woe %>% as.data.frame(), y = X[[1]]$raw$Class %>% as.data.frame(), xValid = X[[2]]$woe %>% as.data.frame(), yValid = X[[2]]$raw$Class %>% as.data.frame() ) %>% darch( ., paramsList = param[[2]], x = X[[2]]$woe %>% as.data.frame(), y = X[[2]]$raw$Class %>% as.data.frame(), xValid = X[[3]]$woe %>% as.data.frame(), yValid = X[[3]]$raw$Class %>% as.data.frame() ) -> Darch return(Darch) }
Repetimos los cálculos para el conjunto DTcut$$woe de forma compacta:
evalq({
require(darch)
require(magrittr)
Ln <- c(0, 16, 8, 0)
nEp_0 <- 25
nEp_1 <- 25
rbm.learnRate = c(0.5,0.3,0.1)
bp.learnRate <- c(0.5,0.3,0.1)
list(par_0, par_1) %>% DNN.train.woe(DTcut) -> Dnn.woe
xValid = DTcut$test$woe %>% as.data.frame()
yValid = DTcut$test$raw$Class %>% as.vector()
Ypredict <- predict(Dnn.woe, newdata = xValid, type = "class")
numIncorrect <- sum(Ypredict != yValid)
cat(paste0("Incorrect classifications on all examples: ", numIncorrect, " (",
round(numIncorrect/nrow(xValid)*100, 2), "%)\n"))
caret::confusionMatrix(yValid, Ypredict) -> cM.woe
}, env)
Realizamos el cálculo para el conjunto DTcut$$raw:
#-------------------------
DNN.train.raw <- function(param, X){
require(darch)
require(magrittr)
darch( darch = NULL, paramsList = param[[1]],
x = X[[1]]$raw %>% tbl_df %>% select(-c(Data, Class)),
y = X[[1]]$raw$Class %>% as.data.frame(),
xValid = X[[2]]$raw %>% tbl_df %>% select(-c(Data, Class)),
yValid = X[[2]]$raw$Class %>% as.data.frame()
) %>%
darch( ., paramsList = param[[2]],
x = X[[2]]$raw %>% tbl_df %>% select(-c(Data, Class)),
y = X[[2]]$raw$Class %>% as.data.frame(),
xValid = X[[3]]$raw %>% tbl_df %>% select(-c(Data, Class)),
yValid = X[[3]]$raw$Class %>% as.data.frame()
) -> Darch
return(Darch)
}
#-------------------------------
evalq({
require(darch)
require(magrittr)
Ln <- c(0, 16, 8, 0)
nEp_0 <- 25
nEp_1 <- 25
rbm.learnRate = c(0.5,0.3,0.1)
bp.learnRate <- c(0.5,0.3,0.1)
list(par_0, par_1) %>% DNN.train.raw(DTcut) -> Dnn.raw
xValid = DTcut$test$raw %>% tbl_df %>% select(-c(Data, Class))
yValid = DTcut$test$raw$Class %>% as.vector()
Ypredict <- predict(Dnn.raw, newdata = xValid, type = "class")
numIncorrect <- sum(Ypredict != yValid)
cat(paste0("Incorrect classifications on all examples: ", numIncorrect, " (",
round(numIncorrect/nrow(xValid)*100, 2), "%)\n"))
caret::confusionMatrix(yValid, Ypredict) -> cM.raw
}, env)
#----------------------------
Echemos un vistazo al resultado y al gráfico de variación del error de clasificación para este conjunto:
> env$cM.raw
Confusion Matrix and Statistics
Referencia
Prediction -1 1
-1 133 73
1 86 209
Accuracy : 0.6826
95% CI : (0.6399, 0.7232)
No Information Rate : 0.5629
P-Value [Acc > NIR] : 2.667e-08
Kappa : 0.3508
Mcnemar's Test P-Value : 0.3413
Sensitivity : 0.6073
Specificity : 0.7411
Pos Pred Value : 0.6456
Neg Pred Value : 0.7085
Prevalence : 0.4371
Detection Rate : 0.2655
Detection Prevalence : 0.4112
Balanced Accuracy : 0.6742
'Positive' Class : -1
#--------------------------------------
plot(env$Dnn.raw, y = "raw")
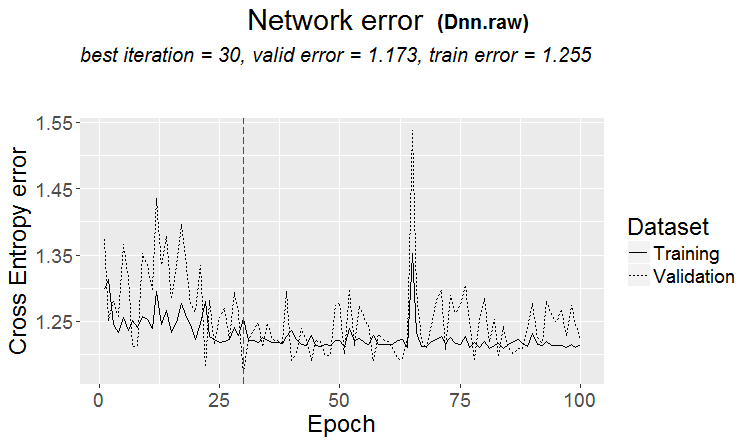
Fig.15. Variación del error de clasificación en la segunda etapa de entrenamiento
No hemos conseguido entrenar la neurored con los datos DTcut$$dum. El lector puede tratar de hacerlo por sí solo, por ejemplo,
intentando aplicar a la entrada los datos DTcut$$bin y previniendo el paso de los predictores a dummy en los parámetros de
entrenamiento.
Entrenamiento sin pre-entrenamiento
Vamos a hacer un experimento con neuroredes entrenadas sin pre-entrenamiento con los mismos datos (woe, raw) en los conjuntos pretrain/train/val. Vamos a ver el resultado.
#-------WOE---------------- evalq({ require(darch) require(magrittr) Ln <- c(0, 16, 8, 0) nEp_1 <- 100 bp.learnRate <- c(0.5,0.7,0.1) #--param---------------- par_1 <- list( layers = Ln, seed = 54321, logLevel = 5, rbm.numEpochs = 0,# ¡No entrenamos SRBM! darch.batchSize = 50, darch.numEpochs = nEp_1, darch.trainLayers = c(T,T,T), #TRUE, darch.unitFunction = c("tanhUnit","maxoutUnit", "softmaxUnit"), bp.learnRate = bp.learnRate, bp.learnRateScale = 1, darch.weightDecay = 0.0002, darch.dither = F, darch.dropout = c(0.0,0.2,0.1), darch.fineTuneFunction = backpropagation, #rpropagation backpropagation normalizeWeights = T, normalizeWeightsBound = 1, darch.weightUpdateFunction = c("weightDecayWeightUpdate", "maxoutWeightUpdate", "weightDecayWeightUpdate"), darch.dropout. T, darch.maxout.poolSize = 2, darch.maxout.unitFunction = exponentialLinearUnit, darch.elu.alpha = 2) #--train--------------------------- darch( darch = NULL, paramsList = par_1, x = DTcut[[1]]$woe %>% as.data.frame(), y = DTcut[[1]]$raw$Class %>% as.data.frame(), xValid = DTcut[[2]]$woe %>% as.data.frame(), yValid = DTcut[[2]]$raw$Class %>% as.data.frame() ) -> Dnn.woe.I #---test-------------------------- xValid = DTcut$val$woe %>% as.data.frame() yValid = DTcut$val$raw$Class %>% as.vector() Ypredict <- predict(Dnn.woe.I, newdata = xValid, type = "class") numIncorrect <- sum(Ypredict != yValid) cat(paste0("Incorrect classifications on all examples: ", numIncorrect, " (", round(numIncorrect/nrow(xValid)*100, 2), "%)\n")) caret::confusionMatrix(yValid, Ypredict) -> cM.woe.I }, env) #---------Ris16------------------------------------ plot(env$Dnn.woe.I, type = "class") env$cM.woe.I
Métricas:
....................................................... INFO [2017-09-14 10:38:01] Classification error on Train set (best model): 28.7% (574/2000) INFO [2017-09-14 10:38:01] Train set (best model) Cross Entropy error: 1.140 INFO [2017-09-14 10:38:02] Classification error on Validation set (best model): 35.86% (359/1001) INFO [2017-09-14 10:38:02] Validation set (best model) Cross Entropy error: 1.299 INFO [2017-09-14 10:38:02] Best model was found after epoch 67 INFO [2017-09-14 10:38:02] Final 0.632 validation Cross Entropy error: 1.241 INFO [2017-09-14 10:38:02] Final 0.632 validation classification error: 33.23% INFO [2017-09-14 10:38:02] Fine-tuning finished after 37.13 secs Incorrect classifications on all examples: 150 (29.94%) > env$cM.woe.I Confusion Matrix and Statistics Referencia Prediction -1 1 -1 144 62 1 88 207 Accuracy : 0.7006 95% CI : (0.6584, 0.7404) No Information Rate : 0.5369 P-Value [Acc > NIR] : 5.393e-14 Kappa : 0.3932 Mcnemar's Test P-Value : 0.04123 Sensitivity : 0.6207 Specificity : 0.7695 Pos Pred Value : 0.6990 Neg Pred Value : 0.7017 Prevalence : 0.4631 Detection Rate : 0.2874 Detection Prevalence : 0.4112 Balanced Accuracy : 0.6951 'Positive' Class : -1
Gráfico de variación del error de clasificación durante el entrenamiento:
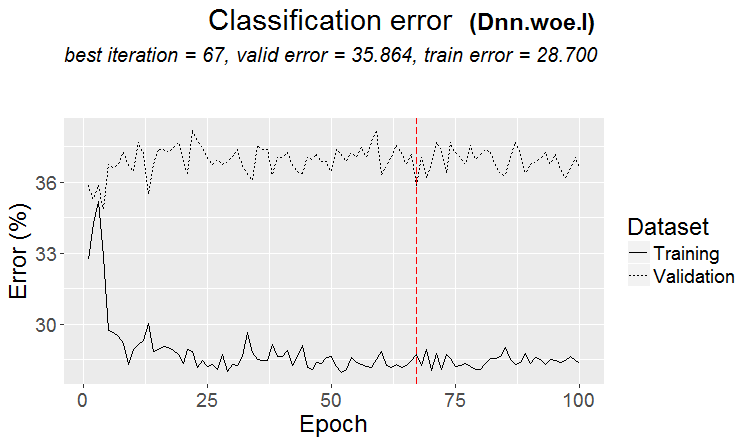
Fig.16. Variación del error de clasificación sin pre-entrenamiento con el conjunto $woe
Lo mismo para el conjunto /raw:
evalq({
require(darch)
require(magrittr)
Ln <- c(0, 16, 8, 0)
nEp_1 <- 100
bp.learnRate <- c(0.5,0.7,0.1)
#--param-----------------------------
par_1 <- list(
layers = Ln,
seed = 54321,
logLevel = 5,
rbm.numEpochs = 0,# ¡No entrenamos SRBM!
darch.batchSize = 50,
darch.numEpochs = nEp_1,
darch.trainLayers = c(T,T,T), #TRUE,
darch.unitFunction = c("tanhUnit","maxoutUnit", "softmaxUnit"),
bp.learnRate = bp.learnRate,
bp.learnRateScale = 1,
darch.weightDecay = 0.0002,
darch.dither = F,
darch.dropout = c(0.1,0.2,0.1),
darch.fineTuneFunction = backpropagation, #rpropagation backpropagation
normalizeWeights = T,
normalizeWeightsBound = 1,
darch.weightUpdateFunction = c("weightDecayWeightUpdate",
"maxoutWeightUpdate",
"weightDecayWeightUpdate"),
darch.dropout. T,
darch.maxout.poolSize = 2,
darch.maxout.unitFunction = exponentialLinearUnit,
darch.elu.alpha = 2)
#---train------------------------------
darch( darch = NULL, paramsList = par_1,
x = DTcut[[1]]$raw %>% tbl_df %>% select(-c(Data, Class)) ,
y = DTcut[[1]]$raw$Class %>% as.vector(),
xValid = DTcut[[2]]$raw %>% tbl_df %>% select(-c(Data, Class)) ,
yValid = DTcut[[2]]$raw$Class %>% as.vector()
) -> Dnn.raw.I
#---test--------------------------------
xValid = DTcut[[3]]$raw %>% tbl_df %>% select(-c(Data, Class))
yValid = DTcut[[3]]$raw$Class %>% as.vector()
Ypredict <- predict(Dnn.raw.I, newdata = xValid, type = "class")
numIncorrect <- sum(Ypredict != yValid)
cat(paste0("Incorrect classifications on all examples: ", numIncorrect, " (",
round(numIncorrect/nrow(xValid)*100, 2), "%)\n"))
caret::confusionMatrix(yValid, Ypredict) -> cM.raw.I
}, env)
#---------Ris17----------------------------------
env$cM.raw.I
plot(env$Dnn.raw.I, type = "class")
Métricas:
INFO [2017-09-14 11:06:13] Classification error on Train set (best model): 30.75% (615/2000) INFO [2017-09-14 11:06:13] Train set (best model) Cross Entropy error: 1.189 INFO [2017-09-14 11:06:13] Classification error on Validation set (best model): 33.67% (337/1001) INFO [2017-09-14 11:06:13] Validation set (best model) Cross Entropy error: 1.236 INFO [2017-09-14 11:06:13] Best model was found after epoch 45 INFO [2017-09-14 11:06:13] Final 0.632 validation Cross Entropy error: 1.219 INFO [2017-09-14 11:06:13] Final 0.632 validation classification error: 32.59% INFO [2017-09-14 11:06:13] Fine-tuning finished after 35.47 secs Incorrect classifications on all examples: 161 (32.14%) > #---------Ris17---------------------------------- > env$cM.raw.I Confusion Matrix and Statistics Referencia Prediction -1 1 -1 140 66 1 95 200 Accuracy : 0.6786 95% CI : (0.6358, 0.7194) No Information Rate : 0.5309 P-Value [Acc > NIR] : 1.283e-11 Kappa : 0.3501 Mcnemar's Test P-Value : 0.02733 Sensitivity : 0.5957 Specificity : 0.7519 Pos Pred Value : 0.6796 Neg Pred Value : 0.6780 Prevalence : 0.4691 Detection Rate : 0.2794 Detection Prevalence : 0.4112 Balanced Accuracy : 0.6738 'Positive' Class : -1
Gráfico de variación del error de clasificación:
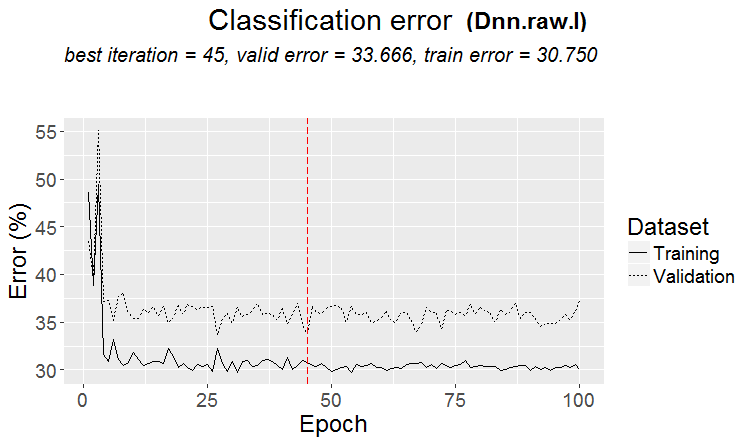
Fig.17. Variación del error de clasificación sin pre-entrenamiento con el conjunto $raw
2.2. Análisis de resultados
Reunimos los resultados de nuestros experimentos en un recuadro:
| Tipo de entrenamiento | Conjunto /woe | Conjunto /raw |
|---|---|---|
| Con pre-entrenamiento | 0.6687 (0.6255 - 0.7098) | 0.6826(0.6399 - 0.7232) |
| Sin pre-entrenamiento | 0.7006(0.6589 - 0.7404) | 0.6786(0.6359 - 0.7194) |
El error de clasificación con pre-entrenamiento en los dos conjuntos es aproximadamente el mismo y se encuentra en los límites 30+/-4%. Durante el entrenamiento sin pre-entrenamiento, a pesar de que el error es más bajo, en el gráfico de variación se precibe claramente el sobre-entrenamiento (el error en los conjuntos de validación y prueba supera significativamente el error de entrenamiento). Por eso, en los experimentos posteriores usaremos el entrenamiento con pre-entrenamiento.
El resultado es muy superior a los índices del modelo básico, pero tenemos la posibilidad de mejorarlos optimizando ciertos
hiperparámetros. De eso nos ocuparemos en el próximo artículo.
Conclusión
A pesar de sus limitaciones (por ejemplo, solo tiene dos métodos de entrenamiento), el paquete darch permite crear neuroredes de
diferentes estructuras y parámetros. Podemos considerarlo una buena herramienta para el estudio profundo de la neuroredes.
Los bajos índices de DNN se deben mayormente a que usamos los parámetros por defecto u otros cercanos a los mismos. El uso del conjunto /woe
no ha mostrado ventajas frente a /raw. Por eso, en el siguiente artículo:
- optimizaremos una parte de los hiperparámetros de la DNN.woe anteriormente creada;
- crearemos una DNN usando la biblioteca TensorFlow, la pondremos a prueba y compararemos los resultados con la DNN (darch);
- crearemos un grupo de neuroredes de distinto tipo (bagging, stacking) y veremos cuánto aumenta este hecho la calidad de las predicciones.
Anexos
En GitHub/PartIV se encuentran:
- FunPrepareData.R — funciones usadas en la preparación de los datos
- RunPrepareData.R — scripts para la preparación de los datos
- Experiment.R — scripts para realizar experimentos
- Part_IV.RData — imagen de la zona de trabajo con todos los objetos obtenidos tras la etapa de preparación de datos
- SessionInfo.txt — información del software utilizado
- Darch_default.txt — lista de parámetros de la estructura DArch con los valores por defecto
Traducción del ruso hecha por MetaQuotes Ltd.
Artículo original: https://www.mql5.com/ru/articles/3473
 R cuadrado como evaluación de la calidad de la curva del balance de la estrategia
R cuadrado como evaluación de la calidad de la curva del balance de la estrategia
 Usando el filtro de Kalman en la predicción del precio
Usando el filtro de Kalman en la predicción del precio
 Asesor Experto multiplataforma: las clases CExpertAdvisor y CExpertAdvisor
Asesor Experto multiplataforma: las clases CExpertAdvisor y CExpertAdvisor
 Nuevo enfoque a la interpretación de la divergencia clásica e inversa
Nuevo enfoque a la interpretación de la divergencia clásica e inversa
- Aplicaciones de trading gratuitas
- 8 000+ señales para copiar
- Noticias económicas para analizar los mercados financieros
Usted acepta la política del sitio web y las condiciones de uso