Генетические алгоритмы - это просто!
Andrey Dik | 25 мая, 2010
Введение
Генетический алгоритм (ГА) относится к эвристическим алгоритмам (ЭА), который даёт приемлемое решение задачи в большинстве практически значимых случаев, однако при этом правильность решения математически не доказана и применяют чаще всего для задач, аналитическое решение которых весьма затруднительно или вовсе невозможно.
Классическим примером задач этого класса (класс NP) является “Задача коммивояжера” (является одной из самых известных задач комбинаторной оптимизации. Задача заключается в отыскании самого выгодного маршрута, проходящего через указанные города хотя бы по одному разу с последующим возвратом в исходный город.). Но ничто не мешает использовать их и для задач, поддающихся формализации.
ЭА широко применяются для решения задач высокой вычислительной сложности, то есть вместо полного перебора вариантов, занимающего существенное время. Применяются в областях искусственного интеллекта, таких, как распознавание образов, в антивирусных программах, инженерии, компьютерных играх и других областях.
Стоит отдельно упомянуть о том, что MetaQuotes Software Corp. использует ГА в своих программных продуктах MetaTrader 4/5. Все мы знаем о тестере стратегий и о том, сколько времени и усилий позволяет сэкономить встроенный оптимизатор стратегий, в котором на ряду с прямым перебором имеется возможность оптимизации с применением ГА. Кроме того, тестер MetaTrader 5 позволяет использовать пользовательские критерии оптимизации. Возможно, читателю будет интересно прочитать статьи на тему ГА и о преимуществах, которые дают ЭА по сравнению с прямым перебором.
1. Немного истории
Чуть больше года назад мне понадобился алгоритм оптимизации для обучения нейронных сетей. Бегло ознакомившись с различными алгоритмами, остановил свой выбор на ГА. В результате поиска готовых реализаций обнаружил, что имеющиеся в открытом доступе либо имеют функциональные ограничения, например на количество оптимизируемых параметров, либо слишком "узко заточены".
Мне же нужен был универсальный гибкий инструмент не только для обучения любых видов нейронных сетей, но и вообще для решения любых задач оптимизации. После длительного изучения чужих "генетических творений" я так и не смог понять, как они работают. Всему виной были либо замысловатый стиль кода, либо недостаток опыта в программировании у меня, а возможно, и то и другое.
Основные трудности вызывало кодирование генов в двоичный код и последующая работа с ними в таком виде. Так или иначе, было решено написать свой генетический алгоритм с нуля, с ориентацией на масштабируемость и легкую модификацию в будущем.
Я не захотел связываться с двоичными преобразованиями, и решил работать с генами напрямую, т.е. представлять хромосому набором генов в виде вещественных чисел. Так появился на свет код моего генетического алгоритма с представлением хромосом вещественными числами. Позднее я узнал, что ничего нового не открыл, и подобные генетические алгоритмы (их называют real-coded GA) существуют уже более 15-и лет с момента первых публикаций о них.
Представленное на суд читателя моё видение подходов к реализации и принципах функционирования ГА основано на личном опыте использования их в практических задачах.
2. Описание ГА
В ГА заложены принципы, заимствованные у самой природы. Эти принципы - наследственность и изменчивость. Наследственность - способность организмов передавать свои признаки и особенности развития потомству. Благодаря этой способности все живые существа сохраняют в своих потомках характерные черты вида.
Изменчивость генов у живых организмов обеспечивает генетическое разнообразие популяции и имеет случайный характер, так как природа заранее "не знает", какие именно признаки особей будут наиболее предпочтительны в будущем (изменение климата, уменьшение/увеличение кормовой базы, появление конкурирующих видов и т.д.). Именно изменчивость позволяет появиться особям с новыми признаками, способным выжить и оставить потомство в новых, изменившихся условиях среды обитания.
В биологии изменчивость, обусловленную возникновением мутаций, называют мутационной, а обусловленную дальнейшим перекомбинированием генов в результате скрещивания - комбинационной. Оба эти вида изменчивости реализованы в ГА. Кроме того, реализован мутагенез, имитирующий природный механизм мутаций(внесение изменений в нуклеотидную последовательность ДНК) – естественный (спонтанный) и искусственный (индуцированный).
Простейшей единицей передачи информации о признаке в алгоритме является ген - структурная и функциональная единица наследственности, контролирующая развитие определенного признака или свойства. Геном называем одну переменную исследуемой функции. Ген представлен вещественным числом. Совокупность генов-переменных исследуемой функции, характеризующая особь - хромосома.
Договоримся изображать хромосому в виде столбика. Тогда хромосома для функции f(x)=x^2 , будет выглядеть так:
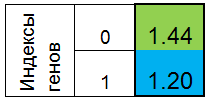
Рисунок 1. Хромосома для функции f(x)=x^2
где 0-й индекс — значение функции f(x), называют приспособленностью особи (функцию будем называть фитнес функцией - FF, а значение функции — VFF). Хромосому удобно хранить в одномерном массиве. Для этого служит массив double Chromosome[].
Все особи одной эпохи развития объединены в популяцию. Причем, популяция условно разделена на две равные колонии — родительскую и колонию потомков. В результате скрещивания родительских особей, которые отбираются из всей популяции, и других операторов ГА, появляется колония новых потомков, равная по численности половине популяции, которая замещает колонию потомков в популяции.
Полная популяция особей при поиске минимума функции f(x)=x^2 может выглядеть, например, так:
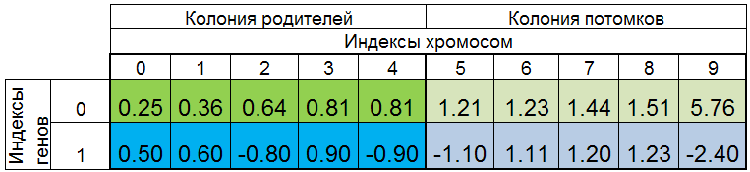
Рисунок 2. Полная популяция особей
Популяция отсортирована по VFF. Здесь 0-й индекс хромосомы занимает особь с наименьшим VFF. Новые потомки полностью замещают только особей в колонии потомков, родительская колония остаётся неприкосновенной. Однако, колония родителей может не всегда быть полной, так как дубликаты особей уничтожаются, тогда новые потомки заполняют свободные места в колонии родителей, оставшиеся помещаются в колонию потомков.
Другими словами, размер популяции редко бывает постоянным и меняется от эпохи к эпохе, почти также как и в живой природе. Например, популяция до размножения и популяция после размножения могут выглядеть так:
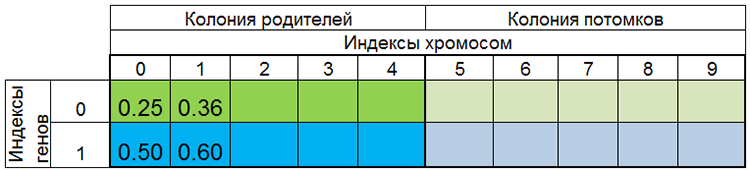
Рисунок 3. Популяция до размножения
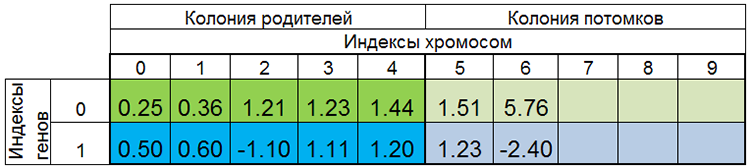
Рисунок 4. Популяция после размножения
Описанный механизм "половинного" заселения потомками популяции, а также уничтожение дубликатов и запрет на скрещивание особей самим с собой, имеют одну цель — избегать "эффекта бутылочного горлышка" (термин из биологии, означает сокращение генофонда популяции в результате критического уменьшения численности по различным причинам, что может привести к полному вымиранию вида, в ГА это грозит прекращением появления уникальных хромосом и “застреванием” в одном из локальных экстремумов.)
3. Описание функций UGAlib
Алгоритм ГА:- Создание протопопуляции. Гены генерируются случайно в заданном диапазоне.
- Определение приспособленности каждой особи, или другими словами, вычисление VFF.
- Подготовка популяции к размножению, удалив дубликаты хромосом.
- Выделение и сохранение эталонной хромосомы (c лучшим VFF).
- Операторы UGA (отбор, скрещивание, мутация). Для каждого скрещивания и мутации каждый раз отбираются родители по-новому. Подготовка популяции к следующей эпохе.
- Сравнение генов лучшего потомка с генами эталонной хромосомы. Если хромосома лучшего потомка лучше эталонной, заменить эталонную хромосому.
Продолжать с п.5 до тех пор, пока перестанут появляться хромосомы лучше эталонной заданное количество эпох.
3.1. Global variables. Глобальные переменные
Объявим на глобальном уровне следующие переменные:
//----------------------Глобальные переменные----------------------------- double Chromosome[]; //Набор оптимизируемых аргументов функции - генов //(например: веса нейронной сети и т.д.)-хромосома int ChromosomeCount =0; //Максимально возможное количество хромосом в колонии int TotalOfChromosomesInHistory=0;//Общее количество хромосом в истории int ChrCountInHistory =0; //Количество уникальных хромосом в базе хромосом int GeneCount =0; //Количество генов в хромосоме double RangeMinimum =0.0;//Минимум диапазона поиска double RangeMaximum =0.0;//Максимум диапазона поиска double Precision =0.0;//Шаг поиска int OptimizeMethod =0; //1-минимум, любое другое - максимум int FFNormalizeDigits =0; //Кол-во знаков после запятой в значении приспособленности int GeneNormalizeDigits =0; //Кол-во знаков после запятой в значении генов double Population [][1000]; //Популяция double Colony [][500]; //Колония потомков int PopulChromosCount =0; //Текущее количество хромосом в популяции int Epoch =0; //Кол-во эпох без улучшения int AmountStartsFF=0; //Количество запусков функции приспособленности //————————————————————————————————————————————————————————————————————————
3.2. UGA. Основная функция ГА
Собственно, основная функция ГА, вызываемая из тела программы - выполнять описанные выше шаги, поэтому, подробно на ней останавливаться не будем.
По завершении алгоритма в лог записывается следующая информация:
- Сколько всего прошло эпох (поколений).
- Сколько всего сбросов.
- Количество уникальных хромосом.
- Общее кол-во запусков FF.
- Общее кол-во хромосом в истории.
- Процентное отношение дубликатов к общему кол-ву хромосом в истории.
- Лучший результат.
"Количество уникальных хромосом" и "Общее кол-во запусков FF" – одни и те же величины, но рассчитываются по-разному. Использовать для контроля.
//———————————————————————————————————————————————————————————————————————— //Основная функция UGA void UGA ( double ReplicationPortion, //Доля Репликации. double NMutationPortion, //Доля Естественной мутации. double ArtificialMutation, //Доля Искусственной мутации. double GenoMergingPortion, //Доля Заимствования генов. double CrossingOverPortion,//Доля Кроссинговера. //--- double ReplicationOffset, //Коэффициент смещения границ интервала double NMutationProbability//Вероятность мутации каждого гена в % ) { //сброс генератора, производится только один раз MathSrand((int)TimeLocal()); //-----------------------Переменные------------------------------------- int chromos=0, gene =0;//индексы хромосом и генов int resetCounterFF =0;//счетчик сбросов "Эпох без улучшений" int currentEpoch =1;//номер текущей эпохи int SumOfCurrentEpoch=0;//сумма "Эпох без улучшений" int MinOfCurrentEpoch=Epoch;//минимальное "Эпох без улучшений" int MaxOfCurrentEpoch=0;//максимальное "Эпох без улучшений" int epochGlob =0;//общее количество эпох // Колония [количество признаков(генов)][количество особей в колонии] ArrayResize (Population,GeneCount+1); ArrayInitialize(Population,0.0); // Колония потомков [количество признаков(генов)][количество особей в колонии] ArrayResize (Colony,GeneCount+1); ArrayInitialize(Colony,0.0); // Банк хромосом // [количество признаков(генов)][количество хромосом в банке] double historyHromosomes[][100000]; ArrayResize (historyHromosomes,GeneCount+1); ArrayInitialize(historyHromosomes,0.0); //---------------------------------------------------------------------- //--------------Проверка корректности входных параметров---------------- //...количество хромосом должно быть не меньше 2 if(ChromosomeCount<=1) ChromosomeCount=2; if(ChromosomeCount>500) ChromosomeCount=500; //---------------------------------------------------------------------- //====================================================================== // 1) Создать протопопуляцию —————1) ProtopopulationBuilding (); //====================================================================== // 2) Определить приспособленность каждой особи —————2) //Для 1-ой колонии for(chromos=0;chromos<ChromosomeCount;chromos++) for(gene=1;gene<=GeneCount;gene++) Colony[gene][chromos]=Population[gene][chromos]; GetFitness(historyHromosomes); for(chromos=0;chromos<ChromosomeCount;chromos++) Population[0][chromos]=Colony[0][chromos]; //Для 2-ой колонии for(chromos=ChromosomeCount;chromos<ChromosomeCount*2;chromos++) for(gene=1;gene<=GeneCount;gene++) Colony[gene][chromos-ChromosomeCount]=Population[gene][chromos]; GetFitness(historyHromosomes); for(chromos=ChromosomeCount;chromos<ChromosomeCount*2;chromos++) Population[0][chromos]=Colony[0][chromos-ChromosomeCount]; //====================================================================== // 3) Подготовить популяцию к размножению ————3) RemovalDuplicates(); //====================================================================== // 4) Выделить эталонную хромосому —————4) for(gene=0;gene<=GeneCount;gene++) Chromosome[gene]=Population[gene][0]; //====================================================================== ServiceFunction(); //Основной цикл генетического алгоритма с 5 по 6 while(currentEpoch<=Epoch) { //==================================================================== // 5) Операторы UGA —————5) CycleOfOperators ( historyHromosomes, //--- ReplicationPortion, //Доля Репликации. NMutationPortion, //Доля Естественной мутации. ArtificialMutation, //Доля Искусственной мутации. GenoMergingPortion, //Доля Заимствования генов. CrossingOverPortion,//Доля Кроссинговера. //--- ReplicationOffset, //Коэффициент смещения границ интервала NMutationProbability//Вероятность мутации каждого гена в % ); //==================================================================== // 6) Сравнить гены лучшего потомка с генами эталонной хромосомы. // Если хромосома лучшего потомка лучше эталонной, // заменить эталонную. —————6) //Если режим оптимизации - минимизация if(OptimizeMethod==1) { //Если лучшая хромосома популяции лучше эталонной if(Population[0][0]<Chromosome[0]) { //Заменим эталонную хромосому for(gene=0;gene<=GeneCount;gene++) Chromosome[gene]=Population[gene][0]; ServiceFunction(); //Сбросим счетчик "эпох без улучшений" if(currentEpoch<MinOfCurrentEpoch) MinOfCurrentEpoch=currentEpoch; if(currentEpoch>MaxOfCurrentEpoch) MaxOfCurrentEpoch=currentEpoch; SumOfCurrentEpoch+=currentEpoch; currentEpoch=1; resetCounterFF++; } else currentEpoch++; } //Если режим оптимизации - максимизация else { //Если лучшая хромосома популяции лучше эталонной if(Population[0][0]>Chromosome[0]) { //Заменим эталонную хромосому for(gene=0;gene<=GeneCount;gene++) Chromosome[gene]=Population[gene][0]; ServiceFunction(); //Сбросим счетчик "эпох без улучшений" if(currentEpoch<MinOfCurrentEpoch) MinOfCurrentEpoch=currentEpoch; if(currentEpoch>MaxOfCurrentEpoch) MaxOfCurrentEpoch=currentEpoch; SumOfCurrentEpoch+=currentEpoch; currentEpoch=1; resetCounterFF++; } else currentEpoch++; } //==================================================================== //Прошла ещё одна эпоха.... epochGlob++; } Print("Прошло всего эпох=",epochGlob," Всего сбросов=",resetCounterFF); Print("MinOfCurrentEpoch",MinOfCurrentEpoch, " AverageOfCurrentEpoch",NormalizeDouble((double)SumOfCurrentEpoch/(double)resetCounterFF,2), " MaxOfCurrentEpoch",MaxOfCurrentEpoch); Print(ChrCountInHistory," - Уникальных хромосом"); Print(AmountStartsFF," - Общее кол-во запусков FF"); Print(TotalOfChromosomesInHistory," - Общее кол-во хромосом в истории"); Print(NormalizeDouble(100.0-((double)ChrCountInHistory*100.0/(double)TotalOfChromosomesInHistory),2),"% дубликатов"); Print(Chromosome[0]," - Лучший результат"); } //————————————————————————————————————————————————————————————————————————
3.3. ProtopopulationBuilding. Создание протопопуляции.
Поскольку в большинстве задач оптимизации заранее неизвестно, где на числовой прямой расположены аргументы функции, то наиболее оптимальным вариантом является случайная генерация в заданном диапазоне.
//———————————————————————————————————————————————————————————————————————— //Создание протопопуляции void ProtopopulationBuilding() { PopulChromosCount=ChromosomeCount*2; //Заполнить популяцию хромосомами со случайными //...генами в диапазоне RangeMinimum...RangeMaximum for(int chromos=0;chromos<PopulChromosCount;chromos++) { //начиная с 1-го индекса (0-ой -зарезервирован для VFF) for(int gene=1;gene<=GeneCount;gene++) Population[gene][chromos]= NormalizeDouble(SelectInDiscreteSpace(RNDfromCI(RangeMinimum,RangeMaximum),RangeMinimum,RangeMaximum,Precision,3),GeneNormalizeDigits); TotalOfChromosomesInHistory++; } } //————————————————————————————————————————————————————————————————————————
3.4. GetFitness. Получение приспособленности
Выполняет по очереди для всех хромосом оптимизируемую функцию.
//———————————————————————————————————————————————————————————————————————— //Получение приспособленности для каждой особи. void GetFitness ( double &historyHromosomes[][100000] ) { for(int chromos=0;chromos<ChromosomeCount;chromos++) CheckHistoryChromosomes(chromos,historyHromosomes); } //————————————————————————————————————————————————————————————————————————
3.5. CheckHistoryChromosomes. Проверка хромосомы по базе хромосом
Хромосома каждой особи проверяется по базе - рассчитывалась ли для неё FF, и если рассчитывалась, то готовое VFF берется из базы, если нет, то для неё вызывается FF. Таким образом, исключаются повторные ресурсоемкие вычисления FF.
//———————————————————————————————————————————————————————————————————————— //Проверка хромосомы по базе хромосом. void CheckHistoryChromosomes ( int chromos, double &historyHromosomes[][100000] ) { //-----------------------Переменные------------------------------------- int Ch1=0; //Индекс хромосомы из базы int Ge =0; //Индекс гена int cnt=0; //Счетчик уникальных генов. Если хоть один ген отличается //- хромосома признается уникальной //---------------------------------------------------------------------- //Если в базе хранится хоть одна хромосома if(ChrCountInHistory>0) { //Переберем хромосомы в базе, чтобы найти такую же for(Ch1=0;Ch1<ChrCountInHistory && cnt<GeneCount;Ch1++) { cnt=0; //Сверяем гены, пока индекс гена меньше кол-ва генов и пока попадаются одинаковые гены for(Ge=1;Ge<=GeneCount;Ge++) { if(Colony[Ge][chromos]!=historyHromosomes[Ge][Ch1]) break; cnt++; } } //Если набралось одинаковых генов столько же, можно взять готовое решение из базы if(cnt==GeneCount) Colony[0][chromos]=historyHromosomes[0][Ch1-1]; //Если нет такой же хромосомы в базе, то рассчитаем для неё FF... else { FitnessFunction(chromos); //.. и если есть место в базе сохраним if(ChrCountInHistory<100000) { for(Ge=0;Ge<=GeneCount;Ge++) historyHromosomes[Ge][ChrCountInHistory]=Colony[Ge][chromos]; ChrCountInHistory++; } } } //Если база пустая, рассчитаем для неё FF и сохраним её в базе else { FitnessFunction(chromos); for(Ge=0;Ge<=GeneCount;Ge++) historyHromosomes[Ge][ChrCountInHistory]=Colony[Ge][chromos]; ChrCountInHistory++; } } //————————————————————————————————————————————————————————————————————————
3.6. CycleOfOperators. Цикл операторов UGA
Здесь, в прямом смысле, вершится судьба целой эпохи искусственной жизни - рождается и умирает целое поколение. Происходит это следующим образом: отбираются два родителя для скрещивания или один для совершения над ним акта мутации. Доля каждого оператора ГА определена соответствующим параметром. В результате появляется один потомок. Всё повторяется снова и снова, пока полностью не заполнится колония потомков. Потом колония потомков выпускается в среду обитания, что бы каждый проявил себя, как может, т.е. вычисляем VFF.
Испытанная "огнем, водой и медными трубами" колония потомков заселяется в популяцию. Следующим шагом искусственной эволюции будет священное убиение клонов, чтобы не допустить обеднения "крови" у будущих поколений, и последующее ранжирование уже обновленной популяции, по степени приспособленности.
//———————————————————————————————————————————————————————————————————————— //Цикл операторов UGA void CycleOfOperators ( double &historyHromosomes[][100000], //--- double ReplicationPortion, //Доля Репликации. double NMutationPortion, //Доля Естественной мутации. double ArtificialMutation, //Доля Искусственной мутации. double GenoMergingPortion, //Доля Заимствования генов. double CrossingOverPortion,//Доля Кроссинговера. //--- double ReplicationOffset, //Коэффициент смещения границ интервала double NMutationProbability//Вероятность мутации каждого гена в % ) { //-----------------------Переменные------------------------------------- double child[]; ArrayResize (child,GeneCount+1); ArrayInitialize(child,0.0); int gene=0,chromos=0, border=0; int i=0,u=0; double p=0.0,start=0.0; double fit[][2]; ArrayResize (fit,6); ArrayInitialize(fit,0.0); //Счетчик посадочных мест в новой популяции. int T=0; //---------------------------------------------------------------------- //Зададим долю операторов UGA double portion[6]; portion[0]=ReplicationPortion; //Доля Репликации. portion[1]=NMutationPortion; //Доля Естественной мутации. portion[2]=ArtificialMutation; //Доля Искусственной мутации. portion[3]=GenoMergingPortion; //Доля Заимствования генов. portion[4]=CrossingOverPortion;//Доля Кроссинговера. portion[5]=0.0; //------------------------Цикл операторов UGA--------- //Заполняем новую колонию потомками while(T<ChromosomeCount) { //============================ for(i=0;i<6;i++) { fit[i][0]=start; fit[i][1]=start+MathAbs(portion[i]-portion[5]); start=fit[i][1]; } p=RNDfromCI(fit[0][0],fit[4][1]); for(u=0;u<5;u++) { if((fit[u][0]<=p && p<fit[u][1]) || p==fit[u][1]) break; } //============================ switch(u) { //--------------------- case 0: //------------------------Репликация-------------------------------- //Если есть место в новой колонии, создадим новую особь if(T<ChromosomeCount) { Replication(child,ReplicationOffset); //Поселим новую особь в новую колонию for(gene=1;gene<=GeneCount;gene++) Colony[gene][T]=child[gene]; //Одно место заняли, счетчик перемотаем вперед T++; TotalOfChromosomesInHistory++; } //--------------------------------------------------------------- break; //--------------------- case 1: //---------------------Естественная мутация------------------------- //Если есть место в новой колонии, создадим новую особь if(T<ChromosomeCount) { NaturalMutation(child,NMutationProbability); //Поселим новую особь в новую колонию for(gene=1;gene<=GeneCount;gene++) Colony[gene][T]=child[gene]; //Одно место заняли, счетчик перемотаем вперед T++; TotalOfChromosomesInHistory++; } //--------------------------------------------------------------- break; //--------------------- case 2: //----------------------Искусственная мутация----------------------- //Если есть место в новой колонии, создадим новую особь if(T<ChromosomeCount) { ArtificialMutation(child,ReplicationOffset); //Поселим новую особь в новую колонию for(gene=1;gene<=GeneCount;gene++) Colony[gene][T]=child[gene]; //Одно место заняли, счетчик перемотаем вперед T++; TotalOfChromosomesInHistory++; } //--------------------------------------------------------------- break; //--------------------- case 3: //-------------Образование особи с заимствованными генами----------- //Если есть место в новой колонии, создадим новую особь if(T<ChromosomeCount) { GenoMerging(child); //Поселим новую особь в новую колонию for(gene=1;gene<=GeneCount;gene++) Colony[gene][T]=child[gene]; //Одно место заняли, счетчик перемотаем вперед T++; TotalOfChromosomesInHistory++; } //--------------------------------------------------------------- break; //--------------------- default: //---------------------------Кроссинговер--------------------------- //Если есть место в новой колонии, создадим новую особь if(T<ChromosomeCount) { CrossingOver(child); //Поселим новую особь в новую колонию for(gene=1;gene<=GeneCount;gene++) Colony[gene][T]=child[gene]; //Одно место заняли, счетчик перемотаем вперед T++; TotalOfChromosomesInHistory++; } //--------------------------------------------------------------- break; //--------------------- } }//Конец цикла операторов UGA-- //Определим приспособленность каждой особи в колонии потомков GetFitness(historyHromosomes); //Поселим потомков в основную популяцию if(PopulChromosCount>=ChromosomeCount) { border=ChromosomeCount; PopulChromosCount=ChromosomeCount*2; } else { border=PopulChromosCount; PopulChromosCount+=ChromosomeCount; } for(chromos=0;chromos<ChromosomeCount;chromos++) for(gene=0;gene<=GeneCount;gene++) Population[gene][chromos+border]=Colony[gene][chromos]; //Подготовим популяцию к следующему размножению RemovalDuplicates(); }//конец ф-ии //————————————————————————————————————————————————————————————————————————
3.7. Replication. Репликация
Оператор наиболее близок к природному явлению, которое в биологии имеет название - Репликация ДНК, хотя и не является одним и тем же по сути. Но поскольку более близкого природного эквивалента я не нашел, решил остановиться на этом названии.
Репликация является важнейшим генетическим оператором генерирующий новые гены, при этом передающий признаки родительских хромосом. Основной оператор, обеспечивающий сходимость алгоритма. ГА может функционировать только с ним без использования других операторов, однако при этом количество запусков FF окажется намного большим.
Рассмотрим принцип работы оператора Replication. Используется две родительские хромосомы. Новый ген потомка случайное число из интервала
[C1-((C2-C1)*ReplicationOffset),C2+((C2-C1)*ReplicationOffset)]
где С1 и С2 родительские гены, ReplicationOffset — коэффициент смещения границ интервала [C1,C2].
К примеру, от отцовской особи (синий цвет) и материнской (розовый цвет) может получиться такой потомок (зеленый цвет):
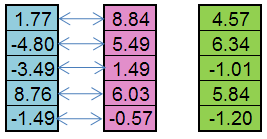
Рисунок 5. Принцип работы оператора Replication
Графически вероятность гена потомка можно представить так:
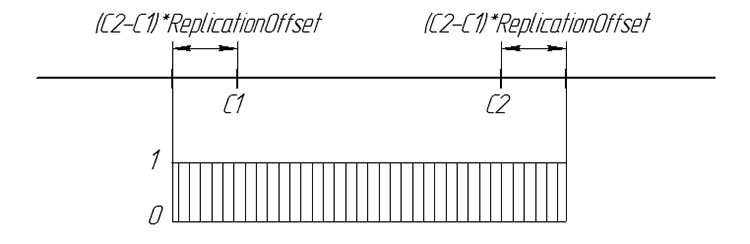
Рисунок 6. Вероятность появления гена потомка на числовой прямой
Аналогично генерируются остальные гены потомка.
//———————————————————————————————————————————————————————————————————————— //Репликация void Replication ( double &child[], double ReplicationOffset ) { //-----------------------Переменные------------------------------------- double C1=0.0,C2=0.0,temp=0.0,Maximum=0.0,Minimum=0.0; int address_mama=0,address_papa=0; //---------------------------------------------------------------------- SelectTwoParents(address_mama,address_papa); //-------------------Цикл перебора генов-------------------------------- for(int i=1;i<=GeneCount;i++) { //----определим откуда мать и отец -------- C1 = Population[i][address_mama]; C2 = Population[i][address_papa]; //------------------------------------------ //Обязательная проверка, что бы поиск не вышел из заданного диапазона if(C1 < RangeMinimum) C1 = RangeMinimum; if(C1 > RangeMaximum) C1 = RangeMaximum; if(C2 < RangeMinimum) C2 = RangeMinimum; if(C2 > RangeMaximum) C2 = RangeMaximum; //------------------------------------------------------------------ //....определим наибольший и наименьший из них, //если С1>C2, поменяем их местами if(C1>C2) { temp = C1; C1=C2; C2 = temp; } //-------------------------------------------- //Назначим границы создания нового гена Minimum = C1-((C2-C1)*ReplicationOffset); Maximum = C2+((C2-C1)*ReplicationOffset); //-------------------------------------------- //Обязательная проверка, что бы поиск не вышел из заданного диапазона if(Minimum < RangeMinimum) Minimum = RangeMinimum; if(Maximum > RangeMaximum) Maximum = RangeMaximum; //--------------------------------------------------------------- temp=RNDfromCI(Minimum,Maximum); child[i]= NormalizeDouble(SelectInDiscreteSpace(temp,RangeMinimum,RangeMaximum,Precision,3),GeneNormalizeDigits); } } //————————————————————————————————————————————————————————————————————————
3.8. NaturalMutation. Естественная мутация
Мутации появляются постоянно в ходе процессов, происходящих в живой клетке, и являются материалом для естественного отбора. Возникают самопроизвольно на протяжении всей жизни организма в нормальных для него условиях окружающей среды с частотой одного раза на 10^10 клеточных генераций.
Нам - любознательным исследователям, не обязательно придерживаться естественного порядка и ждать так долго очередной мутации гена. В этом поможет параметр NMutationProbability, выраженный в процентах, который определяет вероятность мутации каждого гена в хромосоме.
В операторе NaturalMutation мутация заключается в генерации случайного гена в интервале [RangeMinimum,RangeMaximum]. NMutationProbability=100% будет означать 100% мутацию всех генов в хромосоме, а NMutationProbability=0% - полное отсутствие мутаций. Последний вариант в практических задачах использовать не имеет смысла.
//———————————————————————————————————————————————————————————————————————— //Естественная мутация. void NaturalMutation ( double &child[], double NMutationProbability ) { //-----------------------Переменные------------------------------------- int address=0; double prob=0.0; //---------------------------------------------------------------------- if(NMutationProbability<0.0) prob=0.0; if(NMutationProbability>100.0) prob=100.0; //-----------------Отбор родителя------------------------ SelectOneParent(address); //--------------------------------------- for(int i=1;i<=GeneCount;i++) if(RNDfromCI(0.0,100.0)<prob) child[i]=NormalizeDouble( SelectInDiscreteSpace(RNDfromCI(RangeMinimum,RangeMaximum),RangeMinimum,RangeMaximum,Precision,3),GeneNormalizeDigits ); } //————————————————————————————————————————————————————————————————————————
3.9. ArtificialMutation. Искусственная мутация
Основная задача оператора — генерация "свежей крови". Используются два родителя, а гены потомка выбираются из незанятого генами родителей пространства на числовой прямой. Предохраняет ГА от застревания в одном из локальных экстремумов. В небольших долях по сравнению с другими операторами ускоряет сходимость, иначе - замедляет, увеличивая количество запусков FF.
Так же, как и в Replication, используется две родительские хромосомы. Но задача оператора ArtificialMutation не передать потомку признаки родителей, а наоборот, сделать потомка непохожим на них. Потому, являясь полной противоположностью, используя тот же коэффициент смещения границ интервала, но гены генерируются вне интервала, который бы занял Replication. Новый ген потомка случайное число из интервалов [RangeMinimum, C1-(C2-C1)*ReplicationOffset] и [C2+(C2-C1)*ReplicationOffset, RangeMaximum]
Графически вероятность гена потомка при ReplicationOffset=0.25 можно представить так:
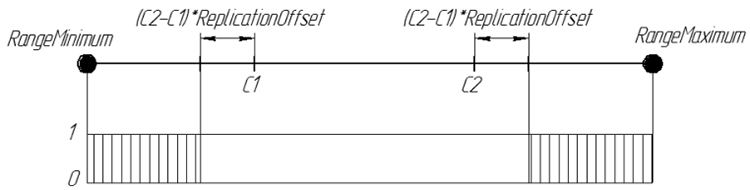
Рисунок 7. Вероятность появления гена потомка при ReplicationOffset=0.25 на числовой прямой интервале [RangeMinimum;RangeMaximum]
//———————————————————————————————————————————————————————————————————————— //Искусственная мутация. void ArtificialMutation ( double &child[], double ReplicationOffset ) { //-----------------------Переменные------------------------------------- double C1=0.0,C2=0.0,temp=0.0,Maximum=0.0,Minimum=0.0,p=0.0; int address_mama=0,address_papa=0; //---------------------------------------------------------------------- //-----------------Отбор родителей------------------------ SelectTwoParents(address_mama,address_papa); //-------------------------------------------------------- //-------------------Цикл перебора генов------------------------------ for(int i=1;i<=GeneCount;i++) { //----определим откуда мать и отец -------- C1 = Population[i][address_mama]; C2 = Population[i][address_papa]; //------------------------------------------ //Обязательная проверка, что бы поиск не вышел из заданного диапазона if(C1 < RangeMinimum) C1 = RangeMinimum; if(C1 > RangeMaximum) C1 = RangeMaximum; if(C2 < RangeMinimum) C2 = RangeMinimum; if(C2 > RangeMaximum) C2 = RangeMaximum; //------------------------------------------------------------------ //....определим наибольший и наименьший из них, //если С1>C2, поменяем их местами if(C1>C2) { temp=C1; C1=C2; C2=temp; } //-------------------------------------------- //Назначим границы создания нового гена Minimum=C1-((C2-C1)*ReplicationOffset); Maximum=C2+((C2-C1)*ReplicationOffset); //-------------------------------------------- //Обязательная проверка, что бы поиск не вышел из заданного диапазона if(Minimum < RangeMinimum) Minimum = RangeMinimum; if(Maximum > RangeMaximum) Maximum = RangeMaximum; //--------------------------------------------------------------- p=MathRand(); if(p<16383.5) { temp=RNDfromCI(RangeMinimum,Minimum); child[i]= NormalizeDouble(SelectInDiscreteSpace(temp,RangeMinimum,RangeMaximum,Precision,3),GeneNormalizeDigits); } else { temp=RNDfromCI(Maximum,RangeMaximum); child[i]= NormalizeDouble(SelectInDiscreteSpace(temp,RangeMinimum,RangeMaximum,Precision,3),GeneNormalizeDigits); } } } //————————————————————————————————————————————————————————————————————————
3.10 GenoMerging. Заимствование генов
Данный оператор ГА не имеет природного эквивалента. Да и трудно представить, как бы этот чудесный механизм работал у живых организмов. Однако, он имеет замечательное свойство переносить гены от множества родителей (число родителей равно числу генов) потомку. Оператор не генерирует новых генов и является комбинаторным механизмом поиска.
Работает так: для первого гена потомка отбирается родитель и берется у него первый ген, далее, для второго гена отбирается второй родитель и берется второй ген и т.д. Целесообразно применять при количестве генов больше одного. Иначе стоит отключить, так как оператор будет генерировать дубликаты хромосом.
//———————————————————————————————————————————————————————————————————————— //Заимствование генов. void GenoMerging ( double &child[] ) { //-----------------------Переменные------------------------------------- int address=0; //---------------------------------------------------------------------- for(int i=1;i<=GeneCount;i++) { //-----------------Отбор родителя------------------------ SelectOneParent(address); //-------------------------------------------------------- child[i]=Population[i][address]; } } //————————————————————————————————————————————————————————————————————————
3.11. CrossingOver. Кроссинговер
Кроссинговер (другое название в биологии перекрёст) — явление обмена участками хромосом. Так же, как и GenoMerging, является комбинаторным механизмом поиска.
Отбираются две родительские хромосомы. Обе “разрезаются” в случайном месте. Хромосома потомка будет состоять из родительских частей хромосом.
Механизм проще всего проиллюстрировать рисунком:
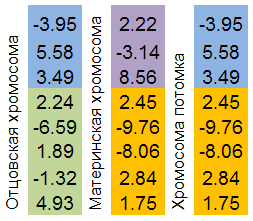
Рисунок 8. Механизм обмена участками хромосом
Целесообразно применять при количестве генов больше одного. Иначе стоит отключить, так как оператор будет генерировать дубликаты хромосом.
//———————————————————————————————————————————————————————————————————————— //Кроссинговер. void CrossingOver ( double &child[] ) { //-----------------------Переменные------------------------------------- int address_mama=0,address_papa=0; //---------------------------------------------------------------------- //-----------------Отбор родителей------------------------ SelectTwoParents(address_mama,address_papa); //-------------------------------------------------------- //Определим точку разрыва int address_of_gene=(int)MathFloor((GeneCount-1)*(MathRand()/32767.5)); for(int i=1;i<=GeneCount;i++) { //----копируем гены матери-------- if(i<=address_of_gene+1) child[i]=Population[i][address_mama]; //----копируем гены отца-------- else child[i]=Population[i][address_papa]; } } //————————————————————————————————————————————————————————————————————————
3.12. SelectTwoParents. Отбор двух родителей
Для предотвращения обеднения генофонда введен запрет на скрещивание сам с собой. Делается 10 попыток найти разных родителей, если так и не удалось найти пару, дается добро на скрещивание особи сам с собой. По сути, появится копия этой особи.
С одной стороны, уменьшается вероятность клонирования особей, с другой – не допускается возможность зацикливания поиска, так как может возникнуть ситуация, когда это сделать практически невозможно (выбрать разных родителей) за приемлемое количество шагов.
Используется в операторах Replication, ArtificialMutation, CrossingOver.
//———————————————————————————————————————————————————————————————————————— //Отбор двух родителей. void SelectTwoParents ( int &address_mama, int &address_papa ) { //-----------------------Переменные------------------------------------- int cnt=1; address_mama=0;//адрес материнской особи в популяции address_papa=0;//адрес отцовской особи в популяции //---------------------------------------------------------------------- //----------------------------Отбор родителей-------------------------- //Десять попыток выбрать разных родителей. while(cnt<=10) { //Для материнской особи address_mama=NaturalSelection(); //Для отцовской особи address_papa=NaturalSelection(); if(address_mama!=address_papa) break; } //--------------------------------------------------------------------- } //————————————————————————————————————————————————————————————————————————
3.13. SelectOneParent. Отбор одного родителя
Здесь всё просто — из всей популяции отбирается один родитель.
Используется в операторах NaturalMutation и GenoMerging.
//———————————————————————————————————————————————————————————————————————— //Отбор одного родителя. void SelectOneParent ( int &address//адрес родительской особи в популяции ) { //-----------------------Переменные------------------------------------- address=0; //---------------------------------------------------------------------- //----------------------------Отбор родителя-------------------------- address=NaturalSelection(); //--------------------------------------------------------------------- } //————————————————————————————————————————————————————————————————————————
3.14. NaturalSelection. Естественный отбор
Естественный отбор — процесс, приводящий к выживанию и преимущественному размножению более приспособленных к данным условиям среды особей, обладающих полезными наследственными признаками.
Оператор похож на традиционный оператор "Рулетка" (Roulette-wheel selection – отбор особей с помощью n "запусков" рулетки. Колесо рулетки содержит по одному сектору для каждого члена популяции. Размер i-ого сектора пропорционален соответствующей величине приспособленности), но имеет существенные отличия. Он учитывает положение особей относительно самой приспособленной и наименее приспособленной. Причем, даже особь, имеющая самые плохие гены, имеет шанс оставить потомка. Справедливо, не правда ли? Хотя дело не в справедливости, а в том, что в природе все особи имеют возможность оставить потомка.
Для примера, возьмем 10 особей, имеющие такие VFF в задаче максимизации: 256, 128, 64, 32, 16, 8, 4, 2, 0,-1 - где наибольшее значение соответствует лучшей приспособленности. Такой пример взят для того, что бы было видно, что "расстояние" между соседними особями в 2 раза больше, чем между двумя предыдущими особями. Однако на круговой диаграмме вероятность того, что каждая особь оставит потомка выглядит так:
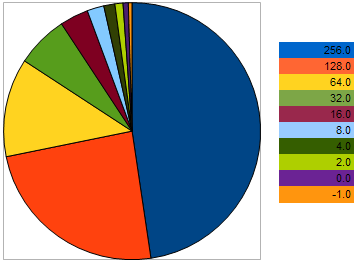
Рисунок 9. Диаграмма вероятности отбора родительский особей
на ней видно, что с приближением положения особей к наихудшей их шансы уравниваются с наихудшей. И наоборот - чем ближе к лучшей особи, тем более равные с лучшей имеет шансы на размножение особь:

Рисунок 10. Диаграмма вероятности отбора родительский особей
//———————————————————————————————————————————————————————————————————————— //Естественный отбор. int NaturalSelection() { //-----------------------Переменные------------------------------------- int i=0,u=0; double p=0.0,start=0.0; double fit[][2]; ArrayResize (fit,PopulChromosCount); ArrayInitialize(fit,0.0); double delta=(Population[0][0]-Population[0][PopulChromosCount-1])*0.01-Population[0][PopulChromosCount-1]; //---------------------------------------------------------------------- for(i=0;i<PopulChromosCount;i++) { fit[i][0]=start; fit[i][1]=start+MathAbs(Population[0][i]+delta); start=fit[i][1]; } p=RNDfromCI(fit[0][0],fit[PopulChromosCount-1][1]); for(u=0;u<PopulChromosCount;u++) if((fit[u][0]<=p && p<fit[u][1]) || p==fit[u][1]) break; return(u); } //————————————————————————————————————————————————————————————————————————
3.15. RemovalDuplicates. Удаление дубликатов
Функция удаляет дубликаты хромосом в популяции, а оставшиеся уникальные хромосомы (уникальные для популяции текущей эпохи) сортируются по VFF в порядке, определяемым типом оптимизации, т.е. по убыванию или по возрастанию.
//———————————————————————————————————————————————————————————————————————— //Удаление дубликатов с сортировкой по VFF void RemovalDuplicates() { //-----------------------Переменные------------------------------------- int chromosomeUnique[1000];//Массив хранит признак уникальности //каждой хромосомы: 0-дубликат, 1-уникальная ArrayInitialize(chromosomeUnique,1); //Предположим, что дубликатов нет double PopulationTemp[][1000]; ArrayResize (PopulationTemp,GeneCount+1); ArrayInitialize(PopulationTemp,0.0); int Ge =0; //Индекс гена int Ch =0; //Индекс хромосомы int Ch2=0; //Индекс второй хромосомы int cnt=0; //Счетчик //---------------------------------------------------------------------- //----------------------Удалим дубликаты---------------------------1 //Выбираем первый из пары для сравнения... for(Ch=0;Ch<PopulChromosCount;Ch++) { //Если не дубликат... if(chromosomeUnique[Ch]!=0) { //Выбираем второй из пары... for(Ch2=0;Ch2<PopulChromosCount;Ch2++) { if(Ch!=Ch2 && chromosomeUnique[Ch2]!=0) { //Обнулим счетчик количества идентичных генов cnt=0; //Сверяем гены, пока попадаются одинаковые гены for(Ge=1;Ge<=GeneCount;Ge++) { if(Population[Ge][Ch]!=Population[Ge][Ch2]) break; else cnt++; } //Если набралось одинаковых генов столько же, сколько всего генов //..хромосома признается дубликатом if(cnt==GeneCount) chromosomeUnique[Ch2]=0; } } } } //Счетчик посчитает количество уникальных хромосом cnt=0; //Скопируем уникальные хромосомы во временный масив for(Ch=0;Ch<PopulChromosCount;Ch++) { //Если хромосома уникальна, скопируем её, если нет, перейдем к следующей if(chromosomeUnique[Ch]==1) { for(Ge=0;Ge<=GeneCount;Ge++) PopulationTemp[Ge][cnt]=Population[Ge][Ch]; cnt++; } } //Назначим переменной "Всего хромосом" значение счетчика уникальных хромосом PopulChromosCount=cnt; //Вернем уникальные хромосомы обратно в массив для временного хранения //..обьединяемых популяций for(Ch=0;Ch<PopulChromosCount;Ch++) for(Ge=0;Ge<=GeneCount;Ge++) Population[Ge][Ch]=PopulationTemp[Ge][Ch]; //=================================================================1 //----------------Ранжирование популяции---------------------------2 PopulationRanking(); //=================================================================2 } //————————————————————————————————————————————————————————————————————————
3.16. PopulationRanking. Ранжирование популяции
Сортировка производится по VFF. Метод похож на "пузырьковый" (Алгоритм состоит в повторяющихся проходах по сортируемому массиву. За каждый проход элементы последовательно сравниваются попарно и, если порядок в паре неверный, выполняется обмен элементов. Проходы по массиву повторяются до тех пор, пока на очередном проходе не окажется, что обмены больше не нужны, что означает — массив отсортирован.
При проходе алгоритма, элемент, стоящий не на своём месте, «всплывает» до нужной позиции как пузырёк в воде, отсюда и название алгоритма, но есть отличие - сортируются только индексы массива, а не содержимое всего массива. Этот способ быстрее, и ненамного отличается по скорости от простого копирования одного массива в другой. И чем больше размер сортируемого массива, тем разница меньше.
//———————————————————————————————————————————————————————————————————————— //Ранжирование популяции. void PopulationRanking() { //-----------------------Переменные------------------------------------- int cnt=1, i = 0, u = 0; double PopulationTemp[][1000]; //Временная популяция ArrayResize (PopulationTemp,GeneCount+1); ArrayInitialize(PopulationTemp,0.0); int Indexes[]; //Индексы хромосом ArrayResize (Indexes,PopulChromosCount); ArrayInitialize(Indexes,0); int t0=0; double ValueOnIndexes[]; //VFF соответствующих //..индексов хромосом ArrayResize (ValueOnIndexes,PopulChromosCount); ArrayInitialize(ValueOnIndexes,0.0); double t1=0.0; //---------------------------------------------------------------------- //Проставим индексы во временном массиве temp2 и //...скопируем первую строку из сортируемого массива for(i=0;i<PopulChromosCount;i++) { Indexes[i] = i; ValueOnIndexes[i] = Population[0][i]; } if(OptimizeMethod==1) { while(cnt>0) { cnt=0; for(i=0;i<PopulChromosCount-1;i++) { if(ValueOnIndexes[i]>ValueOnIndexes[i+1]) { //----------------------- t0 = Indexes[i+1]; t1 = ValueOnIndexes[i+1]; Indexes [i+1] = Indexes[i]; ValueOnIndexes [i+1] = ValueOnIndexes[i]; Indexes [i] = t0; ValueOnIndexes [i] = t1; //----------------------- cnt++; } } } } else { while(cnt>0) { cnt=0; for(i=0;i<PopulChromosCount-1;i++) { if(ValueOnIndexes[i]<ValueOnIndexes[i+1]) { //----------------------- t0 = Indexes[i+1]; t1 = ValueOnIndexes[i+1]; Indexes [i+1] = Indexes[i]; ValueOnIndexes [i+1] = ValueOnIndexes[i]; Indexes [i] = t0; ValueOnIndexes [i] = t1; //----------------------- cnt++; } } } } //Создадим отсортированный массив по полученным индексам for(i=0;i<GeneCount+1;i++) for(u=0;u<PopulChromosCount;u++) PopulationTemp[i][u]=Population[i][Indexes[u]]; //Скопируем отсортированный массив обратно for(i=0;i<GeneCount+1;i++) for(u=0;u<PopulChromosCount;u++) Population[i][u]=PopulationTemp[i][u]; } //————————————————————————————————————————————————————————————————————————
3.17. RNDfromCustomInterval. Генератор случайных чисел из заданного интервала
Просто удобная функция. Пригодилась и в UGA.
//———————————————————————————————————————————————————————————————————————— //Генератор случайных чисел из заданного интервала. double RNDfromCI(double RangeMinimum,double RangeMaximum) { return(RangeMinimum+((RangeMaximum-RangeMinimum)*MathRand()/32767.5));} //————————————————————————————————————————————————————————————————————————
3.18. SelectInDiscreteSpace. Выбор в дискретном пространстве
Служит для уменьшения пространства поиска. При параметре step=0.0 поиск осуществляется в непрерывном пространстве (ограничено лишь языковыми ограничениями, в MQL до 15-го значащего знака включительно). Для использования алгоритма ГА с большей точностью, понадобится писать дополнительную библиотеку для работы с длинными числами.
Работу функции при RoundMode=1 можно проиллюстрировать следующим рисунком:
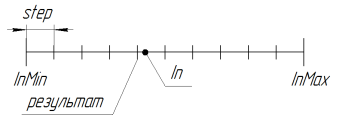
Рисунок 11. Работа функции SelectInDiscreteSpace при RoundMode=1
//———————————————————————————————————————————————————————————————————————— //Выбор в дискретном пространстве. //Режимы: //1-ближайшее снизу //2-ближайшее сверху //любое-до ближайшего double SelectInDiscreteSpace ( double In, double InMin, double InMax, double step, int RoundMode ) { if(step==0.0) return(In); // обеспечим правильность границ if( InMax < InMin ) { double temp = InMax; InMax = InMin; InMin = temp; } // при нарушении - вернем нарушенную границу if( In < InMin ) return( InMin ); if( In > InMax ) return( InMax ); if( InMax == InMin || step <= 0.0 ) return( InMin ); // приведем к заданному масштабу step = (InMax - InMin) / MathCeil ( (InMax - InMin) / step ); switch( RoundMode ) { case 1: return( InMin + step * MathFloor ( ( In - InMin ) / step ) ); case 2: return( InMin + step * MathCeil ( ( In - InMin ) / step ) ); default: return( InMin + step * MathRound ( ( In - InMin ) / step ) ); } } //————————————————————————————————————————————————————————————————————————
3.19. FitnessFunction. Функция приспособленности
Не является частью ГА. В функцию передается индекс положения хромосомы в популяции, для которой будет рассчитано VFF. VFF записывается в нулевой индекс передаваемой хромосомы. Код этой функции индивидуален для каждой задачи.
3.20. ServiceFunction. Сервисная функция
Не является частью ГА. Код этой функции индивидуален для каждой конкретной задачи. Может применяться для осуществления контроля над эпохами. Например, для того чтобы вывести на экран лучшее VFF для текущей эпохи.
4. Примеры работы UGA
Все задачи оптимизации, решаемые с помощью ЭА, делятся на два типа:
- Генотип соответствует фенотипу. Значения генов хромосом напрямую назначаются аргументам оптимизируемой функции. Пример 1.
- Генотип не соответствует фенотипу. Требуется интерпретация значений генов хромосом для вычисления оптимизируемой функции. Пример 2.
4.1. Пример 1
Рассмотрим задачу с заранее известным ответом, для того, что бы убедится, что алгоритм работает, а уже затем решим задачку, решение которой интересует многих трейдеров.
Задача: Найти минимум и максимум функции "Skin":
на участке [-5;5].
Ответ: fmin(3.07021,3.315935)= -4.3182, fmax(-3.315699;-3.072485)= 14.0606.
Рисунок 12. График функции "Skin" на участке [-5;5]
Для решения задачи напишем такой скрипт:
#property script_show_inputs //+——————————————————————————————————————————————————————————————————————+ #include "UGAlib.mqh" #include "Skin.mqh"//тестовая функция //+——————————————————————————————————————————————————————————————————————+ //———————————————————————————————————————————————————————————————————————— //----------------------Входные переменные-------------------------------- input string GenofundParam = "----Параметры генофонда----"; input int ChromosomeCount_P = 50; //Кол-во хромосом в колонии input int GeneCount_P = 2; //Кол-во генов input int FFNormalizeDigits_P = 4; //Кол-во знаков приспособлености input int GeneNormalizeDigits_P = 4; //Кол-во знаков гена input int Epoch_P = 50; //Кол-во эпох без улучшения //--- input string GA_OperatorParam = "----Параметры операторов----"; input double ReplicationPortion_P = 100.0; //Доля Репликации. input double NMutationPortion_P = 10.0; //Доля Естественной мутации. input double ArtificialMutation_P = 10.0; //Доля Искусственной мутации. input double GenoMergingPortion_P = 20.0; //Доля Заимствования генов. input double CrossingOverPortion_P = 20.0; //Доля Кроссинговера. //--- input double ReplicationOffset_P = 0.5; //Коэффициент смещения границ интервала input double NMutationProbability_P= 5.0; //Вероятность мутации каждого гена в % //--- input string OptimisationParam = "----Параметры оптимизации----"; input double RangeMinimum_P = -5.0; //Минимум диапазона поиска input double RangeMaximum_P = 5.0; //Максимум диапазона поиска input double Precision_P = 0.0001;//Требуемая точность input int OptimizeMethod_P = 1; //Оптим.:1-Min,другое-Max //———————————————————————————————————————————————————————————————————————— //———————————————————————————————————————————————————————————————————————— //----------------------Глобальные переменные----------------------------- double ERROR=0.0;//Средняя ошибка на ген //———————————————————————————————————————————————————————————————————————— //———————————————————————————————————————————————————————————————————————— //--------------------------Тело программы-------------------------------- int OnStart() { //-----------------------Переменные------------------------------------- //Подготовка глобальных переменных для UGA ChromosomeCount=ChromosomeCount_P; //Кол-во хромосом в колонии GeneCount =GeneCount_P; //Кол-во генов RangeMinimum =RangeMinimum_P; //Минимум диапазона поиска RangeMaximum =RangeMaximum_P; //Максимум диапазона поиска Precision =Precision_P; //Шаг поиска OptimizeMethod =OptimizeMethod_P; //1-минимум, любое другое-максимум FFNormalizeDigits = FFNormalizeDigits_P; //Кол-во знаков приспособлености GeneNormalizeDigits = GeneNormalizeDigits_P;//Кол-во знаков гена ArrayResize(Chromosome,GeneCount+1); ArrayInitialize(Chromosome,0); Epoch=Epoch_P; //Кол-во эпох без улучшения //---------------------------------------------------------------------- //Локальные переменные int time_start=GetTickCount(),time_end=0; //---------------------------------------------------------------------- //Запуск главной ф-ии UGA UGA ( ReplicationPortion_P, //Доля Репликации. NMutationPortion_P, //Доля Естественной мутации. ArtificialMutation_P, //Доля Искусственной мутации. GenoMergingPortion_P, //Доля Заимствования генов. CrossingOverPortion_P,//Доля Кроссинговера. //--- ReplicationOffset_P, //Коэффициент смещения границ интервала NMutationProbability_P//Вероятность мутации каждого гена в % ); //---------------------------------- time_end=GetTickCount(); //---------------------------------- Print(time_end-time_start," мс - Время исполнения"); //---------------------------------- return(0); } //————————————————————————————————————————————————————————————————————————
Вот и весь код скрипта для решения задачи. Запустив его, получим информацию, выданную функцией Comment() :
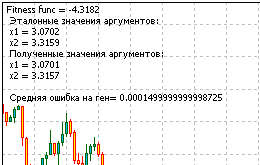
Рисунок 13. Результат решения задачи
Посмотрев на полученные результаты, убеждаемся, что алгоритм работает.
4.2. Пример 2
Широко распространено мнение, что индикатор ZZ показывает идеальные входы переворотной торговой системы. Индикатор очень популярен среди “волновиков” и тех, кто пользуется им для определения размера “фигур”.
Задача: Выяснить, существуют ли иные точки входов для переворотной торговой системы на исторических данных, отличные от вершин ZZ, дающие в сумме больше пунктов теоретической прибыли?
Для экспериментов выберем пару GBPJPY на M1 100 баров. Примем спред равным 80 пунктам (пятизначные котировки). Для начала, нужно определить наилучшие параметры ZZ. Для этого простым перебором найдем лучшее значение параметра ExtDepth с помощью нехитрого скрипта:
#property script_show_inputs //———————————————————————————————————————————————————————————————————————— //----------------------Входные переменные-------------------------------- input int History=100; input double Spred =80.0; input int Depth =5; //Для "одноразового" использования input bool loop =true;//Использовать перебор или нет //———————————————————————————————————————————————————————————————————————— //———————————————————————————————————————————————————————————————————————— //--------------------------Тело программы-------------------------------- void OnStart() { //-----------------------Переменные------------------------------------- double ZigzagBuffer [];//Для хранения буфера индикатора ZZ double PeaksOfZigzag[];//Для хранения значений экстремумов ZZ int Zigzag_handle; //Маркер индикатора ArraySetAsSeries(ZigzagBuffer,true); ArrayResize(PeaksOfZigzag,History); int depth=3; double PipsSum=0.0; int PeaksCount=0; bool flag=true; //---------------------------------------------------------------------- if(loop==true) { while(depth<200 && flag==true) { //----------------------------------------------------------- Zigzag_handle=iCustom(NULL,0,"ZigZag",depth); //--- сбросим код ошибки ResetLastError(); //--- попытаемся скопировать значения индикатора for(int i=0;i<100;i++) { if(BarsCalculated(Zigzag_handle)>0) break; Sleep(1000); } int copied=CopyBuffer(Zigzag_handle,0,0,History,ZigzagBuffer); if(copied<=0) { Print("Не удалось скопировать буфер индикатора. Error =",GetLastError()," copied=",copied); return; } //----------------------------------------------------------- PipsSum=0.0; PeaksCount=0; for(int u=0;u<History;u++) { if(NormalizeDouble(ZigzagBuffer[u],Digits())>0.0) { PeaksOfZigzag[PeaksCount]=NormalizeDouble(ZigzagBuffer[u],Digits()); PeaksCount++; } } //----------------------------------------------------------- for(int V=0;V<PeaksCount-1;V++) PipsSum+=NormalizeDouble((MathAbs(PeaksOfZigzag[V]-PeaksOfZigzag[V+1]))/Point(),Digits())-Spred; //----------------------------------------------------------- if(PeaksCount<=2) flag=false; else { Print(depth," ",PeaksCount," ",PipsSum); depth+=1; } //----------------------------------------------------------- } } else { //----------------------------------------------------------- Zigzag_handle=iCustom(NULL,0,"ZigZag",Depth); //--- сбросим код ошибки ResetLastError(); //--- попытаемся скопировать значения индикатора for(int i=0;i<History;i++) { if(BarsCalculated(Zigzag_handle)>0) break; Sleep(1000); } int copied=CopyBuffer(Zigzag_handle,0,0,History,ZigzagBuffer); if(copied<=0) { Print("Не удалось скопировать буфер индикатора. Error =",GetLastError()," copied=",copied); return; } //----------------------------------------------------------- for(int u=0;u<History;u++) { if(NormalizeDouble(ZigzagBuffer[u],Digits())>0.0) { PeaksOfZigzag[PeaksCount]=NormalizeDouble(ZigzagBuffer[u],Digits()); PeaksCount++; } } //----------------------------------------------------------- for(int V=0;V<PeaksCount-1;V++) { PipsSum+=NormalizeDouble((MathAbs(PeaksOfZigzag[V]-PeaksOfZigzag[V+1]))/Point(),Digits())-Spred; } Print(Depth," ",PeaksCount," ",PipsSum); //----------------------------------------------------------- } } //————————————————————————————————————————————————————————————————————————
Запустив скрипт, получили 4077 пунктов при ExtDepth=3. На 100 барах “уместилось” 19 вершин индикатора. С увеличением ExtDepth количество вершин ZZ уменьшается, уменьшается и прибыльность.
Теперь найдем вершины альтернативного ZZ с помощью UGA. У вершин ZZ может быть три положения на каждом баре: 1) High, 2) Low, 3) Отсутствие вершины. Признак наличия и положения вершины и будет нести каждый ген для каждого бара. Таким образом, размер хромосомы – 100 генов.
По моим расчетам (а при необходимости математики меня поправят) на 100 барах можно построить 3^100, или 5.15378e47 вариантов альтернативных “зигзагов”. Именно столько вариантов нужно рассмотреть, используя прямой перебор. При вычислениях со скоростью 100000000 вариантов в секунду, понадобится 1.6e32 года! Это больше, чем возраст Вселенной. Тут у меня возникли сомнения в возможности решения этой задачки.
Но все же, приступим, пожалуй.
Так как UGA использует представление хромосомы вещественными числами, нам нужно как-то закодировать положения вершин. Это как раз тот случай, когда генотип хромосомы не соответствует фенотипу. Зададим интервал поиска для генов[0;5]. Условимся считать интервал [0;1] соответствием вершине ZZ на High, интервал [4;5] соответствием вершине на Low, и интервал (1;4) соответствием отсутствия вершины.
Необходимо остановится на одном важном моменте. Так как протопопуляция генерируется с генами случайно в заданном интервале, первые особи будут с очень плохими результатами, может быть даже с несколькими сотнями пунктами со знаком минус. Через несколько поколений появится (хотя есть вероятность появления сразу в первом поколении) особь, у которой будут гены, соответствующие отсутствию вершин вообще. Это означало бы отсутствие торговли и уплаты неизбежного спреда.
Как говорят некоторые бывшие трейдеры: “Лучшая стратегия торговли – не торговать”. Такая особь будет вершиной искусственной эволюции. Что бы заставить эту “настольную” эволюцию рождать торгующих особей, т.е. заставить расставлять вершины альтернативного ZZ, назначим приспособленность особи с отсутствием вершин значение “-10000000.0”, заведомо поставить её на самую низшую ступеньку эволюции по сравнению с любыми другими особями.
Вот код скрипта, использующего UGA для нахождения вершин альтернативного ZZ:
#property script_show_inputs //+——————————————————————————————————————————————————————————————————————+ #include "UGAlib.mqh" //+——————————————————————————————————————————————————————————————————————+ //———————————————————————————————————————————————————————————————————————— //----------------------Входные переменные-------------------------------- input string GenofundParam = "----Параметры генофонда----"; input int ChromosomeCount_P = 100; //Кол-во хромосом в колонии input int GeneCount_P = 100; //Кол-во генов input int FFNormalizeDigits_P = 0; //Кол-во знаков приспособлености input int GeneNormalizeDigits_P= 0; //Кол-во знаков гена input int Epoch_P = 50; //Кол-во эпох без улучшения //--- input string GA_OperatorParam = "----Параметры операторов----"; input double ReplicationPortion_P = 100.0; //Доля Репликации. input double NMutationPortion_P = 10.0; //Доля Естественной мутации. input double ArtificialMutation_P = 10.0; //Доля Искусственной мутации. input double GenoMergingPortion_P = 20.0; //Доля Заимствования генов. input double CrossingOverPortion_P = 20.0; //Доля Кроссинговера. input double ReplicationOffset_P = 0.5; //Коэффициент смещения границ интервала input double NMutationProbability_P= 5.0; //Вероятность мутации каждого гена в % //--- input string OptimisationParam = "----Параметры оптимизации----"; input double RangeMinimum_P = 0.0; //Минимум диапазона поиска input double RangeMaximum_P = 5.0; //Максимум диапазона поиска input double Precision_P = 1.0; //Требуемая точность input int OptimizeMethod_P = 2; //Оптим.:1-Min,другое-Max input string Other = "----Прочее----"; input double Spred = 80.0; input bool Show = true; //———————————————————————————————————————————————————————————————————————— //———————————————————————————————————————————————————————————————————————— //----------------------Глобальные переменные----------------------------- double Hight []; double Low []; datetime Time []; datetime Ti []; double Peaks []; bool show; //———————————————————————————————————————————————————————————————————————— //--------------------------Тело программы-------------------------------- int OnStart() { //-----------------------Переменные------------------------------------- //Подготовка глобальных переменных для UGA ChromosomeCount=ChromosomeCount_P; //Кол-во хромосом в колонии GeneCount =GeneCount_P; //Кол-во генов RangeMinimum =RangeMinimum_P; //Минимум диапазона поиска RangeMaximum =RangeMaximum_P; //Максимум диапазона поиска Precision =Precision_P; //Шаг поиска OptimizeMethod =OptimizeMethod_P; //1-минимум, любое другое-максимум FFNormalizeDigits = FFNormalizeDigits_P; //Кол-во знаков приспособлености GeneNormalizeDigits = GeneNormalizeDigits_P;//Кол-во знаков гена ArrayResize(Chromosome,GeneCount+1); ArrayInitialize(Chromosome,0); Epoch=Epoch_P; //Кол-во эпох без улучшения //---------------------------------------------------------------------- //Подготовка глобальных переменных ArraySetAsSeries(Hight,true); CopyHigh (NULL,0,0,GeneCount+1,Hight); ArraySetAsSeries(Low,true); CopyLow (NULL,0,0,GeneCount+1,Low); ArraySetAsSeries(Time,true); CopyTime (NULL,0,0,GeneCount+1,Time); ArrayResize (Ti,GeneCount+1);ArrayInitialize(Ti,0); ArrayResize(Peaks,GeneCount+1);ArrayInitialize(Peaks,0.0); show=Show; //---------------------------------------------------------------------- //Локальные переменные int time_start=GetTickCount(),time_end=0; //---------------------------------------------------------------------- //Очистим экран ObjectsDeleteAll(0,-1,-1); ChartRedraw(0); //Запуск главной ф-ии UGA UGA ( ReplicationPortion_P, //Доля Репликации. NMutationPortion_P, //Доля Естественной мутации. ArtificialMutation_P, //Доля Искусственной мутации. GenoMergingPortion_P, //Доля Заимствования генов. CrossingOverPortion_P,//Доля Кроссинговера. //--- ReplicationOffset_P, //Коэффициент смещения границ интервала NMutationProbability_P//Вероятность мутации каждого гена в % ); //---------------------------------- //Выведем последний результат на экран show=true; ServiceFunction(); //---------------------------------- time_end=GetTickCount(); //---------------------------------- Print(time_end-time_start," мс - Время исполнения"); //---------------------------------- return(0); } //———————————————————————————————————————————————————————————————————————— //———————————————————————————————————————————————————————————————————————— //-----------------------------------------------------------------------+ // Сервисная функция. Вызывается из UGA. | | //Если в ней нет обходимости, оставить функцию пустой так: | // void ServiceFunction() | // { | // } | //-----------------------------------------------------------------------+ void ServiceFunction() { if(show==true) { //-----------------------Переменные----------------------------------- double PipsSum=0.0; int PeaksCount=0; double temp=0.0; //-------------------------------------------------------------------- for(int u=1;u<=GeneCount;u++) { temp=Chromosome[u]; if(temp<=1.0 ) { Peaks[PeaksCount]=NormalizeDouble(Hight[u],Digits()); Ti [PeaksCount]=Time[u]; PeaksCount++; } if(temp>=4.0) { Peaks[PeaksCount]=NormalizeDouble(Low[u],Digits()); Ti [PeaksCount]=Time[u]; PeaksCount++; } } ObjectsDeleteAll(0,-1,-1); for(int V=0;V<PeaksCount-1;V++) { PipsSum+=NormalizeDouble((MathAbs(Peaks[V]-Peaks[V+1]))/Point(),FFNormalizeDigits)-Spred; ObjectCreate (0,"BoxBackName"+(string)V,OBJ_TREND,0,Ti[V],Peaks[V],Ti[V+1],Peaks[V+1]); ObjectSetInteger(0,"BoxBackName"+(string)V,OBJPROP_COLOR,Black); ObjectSetInteger(0,"BoxBackName"+(string)V,OBJPROP_SELECTABLE,true); } ChartRedraw(0); Comment(PipsSum); } //---------------------------------------------------------------------- else return; } //———————————————————————————————————————————————————————————————————————— //———————————————————————————————————————————————————————————————————————— //-----------------------------------------------------------------------+ // Функция оценки приспособленности особи. Вызывается из UGA. | //-----------------------------------------------------------------------+ void FitnessFunction(int chromos) { //-----------------------Переменные------------------------------------- double PipsSum=0.0; int PeaksCount=0; double temp=0.0; //---------------------------------------------------------------------- for(int u=1;u<=GeneCount;u++) { temp=Colony[u][chromos]; if(temp<=1.0) { Peaks[PeaksCount]=NormalizeDouble(Hight[u],Digits()); PeaksCount++; } if(temp>=4.0) { Peaks[PeaksCount]=NormalizeDouble(Low[u],Digits()); PeaksCount++; } } if(PeaksCount>1) { for(int V=0;V<PeaksCount-1;V++) PipsSum+=NormalizeDouble((MathAbs(Peaks[V]-Peaks[V+1]))/Point(),FFNormalizeDigits)-Spred; Colony[0][chromos]=PipsSum; } else Colony[0][chromos]=-10000000.0; AmountStartsFF++; } //————————————————————————————————————————————————————————————————————————
Запустив скрипт, получаем вершины, с суммарным профитом в 4939 пунктов. Причем, понадобилось всего лишь 17929 раз посчитать пункты против 3^100 прямого перебора, на моем компьютере 21,7 секунды против 1.6e32 года!
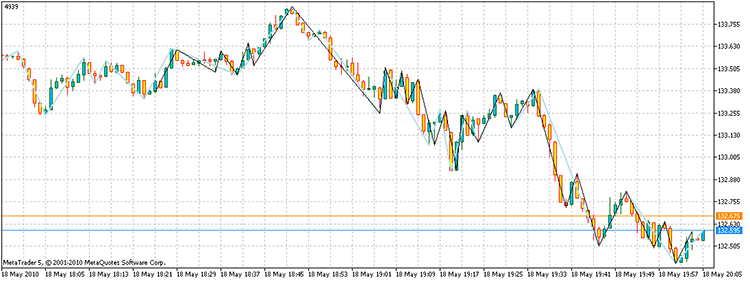
Рисунок 14. Результат решения задачи. Отрезки черного цвета – альтернативный ZZ, небесного цвета – индикатор ZZ
Итак, ответ на вопрос задачи будет звучать следующим образом: “Существуют”.
5. Рекомендации по работе с UGA
- Старайтесь корректно задавать оценочные условия в FF, чтобы ожидать адекватный результат работы алгоритма. Вспомните про Пример2. Это, пожалуй, главный совет.
- Не используйте слишком малое значение параметра Precision. Хотя алгоритм и способен работать с шагом 0, всё же, следует требовать разумную точность решения задачи. Именно для снижения размерности задач и задуман этот параметр.
- Варьируйте размер популяции и пороговое значение количества эпох. Хорошим решением будет назначать параметр Epoch в два раза большим, чем показал MaxOfCurrentEpoch. Не стоит выбирать слишком большие значения, это не ускорит решение задачи.
- Экспериментируйте с параметрами генетических операторов. Универсальных параметров нет, и нужно задавать их исходя из условий поставленной задачи.
Выводы
Наряду с очень мощным штатным тестером стратегий терминала, язык MQL5 позволяет создавать не менее мощные инструменты для работы трейдера, позволяющие решать поистине грандиозные поставленные задачи. Получен очень гибкий и масштабируемый алгоритм оптимизации. И я, без стеснения, горжусь своим “велосипедом”, открытым заново.
Поскольку UGA изначально разрабатывался так, что бы иметь возможность легкой модификации и расширения за счет дополнительных операторов и расчетных блоков, читатель с легкостью сможет сделать свой вклад в развитие “настольной” эволюции.
Желаю читателю успехов в поиске оптимальных решений. Надеюсь я, хоть немного, помог ему в этом. Удачи!
Примечание
В статье использовался индикатор ZigZag. Все исходные коды UGA прилагаются.
Лицензии: Исходные коды, прилагаемые к статье (код UGA), распространяются на условиях BSD.