Redes Neurais de Maneira Fácil (Parte 9): Documentação do trabalho
Dmitriy Gizlyk | 24 março, 2021
Conteúdo
- Introdução
- 1. Os princípios básicos da criação de uma documentação
- 2. Seleção das ferramentas
- 3. Documentando o código
- 4. Preparação do arquivo de código-fonte
- 5. Gerando a documentação
- Conclusões
- Referências
- Programas utilizados no artigo
Introdução
Durante os 8 artigos, nós adicionamos novos objetos e expandimos a funcionalidade dos já existentes. Todas essas adições expandiram a nossa biblioteca. Nós também adicionamos um arquivo de programa OpenCL. Agora o código está 10 vezes maior que o primeiro. Está se tornando difícil de entender os relacionamentos entre os objetos no código. Os leitores podem achar o código muito confuso e difícil de entender. Eu tento fornecer uma descrição detalhada da lógica de ação em cada artigo. Mas a demonstração das cadeias de ação separadas não fornece uma compreensão geral do programa.
Por isso eu decidi demonstrar a criação da documentação para o código, o que permitiria olhar o código de outra perspectiva. O objetivo da documentação é generalizar todos os objetos e métodos na biblioteca e construir uma hierarquia de herança de objetos e métodos. Isso deve nos dar uma ideia geral do que nós já fizemos.
1. Os princípios básicos da criação de uma documentação
Qual é a finalidade da documentação técnica nos desenvolvimentos de TI? Em primeiro lugar, a documentação dá uma ideia geral da arquitetura e operação do programa. A documentação adequada permite que as equipes de desenvolvimento distingam corretamente as áreas de responsabilidade, acompanhem todas as alterações no código e avaliem sua influência em todo o algoritmo e na integridade da arquitetura. Ele também facilita o compartilhamento de conhecimento. Compreender a integridade da arquitetura do programa torna possível analisar e elaborar formas de desenvolvimento do projeto.
A documentação técnica escrita adequadamente deve levar em consideração as qualificações de seu usuário-alvo. As informações devem ser claras e evitar explicações excessivas. A documentação deve conter todas as informações de que o usuário precisa. Ao mesmo tempo, deve ser conciso e fácil de ler. O conteúdo excessivo leva mais tempo para ser lido e irrita o leitor. É ainda mais irritante se o usuário lê uma documentação extensa e não consegue encontrar as informações necessárias. Isso leva à próxima regra: a documentação deve ter ferramentas convenientes para a busca de informações. Uma interface amigável e referências cruzadas facilitam a localização das informações de que você precisa.
A documentação deve conter a arquitetura completa da solução e uma descrição das soluções técnicas implementadas. A descrição completa e detalhada da solução facilita o desenvolvimento e suporte posterior. E é muito importante manter a documentação sempre atualizada. Informações desatualizadas podem levar a decisões de gestão contraditórias e, como resultado, podem desequilibrar todo o desenvolvimento.
Além disso, a documentação deve necessariamente descrever as interfaces entre os componentes.
2. Seleção das ferramentas
Existem alguns programas especializados que podem ajudar na criação da documentação. Eu acho que os mais comuns são o Doxygen, Esfinge, Látex (existem também algumas outras ferramentas). Todos eles visam reduzir os custos de mão de obra para a elaboração da documentação. Claro, cada programa foi criado por desenvolvedores para resolver problemas específicos. Por exemplo, o Doxygen é um programa para a criação de documentação de programas escritos em C++ e linguagens de programação semelhantes. Sphinx foi criado para a documentação para Python. Mas isso não significa que eles sejam altamente especializados em linguagens de programação. Ambos os programas funcionam bem com várias linguagens de programação. Os sites relevantes do programa fornecem referências detalhadas sobre como usá-los, para que você possa escolher aquele que melhor se adapta a você.
A documentação para a MQL5 já foi discutida anteriormente, no artigo "Documentação gerada automaticamente para o código MQL5". Este artigo sugeriu o uso do Doxygen. Eu também uso este programa para meus desenvolvimentos. A sintaxe MQL5 é próxima da C++ e, portanto, o Doxygen é adequado para os programas em MQL5. Eu gosto do fato de que, para criar a documentação, você só precisa adicionar os comentários apropriados ao código do programa, enquanto o software especializado fará o resto. Além disso, o Doxygen permite inserir hiperlinks e fórmulas matemáticas, o que é importante dado o tema dos artigos. Nós vamos considerar as especificações de uso da funcionalidade mais adiante neste artigo, usando exemplos específicos.
3. Documentando o código
Conforme mencionado acima, para gerar a documentação, você precisa adicionar os comentários no código do programa. O Doxygen cria a documentação com base nesses comentários. Naturalmente, nem todos os comentários de código devem ser incluídos na documentação. Alguns dos comentários podem conter notas do desenvolvedor, desse modo, o código não utilizado é comentado. Os desenvolvedores do Doxygen forneceram maneiras de marcar comentários a serem incluídos na documentação. Existem várias opções e você pode escolher a que for mais conveniente para você.
Da mesma forma que o MQL5, os comentários para a documentação podem ser de uma ou de várias linhas. Para não interferir no uso direto do código no futuro, nós usaremos as opções padrão para inserir os comentários e usaremos uma barra adicional para comentários de linha única ou um asterisco para comentários de várias linhas. Opcionalmente, um ponto de exclamação pode ser usado para identificar os blocos de comentários para a documentação.
/// A single-line comment for documentation /** A multi-line block for documentation */ //! An alternative single-line comment for documentation /*! An alternative multi-line block for documentation */
Observe que um bloco de comentário com várias linhas não significa que a mesma apresentação com várias linhas será usada na documentação. Se você precisar separar uma descrição curta e outra detalhada de um objeto do programa, você pode adicionar blocos de comentários diferentes ou usar comandos especiais, que são indicados pelo caractere "\" ou "@". O comando "\n" pode ser usado para a quebra de linha forçada.
Option 1: Separate blocks /// Short description /** Detailed description */ Option 2: Use of special commands /** \brief Brief description \details Detailed description */
Em geral, presume-se que o objeto da documentação esteja localizado no arquivo próximo ao bloco de comentário. Mas, na prática, pode ser necessário comentar sobre o objeto localizado antes do bloco de comentário. Neste caso, use o caractere "<" que informa ao Doxygen que o objeto comentado está localizado antes do bloco. Para criar as referências cruzadas nos comentários, precedemos o objeto de referência com "#". Abaixo está um exemplo de código e de um bloco gerado na documentação. No modelo gerado, a "CConnection" é uma referência que aponta para a página de documentação da classe correspondente.
#define defConnect 0x7781 ///<Connection \details Identified class #CConnection
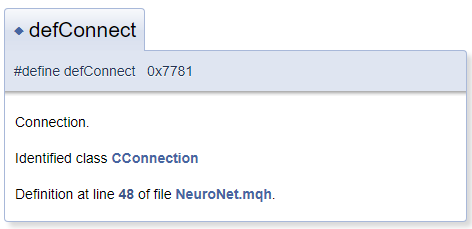
Os recursos do Doxygen são extensos. A lista completa de comandos e suas descrições estão disponíveis na página do programa, sob a seção documentation. Além disso, o Doxygen entende marcações em HTML e XML. Todos esses recursos permitem resolver uma variedade de tarefas ao criar a documentação.
4. Preparação do arquivo de código-fonte
Agora que nós revisamos os recursos da ferramenta, nós podemos começar a trabalhar na documentação. Primeiro, vamos descrever nossos arquivos.
/// \file /// \brief NeuroNet.mqh /// Library for creating Neural network for use in MQL5 experts /// \author [DNG](https://www.mql5.com/en/users/dng) /// \copyright Copyright 2019, DNG
e
/// \file /// \brief NeuroNet.cl /// Library consist OpenCL kernels /// \author <A HREF="https://www.mql5.com/en/users/dng"> DNG </A> /// \copyright Copyright 2019, DNG
Preste atenção que no primeiro caso o ponteiro \autor é seguido pela marcação fornecida pelo Doxygen e, no segundo caso, a marcação HTML é usada. Isso é usado aqui para demonstrar as diferentes opções para a criação de hiperlinks. O resultado é o mesmo nesses casos - ele cria um link para o meu perfil em MQL5.com.
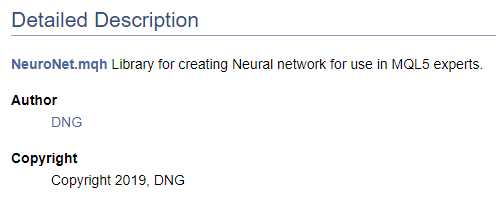
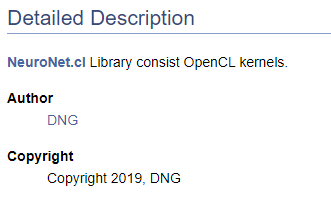
Claro, ao iniciar a criação da documentação do código, é necessário ter pelo menos uma estrutura de alto nível do resultado desejado. A compreensão da estrutura final permite um agrupamento correto dos objetos da documentação. Vamos combinar as enumerações criadas em um grupo separado. Para declarar um grupo, usamos o comando "\defgroup". Os limites do grupo são indicados pelos caracteres "@ {" e "@}".
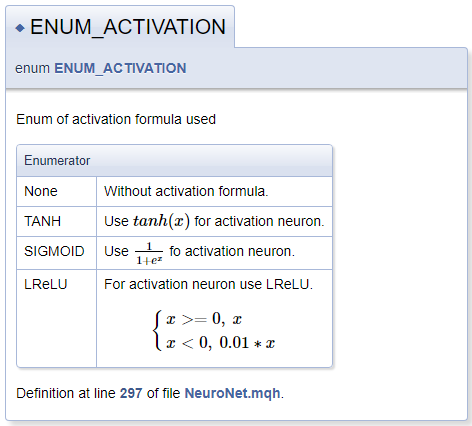
///\defgroup enums ENUM ///@{ //+------------------------------------------------------------------+ /// Enum of activation formula used //+------------------------------------------------------------------+ enum ENUM_ACTIVATION { None=-1, ///< Without activation formula TANH, ///< Use \f$tanh(x)\f$ for activation neuron SIGMOID, ///< Use \f$\frac{1}{1+e^x}\f$ fo activation neuron LReLU ///< For activation neuron use LReLU \f[\left\{ \begin{array} a x>=0, \ x \\x<0, \ 0.01*x \end{array} \right.\f] }; //+------------------------------------------------------------------+ /// Enum of optimization method used //+------------------------------------------------------------------+ enum ENUM_OPTIMIZATION { SGD, ///< Stochastic gradient descent ADAM ///< Adam }; ///@}
Ao descrever as funções de ativação, eu demonstrei a funcionalidade para declarar as fórmulas matemáticas por meio da MathJax. As descrições de tais fórmulas devem ser colocadas entre um par de comandos "\f$", se você desejar exibir a fórmula em uma linha de texto ou entre comandos "\f[" e "\f]" se você quiser que a fórmula apareça em uma linha separada. O comando "\frac" permite descrever uma fração. O comando é seguido pelo numerador e denominador da fração entre chaves.
Ao descrever a LReLU, nós precisamos de uma chave esquerda unificadora. Para criá-la, nós usamos os comandos "\left\{" e "\right\.". O comando "\right" é seguido por "\.", porque a chave direita não é necessária na fórmula. Caso contrário, o ponto final seria substituído por uma chave de fechamento. Um array de strings é declarado dentro do bloco usando os comandos "\begin{array} a" e "\end{array}", a separação dos elementos do array é realizada pelo comando "\\". Os caracteres "\ " permitem adicionar um espaço forçado.
O bloco da documentação gerado é mostrado abaixo.
Na próxima etapa, vamos criar um grupo separado para os identificadores de classe na biblioteca. Dentro do grupo, nós vamos alocar os subgrupos de matrizes, neurônios calculando as operações na CPU e os neurônios calculando as operações na GPU. Um link para a classe apropriada é adicionado conforme explicado anteriormente.
///\defgroup ObjectTypes Defines Object types identified ///Used to identify classes in a library ///@{ //+------------------------------------------------------------------+ ///\defgroup arr Arrays ///Used to identify array classes ///\{ #define defArrayConnects 0x7782 ///<Array of connections \details Identified class #CArrayCon #define defLayer 0x7787 ///<Layer of neurons \details Identified class #CLayer #define defArrayLayer 0x7788 ///<Array of layers \details Identified class #CArrayLayer #define defNet 0x7790 ///<Neuron Net \details Identified class #CNet ///\} ///\defgroup cpu CPU ///Used to identify classes with CPU calculation ///\{ #define defConnect 0x7781 ///<Connection \details Identified class #CConnection #define defNeuronBase 0x7783 ///<Neuron base type \details Identified class #CNeuronBase #define defNeuron 0x7784 ///<Full connected neuron \details Identified class #CNeuron #define defNeuronConv 0x7785 ///<Convolution neuron \details Identified class #CNeuronConv #define defNeuronProof 0x7786 ///<Proof neuron \details Identified class #CNeuronProof #define defNeuronLSTM 0x7791 ///<LSTM Neuron \details Identified class #CNeuronLSTM ///\} ///\defgroup gpu GPU ///Used to identify classes with GPU calculation ///\{ #define defBufferDouble 0x7882 ///<Data Buffer OpenCL \details Identified class #CBufferDouble #define defNeuronBaseOCL 0x7883 ///<Neuron Base OpenCL \details Identified class #CNeuronBaseOCL #define defNeuronConvOCL 0x7885 ///<Convolution neuron OpenCL \details Identified class #CNeuronConvOCL #define defNeuronProofOCL 0x7886 ///<Proof neuron OpenCL \details Identified class #CNeuronProofOCL #define defNeuronAttentionOCL 0x7887 ///<Attention neuron OpenCL \details Identified class #CNeuronAttentionOCL ///\} ///@}
A divisão em grupos na documentação gerada é a seguinte.
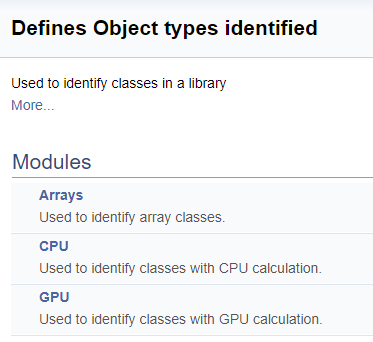
A seguir, nós iremos trabalhar em um grande grupo de definições para trabalhar com os kernels em OpenCL. Neste bloco, nomes mnemônicos são atribuídos aos índices do kernel e seus parâmetros, que são usados ao chamar os kernels do programa principal. Usando a tecnologia acima, nós iremos dividir este grupo pela classe de neurônios a partir da qual o kernel é chamado e, em seguida, pelo conteúdo das operações no kernel (feed-forward, retropropagação do gradiente, atualização dos coeficientes de peso). Eu não irei fornecer o código completo aqui - ele está disponível no anexo abaixo. A lógica para construir os subgrupos é semelhante ao exemplo acima. A imagem abaixo mostra a estrutura completa do grupo.
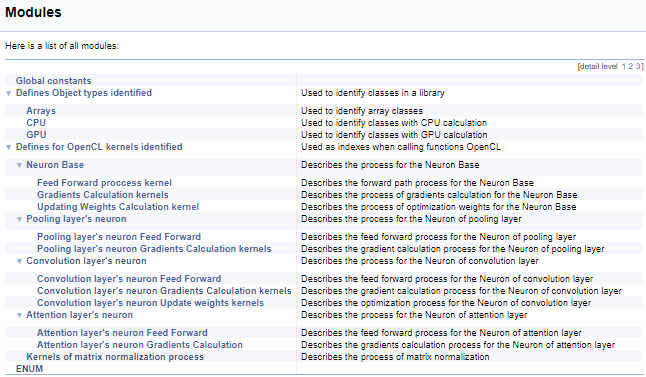
Continuando com os kernels, vamos passar a comentar sobre o programa em OpenCL. Para criar uma estrutura de documentação coerente e para obter uma imagem geral, nós usaremos outro comando do Doxygen "\ingroup", que permite adicionar novos objetos de documentação aos grupos criados anteriormente. Vamos usá-lo para adicionar os kernels aos grupos de índices criados anteriormente para trabalhar com os kernels. Na descrição do kernel, adicionamos um link para a chamada da classe e para um artigo neste site com uma descrição do processo. A seguir, vamos descrever os parâmetros do kernel. O uso dos ponteiros "[in]" e "[out]" mostrará a direção do fluxo de informações. As referências cruzadas mostrarão o formato dos dados.
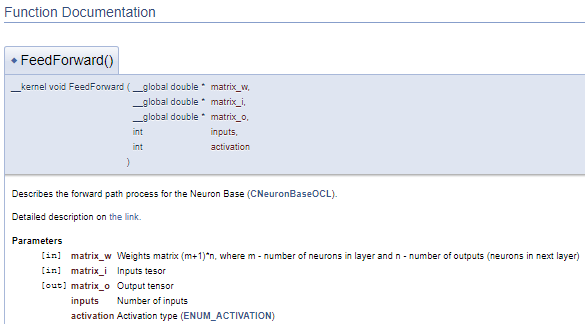
///\ingroup neuron_base_ff Feed forward process kernel /// Describes the forward path process for the Neuron Base (#CNeuronBaseOCL). ///\details Detailed description on <A HREF="https://www.mql5.com/en/articles/8435#para41">the link.</A> //+------------------------------------------------------------------+ __kernel void FeedForward(__global double *matrix_w,///<[in] Weights matrix (m+1)*n, where m - number of neurons in layer and n - number of outputs (neurons in next layer) __global double *matrix_i,///<[in] Inputs tesor __global double *matrix_o,///<[out] Output tensor int inputs,///< Number of inputs int activation///< Activation type (#ENUM_ACTIVATION) )
O código acima irá gerar o seguinte bloco de documentação.
No exemplo acima, a descrição dos parâmetros é fornecida imediatamente após a sua declaração. Mas essa abordagem pode tornar o código desajeitado. Nesses casos, sugere-se o uso do "\param" para descrever os parâmetros. Usando este comando, nós podemos descrever os parâmetros em qualquer parte do arquivo, mas nós precisamos especificar diretamente o nome do parâmetro.
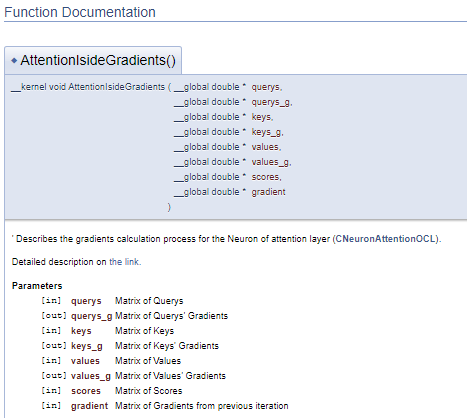
///\ingroup neuron_atten_gr Attention layer's neuron Gradients Calculation kernel /// Describes the gradients calculation process for the Neuron of attention layer (#CNeuronAttentionOCL). ///\details Detailed description on <A HREF="https://www.mql5.com/ru/articles/8765#para44">the link.</A> /// @param[in] querys Matrix of Querys /// @param[out] querys_g Matrix of Querys' Gradients /// @param[in] keys Matrix of Keys /// @param[out] keys_g Matrix of Keys' Gradients /// @param[in] values Matrix of Values /// @param[out] values_g Matrix of Values' Gradients /// @param[in] scores Matrix of Scores /// @param[in] gradient Matrix of Gradients from previous iteration //+------------------------------------------------------------------+ __kernel void AttentionIsideGradients(__global double *querys,__global double *querys_g, __global double *keys,__global double *keys_g, __global double *values,__global double *values_g, __global double *scores, __global double *gradient)
Esta abordagem gera um bloco de documentação semelhante, mas permite separar o bloco de comentários do código do programa. Assim, o código fica mais fácil de ler.
O principal trabalho diz respeito à documentação de nossas classes de biblioteca e seus métodos. Nós precisamos descrever todas as classes usadas e seus métodos. Para fazer isso, nós usaremos todos os comandos descritos acima em diferentes variações e adicionaremos alguns novos. Primeiro, vamos adicionar a classe ao grupo apropriado, como nós fizemos anteriormente com os kernels (o comando \ingroup). O comando "\class" informa ao Doxygen que a descrição abaixo se aplica à classe. Nos parâmetros de comando, especificamos o nome da classe para vincular a descrição ao objeto certo
Usando os comandos "\brief" e "\details", fornecem uma descrição breve e extensa da classe. Na descrição detalhada, adicionamos um hiperlink ao artigo correspondente. Aqui, nós adicionaremos um link âncora a uma seção específica do artigo, o que permitirá aos usuários encontrar as informações necessárias com mais rapidez.
Adicionamos suas descrições diretamente à linha de declaração da variável. Se necessário, adicionamos os links para os objetos explicativos. Não há necessidade de definir ponteiros para as classes de objetos declarados nos comentários, enquanto o Doxygen os adicionará automaticamente.
Da mesma forma, descreva os métodos das classes. No entanto, ao contrário das variáveis, uma descrição dos parâmetros deve ser incluída nos comentários. Para fazer isso, usamos o comando descrito anteriormente "\param" junto com os ponteiros "[in]", "[out]", "[in,out]". Descrevemos o resultado da execução do método usando o comando "\return".
Também é possível anexar métodos individuais a grupos por determinados recursos. Por exemplo, eles podem ser combinados por funcionalidade.
O código abaixo mostra todas as etapas acima.
///\ingroup neuron_base ///\class CNeuronBaseOCL ///\brief The base class of neuron for GPU calculation. ///\details Detailed description on <A HREF="https://www.mql5.com/ru/articles/8435#para45">the link.</A> //+------------------------------------------------------------------+ class CNeuronBaseOCL : public CObject { protected: COpenCLMy *OpenCL; ///< Object for working with OpenCL CBufferDouble *Output; ///< Buffer of Output tenzor CBufferDouble *PrevOutput; ///< Buffer of previous iteration Output tenzor CBufferDouble *Weights; ///< Buffer of weights matrix CBufferDouble *DeltaWeights; ///< Buffer of last delta weights matrix (#SGD) CBufferDouble *Gradient; ///< Buffer of gradient tenzor CBufferDouble *FirstMomentum; ///< Buffer of first momentum matrix (#ADAM) CBufferDouble *SecondMomentum; ///< Buffer of second momentum matrix (#ADAM) //--- const double alpha; ///< Multiplier to momentum in #SGD optimization int t; ///< Count of iterations //--- int m_myIndex; ///< Index of neuron in layer ENUM_ACTIVATION activation; ///< Activation type (#ENUM_ACTIVATION) ENUM_OPTIMIZATION optimization; ///< Optimization method (#ENUM_OPTIMIZATION) //--- ///\ingroup neuron_base_ff virtual bool feedForward(CNeuronBaseOCL *NeuronOCL); ///< \brief Feed Forward method of calling kernel ::FeedForward().@param NeuronOCL Pointer to previos layer. ///\ingroup neuron_base_opt virtual bool updateInputWeights(CNeuronBaseOCL *NeuronOCL); ///< Method for updating weights.\details Calling one of kernels ::UpdateWeightsMomentum() or ::UpdateWeightsAdam() in depends of optimization type (#ENUM_OPTIMIZATION).@param NeuronOCL Pointer to previos layer. public: /** Constructor */CNeuronBaseOCL(void); /** Destructor */~CNeuronBaseOCL(void); virtual bool Init(uint numOutputs, uint myIndex, COpenCLMy *open_cl, uint numNeurons, ENUM_OPTIMIZATION optimization_type); ///< Method of initialization class.@param[in] numOutputs Number of connections to next layer.@param[in] myIndex Index of neuron in layer.@param[in] open_cl Pointer to #COpenCLMy object. #param[in] numNeurons Number of neurons in layer @param optimization_type Optimization type (#ENUM_OPTIMIZATION)@return Boolen result of operations. virtual void SetActivationFunction(ENUM_ACTIVATION value) { activation=value; } ///< Set the type of activation function (#ENUM_ACTIVATION) //--- virtual int getOutputIndex(void) { return Output.GetIndex(); } ///< Get index of output buffer @return Index virtual int getPrevOutIndex(void) { return PrevOutput.GetIndex(); } ///< Get index of previous iteration output buffer @return Index virtual int getGradientIndex(void) { return Gradient.GetIndex(); } ///< Get index of gradient buffer @return Index virtual int getWeightsIndex(void) { return Weights.GetIndex(); } ///< Get index of weights matrix buffer @return Index virtual int getDeltaWeightsIndex(void) { return DeltaWeights.GetIndex(); } ///< Get index of delta weights matrix buffer (SGD)@return Index virtual int getFirstMomentumIndex(void) { return FirstMomentum.GetIndex(); } ///< Get index of first momentum matrix buffer (Adam)@return Index virtual int getSecondMomentumIndex(void) { return SecondMomentum.GetIndex();} ///< Get index of Second momentum matrix buffer (Adam)@return Index //--- virtual int getOutputVal(double &values[]) { return Output.GetData(values); } ///< Get values of output buffer @param[out] values Array of data @return number of items virtual int getOutputVal(CArrayDouble *values) { return Output.GetData(values); } ///< Get values of output buffer @param[out] values Array of data @return number of items virtual int getPrevVal(double &values[]) { return PrevOutput.GetData(values); } ///< Get values of previous iteration output buffer @param[out] values Array of data @return number of items virtual int getGradient(double &values[]) { return Gradient.GetData(values); } ///< Get values of gradient buffer @param[out] values Array of data @return number of items virtual int getWeights(double &values[]) { return Weights.GetData(values); } ///< Get values of weights matrix buffer @param[out] values Array of data @return number of items virtual int Neurons(void) { return Output.Total(); } ///< Get number of neurons in layer @return Number of neurons virtual int Activation(void) { return (int)activation; } ///< Get type of activation function @return Type (#ENUM_ACTIVATION) virtual int getConnections(void) { return (CheckPointer(Weights)!=POINTER_INVALID ? Weights.Total()/(Gradient.Total()) : 0); } ///< Get number of connections 1 neuron to next layer @return Number of connections //--- virtual bool FeedForward(CObject *SourceObject); ///< Dispatch method for defining the subroutine for feed forward process. @param SourceObject Pointer to the previous layer. virtual bool calcHiddenGradients(CObject *TargetObject); ///< Dispatch method for defining the subroutine for transferring the gradient to the previous layer. @param TargetObject Pointer to the next layer. virtual bool UpdateInputWeights(CObject *SourceObject); ///< Dispatch method for defining the subroutine for updating weights.@param SourceObject Pointer to previos layer. ///\ingroup neuron_base_gr ///@{ virtual bool calcHiddenGradients(CNeuronBaseOCL *NeuronOCL); ///< Method to transfer gradient to previous layer by calling kernel ::CalcHiddenGradient(). @param NeuronOCL Pointer to next layer. virtual bool calcOutputGradients(CArrayDouble *Target); ///< Method of output gradients calculation by calling kernel ::CalcOutputGradient().@param Target target value ///@} //--- virtual bool Save(int const file_handle);///< Save method @param[in] file_handle handle of file @return logical result of operation virtual bool Load(int const file_handle);///< Load method @param[in] file_handle handle of file @return logical result of operation //--- virtual int Type(void) const { return defNeuronBaseOCL; }///< Identifier of class.@return Type of class };
Para terminar de trabalhar com o código, vamos criar uma página de rosto. O comando "\mainpage" é usado para identificar o bloco da página de rosto. O comando deve ser seguido pelo título da página de rosto. Abaixo, vamos adicionar a descrição do projeto e criar uma lista de referências. Os itens da lista serão marcados pelo caractere "-". Para criar links para grupos criados anteriormente, usamos o comando "\ref". Quando o Doxygen gera a documentação, são geradas as páginas da hierarquia de classes (hierarchy.html) e dos arquivos usados (files.html). Adicionamos os links para as páginas especificadas à lista. O código final da página de rosto é mostrado abaixo.
///\mainpage NeuronNet /// Library for creating Neural network for use in MQL5 experts. /// - \ref const /// - \ref enums /// - \ref ObjectTypes /// - \ref group1 /// - [<b>Class Hierarchy</b>](hierarchy.html) /// - [<b>Files</b>](files.html)
A página a seguir será gerada com base no código acima.
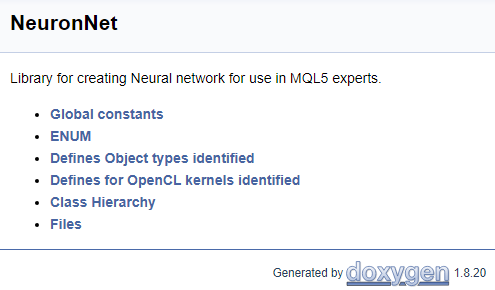
O código completo de todos os comentários é fornecido em anexo.
5. Gerando a documentação
Depois de concluir o trabalho com o código, prosseguimos para a próxima etapa. A instalação e configuração do Doxygen são descritas em detalhes no artigo [9]. Vamos considerar a configuração de alguns parâmetros do programa. Primeiro, informe ao Doxygen com quais arquivos ele deve trabalhar: na guia Expert, no tópico Input, adicione as máscaras de arquivo necessárias ao parâmetro FILE_PATTERNS. Neste caso, eu adicionei "*.mqh" e "*.cl".
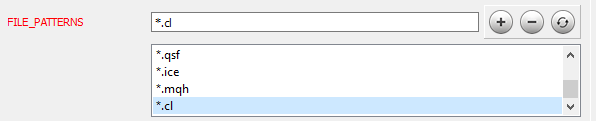
Agora nós precisamos informar ao Doxygen como analisar os arquivos adicionados. Vamos para o tópico Project na mesma guia Expert e editamos o parâmetro EXTENSION_MAPPING conforme mostrado na figura abaixo.
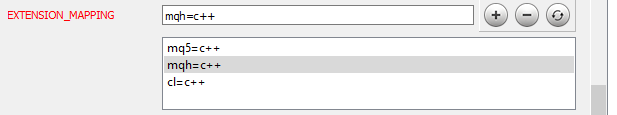
Para permitir que o Doxygen gere fórmulas matemáticas, ativamos o uso de MathJax. Para fazer isso, ativamos o parâmetro USE_MATHJAX no tópico HTML da guia Expert, conforme mostrado na figura abaixo.
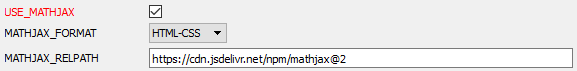
Após configurar o programa, acessamos a aba Wizard e especificamos o nome do projeto, o caminho para os arquivos fonte e o caminho para a exibição da documentação gerada (todas essas etapas são exibidas no artigo [9]). Vá para a guia Run e execute o programa de geração de documentação.
Assim que o programa for concluído, você receberá uma documentação pronta para uso. Algumas imagens são mostradas abaixo. A documentação completa é fornecida em anexo.
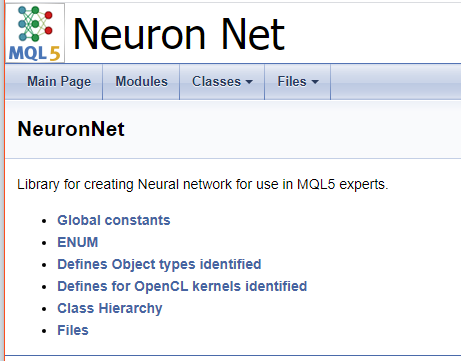
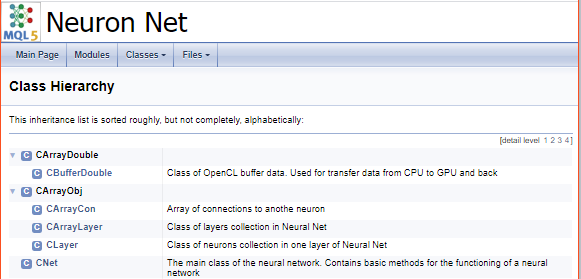
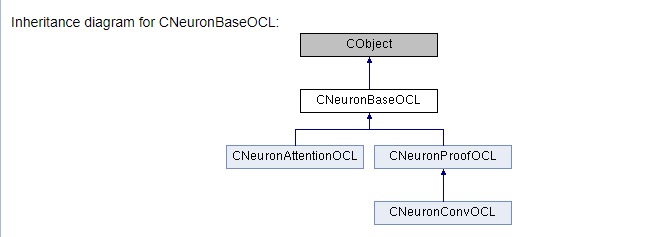
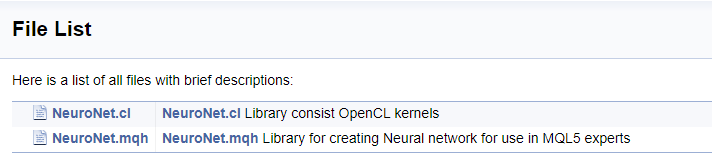
Conclusões
A documentação dos programas desenvolvidos não é a principal tarefa do programador. No entanto, essa documentação é essencial no desenvolvimento de projetos complexos. Ele ajuda no acompanhamento da execução de tarefas, na coordenação do trabalho de uma equipe de desenvolvimento e fornece uma visão holística do desenvolvimento. A documentação é uma obrigação ao compartilhar conhecimento.
O artigo descreve um mecanismo para documentar os desenvolvimentos na linguagem MQL5. Ele fornece uma descrição detalhada de todas as etapas do mecanismo. Os resultados do trabalho realizado estão disponíveis em anexo, para que todos possam avaliá-los.
Espero que minha experiência seja útil.
Referências
- Redes neurais de maneira fácil
- Redes neurais de maneira fácil (Parte 2): Treinamento e teste da rede
- Redes Neurais de Maneira Fácil (Parte 3): Redes Convolucionais
- Redes Neurais de Maneira Fácil (Parte 4): Redes Recorrentes
- Redes Neurais de Maneira Fácil (Parte 5): Cálculos em Paralelo com o OpenCL
- Redes neurais de Maneira Fácil (Parte 6): Experimentos com a taxa de aprendizado da rede neural
- Redes Neurais de Maneira Fácil(Parte 7): Métodos de otimização adaptativos
- Redes Neurais de Maneira Fácil (Parte 8): Mecanismos de Atenção
- Documentação gerada automaticamente para o código MQL5
- Doxygen
Programas utilizados no artigo
| # | Nome | Tipo | Descrição |
|---|---|---|---|
| 1 | NeuroNet.mqh | Biblioteca de classe | Uma biblioteca de classes para a criação de uma rede neural |
| 2 | NeuroNet.cl | Código Base | Biblioteca do código do programa OpenCL |
| 3 | html.zip | Arquivo zip | Arquivo de documentação gerado pelo Doxygen |
| 4 | NN.chm | Ajuda HTML | O arquivo de ajuda HTML convertido. |
| 5 | Doxyfile | Arquivo de parâmetros do Doxygen |