Redes Neurais Profundas (Parte VII). Ensemble de redes neurais: stacking
Vladimir Perervenko | 25 setembro, 2018
Conteúdo
- Introdução
- 1. Preparação dos dados de entrada para o combinador treinável
- 2. Modelos base de comparação
- 3 Bibliotecas Keras/TensorFlow. Descrição geral e instalação
- 4 Combinador de saídas do bagging de ensemble — rede neural
- 5. Análise de resultados experimentais
- Conclusão
- Anexos
Introdução
Os modelos de nível base do conjunto (classificadores individuais) são treinados em um conjunto completo. Em seguida, o metamodelo é treinado nas saídas obtidas do ensemble durante a previsão com base no conjunto de testes. Nesse caso, as saídas dos classificadores de base do conjunto se tornam os dados de entrada para o novo classificador treinado, que em si é um combinador. Esta abordagem é chamada de "combinação complexa" ou "generalização através da aprendizagem", sendo mais frequente o uso da palavra "stacking".
Um dos principais problemas deste combinador é a construção de um conjunto de treinamento para o metaclassificador.
Vamos experimentar com a construção e teste do stacking de ensembles. Eles usarão um conjunto de classificadores de redes neurais ELM com os hiperparâmetros ótimos obtidos anteriormente. As saídas do ensemble podado serão usadas no primeiro experimento e todas as entradas do conjunto no segundo experimento. Como combinadores, ambas as variantes terão redes neurais totalmente conectadas, mas com estruturas diferentes. Em experimentos futuros, nós verificaremos como a multimodalidade e a multitarefa afetam a qualidade da classificação da rede neural.
Os modelos base de comparação serão usados para estimar a qualidade de previsão nessas variantes.
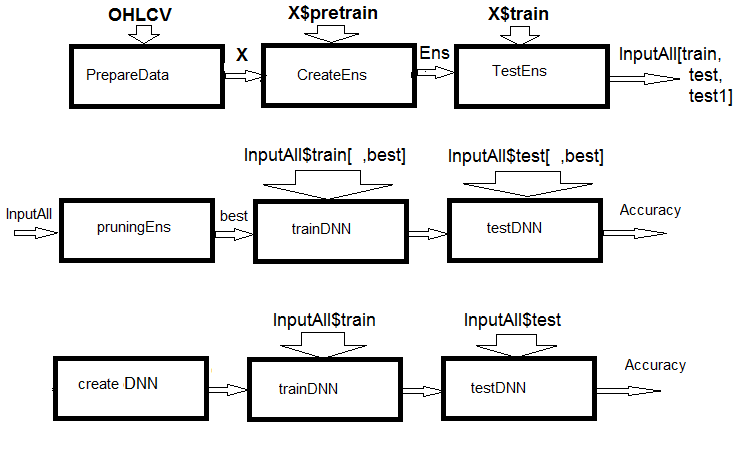
Fig.1. Esquema estrutural dos cálculos
Como você pode ver na figura, o experimento consiste de três partes.
- Preparação dos dados de entrada para o conjunto, treinamento do conjunto ELM e a obtenção de previsões nos conjuntos train/test/test1. Esses conjuntos servirão como entradas InputAll para os combinadores treináveis.
- Poda do ensemble: escolhemos as melhores previsões da ELM por importância da informação. Testamos os módulos base de comparação para obtenção das métricas de referência. Treinamos e testamos o DNN nesses dados, calculamos as métricas dos modelos e comparamos eles com as métricas do modelo base.
- Criaremos as redes neurais multimodais e multitarefas, treinaremos e testaremos eles nos conjuntos InputAll. Calcularemos as métricas dos modelos obtidos e faremos a comparação com as métricas do modelo base.
1. Preparação dos dados de entrada para o combinador treinável
Para os experimentos, o R versão 3.4.4 será usado. Ele contém vários novos pacotes que ainda nós não usamos.
Iniciamos o RStudio. Faça o download do arquivo Cotir.RData contendo as cotações do terminal localizado no link GitHub/Part_I e os arquivos Importar.R, Libary.R, FunPrepareData_VII.R, FUN_Stacking.R com as funções de preparação dos dados localizados emGitHub/Part_VII. Atenção: a sequência do download dos arquivos é importante! Eu modifiquei um pouco as funções para acelerar os cálculos e melhorar a legibilidade dos scripts. Além disso, foi adicionado vários indicadores necessários para os experimentos.
Nós usaremos as mesmas cotações e as dividiremos nas mesmas amostras dos artigos anteriores desta série. Vamos escrever um script para preparar os dados iniciais. Os detalhes do cálculo não serão considerados novamente — eles foram descritos anteriormente. As alterações feitas estão relacionadas ao uso do pacote dplyr e a importação dos pacotes e funções para o ambiente de trabalho. dplyr é um pacote muito útil que facilita a manipulação dos dados. Às vezes, ele apresenta surpresas durante a depuração, o que exige muitas horas de busca por erros.
Deixe-me esclarecer isso. Ao carregar a biblioteca dplyr, os seguintes avisos aparecem no console:
> library(dplyr) Attaching package: ‘dplyr’ The following objects are masked from ‘package:stats’: filter, lag The following objects are masked from ‘package:base’: intersect, setdiff, setequal, union
Um conflito de nomes de funções é evidente. Para resolver o problema, foi necessário especificar explicitamente o pacote de uma função específica. Por exemplo, dplyr::filter(), dplyr::lag. O segundo problema: muitas vezes, apenas uma ou duas funções do pacote são necessárias, mas é necessário carregar a biblioteca inteira. E certos pacotes (por exemplo, caret) são massivos e são acompanhados por pacotes dependentes que nós não precisamos. Nesse sentido, o estilo para importar as funções e pacotes em Python é mais lógico. Por exemplo:
from theano import function, config, shared, tensor import numpy as np import time
Na primeira linha, uma série de funções do pacote theano são importados; na segunda linha, o pacote numpy é importado e apelidado de np; e no terceiro — o pacote time. O mesmo recurso no R é implementado no pacote importar, que tem apenas duas funções — import() e import_fun(). O primeiro permite importar os pacotes e o segundo é para importar funções. No entanto, o primeiro teve que ser renomeado para import_pack(), para que não entre em conflito com reticulate::import().
Além disso, novas variáveis foram introduzidas, o que será necessário para os experimentos. Gere dois conjuntos de dados — data1 e data2. Organize seus preditores por importância da informação.
Abaixo está um script para preparar os dados iniciais para os experimentos. Ele está disponível no arquivo Prepare.R.
#--0--Library------------- # source(file = "importar.R") # source(file = "Library.R") # source(file = "FunPrepareData_VII.R") # source(file = "FUN_Stacking.R") #--1-prepare---- evalq({ # combina as cotações OHLCV, Med, Typ, W no quadro de dados # calcula os preditores e o alvo dt <- PrepareData(Data, Open, High, Low, Close, Volume) # divide os dados iniciais em pretrain/train/val/test DT <- SplitData(dt$feature, 4000, 1000, 500, 250, start = 1) # define os parâmetros de outliers pre.outl <- PreOutlier(DT$pretrain) # importa os outliers em todos os conjuntos DTcap <- CappingData(DT, impute = T, fill = T, dither = F, pre.outl = pre.outl) # define o método para normalizar os preditores meth <- "spatialSign" #"expoTrans" "range" "spatialSign", # define os parâmetros de normalização preproc <- PreNorm(DTcap$pretrain, meth = meth, rang = c(-0.95, 0.95)) # normaliza os preditores em todos os conjuntos DTcap.n <- NormData(DTcap, preproc = preproc) }, env)
No bloco 0 (Library), é carregado as bibliotecas e funções necessárias. Quatro arquivos com os scripts devem ser carregados na sequência especificada. No bloco 1 (prepare), são criados e normalizado os preditores, removendo os outliers. O método de normalização pode ser alterado.
Agora, forma-se dois conjuntos de dados — data1 e data2. No primeiro conjunto, os filtros digitais e suas diferenças de primeira ordem serão usadas como preditores, e o sinal de mudança na diferença de primeira ordem do ZigZag serve como alvo. No segundo conjunto, os preditores serão as diferenças da primeira ordem das cotações High/Low/Close e as diferenças das cotações CO/HO/LO/HL, enquanto que a diferença de primeira ordem do ZigZag será o alvo. O script é exibido abaixo e está disponível no arquivo Prepare.R.
#--2-Data X------------- evalq({ foreach(i = 1:length(DTcap)) %do% { DTcap.n[[i]] ->.; dp$select(., Data, ftlm, stlm, rbci, pcci, fars, v.fatl, v.satl, v.rftl, v.rstl,v.ftlm, v.stlm, v.rbci, v.pcci, Class)} -> data1 X1 <- vector(mode = "list", 4) foreach(i = 1:length(X1)) %do% { data1[[i]] %>% dp$select(-c(Data, Class)) %>% as.data.frame() -> x data1[[i]]$Class %>% as.numeric() %>% subtract(1) -> y list(x = x, y = y)} -> X1 list(pretrain = X1[[1]] , train = X1[[2]] , test = X1[[3]] , test1 = X1[[4]] ) -> X1 }, env) #----------------- evalq({ foreach(i = 1:length(DTcap.n)) %do% { DTcap.n[[i]] ->.; dp$select(., Data, CO, HO, LO, HL, dC, dH, dL)} -> data2 X2 <- vector(mode = "list", 4) foreach(i = 1:length(X2)) %do% { data2[[i]] %>% dp$select(-Data) %>% as.data.frame() -> x DT[[i]]$dz -> y list(x = x, y = y)} -> X2 list(pretrain = X2[[1]] , train = X2[[2]] , test = X2[[3]] , test1 = X2[[4]] ) -> X2 }, env)
Os preditores são ordenados em ambos os conjuntos em ordem decrescente de importância da informação. Vamos ver como eles são classificados. O script é exibido abaixo, ele está disponível no arquivo Prepare.R.
#---3--bestF----------------------------------- #require(clusterSim) evalq({ orderF(x = X1$pretrain$x %>% as.matrix(), type = "metric", s = 1, 4, distance = NULL, # "d1" - Manhattan, "d2" - Euclidean, #"d3" - Chebychev (max), "d4" - squared Euclidean, #"d5" - GDM1, "d6" - Canberra, "d7" - Bray-Curtis method = "kmeans" ,#"kmeans" (default) , "single", #"ward.D", "ward.D2", "complete", "average", "mcquitty", #"median", "centroid", "pam" Index = "cRAND") %$% stopri[ ,1] -> orderX1 }, env) colnames(env$X1$pretrain$x)[env$orderX1] [1] "v.fatl" "v.rbci" "v.ftlm" "fars" "v.satl" "stlm" [7] "rbci" "ftlm" "v.stlm" "v.rftl" "pcci" "v.rstl" [13] "v.pcci evalq({ orderF(x = X2$pretrain$x %>% as.matrix(), type = "metric", s = 1, 4, distance = NULL, # "d1" - Manhattan, "d2" - Euclidean, #"d3" - Chebychev (max), "d4" - squared Euclidean, #"d5" - GDM1, "d6" - Canberra, "d7" - Bray-Curtis method = "kmeans" ,#"kmeans" (default) , "single", #"ward.D", "ward.D2", "complete", "average", "mcquitty", #"median", "centroid", "pam" Index = "cRAND") %$% stopri[ ,1] -> orderX2 }, env) colnames(env$X2$pretrain$x)[env$orderX2] [1] "dC" "CO" "HO" "LO" "dH" "dL" "HL"
A ordem dos preditores da cotação é de particular interesse.
Para preparar os dados de entrada para os combinadores, é necessário:
- criar um conjunto ELM e treiná-lo no conjunto de treinamento X1$pretrain;
- fazer uma previsão do conjunto X1$train usando o ensemble treinado. Este será o conjunto de treinamento InputTrain.
- fazer uma previsão do conjunto X1$test usando o ensemble treinado. Este será o conjunto de testes InputTest;
- fazer uma previsão do conjunto X1$test1 usando o ensemble treinado. Este será o conjunto de testes InputTest1.
Foi definido as variáveis e constantes, escrito as funções createEns() e GetInputData() — elas retornarão o valor de todas as saídas do ensemble. Os valores dos parâmetros da função createEns() já foram obtidos após a otimização do conjunto. Você pode ter outros valores. O script fornecido abaixo pode ser encontrado no arquivo FUN_Stacking().
#----Library------------- import_fun(rminer, holdout, holdout) #source(file = "FunPrepareData_VII.R") #----Input------------- evalq({ #type of activation function. Fact <- c("sig", #: sigmoid "sin", #: sine "radbas", #: radial basis "hardlim", #: hard-limit "hardlims", #: symmetric hard-limit "satlins", #: satlins "tansig", #: tan-sigmoid "tribas", #: triangular basis "poslin", #: positive linear "purelin") #: linear n <- 500 #---createENS---------------------- createEns <- function(numFeature = 8L, r = 7L, nh = 5L, fact = 7L, order, X){ # determina os índices dos melhores preditores bestF <<- order %>% head(numFeature) # escolhe os melhores preditores para o conjunto de treinamento Xtrain <- X$pretrain$x[ , bestF] #setMKLthreads(1) k <- 1 rng <- RNGseq(n, 12345) #---creste Ensemble--- Ens <<- foreach(i = 1:n, .packages = "elmNN") %do% { rngtools::setRNG(rng[[k]]) idx <- rminer::holdout(Ytrain, ratio = r/10, mode = "random")$tr k <- k + 1 elmtrain(x = Xtrain[idx, ], y = Ytrain[idx], nhid = nh, actfun = Fact[fact]) } return(Ens) } #---GetInputData -FUN----------- GetInputData <- function(Ens, X){ #---predict-InputTrain-- Xtest <- X$train$x[ , bestF] foreach(i = 1:n, .packages = "elmNN", .combine = "cbind") %do% { predict(Ens[[i]], newdata = Xtest) } -> predEns #[ ,n] #---predict--InputTest---- Xtest1 <- X$test$x[ , bestF] foreach(i = 1:n, .packages = "elmNN", .combine = "cbind") %do% { predict(Ens[[i]], newdata = Xtest1) } -> InputTest #[ ,n] #---predict--InputTest1---- Xtest2 <- X$test1$x[ , bestF] foreach(i = 1:n, .packages = "elmNN", .combine = "cbind") %do% { predict(Ens[[i]], newdata = Xtest2) } -> InputTest1 #[ ,n] #---res------------------------- return(list(InputTrain = predEns, InputTest = InputTest, InputTest1 = InputTest1)) } }, env)
Foi criado um ensemble e calculado as entradas para os combinadores treináveis:
#---4--createEns---------------- evalq({ Ytrain <- X1$pretrain$y Ytest <- X1$train$y Ytest1 <- X1$test$y Ytest2 <- X1$test1$y Ens <- vector(mode = "list", n) createEns(order = orderX1, X = X1) -> Ens GetInputData(Ens, X1) -> res }, env)
Estrutura do resultado:
> env$res %>% str() List of 3 $ InputTrain: num [1:1001, 1:500] 0.811 0.882 0.924 0.817 0.782 ... $ InputTest : num [1:501, 1:500] 0.5 0.383 0.366 0.488 0.359 ... $ InputTest1: num [1:251, 1:500] 0.32 0.246 0.471 0.563 0.451 ...
2. Modelos base de comparação
Dois combinadores treináveis serão criados. Um substituirá a média das saídas das melhores redes neurais do conjunto e a segunda substituirá a poda e a média. Portanto, os escores de qualidade da classificação serão necessários para as duas opções.
Ensemble de redes neurais
Para a primeira opção, o ensemble ELM com os parâmetros ótimos será o modelo base de comparação.
Como a segunda opção implica em 500 entradas, o pacote varbvs será usado. Ele fornece algoritmos rápidos para a seleção dos modelos Bayesianos para escolher as variáveis e calcular os coeficientes Bayesianos, onde o resultado é modelado usando regressão linear ou logística. Esses algoritmos são baseados nas aproximações variacionais descritas no artigo "Scalable Variational Inference for Bayesian Variable Selection in Regression, and Its Accuracy in Genetic Association Studies". Este software foi usado para trabalhar com grandes conjuntos de dados contendo mais de um milhão de variáveis e milhares de amostras.
Para a primeira opção, foi escrito as funções adicionais getBest(), testAver(), testVot() e calculado as métricas. As funções estão disponíveis no arquivo FUN_Stacking.R.
evalq({
getBest <- function(Ens, x, y, nb){
n <- length(Ens)
foreach(i = 1:n, .packages = "elmNN", .combine = "cbind") %do% {
predict(Ens[[i]], newdata = x)} -> y.pr
foreach(i = 1:n, .combine = "c") %do% {
median(y.pr[ ,i])} ->> th
foreach(i = 1:n, .combine = "c") %do% {
ifelse(y.pr[ ,i] > th[i], 1, 0) -> Ypred
Evaluate(actual = y, predicted = Ypred)$Metrics$F1 %>%
mean()
} -> Score
Score %>% order(decreasing = TRUE) %>% head(2*nb + 1) -> best
y.pr[ ,best] %>%
apply(1, sum) %>%
divide_by(length(best)) %>%
median() -> med
return(list(Score = Score, bestNN = best, med = med))
}
testAver <- function(Ens, x, y, best, med){
n <- length(Ens)
foreach(i = 1:n, .packages = "elmNN", .combine = "cbind") %:%
when(i %in% best) %do% {
predict(Ens[[i]], newdata = x)} %>%
apply(1, sum) %>% divide_by(length(best)) -> ensPred
ifelse(ensPred > med, 1, 0) -> clAver
Evaluate(actual = y, predicted = clAver)$Metrics[ ,2:5] %>%
round(3) -> Score
return(list(Score = Score, Ypred = ensPred, clAver = clAver))
}
testVot <- function(Ens, x, y, best){
n <- length(Ens)
foreach(i = 1:n, .packages = "elmNN", .combine = "cbind") %:%
when(i %in% best) %do% {
predict(Ens[[i]], newdata = x)} %>%
apply(2, function(x) ifelse(x > th[i], 1, -1)) %>%
apply(1, function(x) sum(x)) -> vot
ifelse(vot > 0, 1, 0) -> ClVot
Evaluate(actual = y, predicted = ClVot)$Metrics[ ,2:5] %>%
round(3) -> Score
return(list(Score = Score, Ypred = ClVot))
}
}, env)
Estas funções já foram analisadas. Portanto, nós não deteremos em sua descrição. No entanto, seus resultados merecem atenção.
- A função getBest() retorna as métricas (Score), os índices dos melhores classificadores individuais do conjunto (bestNN), a mediana da saída média do ensemble (med), que será usado ao testar o modelo. O vetor mediana th[500] de todas as saídas do ensemble é inserido no ambiente.
- A função testAver() retorna as métricas (Score), a previsão contínua média do ensemble (Ypred) e a previsão nominal do ensemble (clAver).
- A função testVot() retorna as métricas (Score) e a previsão nominal do conjunto (Ypred).
Foi testado o ensemble criado em dois conjuntos de testes usando a média e a votação por maioria e, em seguida, analisado as métricas.
#--2---test---- evalq({ Ytrain <- X1$pretrain$y Ytest <- X1$train$y Ytest1 <- X1$test$y Ytest2 <- X1$test1$y Ens <- vector(mode = "list", n) Ens <- createEns(order = orderX1, X = X1) #---3------ resBest <- getBest(Ens, x = X1$train$x[ , bestF], y = Ytest, nb = 3) #---4--averaging--- ScoreAver <- testAver(Ens, x = X1$test$x[ , bestF], y = Ytest1, best = resBest$bestNN, med = resBest$med) ScoreAver1 <- testAver(Ens, x = X1$test1$x[ , bestF], y = Ytest2, best = resBest$bestNN, med = resBest$med) #---5--voting---- ScoreVot <- testVot(Ens, x = X1$test$x[ , bestF], y = Ytest1, best = resBest$bestNN) ScoreVot1 <- testVot(Ens, x = X1$test1$x[ , bestF], y = Ytest2, best = resBest$bestNN) }, env) > env$ScoreAver$Score Accuracy Precision Recall F1 0 0.75 0.708 0.778 0.741 1 0.75 0.794 0.727 0.759 > env$ScoreAver1$Score Accuracy Precision Recall F1 0 0.753 0.750 0.826 0.786 1 0.753 0.758 0.664 0.708 > env$ScoreVot$Score Accuracy Precision Recall F1 0 0.752 0.702 0.800 0.748 1 0.752 0.808 0.712 0.757 > env$ScoreVot1$Score Accuracy Precision Recall F1 0 0.741 0.739 0.819 0.777 1 0.741 0.745 0.646 0.692
Há um bom desempenho nos dois conjuntos de testes. Abaixo, na análise dos resultados, o erro de classificação é decomposto em bias/variance/noise, e é estimado a contribuição de cada componente para o erro total.
Para a segunda opção (500 entradas), o script para treinamento do modelo é fornecido abaixo. Ele pode ser encontrado no arquivo varb.R.
library(varbvs) evalq({ vr <- varbvs(X = res$InputTrain, Z = NULL, y = Ytest, family = "binomial", optimize.eta = TRUE, logodds = seq(-6,-2, 0.25), nr = 250, initialize.params = TRUE, maxiter = 1e5, verbose = FALSE) summary(vr, cred.int = 0.95, nv = 7, nr = 1e5) %>% print() }, env) Summary of fitted Bayesian variable selection model: family: binomial num. hyperparameter settings: 17 samples: 1001 iid variable selection prior: yes variables: 500 fit prior var. of coefs (sa): yes covariates: 1 fit approx. factors (eta): yes maximum log-likelihood lower bound: -579.4602 Hyperparameters: estimate Pr>0.95 candidate values sa 8.09 [7.63,8.54] NA--NA logodds -2.26 [-2.75,-2.00] (-6.00)--(-2.00) Selected variables by probability cutoff: >0.10 >0.25 >0.50 >0.75 >0.90 >0.95 3 3 3 3 3 3 Top 7 variables by inclusion probability: index variable prob PVE coef* Pr(coef.>0.95) X18 18 X18 1.00000 NA 4.529 [+3.861,+5.195] X5 5 X5 1.00000 NA 1.955 [+1.543,+2.370] X255 255 X255 1.00000 NA 2.097 [+1.537,+2.660] X109 109 X109 0.00948 NA -1.033 [-2.008,-0.057] X404 404 X404 0.00467 NA -0.665 [-1.350,+0.024] X275 275 X275 0.00312 NA -0.726 [-1.735,+0.286] X343 343 X343 0.00299 NA -0.604 [-1.353,+0.149] *See help(varbvs) about interpreting coefficients in logistic regression.
Usando o modelo obtido, é calculado a previsão em dois conjuntos de testes e suas métricas.
env$vr$pip %>% order() %>% tail(7) -> bestNN_vr evalq({ predict(vr, res$InputTest) -> pr.vr1 Evaluate(actual = Ytest1, predicted = pr.vr1)$Metrics[ ,2:5] %>% round(3) -> metr.test confus(table(Ytest1, pr.vr1)) -> cm1 predict(vr, res$InputTest1) -> pr.vr2 Evaluate(actual = Ytest2, predicted = pr.vr2)$Metrics[ ,2:5] %>% round(3) -> metr.test1 confus(table(Ytest2, pr.vr2)) -> cm2 }, env) > env$metr.test Accuracy Precision Recall F1 0 0.78 0.750 0.783 0.766 1 0.78 0.808 0.779 0.793 > env$metr.test1 Accuracy Precision Recall F1 0 0.729 0.765 0.732 0.748 1 0.729 0.689 0.726 0.707
O resultado é excelente, muito melhor que as métricas do ensemble.
Todos os dados necessários para continuar os experimentos estão prontos.
Já que as bibliotecas keras/tensorflow devem ser usadas no futuro, elas são brevemente consideradas abaixo.
3 Bibliotecas Keras/TensorFlow. Descrição geral e instalação
O campo, em rápida expansão, das redes neurais profundas foi complementado com um número de bibliotecas de código aberto. Esses incluem — TensorFlow (Google), CNTK (Microsoft), Apache MXNet e muitos outros. Devido ao fato de que todos esses e outros grandes desenvolvedores de software são membros do R Consortium, todas essas bibliotecas são fornecidas com APIs para R.
Todas as bibliotecas acima são de baixo nível. Elas são difíceis de aprender e usar para quem é iniciante. Com isso em mente, a equipe do Rstudio desenvolveu o pacote keras para o R.
Keras é uma API de rede neural de alto nível. O pacote é projetado com ênfase na capacidade de criar rapidamente protótipos e experimentalmente testar o desempenho de um modelo. Aqui estão as principais características do Keras:
- Permite trabalhar igualmente em uma CPU ou GPU.
- API amigável, que permite criar protótipos de modelos de aprendizagem profunda com facilidade.
- Suporte embutido para redes convolucionais (para visão computacional), redes recorrentes (para processamento de sequências) e qualquer combinação delas.
- Suporta arquiteturas de rede arbitrárias: modelos com múltiplas entradas ou múltiplas saídas, compartilhamento de camadas, compartilhamento de modelos, etc. Isso significa que o Keras é essencialmente adequado para a construção de qualquer modelo de aprendizagem profunda, desde uma rede de memória até uma máquina de Turing Neural.
- Ele é capaz de trabalhar em vários backends, incluindo TensorFlow, CNTK ou Theano.
Keras é uma API projetada para humanos, não para máquinas. O pacote reduz a carga cognitiva: oferece APIs consistentes e simples, minimiza o número de ações do usuário e fornece feedback efetivo sobre erros do usuário. Tudo isso faz o Keras ser fácil de aprender e de usar. Mas isso não é causado por uma diminuição na flexibilidade: já que o Keras faz a integração com linguagens de baixo nível de aprendizagem profunda (em particular o TensorFlow), ele permite implementar tudo o que você pode criar na linguagem base.
Você pode desenvolver um modelo Keras usando vários módulos de aprendizagem profunda. Qualquer modelo Keras que usa apenas camadas incorporadas pode ser transferido entre todos esses backends sem alterações: você pode treinar um modelo com um backend e carregá-lo em outro backend. Os backends disponíveis incluem:
- TensorFlow backend (do Google)
- CNTK backend (da Microsoft)
- Theano backend
Você pode treinar um modelo Keras em várias plataformas de hardware diferentes, não apenas a CPU:
- GPUs NVIDIA
- TPUs da Google, através do backend TensorFlow e Google Cloud
- GPUs compatíveis com OpenCL, como as da AMD, através do backend do PlaidML Keras
Instalação do backend Keras e TensorFlow
O Keras e TensorFlow podem ser configurados para trabalhar em uma CPU ou GPU. A versão da CPU é muito mais fácil de instalar e configurar, portanto, é a melhor escolha para começar a usar o pacote. Aqui estão os manuais para as versões de CPU e GPU do site do TensorFlow:
- TensorFlow apenas com suporte a CPU. Se o seu sistema não tiver uma GPU NVIDIA®, você deverá instalar esta versão.
- TensorFlow com suporte a GPU. Os programas TensorFlow geralmente são executados significativamente mais rápido em uma GPU do que em uma CPU. Portanto, se o seu sistema tiver uma GPU NVIDIA® em conformidade com todos os pré-requisitos e você precisar executar aplicativos críticos para o desempenho, você deve instalar essa versão.
O único método de instalação suportado no Windows é "conda". Isso significa que você deve instalar o Anaconda 3.x (Python 3.5.x/3.6.x) para o Windows antes de instalar o Keras. Eu instalei a Anaconda3 (Python3.6).
Primeiro, instale o pacote keras do CRAN:
install.packages("keras")
A interface Keras R usa o TensorFlow por padrão. Para instalar ambos a biblioteca principal Keras e o backend TensorFlow, use a função install_keras():
# default installation library(keras) install_keras()
Assim, as versões para CPU do Keras e TensorFlow será instalado. Se você precisar de uma configuração personalizada — por exemplo, com uma GPU NVIDIA, consulte a documentação. Para instalar o TensorFlow de uma versão específica ou com suporte a GPU, faça o seguinte:
# install with GPU version of TensorFlow # (NOTE: only do this if you have an NVIDIA GPU + CUDA!) install_keras(tensorflow = "gpu") # install a specific version of TensorFlow install_keras(tensorflow = "1.5") install_keras(tensorflow = "1.5-gpu")
Para mais detalhes, veja aqui.
O pacote de suporte tfruns é projetado para experimentos com o TensorFlow. Este é um kit de ferramentas para gerenciar o treinamento e experimentos do TensorFlow a partir do R.
- Monitorar os hiperparâmetros, métricas, dados de saída e código-fonte de cada execução de treinamento.
- Comparar os hiperparâmetros e as métricas entre as execuções para encontrar o modelo de melhor desempenho.
- Gerar relatórios automaticamente para visualizar as execuções de treinamento individuais ou comparações entre execuções.
- Não são necessárias alterações no código-fonte (os dados de execução são automaticamente capturados para todos os modelos Keras e tfestimators).
A melhor qualidade de visualização do processo de treinamento e resultados da DNN é fornecida por TensorBoard.
Especialistas em aprendizado profundo têm a oportunidade de trabalhar diretamente com a biblioteca TensorFlow usando o pacote tensorflow.
Todos esses pacotes são baseados no pacote principal reticulate, que é uma interface R para os módulos, funções e classes em Python. Quando chamado no Python, os tipos de dados em R são convertidos automaticamente nos tipos equivalentes em Python. Os valores retornados do Python são convertidos novamente nos tipos em R.
Todos esses pacotes são bem documentados, fornecidos com inúmeros exemplos e em constante evolução. Isto torna possível usar os modelos mais avançados de aprendizagem profunda (DNN, RNN, CNN, LSTM, VAE, etc.), aprendizado por reforço (RL) e muitos outros desenvolvimentos em Python nos experts e indicadores do terminal. A única limitação é o conhecimento e a experiência do desenvolvedor.
Aqui estão mais dois pacotes interessantes que merecem atenção: kerasR e kerasformula. Existem testes para o primeiro, confirmando uma velocidade de operação maior que a do original "tensorflow-1.5". O segundo oferece uma versão simplificada do modelo usando uma fórmula.
Este artigo visa apenas dar exemplos para um simples início em um novo campo. Sua tarefa não é cobrir toda a diversidade de oportunidades e obter pontuações de alta qualidade do modelo.
Antes de iniciar os experimentos, é necessário verificar se o Python está instalado e se o R interage com ele.
> library(reticulate)
> py_config()
python: K:\Anaconda3\envs\r-tensorflow\python.exe
libpython: K:/Anaconda3/envs/r-tensorflow/python36.dll
pythonhome: K:\ANACON~1\envs\R-TENS~1
version: 3.6.5 | packaged by conda-forge | (default, Apr 6 2018, 16:13:55)
[MSC v.1900 64 bit (AMD64)]
Architecture: 64bit
numpy: K:\ANACON~1\envs\R-TENS~1\lib\site-packages\numpy
numpy_version: 1.14.2
tensorflow: K:\ANACON~1\envs\R-TENS~1\lib\site-packages\tensorflow
python versions found:
K:\Anaconda3\envs\r-tensorflow\python.exe
K:\ANACON~1\python.exe
K:\Anaconda3\python.exe
Let us see the version of tensorflow used:
> library(tensorflow) > tf_config() TensorFlow v1.5.1 (K:\ANACON~1\envs\R-TENS~1\lib\site-packages\tensorflow) Python v3.6 (K:\Anaconda3\envs\r-tensorflow\python.exe)
Tudo está pronto para a continuação dos experimentos.
4 Combinador de saídas do bagging de ensemble — rede neural
Vamos realizar dois experimentos. No primeiro, é aplicado a função softmax em vez de calcular a média das melhores saídas do ensemble. No segundo, é substituído a poda e a média por uma rede neural, alimentando todas as 500 saídas do conjunto como entrada. O esquema da estrutura do experimento é exibido na figura abaixo.
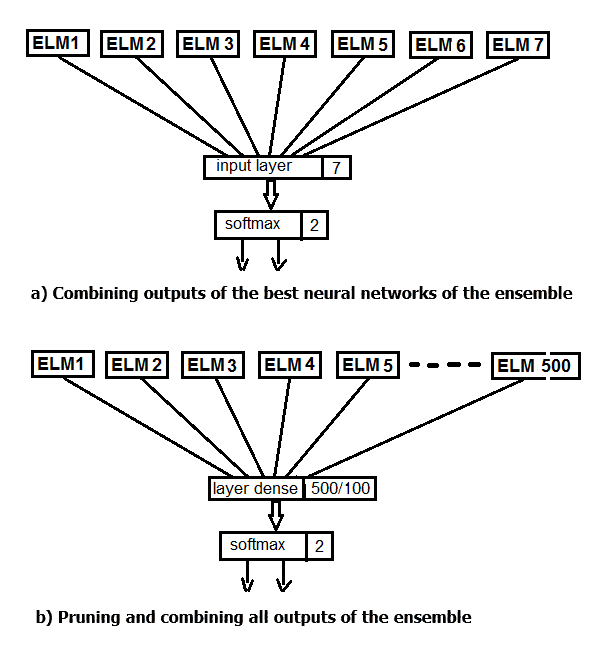
Fig.2. Substituição da média das saídas do ensemble com uma rede neural
A estrutura de dados principal em Keras é um modelo, uma maneira de organizar as camadas. O tipo mais simples é um modelo Sequencial, representando uma pilha linear de camadas.
Primeiro, é criado um modelo sequencial simples e, em seguida, eles começam a adicionar camadas usando o operador pipe (%>%). Para o primeiro experimento, é criado uma rede neural, consistindo apenas da camada de entrada e saída. As saídas do ensemble após a poda, obtidas no conjunto de testes, serão alimentadas como entrada. 20% do conjunto de treinamento será usado para validação. A criação, o treinamento e o teste de redes neurais neste pacote são extremamente fáceis.
É definido os parâmetros constantes do modelo, os conjuntos de treinamento e teste para o DNN.
#===========Keras=========================================== library(keras) num_classes <- 2L batch_size <- 32L epochs <- 300L #--------- bestNN <- env$resBest$bestNN x_train <- env$res$InputTrain[ ,bestNN] y_train <- env$Ytest %>% to_categorical() x_test <- env$res$InputTest[ ,bestNN] y_test <- env$Ytest1 %>% to_categorical() x_test1 <- env$res$InputTest1[ ,bestNN] y_test1 <- env$Ytest2 %>% to_categorical()
O modelo é criado. O código do script é fornecido abaixo. Modelo compilado e definido com uma estrutura NN(7, 2) — 7 neurônios na entrada e 2 na saída. Otimizador — a função "rmsprop", função de perda — 'binary'_crossentropy', métricas para o resultado dos testes — "accuracy".
##----model--keras------------------------- # define model model <- keras_model_sequential() # add layers and compile model %>% layer_dense(units = num_classes, input_shape = dim(x_train)[2]) %>% layer_activation(activation = 'softmax') %>% compile( loss = 'binary_crossentropy', optimizer = optimizer_rmsprop(), metrics = 'accuracy' )
O modelo é treinado e testado (o script é fornecido abaixo). O histórico do treinamento é salvo. Além disso, especifique:
- não é necessário enviar o resultado de cada iteração para o terminal;
- não é necessário mostrar os gráficos em tempo real no Viewer/Rstudio;
- é necessário misturar os dados de entrada após cada época de treinamento.
Para validação, use os 20% do conjunto de treinamento.
## Training & Evaluation --------------------------- # Fit model to data model %>% fit( x_train, y_train, batch_size = batch_size, epochs = epochs, verbose = 0, view_metrics = FALSE, shuffle = TRUE, validation_split = 0.2) -> history # Output metrics score <- model %>% evaluate(x_test, y_test, verbose = 0) cat('Test loss:', score[[1]] %>% round(3), '\n') Test loss: 0.518 cat('Test accuracy:', score[[2]] %>% round(3), '\n') Test accuracy: 0.754 #--------------- score1 <- model %>% evaluate(x_test1, y_test1, verbose = 0) cat('Test loss:', score1[[1]] %>% round(3), '\n') Test loss: 0.55 cat('Test accuracy:', score1[[2]] %>% round(3), '\n') Test accuracy: 0.737
Os resultados quantitativos não são ruins, eles são virtualmente iguais aos resultados da média do conjunto. Vamos ver o histórico de treinamento e teste deste modelo:
#--plot------------------------
plot(history)
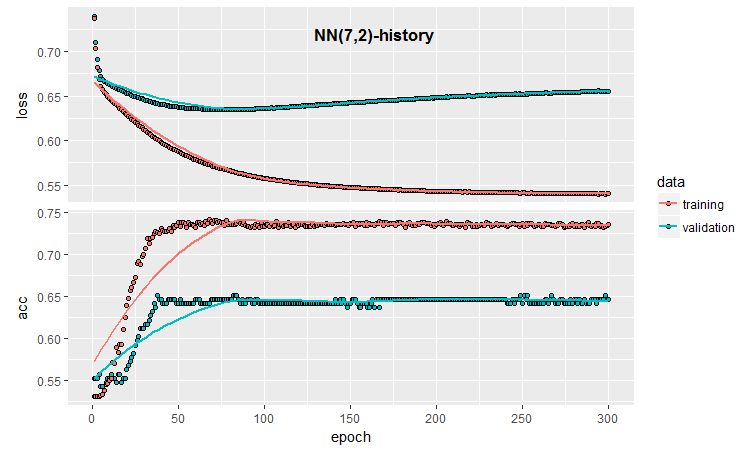
Fig.3. Histórico de treinamento do modelo (7, 2)
O gráfico mostra que após 30 épocas o modelo está claramente sofrendo overfitting (sobre treinado), e depois de 50 épocas a Accuracy (precisão) atinge um platô.
Como você se lembra, o processo de inicialização da rede neural é aleatório. Ou seja, cada modelo recém-criado, treinado e testado produzirá resultados diferentes.
Mesmo nessa configuração mínima de uma rede neural, há muitas oportunidades de influenciar a qualidade da classificação e o overfitting. Aqui estão alguns deles: parada antecipada; método de inicialização de neurônios; regularização da função de ativação, etc. Destes, nós verificaremos somente a parada antecipada, adicionando ruído aos dados de entrada, regularizando a função de ativação e gerando uma representação gráfica mais detalhada dos resultados do treinamento. Para fazer isso, use a capacidade de aplicar um callback durante o treinamento, fornecido pelo keras.
callback_early_stopping(monitor = "val_loss", min_delta = 0, patience = 0, verbose = 0, mode = c("auto", "min", "max")) callback_tensorboard(log_dir = NULL, histogram_freq = 0, batch_size = 32, write_graph = TRUE, write_grads = FALSE, write_images = FALSE, embeddings_freq = 0, embeddings_layer_names = NULL, embeddings_metadata = NULL)
As funções de retorno de chamada são definidas.
early_stopping <- callback_early_stopping(monitor = "val_acc", min_delta = 1e-5, patience = 20, verbose = 0, mode = "auto") log_dir <- paste0(getwd(),"/run_1") tensboard <- callback_tensorboard(log_dir = log_dir, histogram_freq = 1, batch_size = 32, write_graph = TRUE, write_grads = TRUE, write_images = FALSE)
Na primeira, nós indicamos que é necessário rastrear o valor de Accuracy. Se este valor se tornar menor que min_delta em épocas patiente, o treinamento deve ser interrompido. Na segunda, nós definimos o caminho para o diretório onde os resultados do treinamento devem ser armazenados para posterior reprodução e também indicamos onde armazená-los exatamente. Vamos escrever um script completo usando essas funções e ver o resultado.
##=====Variant earlystopping================================= #--prepare data-------------------------- library(reticulate) library(keras) py_set_seed(12345) num_classes <- 2L batch_size <- 32L learning_rate <- 0.005 epochs <- 100L #--------- bestNN <- env$resBest$bestNN x_train <- env$res$InputTrain[ ,bestNN] y_train <- env$Ytest %>% to_categorical() x_test <- env$res$InputTest[ ,bestNN] y_test <- env$Ytest1 %>% to_categorical() x_test1 <- env$res$InputTest1[ ,bestNN] y_test1 <- env$Ytest2 %>% to_categorical() ##----model--keras------------------------- # define model model <- keras_model_sequential() # add layers and compile model %>% layer_gaussian_noise(stddev = 0.05, input_shape = dim(x_train)[2], name = "GN") %>% layer_dense(units = num_classes, name = "dense1") %>% layer_activation_softmax(name = "soft") %>% layer_activity_regularization(l2 = 1.0, name = "reg") %>% #l1 = 0.01, compile( loss = 'binary_crossentropy', optimizer = optimizer_rmsprop(lr = learning_rate, decay = 0.01), metrics = 'accuracy' ) ## Training & Evaluation --------------------------- # Fit model to data model %>% fit( x_train, y_train, batch_size = batch_size, epochs = epochs, verbose = 0, view_metrics = TRUE , shuffle = TRUE, validation_split = 0.2, callbacks = list(early_stopping, tensboard)) -> history
Métricas em dois conjuntos de testes e no histórico de treinamento com a parada antecipada.
# Output metrics > score <- model %>% evaluate(x_test, y_test, verbose = 0) > cat('Test loss:', score[[1]] %>% round(3), '\n') Test loss: 0.539 > cat('Test accuracy:', score[[2]] %>% round(3), '\n') Test accuracy: 0.756 > #--------------- > score1 <- model %>% evaluate(x_test1, y_test1, verbose = 0) > cat('Test loss:', score1[[1]] %>% round(3), '\n') Test loss: 0.571 > cat('Test accuracy:', score1[[2]] %>% round(3), '\n') Test accuracy: 0.713
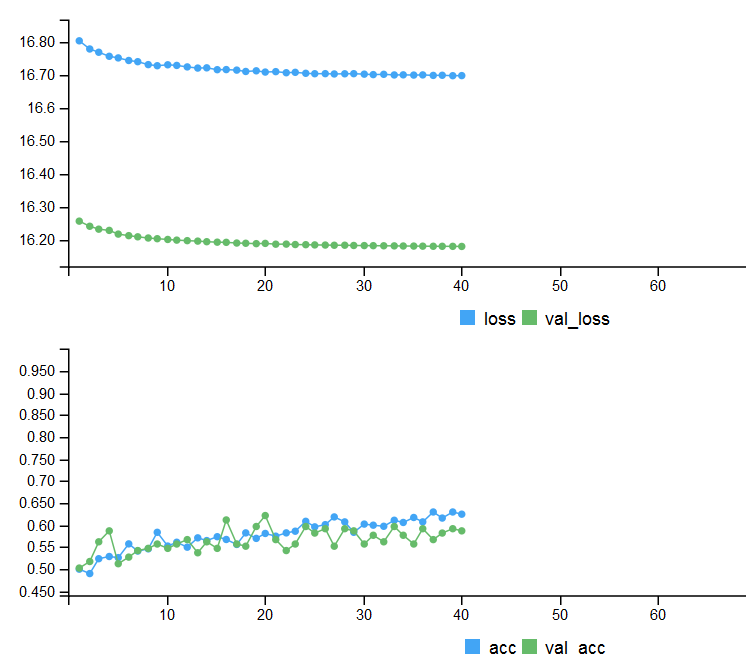
Fig. 4. Histórico de treinamento com a parada antecipada
Para exibir as informações gráficas detalhadas sobre o processo de treinamento de uma rede neural, foi usado os recursos tensorboard:
> tensorboard(log_dir = log_dir) TensorBoard 1.7.0 at http://127.0.0.1:7451 (Press CTRL+C to quit) Started TensorBoard at http://127.0.0.1:7451
O navegador irá abrir uma página onde você pode visualizar todos os detalhes internos da rede neural. Aqui estão algumas capturas de tela:
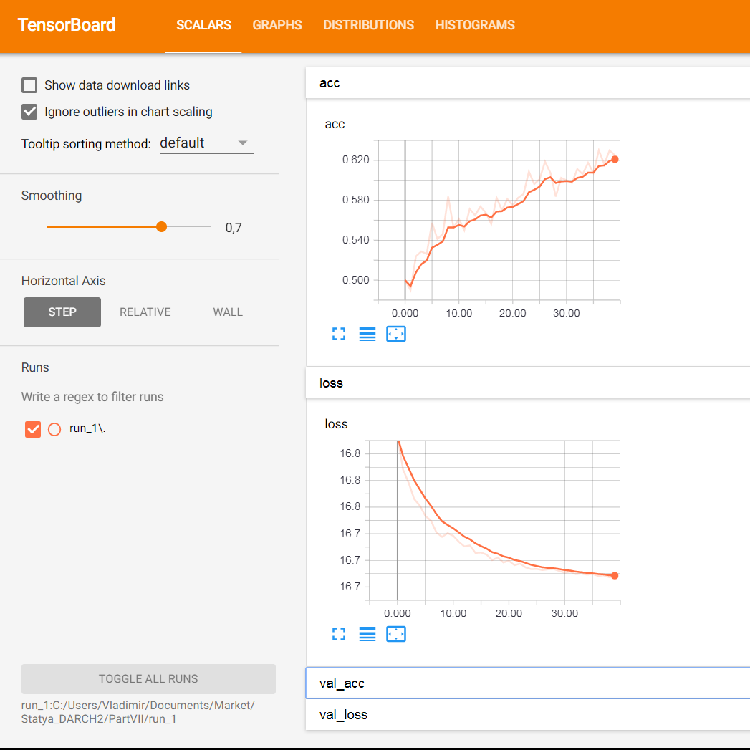
Fig. 5. Gráficos de perda e precisão para o treinamento do conjunto de treinamento, dados de validação baseados em val_acc e val_loss
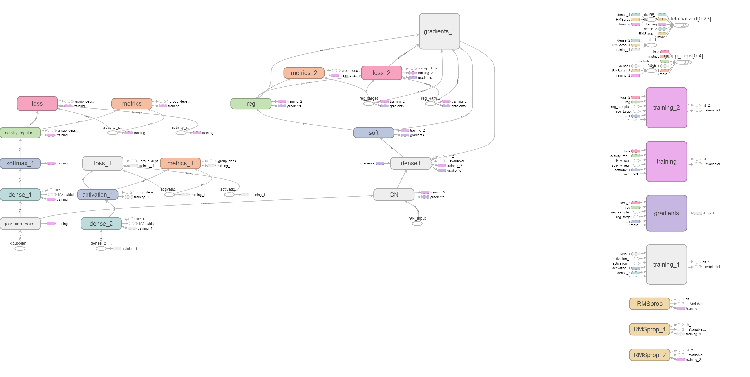
Fig. 6. Grafo computacional da rede neural
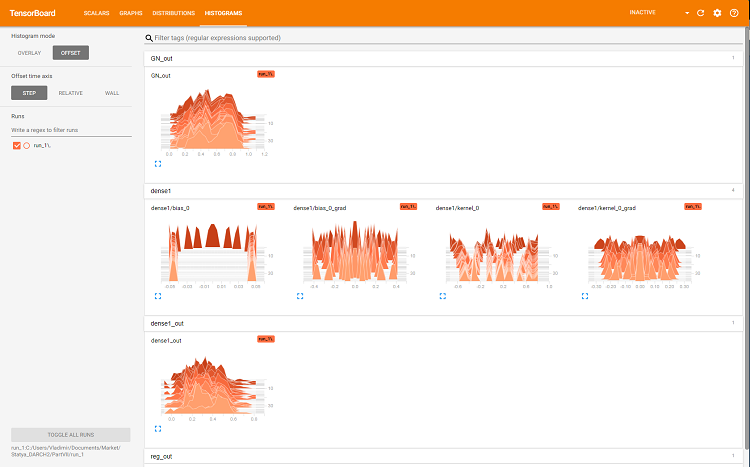
Fig. 7. Histogramas da camada "dense"
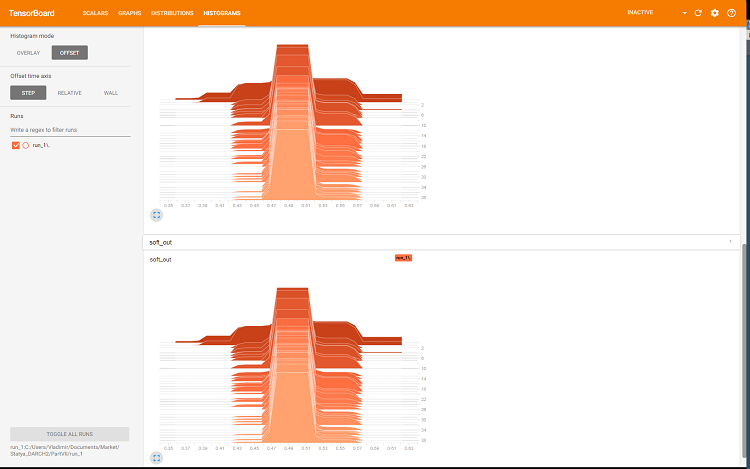
Fig. 8. Histogramas das saídas softmax e regularization
Esses gráficos são uma ferramenta poderosa para o ajuste dos parâmetros da rede neural, mas sua análise detalhada está além do escopo deste artigo.
A cada novo início do tensorboard, é necessário alterar o caminho de armazenamento log_dir ou excluir o que foi usado anteriormente.
Vamos ver como a qualidade da classificação com os mesmos parâmetros mudaria, mas usando o conjunto de testes para validação. O script é exibido abaixo e está disponível no arquivo:
library(reticulate) library(keras) py_set_seed(12345) num_classes <- 2L batch_size <- 32L learning_rate <- 0.005 epochs <- 100L #--------- bestNN <- env$resBest$bestNN x_train <- env$res$InputTrain[ ,bestNN] y_train <- env$Ytest %>% to_categorical() x_test <- env$res$InputTest[ ,bestNN] y_test <- env$Ytest1 %>% to_categorical() x_test1 <- env$res$InputTest1[ ,bestNN] y_test1 <- env$Ytest2 %>% to_categorical() #---------------------------------------- early_stopping <- callback_early_stopping(monitor = "val_acc", min_delta = 1e-5, patience = 20, verbose = 0, mode = "auto") log_dir <- paste0(getwd(),"/run_2") tensboard <- callback_tensorboard(log_dir = log_dir, histogram_freq = 1, batch_size = 32, write_graph = TRUE, write_grads = TRUE, write_images = FALSE) ##----model--keras------------------------- # define model model <- keras_model_sequential() # add layers and compile model %>% layer_gaussian_noise(stddev = 0.05, input_shape = dim(x_train)[2], name = "GN") %>% layer_dense(units = num_classes, name = "dense1") %>% layer_activation_softmax(name = "soft") %>% layer_activity_regularization(l2 = 1.0, name = "reg") %>% #l1 = 0.01, compile( loss = 'binary_crossentropy', optimizer = optimizer_rmsprop(lr = learning_rate, decay = 0.01), metrics = 'accuracy' ) ## Training & Evaluation --------------------------- # Fit model to data model %>% fit( x_train, y_train, batch_size = batch_size, epochs = epochs, verbose = 0, view_metrics = TRUE , shuffle = TRUE, validation_data = list(x_test, y_test), callbacks = list(early_stopping, tensboard)) -> history
Vamos ver as métricas no segundo conjunto de testes:
#--model--test1------------------------------------------------- predict(model, x_test1) -> Ypr.test1 Ypr.test1 %>% max.col()-1 -> y_pr_test1 #Ypr.test1 %>% apply(1, function(x) which.max(x)) %>% subtract(1) -> y_pr_test1 evalq(res_mod_test1 <- Eval(Ytest2, y_pr_test1), env) > env$res_mod_test1 $metrics Accuracy Precision Recall F1 0 0.713 0.704 0.826 0.760 1 0.713 0.730 0.575 0.644 $confMatr Confusion Matrix and Statistics predicted actual 0 1 0 114 24 1 48 65 Accuracy : 0.7131 95% CI : (0.6529, 0.7683) No Information Rate : 0.6454 P-Value [Acc > NIR] : 0.013728 Kappa : 0.4092 Mcnemar's Test P-Value : 0.006717 Sensitivity : 0.7037 Specificity : 0.7303 Pos Pred Value : 0.8261 Neg Pred Value : 0.5752 Prevalence : 0.6454 Detection Rate : 0.4542 Detection Prevalence : 0.5498 Balanced Accuracy : 0.7170 'Positive' Class : 0
Eles são praticamente idênticos aos da primeira variante. Nós podemos comparar visualmente as duas variantes usando o tensorboard.
#-----plot------------------ tensorboard(log_dir = c(paste0(getwd(),"/run_1"), paste0(getwd(),"/run_2")))
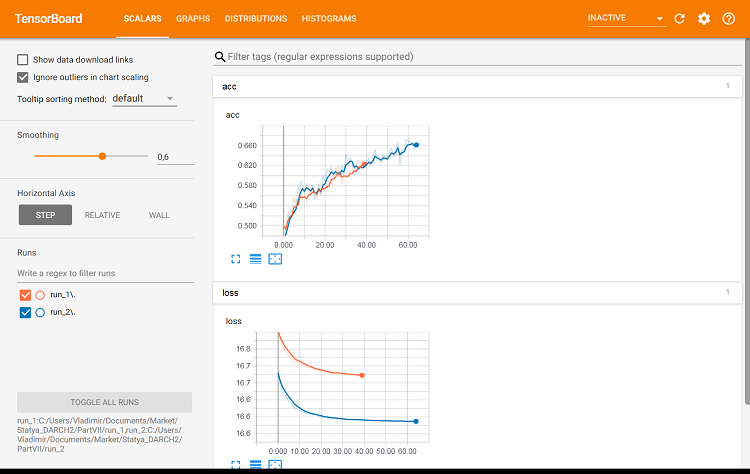
Fig. 9. Métricas do conjunto de treinamento
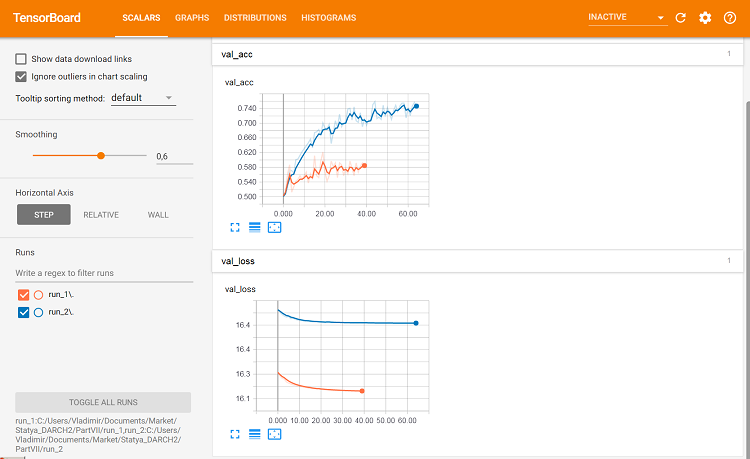
Fig. 10. Métricas do conjunto de validação
É aqui que as diferenças podem ser vistas.
Vamos realizar o último experimento. Todas as 500 saídas do ensemble serão alimentadas como entrada para a rede neural multicamadas. Assim, a rede neural realiza a poda e a combinação simultaneamente. O script é fornecido abaixo e também está disponível no arquivo modelDNN_500.R.
library(reticulate) library(keras) py_set_seed(12345) num_classes <- 2L batch_size <- 32L learning_rate <- 0.0001 epochs <- 100L #--------- x_train <- env$res$InputTrain y_train <- env$Ytest %>% to_categorical() x_test <- env$res$InputTest y_test <- env$Ytest1 %>% to_categorical() x_test1 <- env$res$InputTest1 y_test1 <- env$Ytest2 %>% to_categorical() #---------------------------------------- early_stopping <- callback_early_stopping(monitor = "val_acc", min_delta = 1e-5, patience = 20, verbose = 0, mode = "auto")
Nós carregamos as bibliotecas, constantes, definimos os conjuntos para treinamento e teste, bem como a função de parada antecipada.
##----modelDNN--keras------------------------- # define model modDNN <- keras_model_sequential() # add layers and compile modDNN %>% layer_gaussian_noise(stddev = 0.001, input_shape = dim(x_train)[2], name = "GN") %>% layer_batch_normalization() %>% layer_dense(units = 100, activation = "elu", name = "dense1") %>% layer_dropout(rate = 0.5, name = "dp1") %>% layer_batch_normalization() %>% layer_dense(units = 50, activation = "elu", name = "dense2") %>% layer_batch_normalization() %>% layer_dropout(rate = 0.5, name = "dp2") %>% layer_dense(units = 10, activation = "elu", name = "dense3") %>% layer_batch_normalization() %>% layer_dropout(rate = 0.2, name = "dp3") %>% layer_dense(units = num_classes, activation = "softmax", name = "soft") %>% compile( loss = 'binary_crossentropy', optimizer = optimizer_rmsprop(lr = learning_rate, decay = 0.0001), metrics = 'accuracy' )
Assim, nós definimos a rede neural, especificando a sequência e os parâmetros das camadas. Nós também instruímos o compilador em qual função de perda, otimizador e métrica usar ao treinar o modelo. Agora, o modelo é treinado:
## Training & Evaluation --------------------------- # Fit model to data modDNN %>% fit( x_train, y_train, batch_size = batch_size, epochs = epochs, verbose = 0, view_metrics = TRUE , shuffle = TRUE, validation_split = 0.2, #validation_data = list(x_test, y_test), callbacks = list(early_stopping)) -> history
Durante o treinamento, nós vamos emitir as métricas e misturar os dados de entrada, 20% do conjunto de treinamento será usado para validação e a interrupção antecipada será aplicada. O modelo é testado no conjunto de testes:
#--model--test------------------------- predict(modDNN, x_test) -> Ypr.test Ypr.test %>% apply(1, function(x) which.max(x)) %>% subtract(1) -> y_pr_test evalq(res_mod_test <- Eval(Ytest1, y_pr_test), env)
Pode-se ver que os números dos resultados estão no mesmo nível da média do conjunto:
> env$res_mod_test $metrics Accuracy Precision Recall F1 0 0.752 0.702 0.800 0.748 1 0.752 0.808 0.712 0.757 $confMatr Confusion Matrix and Statistics predicted actual 0 1 0 184 46 1 78 193 Accuracy : 0.7525 95% CI : (0.7123, 0.7897) No Information Rate : 0.523 P-Value [Acc > NIR] : < 2.2e-16 Kappa : 0.5068 Mcnemar's Test P-Value : 0.005371 Sensitivity : 0.7023 Specificity : 0.8075 Pos Pred Value : 0.8000 Neg Pred Value : 0.7122 Prevalence : 0.5230 Detection Rate : 0.3673 Detection Prevalence : 0.4591 Balanced Accuracy : 0.7549 'Positive' Class : 0
Vamos traçar o histórico do treinamento:
plot(history)
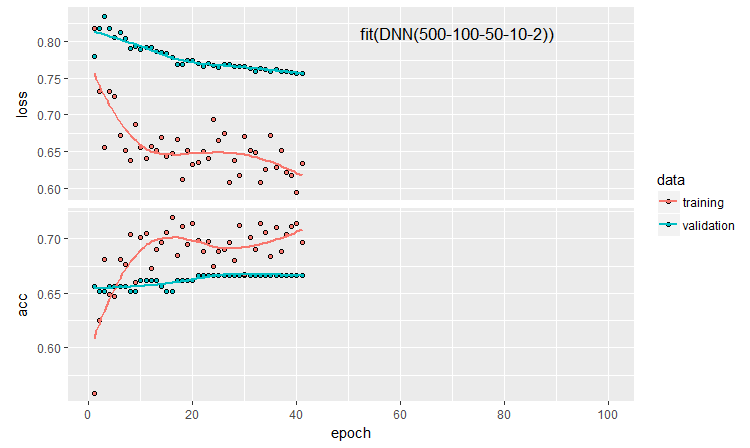
Fig. 11. O histórico de treinamento da rede neural DNN500
Para melhorar a qualidade da classificação, vários hiperparâmetros podem ser modificados: método de inicialização do neurônio, regularização da ativação dos neurônios e seus pesos, etc. Os resultados obtidos com os parâmetros selecionados quase intuitivamente têm uma qualidade promissora, mas também um limite decepcionante. Sem otimização, não foi possível aumentar a Accuracy (precisão) acima de 0.82. Conclusão: é necessário otimizar os hiperparâmetros da rede neural. Nos artigos anteriores, nós experimentamos a otimização bayesiana. Ela pode ser aplicada aqui também, mas é um tópico difícil e separado.
Definir um modelo sequencialmente permite testar e configurar modelos de qualquer complexidade e profundidade. Mas usando a API funcional do keras, é possível criar estruturas mais complexas de redes neurais: por exemplo, com várias entradas e saídas. Isso será discutido no próximo artigo.
5. Análise de resultados experimentais
Então, nós temos os resultados e teste de treinamento de cinco modelos:
- ensemble com suavização (EnsAver);
- ensemble com votação por maioria (EnsVot);
- modelo de regressão logística varb;
- rede neural DNN(7,2);
- rede neural DNN500.
Vamos reunir os índices de qualidade de todos esses modelos em uma tabela, decompor o erro de classificação em componentes e estimar sua contribuição para o erro global. Nós usamos a função randomUniformForest::biasVarCov() (Bias-Variance-Covariance Decomposition). Veja a descrição do pacote para mais detalhes sobre esta função. O código para decomposição do erro de classificação dos conjuntos EnsAver e EnsVot é mostrado abaixo. Os scripts são semelhantes aos outros modelos.
#---bias--test------------------------------- import_fun(randomUniformForest, biasVarCov, BiasVar) evalq({ target = Ytest1 biasAver <- BiasVar(predictions = ScoreAver$clAver, target = target, regression = FALSE, idx = 1:length(target)) biasVot <- BiasVar(predictions = ScoreVot$ClVot, target = target, regression = FALSE, idx = 1:length(target)) }, env) ----------------------------- Noise: 0.2488224 Squared bias: 0.002107561 Variance of estimator: 0.250475 Covariance of estimator and target: 0.1257046 Assuming binary classification with classes {0,1}, where '0' is the majority class. Misclassification rate = P(Y = 1)P(Y = 0) + {P(Y = 1) - P(Y_hat = 1)}^2 + P(Y_hat = 0)P(Y_hat = 1) - 2*Cov(Y, Y_hat) Misclassification rate = P(Y = 1) + P(Y_hat = 1) - 2*E(Y*Y_hat) = 0.2499958 --------------------- Noise: 0.2488224 Squared bias: 0.004079665 Variance of estimator: 0.2499721 Covariance of estimator and target: 0.1274411 Assuming binary classification with classes {0,1}, where '0' is the majority class. Misclassification rate = P(Y = 1)P(Y = 0) + {P(Y = 1) - P(Y_hat = 1)}^2 + P(Y_hat = 0)P(Y_hat = 1) - 2*Cov(Y, Y_hat) Misclassification rate = P(Y = 1) + P(Y_hat = 1) - 2*E(Y*Y_hat) = 0.2479918
Saída compacta:
> env$biasAver $predError [1] 0.2499958 $squaredBias [1] 0.002107561 $predictionsVar [1] 0.250475 $predictionsTargetCov [1] 0.1257046
| 95%CI Acc | Precision | Recall | F1 | PredErr | sqBias | predVar | predTargCov | |
|---|---|---|---|---|---|---|---|---|
| EnsAver | 0.7102, 0.7880 (0.7500) | 0.708 0.794 |
0.778 0.727 |
0.741 0.759 |
0.2499 | 0.0021 | 0.2505 | 0.1257 |
| EnsVot | 0.7123, 0.7897 (0.7525) | 0.702 0.808 |
0.800 0.712 |
0.748 0.757 |
0.248 | 0.0041 | 0.25 | 0.1274 |
| varb | 0.7416, 0.8159 (0.7804) | 0.790 0.808 |
0.783 0.779 |
0.766 0.793 |
0.2199 | 0.000398 | 0.25 | 0.13964 |
| DNN(7, 2) | 0.7165, 0.7935 (0.7565) | 0.765 0.751 |
0.678 0.823 |
0.719 0.785 |
0.2460 | 0.000195 | 0.2498 | 0.1264 |
| DNN500 | 0.7123, 0.7897 (0.7525) | 0.702 0.808 |
0.800 0.712 |
0.748 0.757 |
0.2779 | 0.01294 | 0.2452 | 0.1145 |
Informações na tabela de resumo:
- O melhor dado de Accuracy entre os modelos base foi obtido por varb, combinando 500 saídas do ensemble. O melhor modelo entre os combinadores treináveis de acordo com essa pontuação é DNN(7,2), combinando os 7 melhores resultados do conjunto.
- O menor erro de teste para a amostra de teste (PredErr) foi alcançado por varb e DNN(7,2).
- O quadrado do viés (sqBias) dos mesmos dois modelos é uma ordem de magnitude melhor que os outros.
- A variação de erro (PredVar) de todos os modelos é quase a mesma. Isso parece estranho: o ensemble deveria ter fornecido uma diminuição na variância, mas nós recebemos um viés baixo.
- A melhor covariância entre a estimativa e a resposta (predictionTargetCov) é mostrada por varb. Isso não significa nada sozinho como uma variável individual, é usado apenas para comparação de modelos.
- Todas as pontuações do modelo DNN500 são as mais baixas. Conclusão: aumentar a complexidade dos modelos para tarefas simples não leva a melhores resultados.
Conclusão
O conjunto de classificadores de redes neurais ELM com suavização ou com votação simples por maioria mostra uma boa qualidade de classificação em uma velocidade computacional muito alta. É possível melhorar a qualidade otimizando o limite para converter as saídas das variáveis contínuas em nominais e calibrando as saídas antes da média. Nenhuma diminuição perceptível na variância do erro foi detectada.
Substituir a média das saídas do conjunto com a função softmax de uma rede neural simples reduz a polarização em uma ordem de magnitude sem qualquer diminuição perceptível na variação. O uso de modelos de rede neural mais complexos para substituir a poda e a média não produziu bons resultados.
O modelo de regressão logística obtido com a ajuda da seleção de variáveis bayesianas (pacote varbvs) mostra resultados muito bons. Você pode usar as melhores saídas determinadas por este pacote para a rede neural.
24% do ruído, que parece ser irremovível desde o pré-processamento, mais uma vez sugere que as amostras de ruído sejam remarcadas para uma classe separada em algum momento.
É necessário usar os recursos do keras e trabalhar com as sequências (séries temporais), que são os nossos dados. Isso pode melhorar a qualidade da classificação.
Anexos
GitHub/PartVII contêm os seguintes arquivos:
- Importar.R — funções de importação de pacotes.
- Library.R — bibliotecas necessárias.
- FunPrepareData_VII.R — funções para a preparação dos dados iniciais.
- FunStacking.R — funções para a criação e teste do ensemble.
- Prepare.R — funções e scripts para preparar os dados iniciais para os combinadores treináveis.
- Varb.R — scripts do modelo base de varb.
- model_DNN7_2.R — scripts da rede neural DNN(7-2).
- model_DNN_500.R — scripts da rede neural DNN500.
- SessionInfo_VII.txt — lista de pacotes usados nos scripts do artigo.